基于2D模型的药物小分子筛选方法
徐其凤 冯 林* 余 游 罗桂林
1(四川师范大学计算机科学学院 四川 成都 610101)2(四川大学生物治疗国家重点实验室 四川 成都 610041)
0 引 言
分子是化合物的基础,化合物的合成和活性化合物的筛选是药物研发的重点和难点,即使在生物化学技术飞速发展的今天,如何筛选出最合适的分子,一直是药物研发领域面临的挑战性问题之一。
一方面,在生物领域中,Jencks[1]构建了基于片段的筛选方法(FBDD)理论框架;被批准上市的BRAF-V600E激酶抑制剂Vemurafenib[2]则是FBDD方法的一个典型成功案例,Vemurafenib从初始的片段筛选到被FDA批准上市仅耗费了6年;Winter等[3]描述了一种使用X射线结晶学的片段筛选活动,在Ras:SOS复合体上发现了三个片段结合位点;Liang等[4]使用FBDD和QSAR研究,首次完全合成了一种溴化酪氨酸次级代谢产物,它是一种有效的p38α抑制剂,具有抗癌作用;Erlanson等[5]对近20年来FBDD的发展进行研究,讨论了其研究步骤,并显示FBDD概念如何渗透和加强药物发现工作的,指出FBDD已成为药物研发的主流方法之一。
另一方面,随着计算机技术的发展和大数据技术的应用,提升生物化学数据处理能力,缩短药物研发周期,成为了众多研究者关注的热点[6-7],并成功利用计算机技术解决了部分难题,如预测分子属性[8-9]、检验化学反应结果[10]、处理医学图像和数据[11-12]。Lusci等[13]通过考虑与分子图的所有可能的顶点中心非循环方向相关联的递归神经网络的集合,解决分子的无向循环图转化为有向无环图的问题,并在四个基准数据上进行了测试;Urban等[14]则将视线聚焦于输入数据问题,提出了Inner和Outer两种方法;Olivecrona等[15]提出了一种基于序列的生成模型方法,用于生成不同类型分子的生成模型,给出了结合无监督机器学习方法设计新化合物的新思路。
以上大多数研究方法解决了各种生物化学和生物医学上的一些问题,如医学图像识别和分类、分子性质预测、抑制剂的发现。但是,在解决药物小分子筛选方面还存在不足:(1) 现有研究大多未进行分子分量区分,研究过程将大分子、中分子和小分子一概而论,不利于挖掘小分子的特有特征。(2) 存在研究复杂、耗时长、解决问题单一和算法复杂度高等问题。(3) FBDD大多只从生物化学角度进行研究。针对上述问题,本文从小分子2D存储的SDF文件出发,提出一种基于2D模型的药物小分子筛选方法(SMS-2D),利用计算机技术进行片段筛选。首先,输入分子片段P和小分子数据库文件,对数据进行预处理,将分子片段P和数据库文件转化为比对小分子信息MP和小分子数据集W;其次,每次取小分子数据集W中的一个小分子信息Mi,计算Mi与比对小分子Mp的包含度αi;然后,筛选出包含度αi大于或等于阈值α的小分子,并存入小分子数据集W′;最后,输出小分子数据集W′中的所有小分子信息,并进行可视化处理。
1 理论基础
1.1 FBDD
目前高通量筛选(HTS)是药物筛选的主要方法,但是大型复合库的收集、维护和筛选较为复杂和困难,且HTS在新目标筛选时命中率低。针对上述问题,基于片段的筛选方法FBDD开始逐渐成为药物研发的主流方法。不同于HTS,FBDD从分子结构的一个片段结构出发。在药物分子结构中,分子的每一个片段都有其特殊作用。近年来,研究者们将目光聚焦在分子片段上,旨在通过研究分子片段来得到新的药物分子。FBDD的优势在于可以结合一个蛋白的多个位点或者多个蛋白,在这样的情况下,即使分子片段是弱结合,但具有高筛选命中率,这种优势在面对复杂靶标时尤为明显,并且使片段库的收集和维护更便捷,这使得小型学术机构也能从事药物发现工作。FBDD的应用范围非常广泛,从化学生物学到计算化学,从抑制剂的发现到潜在位点的寻找,FBDD都取得了不错的进展。
1.2 分子表示
分子常用的表示分为以下三种:图像表示(分子结构式)、线性编码(如SMILES串、WLN等)和文本信息编码(如MOL、SDF等)。
1) 图像表示。图像表示方法的优点是能够直观的观察分子的结构,缺点在于所需存储空间大,且小分子信息遗失多,且无法对分子的某一物理化学属性进行分析。
2) 线性编码表示。它将化学结构转化为一棵树,使用一串字符来描述一个三维的化学结构。线性编码表示的优点是具有唯一性、所需存储空间少,缺点是不利于子结构检索。
3) 文本信息编码表示。文本信息编码指使用文本方式存储分子的相关信息,其描述如表1所示。它具有存储空间少、便于提取各种细节信息等优点,缺点在于不够直观。本文使用文本信息编码表示的SDF文件进行实验。SDF是由MDL公司开发的、最常见的化学数据文件存储格式,专门用于分子结构信息表示。SDF文件分为结构数据和理化数据两部分,结构数据包括原子信息和键值信息,以字符“END”作为结束标志;理化数据包括分子ID、分子物理属性等信息,以字符“$$$$”作为结束标志。SDF可以以二维和三维两种不同的形式存储分子信息。

表1 NADPH的SDF文件及描述

续表1
2 SMS-2D描述
2.1 问题表述
前期基础研究中,四川大学生物治疗国家重点实验室通过肠道EV68病毒3C蛋白的酶活实验研究发现某小分子B中的片段结构H对肠道EV68病毒3C蛋白的活性有抑制作用,可能小分子B中的分子片段H起关键作用,然而细胞毒性实验结果表明小分子B细胞毒性较大,不能直接应用于临床实验。实验室根据此结果提出一种假设,在现有的药物大数据中还存在尚未被发现的包含了分子片段H的小分子,并合理猜测这些小分子可能具备同样的效果。但是,如何从海量药物大数据中筛选出包含与分子片段H相似分子片段的药物小分子成为难点。
目前,实验室主要采用人工筛选方法,但人工筛选存在耗时、效率低、药物筛选周期长等问题,因此利用计算机技术解决该问题成为新思路。计算机技术不仅能够降低新药开发成本,减少人力损耗,更能缩短新药研发周期,对促进降糖药物研发具有重要意义。
本文利用计算机技术对药物自动化筛选进行了研究,利用计算机技术在海量药物大数据中筛选出包含与分子片段H具有相似分子片段的小分子,并将筛选结果交由四川大学生物治疗国家重点实验室进行生物实验验证,测试这些小分子是否具有效果。
由于保密要求,所以本文以还原型烟酰胺腺嘌呤二核苷酸磷酸NADPH为例进行实验,NADPH的分子结构如图1所示。为便于对算法性能进行测试,选取NADPH中的两个不同分子片段P1和P2进行实验。P1和P2的分子结构如图2所示。

图1 NADPH2D分子结构式

(a) 片段P1 (b) 片段P2图2 NADPH的分子片段
2.2 相关定义
为了便于叙述和理解,本文将以数学形式定义与SMS-2D方法相关的基本概念。
定义1小分子数据集。一个小分子数据集W是一个二元组W=(U,M),其中:U表示小分子数据集名称,M=(M1,M2,…,MN)表示小分子信息的集合。
定义2小分子信息。一个小分子信息M是一个四元组M=(id,K,m,n),其中:id表示该小分子的查询ID;K=(k1,k2,…,kn)表示小分子化学键信息的集合;m表示小分子含有原子的个数;n表示小分子含有的化学键的个数。
定义3化学键信息。一个化学键信息对k是一个四元组k=(X1,X2,B,T),其中:X1、X2表示形成化学键的两个原子;B表示两个原子形成化学键的数目,例如单键表示为1,双键表示为2;T表示化学键在小分子中的转向。

定义5查全率与准确率。给定小分子数据集W=(U,M)、比对小分子Mb=(idb,Kb,mb,nb)与包含度阈值α,设小分子数据集W=(U,M)中含有与Mb=(idb,Kb,mb,nb)包含度大于等于α的个数为Z,从W=(U,M)中筛选出小分子数据集W′=(U′,M′):


2.3 算法流程
SMS-2D流程如图3所示,总共分为四个步骤:第一步输入分子片段P和小分子数据库文件;第二步对数据进行转换,从化学描述方式转换为小分子信息和小分子数据集;第三步进行包含度计算,筛选符合条件的小分子;第四步输出结果。

图3 SMS-2D算法流程
2.4 算法步骤
SMS-2D算法步骤如下:
输入:分子片段P及小分子数据集,两者都以SDF文件格式输入。
输出:小分子数据集。
Step1读取分子片段P和小分子数据集。
(1) 读取分子片段的原子信息、键值信息和ID,存入line中;
(2) 读取数据集中每一个小分子的原子信息、键值信息和ID,以“$$$$”作为小分子信息的结束符号。
Step2将输入的分子片段P和小分子数据库文件转化成小分子信息Mp=(idp,Kp,mp,np)和小分子数据集W=(U,M)。
(1) 读取小分子P的原子信息第四列数据,即元素信息,存入线性表atom[]中;
(2) 读取键值信息的全部数据,存入线性表bond[]中;
(3) 将数组bond[]中第一列和第二列数据按序号替换为数组atom[]中相应的原子;
(4) Foriinbond[]:
kpi=bond[i]
End For
(5) 读取小分子P的数据ID设为idp,原子个数设为mp,原子键个数设为np;
(6) 输出小分子信息Mp=(idp,Kp,mp,np);
(7) 重复Step 2中的步骤(1)-步骤(4),将小分子数据集中的所有小分子数据转化小分子信息,输出小分子数据集W=(U,M)。
Step3依次计算小分子数据集中每个小分子信息Mi=(idi,Ki,mi,ni)与Mp=(idp,Kp,mp,np)的包含度。
ForMiinM:
按照定义4计算出αi=η(M1,M2)
End For
输出所有小分子与Mp的包含度:(α1,α2,…,αn)。
Step4筛选出包含度大于等于阈值α的小分子信息,并存入小分子数据集W′=(U′,M′)中。
Step5输出筛选出的小分子数据集W′=(U′,M′)。算法停止。
2.5 算法复杂度分析
对于本文的SMS-2D,数据转化的时间复杂度为O(Ki×Kn) ,Ki为键值信息的总行数,Kn为键值信息的列数;小分子包含度计算的时间复杂度为O(N×Mn),N为数据集的数据量,Mn为小分子信息Mp的大小。在药物小分子数据集中,由于O(N×Mn)>O(Ki×Kn),因此SMS-2D的时间复杂度为O(N×Mn)。SMS-2D的空间复杂度由Step 2中的atom[]和bond[]的大小决定,atom[]的空间复杂度为mp,bond[]的空间复杂度为4np,所以SMS-2D的空间复杂度为O(Mp+4np)。
3 实 验
3.1 实验环境
本文实验所采用的硬件环境为AMD Ryzen 3 PRO 2200G with Radeon Vega Graphics 3.50 GHz处理器,RAM大小为8 GB;软件环境为Windows 10系统,Python编程语言,ChemDraw14.0软件。
3.2 数据源
本文实验所使用小分子数据集如表2所示。

表2 数据集信息
3.3 实验设计
为了验证本文SMS-2D的效果,实验分成实验一和实验二两个部分。实验一为SMS-2D性能测试:在四个数据集上进行了两次实验,第一次实验筛选的分子片段为片段P1,第二次实验筛选的分子片段为片段P2。每次实验都记录下算法的运行时间和符合要求的小分子数量,输出结果并分析。实验二为算法查全率测试:为验证SMS-2D算法的查全率,将事先准备的60个虚构小分子作为验证数据加入数据集,输出结果进行比较分析。
3.4 实验一:算法性能测试
实验一对SMS-2D算法的性能进行测试,包括算法的运行时间和输出结果。本文分别对分子片段P1、P2进行实验,对比分析了SMS-2D算法在四个规模不同的数据集上的表现,并给出在包含度阈值α=1和α=0.75下的实验结果,实验结果如表3、表4所示。
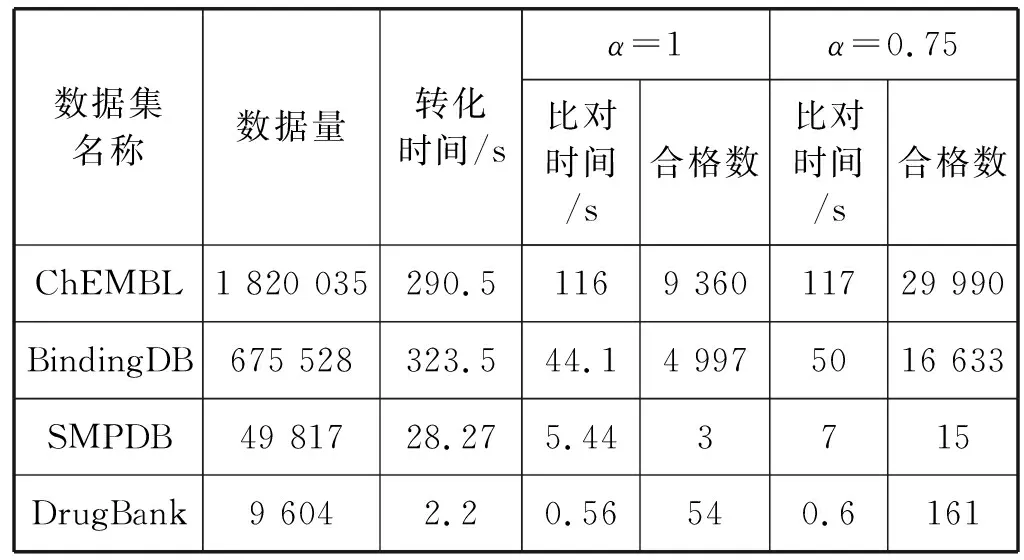
表3 分子片段P1实验结果表

表4 分子片段P2实验结果表
从表3和表4可以看出,不同数据集中符合要求的小分子数量不一,且结果集数量与数据集数量不成正比关系。包含度阈值α的大小决定了结果集的规模,α越大,结果集规模越小;反之,结果集规模越大。但α越小,输出结果与分子片段P的包含程度就越低,实验结果的实际意义就越小,所以选用合适的α至关重要。从输出的小分子数量分析,符合要求的小分子数量相较于数据集而言占比非常小,即需要在海量数据中筛选出少量符合要求的小分子。SMS-2D算法的转化时间和比对时间较短,通过表中数据可得出数据集数量与时间的关系:t≈n(万条)×1.5(秒)。算法所用时间与数据集大小成正比,与输出结果数无紧密联系。总体而言,无论数据量的大小,算法运行时间都非常短暂,即使是百万级的数据也仅仅只需要300 s,如果采用人工筛选的方法处理同样规模的数据则会耗费长达几月的时间。因此,相较于人工方法,SMS-2D算法能高效筛选出符合要求的药物小分子,节省大量时间。
3.5 实验二
通过SMS-2D算法可以找出各个数据集中包含有与分子片段P1具有相似分子片段的小分子,但是这样无法确定算法的查全率,不能保证是否存在漏选。为了对算法的查全率进行测试,本文在实验一的基础上增加一个测试步骤,加入了验证数据,其步骤如下:
Step1利用化学软件ChemDraw设计了30个包含分子片段P1的小分子和30个从其他数据库获取的未包含分子片段P1的小分子作为验证数据,设计时尽量保证除分子片段P1之外的其余结构具有较大的差异性。
Step2将60个小分子随机插入到数据集中。
Step3设定包含度阈值α=1,运行算法,输出符合要求的小分子在数据集中的ID,查看输出结果中包含多少验证数据,以此对SMS-2D算法的查全率进行估计。
实验二结果如表5所示。

表5 实验二验证结果表(α=1)
实验二在四个数据集中随机插入了60个的验证数据,其中自主设计的30个验证数据复杂度不一,分子片段P1的位置随机。从表5可以看出,对于不同大小的数据集,SMS-2D算法都能找出30个包含了分子片段P1的验证数据,且未找出其余30个未包含分子片段P1的验证数据,由此可得推论:在包含度阈值α=1时,SMS-2D算法具备100%的查全率。
为对SMS-2D算法的准确率进行测试,本文对DrugBank数据集在包含度阈值α=1时的输出结果进行评估。评估发现,输出的结果中包含完整的分子片段P1和P2,由此推论:在包含度阈值α=1时,SMS-2D算法具备100%的准确率。
3.6 结果可视化
为了便于后续生物实验的进行,利用软件ChemDraw对输出结果进行可视化操作。首先,根据小分子ID在对应的数据集中找到小分子,输出其SDF文件;其次,将SDF文件导入ChemDraw软件,转存为PNG格式的分子结构图。分子结构图相较于SDF文件更加直观明了。表6和表7分别给出了分子片段P1和P2在不同阈值下的部分可视化输出结果。

表6 输出结果可视化图(α=1)

表7 输出结果可视化图(α=0.75)
表6和表7中黑色方框内为与分子片段P1或P2相似的部分。由结果可视化图可知,当α=1时,输出的小分子中包含有完整的分子片段;当α=0.75时,输出的小分子中包含分子片段的部分结构。
4 结 语
化合物的合成和活性化合物的筛选是药物研发的重点和难点,在此过程中,药物小分子的筛选至关重要。本文的主要贡献如下:基于分子的2DSDF存储文本,提出了一种基于2D模型的药物小分子筛选方法SMS-2D。仿真实验结果证明:SMS-2D能够应用于各个数据集的2DSDF文件药物筛选,并能够快速全面地搜索出目标小分子,保证高查全率。目前,SMS-2D药物筛选方法正用于四川大学生物治疗国家重点实验室的药物小分子筛选中,与实验室先前的人工筛选方法相比,SMS-2D大幅度地减少了药物研发过程中的小分子筛选时间,缩短药物研发周期。SMS-2D仍存在两个不足之处:一是其只能应用于2D的SDF文件;二是分子片段不宜太大,所涉及的原子最好不超过12个。未来将把研究重点放在分子的三维结构上,以三维SDF文件为研究对象,深入挖掘分子结构关系,期望能更高效率地进行小分子筛选。

