玉米生长素响应因子家族基因的表达模式分析
李文兰 李文才 孙 琦 于彦丽 赵 勐 鲁守平 李艳娇 孟昭东
玉米生长素响应因子家族基因的表达模式分析
李文兰 李文才 孙 琦 于彦丽 赵 勐 鲁守平 李艳娇 孟昭东*
山东省农业科学院玉米研究所 / 小麦玉米国家工程实验室 / 农业部黄淮海北部玉米生物学与遗传育种重点实验室, 山东济南 250100
生长素响应因子(auxin response factor, ARF)是一类重要的转录因子, 通过特异性地结合生长素响应元件调节下游靶基因的转录, 参与诸多植物生长发育过程的调控。玉米中有许多家族基因, 但其表达模式有待深入研究。本研究分析了玉米家族基因在不同组织器官中的表达, 发现除、和组成型表达外, 其余32个基因的表达水平在生殖器官中要明显高于营养器官。对基因启动子区的顺式作用元件分析显示, 28个基因的启动子区含有逆境胁迫相关顺式元件, 实时定量PCR分析结果显示, 多个基因分别响应冷、热、盐和渗透胁迫。研究结果不仅暗示了家族基因在玉米生殖生长和非生物逆境胁迫响应中的重要性, 也为全面解析基因在玉米中的生物学功能提供有用信息。
; 表达; 逆境胁迫; 玉米
生长素响应因子(auxin response factor, ARF)是植物所特有的一类转录因子[1], 通过特异性地结合生长素响应元件(AuxREs)调节下游靶基因的转录, 参与诸多植物生长发育过程的调控, 例如向性运动、顶端优势、干旱胁迫响应等。研究基因的表达模式对于全面了解玉米基因家族的生物学功能具有重要意义。
生长素信号转导过程涉及早期响应基因(、和基因家族等)和与AuxREs相互作用的家族基因[2]。ARF蛋白结合到生长素调控基因启动子区的生长素响应元件(5'-TGTCTC-3')上, 抑制或激活这些基因的转录[3], 从而影响植物的生长发育。拟南芥、水稻、二穗短柄草、鹰嘴豆和丹参等多种植物中的基因已有相关研究[4-9]。拟南芥中第一个被发现的转录因子是, 是以人工合成的生长素响应元件为诱饵,通过活性很高的酵母单杂系统克隆得到的[3]。ARF1蛋白包括665个氨基酸, N端包含1个DNA结合结构域; 中间的一段区域富含脯氨酸, 丝氨酸和苏氨酸含量也较高[3], 与其他富含脯氨酸结构域的转录抑制子和转录激活子序列类似[4], 该区域可能是转录抑制或转录激活结构域; C端与基因编码蛋白的结构域III和IV有类似结构[3,10]。拟南芥中有22个基因编码ARF蛋白[11], 其中参与胚胎发生与维管形成[12];参与植物向光性和向重力性[13];控制叶扩展和侧根生长, 并与表现出功能冗余性[14-15];和参与花成熟调控, 且呈现功能冗余性[16]。此外, 马铃薯参与顶端休眠, 调控与顶端分生组织连接维管束的发育[17]; 水稻受生长素诱导, 参与胚芽鞘的向性运动[18]; 番茄也是家族基因, 参与种子发育、幼苗生长和果实成熟[19]。
玉米有35个基因[20], 基因结构和进化关系等已有相关分析, 但其表达模式还有待深入研究。关于基因在玉米不同组织器官中的表达模式以及在冷、热、盐和渗透胁迫下的表达模式还未见详细报道。Liu等[20]通过全基因组序列分析, 找到了35个玉米基因, 并对基因结构和进化关系等进行了分析。本研究拟通过分析基因在玉米不同组织器官中的表达模式以及在冷、热、盐和渗透胁迫下的表达模式, 探究基因在玉米生长发育和逆境胁迫响应过程中的作用, 为全面解析玉米基因的生物学功能提供有用信息。
1 材料与方法
1.1 试验材料与生长条件
试验所用的玉米材料是B73自交系。分别取材播种后14 d (V3时期)的初级根、第2片叶、整个幼苗植株、播种后25 d (V5时期)的中部茎秆和播种后34 d (V7时期)的整个幼嫩雄花序及雌花序, 用于实时定量PCR分析。逆境胁迫试验所用的材料在光照培养箱中进行长日照培养(16 h光照/8 h黑暗), 生长14 d。冷胁迫处理是将幼苗置于4℃培养箱中进行处理, 热胁迫处理是将幼苗置于40℃培养箱中进行处理, 盐胁迫处理是利用250 mmol L–1的NaCl对幼苗进行处理, 渗透胁迫处理是利用15% PEG-6000对幼苗进行处理。每种胁迫处理均在处理0、1、2、4和8 h后, 收集对照植株和处理植株的第2片叶用于实时定量PCR分析。
1.2 RNA提取和实时定量PCR分析
利用天根的多糖多酚总RNA提取试剂盒提取各样品的总RNA, 按照反转录试剂盒说明书完成cDNA的合成和纯化。实时定量PCR分析利用Bio-Rad CFX96实时定量检测系统(Bio-Rad, Hercules, CA, USA), 引物序列见表1,基因作为内参基因, 根据2-DDCT计算基因相对表达量。对于胁迫处理下玉米幼苗的qRT-PCR分析, 将各处理0 h各基因表达量设为1, 并以此对照计算其在其他时间点的相对表达量。实验数据通过SPSS18.0软件进行差异性分析。
1.3 利用转录组数据对ARF基因进行表达分析
利用从MaizeGDB数据库获得的玉米基因的ID号在Genevestigator网站(http://www.genevestigator. com/gv/)搜索基因在不同组织器官中的转录表达数据, 然后利用Genevestigator网站的系统软件对转录组表达数据进行分析, 获得基因在不同组织器官中的表达模式热点分布图[21]。
1.4 启动子区顺式作用元件分析
利用Gramene网站(http://ensembl.gramene.org/ Zea_mays/Info/Index)搜索确定玉米家族基因起始密码子上游1500 bp的启动子区序列, 利用PlantCare数据库(http://bioinformatics.psb.ugent.be/ webtools/plantcare/html/)的顺式作用元件分析工具, 对玉米家族基因启动子区序列进行顺式作用元件分析[22]。
2 结果与分析
2.1 玉米ARF家族基因在不同组织器官中的表达分析
为探究生长素响应因子家族成员在玉米中的表达模式, 我们利用实时定量PCR方法对基因在6种不同组织器官(根、茎、叶、幼苗、幼嫩雄花序和幼嫩雌花序)中的表达量进行了检测(图1)。、和在玉米中组成型表达,在茎、幼苗和雄花序中高表达,在叶和雌花序中高表达, 而剩余的30个基因均在雄花序和雌花序中优势表达。从整体空间表达上看, 各基因的表达量高低呈现生殖器官>营养器官。玉米花序发育涉及小穗对原基、小穗原基、花器官原基等各类原基发育起始过程, 在这些原基起始部位都存在生长素积累和转运[23]。作为生长素响应因子,基因在雌花序和雄花序中高水平表达, 暗示基因参与花序发育调控过程。
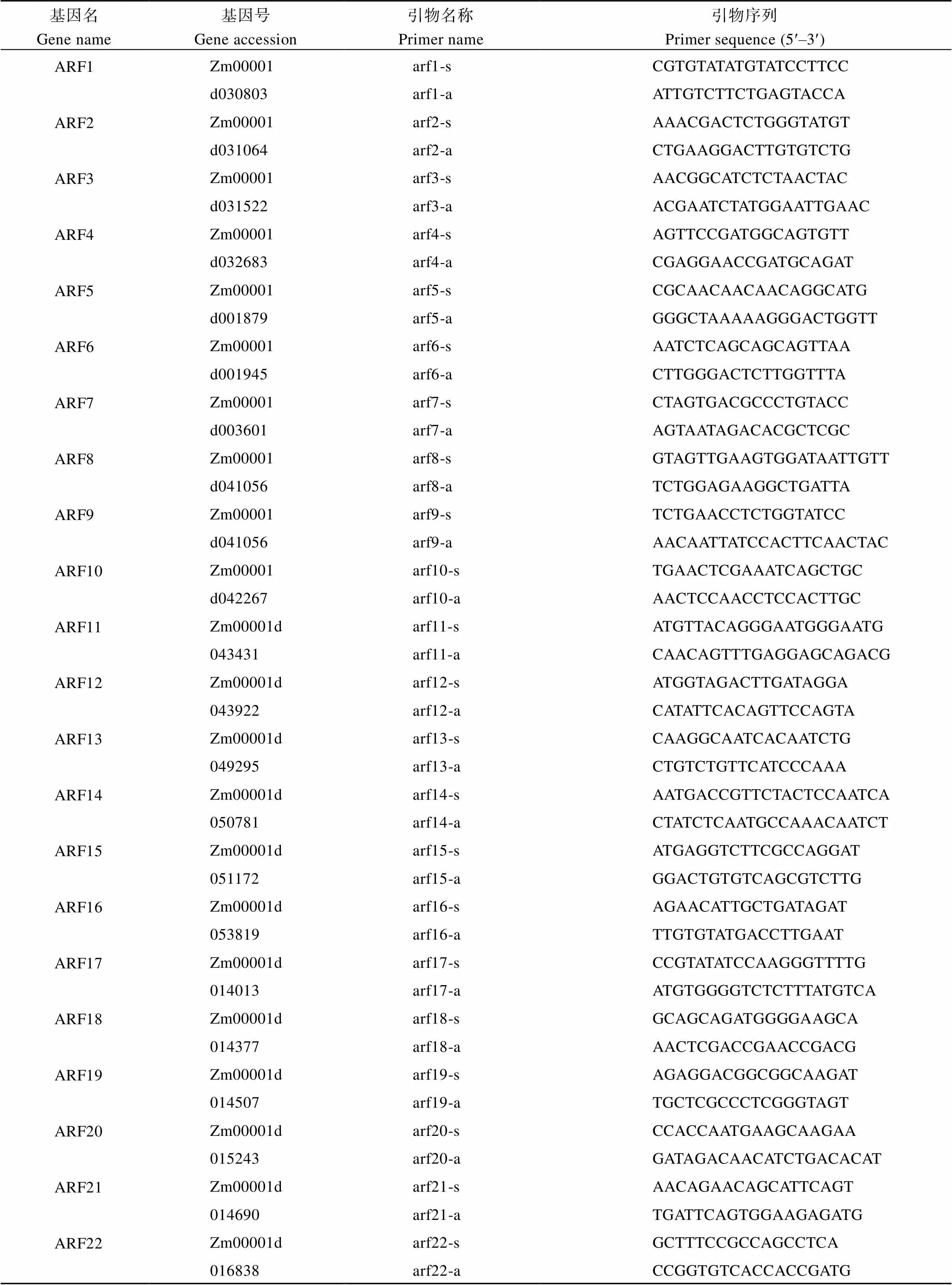
表1 实时定量PCR的引物序列
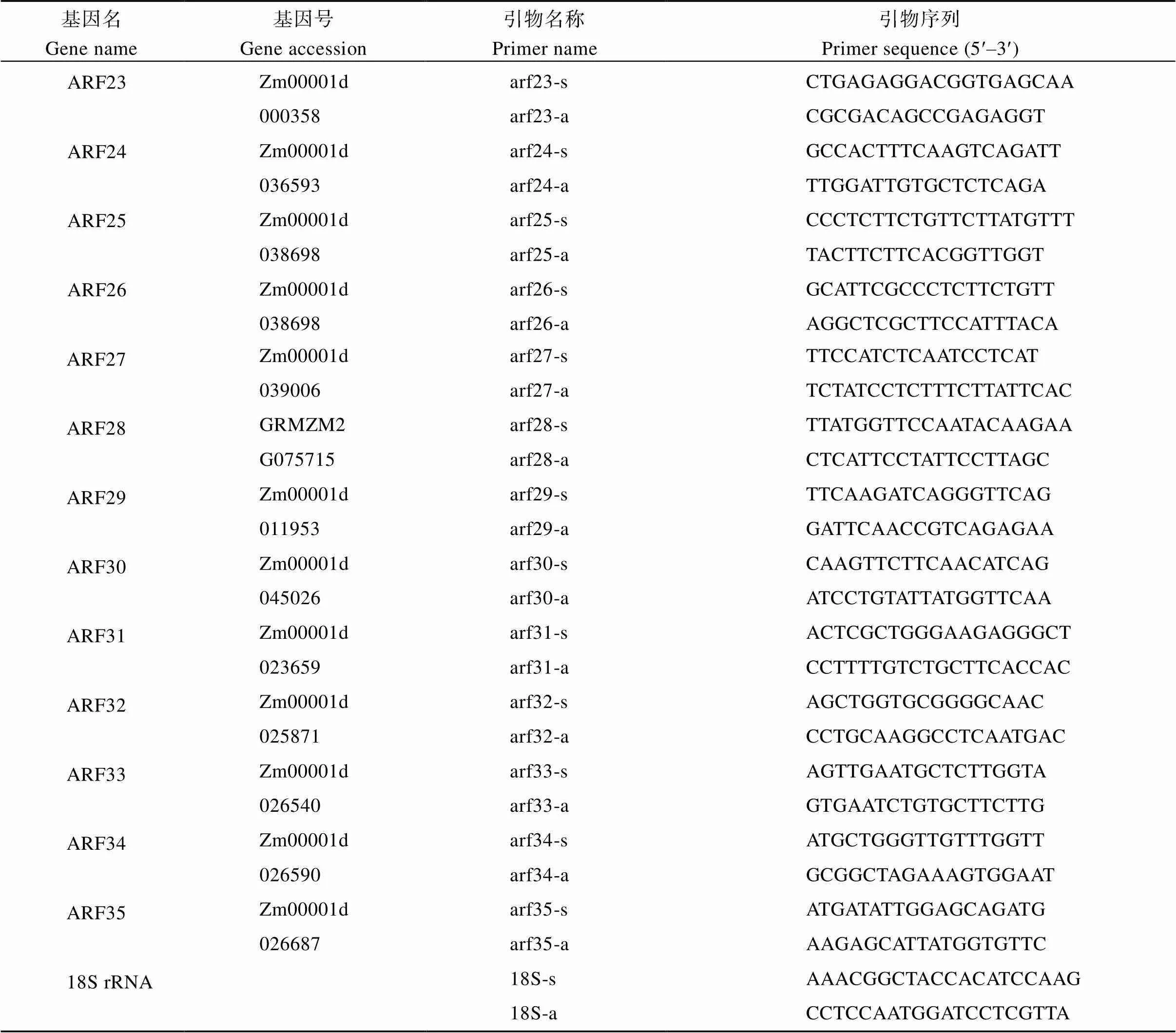
(续表1)
2.2 转录组数据库中玉米ARF家族基因在不同组织器官中的表达分析
为进一步了解玉米家族基因的表达模式, 我们利用Genevestigator数据库中的转录组数据对家族基因在更多不同组织器官(41个组织器官)中的表达模式进行了分析, 共获得31个玉米基因(未获得、、和这4个基因的表达信息)的表达信息, 结果如图2所示。除了花粉,、和在所检测的其余组织器官中均有表达, 这与实时定量PCR结果基本吻合。
和在花药中优势表达, 这与定量结果显示其在雄花序中优势表达是一致的。此外, 除了和, 其余29个玉米基因在雌花序发育(pistil, ovary, ear, spikelet)和种子发育过程(embryo, endosperm, embryo sac, embryo-surrounding region, basal endosperm transfer layer)中均有较高的表达量。这与实时定量PCR结果显示基因在生殖器官发育过程中优势表达也是一致的。然而, 与定量结果显示基因在茎中低表达(除了和)相反, 有29个基因(除和)在茎中具有较高的表达水平。这可能是由于取材时期和试验材料不同所造成的结果差异。结合转录组数据和实时定量PCR结果, 我们可以推测基因在玉米生长发育和生殖器官发育过程中均发挥着重要的作用。
R: 根; ST: 茎; L: 叶; S: 幼苗; T: 雄花序; E: 雌花序。图柱上的不同字母代表Duncan’s multiple range tests的显著差异(< 0.05)。
R: root; ST: stem; L: leaf; S: seedling; T: tassel; E: ear. Values marked with different letters indicate significant differences at< 0.05 by Duncan’s multiple range tests.
图中每个基因在所有检测组织器官中的表达量设为100%, 颜色的深浅代表表达百分率。
The relative expression of each gene in all detected tissues and organs was set at 100%, and the shade of color represents the percentage of relative expression levels.
2.3 玉米ARF家族基因的顺式作用元件分析
为更好了解玉米家族基因在逆境胁迫响应过程中的作用, 我们对玉米家族基因起始密码子上游1500 bp的启动子区进行顺式作用元件分析(表2), 除转录和光响应相关必需顺式作用元件, 在玉米家族基因的启动子区找到大量顺式作用元件, 根据功能可分为: 植物激素响应相关顺式作用元件(Element A)、生长发育相关顺式作用元件(Element B)和逆境胁迫相关顺式作用元件(Element C)。Element A中的脱落酸响应元件(ABRE)分布于除和ARF19之外的每一个基因启动子区, 茉莉酸甲酯响应元件(CGTCA-motif, TGACG-motif, MeJA-responsive element)分布在除ARF14、、、和ARF35之外的30个基因启动子区, 赤霉素响应元件P-box (gibberellin-responsive element)和GARE-motif (gibberellin-responsive element)分别位于、、、、、、、、、和、、、、、、、、、的启动子区, 生长素响应元件AuxRR-core和TGA-element分别位于、、、和、、、、、、、、、、和的启动子区。Xing等[24]分析了31个基因启动子区的生长素响应元件, 与本文结果基本吻合。Element B中的分生组织相关响应元件(CAT-box)位于、、、、、、、、、、、、、、、、和的启动子区, 种子特异调控响应元件(RY-element)分布在、、、和的启动子区, 生物钟相关响应元件(circadian)只分布在和的启动子区, 胚乳表达相关元件(GCN4_motif)只分布在和的启动子区。Element C中的低温响应元件(low temperature- responsive element, LTR)位于、、、、、、、、、、、、、和的启动子区, 干旱胁迫响应元件(MYB binding site involved in drought-inducibility, MBS)位于、、、、、、、、、、、、、、、和的启动子区, 防御胁迫响应元件(TC-rich repeats)位于、、、、和的启动子区, 水杨酸响应元件(TCA-element)位于、、、、、、、、、和的启动子区。其中, 植物激素响应相关顺式作用元件最多, 每个基因都含有2~15个; 生长发育相关顺式作用元件分布在23个基因的启动子区, 主要包括RY-element、GCN4-motif和circadian等; 逆境胁迫相关顺式作用元件主要分布在29个基因的启动子区, 主要包括MBS、LTR和TC-rich repeats等。每个基因的启动子区都包含多个顺式作用元件, 表明玉米基因在植物激素响应、生长发育和逆境胁迫响应等方面均发挥重要作用。
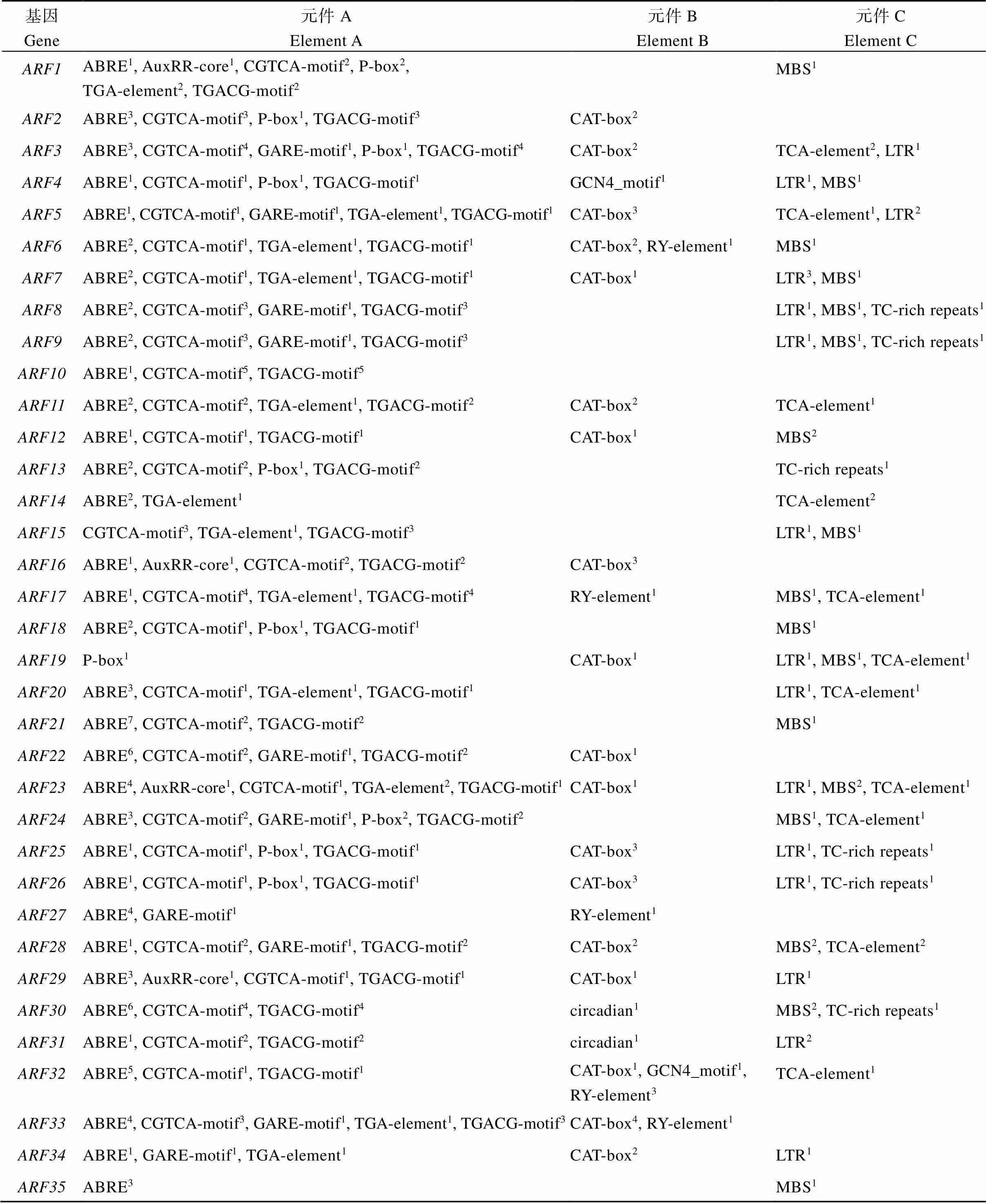
表2 玉米ARF家族基因启动子区的顺式作用元件
ABRE: 脱落酸响应元件; AuxRR-core和TGA-element: 生长素响应元件; CGTCA-motif和TGACG-motif: 茉莉酸甲酯响应元件; GARE-motif和P-box: 赤霉素响应元件; CAT-box: 分生组织相关响应元件; circadian: 生物钟相关响应元件; GCN4_motif: 胚乳表达相关元件;RY-element: 种子特异调控响应元件; LTR: 低温响应元件; MBS: 干旱胁迫响应元件; TC-rich repeats: 防御胁迫响应元件; TCA-element: 水杨酸响应元件。肩标数字代表每个顺式作用元件在基因启动区的拷贝数。
ABRE: ABA-responsive element; LTR: low temperature-responsive element; MBS: MYB binding site involved in drought-inducibility. The shoulder number represents the number of copies of each cis-acting element in thegene promoter region.
2.4 玉米ARF家族基因在冷、热、盐和渗透胁迫条件下的表达分析
顺式作用元件分析显示有29个玉米基因启动子区包含逆境胁迫相关顺式作用元件。为进一步研究玉米家族基因在逆境胁迫响应过程中的作用, 我们选取了9个在叶片中表达量较高的基因(、、、、、、、和), 利用实时定量荧光PCR方法检测玉米家族基因在冷、热、盐及渗透胁迫下的表达量变化(图3)。在冷胁迫处理下, 所有被检测基因的表达模式都是类似的, 在处理1 h时表达量迅速上升, 随后降到较低水平, 处理4 h后又开始上升。在热胁迫处理下,基因1 h时表达量达到峰值, 是未处理时的25倍, 随后又逐渐下降, 而其余8个基因的表达量则是随着时间逐渐上升。在盐胁迫处理下, 多数基因的表达量变化不大(除了在处理4 h时表达量达到峰值随后又开始下降外), 比较有趣的一点是,的表达量在处理后迅速下降并维持在较低水平, 暗示该基因可能是通过与其他基因相反的途径参与盐胁迫调控过程。在渗透胁迫处理下,、和的表达量是先上升后下降, 在处理8 h时表达量达到峰值;、、和的表达模式是相似的, 处理1 h时表达量达到峰值, 随后下降并维持在较低水平;和的表达量变化不大。这些结果表明,基因涉及参与多种逆境胁迫响应过程。
3 讨论
作为重要的植物激素, 生长素几乎参与了植物生长发育的每个阶段。而基因作为生长素响应因子, 通过特异性地结合生长素响应元件调节下游靶基因的转录, 参与诸多植物生长发育过程[2]。根据标记基因表达情况可将家族分为3类: 一类(Clade A)具有转录激活功能, 一类(Clade B)具有转录抑制功能, 一类(Clade C)既不具有转录激活功能也不具有转录抑制功能[25]。这3类基因的功能分类与进化关系是一致的[26]。结合不同数据库分析得到的转录组数据和实时定量PCR结果可以看出, Clade C主要在幼嫩雄花序中优势表达, 而Clade A和Clade B则主要在幼嫩花序中高水平表达, 有的在幼嫩雌花序中优势表达, 有的在雄花序中优势表达。不管基因通过转录激活(Clade A)还是转录抑制(Clade B), 又或者通过别的调控途径(Clade C), 都会通过参与调控生长素相关基因影响玉米花序发育过程。
A: 冷胁迫处理; B: 热胁迫处理; C: NaCl胁迫处理; D: 渗透胁迫处理。
A: cold stress treatment; B: hot stress treatment; C: NaCl stress treatment; D: osmotic stress treatment.
基因参与了诸多生物学过程。拟南芥中的突变体花期、叶衰亡和花脱落时间推迟[27], 推测与之同源的玉米和也参与花发育和叶片发育, 而这2个基因都在幼嫩花序中优势表达, 也从侧面印证这一推测;参与胚模式建成和维管组织形成[12,28-29], 而作为同源基因的玉米在幼嫩花序和中柱中高表达,在幼嫩花序和果皮中高表达, 这也暗示这2个基因可能参与胚发育和微管形成;调控花成熟[16,30], 同源基因玉米和在幼嫩花序和胚中高表达, 可能参与花和果穗形成过程;是果实起始发育的负调控因子[16,30], 黄瓜的同源基因和在单性结实和非单性结实品种的表达差异大[31], 而玉米的同源基因和在幼嫩花序和胚中高表达, 推测参与果穗的发育和形态建成。然而玉米基因众多, 通过DAP-seq测序技术获得的顺式作用元件分析结果显示,基因在Clade A、Clade B和Clade C组间的结合位点特异性和靶基因等差异不大[32], 暗示基因功能冗余现象严重, 这也是至今还未发现有表型的玉米突变体的原因[25], 开展功能研究还需新的思路和方法。
随着玉米基因组测序完成, 高通量测序技术被广泛应用于基因功能研究。通过分析数据库收录的测序数据, 可以获得不同部位组织器官在不同发育时期、不同环境条件下的表达模式。Liu等[20]通过EST数据库预测基因主要在雄穗和果穗中表达, Gallavotti等[26]通过数据库数据分析基因在胚、幼嫩花序和成熟花序中优势表达, 而本文结合实时定量PCR方法和现有数据库更新的转录组数据分析了玉米基因在不同组织器官的表达模式, 发现基因主要在雄花序和雌花序中优势表达, 充分说明基因在花序发育过程中发挥重要功能。
基因在植物的生长发育过程中发挥了重要作用, 也参与了许多逆境胁迫响应过程。拟南芥参与维管形成, 而干旱条件下植株的维管合成能力加强, 暗示通过参与维管合成而影响植株的抗旱性[6];和通过控制根冠的发育来影响植株的抗旱性[33]; 大豆基因的表达量在水分胁迫下发生明显变化, 暗示基因参与了大豆水分胁迫响应[34]。玉米中的基因在冷、热、盐和渗透胁迫处理下, 表达量均发生了明显变化, 这暗示基因参与了玉米多种胁迫响应过程。
4 结论
玉米基因家族的、和呈组成型表达, 其余32个基因均在幼嫩雄花序和雌花序中高表达, 参与了玉米花序发育过程; 玉米基因家族在冷、热、盐和渗透胁迫处理下表达量发生明显变化, 暗示玉米基因家族参与了多种逆境胁迫响应过程。
[1] 吴蓓, 吴建勇, 蔡刘体, 李运合, 黄学林. 生长素反应因子. 植物生理学通讯, 2005, 41: 273–278.Wu B, Wu J Y, Cai L T, Li Y H, Huang X L. Auxin response factor., 2005, 41: 273–278 (in Chinese).
[2] Abel S, Theologis A. Early genes and auxin action., 1996, 111: 9.
[3] Ulmasov T, Hagen G, Guifoyle T J. Dimerization and DNA binding of auxin response factors., 1999, 19: 309–319.
[4] Liu Z B, Hagen G, Guifoyle T J. A G-box-binding protein from soybean binds to the E1 auxin-response element in the soybean GH3 promoter and contains a proline-rich repression domain., 1997, 115: 397–407.
[5] Wang D, Pei K, Fu Y, Sun Z, Li S, Liu H, Tang K, Han B, Tao Y Z. Genome-wide analysis of the auxin response factors () gene family in rice ()., 2007, 394: 13–24.
[6] Singh V K, Rajkumar M S, Garg R, Jain M. Genome-wide identification and co-expression network analysis provide insights into the roles of auxin response factor gene family in chickpea., 2017, 7: 10895.
[7] Die J V, Gil J, Millan T. Genome-wide identification of the auxin response factor gene family in., 2018, 19: 301.
[8] Zhou X, Wu X, Li T, Jia M, Liu X, Zou Y, Liu Z X, Wen F. Identification, characterization, and expression analysis of auxin response factor () gene family in., 2018, 18: 709–724.
[9] Xu Z, Ji A, Song J, Chen S. Genome-wide analysis of auxin response factor gene family members in medicinal model plant., 2016, 5: 848–857.
[10] Wang S, Hagen G, Guilfoyle T J. ARF-Aux/IAA interactions through domain III/IV are not strictly required for auxin- responsive gene expression., 2013, 8: e24526.
[11] Remington D L, Vision T J, Guilfoyle T J, Reed J W. Contrasting modes of diversification in theandgene families., 2004, 135: 1738–1752.
[12] Hardtke C S, Berleth T. Thegeneencodes a transcription factor mediating embryo axis formation and vascular development., 1998, 17: 1405–1411.
[13] Harper R M, Stowe-Evans E L, Luesse D R, Muto H, Tatematsu K, Watahiki M K, Yamamoto K, Liscum E. The NPH4 locus encodes the auxin response factor, a conditional regulator of differential growth in aerialtissue., 2000, 12: 757–770.
[14] Okushima Y, Overvoorde P J, Aeima K, Alonso J M, Chan A, Chang C, Ecker J R, Hughes B. Lui A, Nguyen D. Functional genomic analysis of thegene family members in: unique and overlapping functions ofand., 2005, 17: 444–463.
[15] Wilmoth J C, Wang S, Tiwari S B, Joshi A D, Hagen G, Guifoyle T J, Alonso J M, Ecker J R, Reed J W. NPH4/ARF7 and ARF19 promote leaf expansion and auxin-induced lateral root formation., 2005, 43: 118–130.
[16] Nagpal P, Ellis C M, Weber H, Ploense S E, Barkawi L S, Guilfoyle T J, Hagen G, Alonso J M, Cohen J D, Farmer E E. Auxin response factors ARF6 and ARF8 promote jasmonic acid production and flower maturation., 2005, 132: 4107–4118.
[17] Faivre-rampant O, Cardle L, Marshall D, Viola R, Taylor M A. Changes in gene expression during meristem activation processes inwith a focus on the regulation of an auxin response factor gene., 2004, 55: 613–622.
[18] Waller F, Furuya M, Nick P., an auxin response factor from rice, is auxin-regulated and classifies as a primary auxin responsive gene., 2002, 50: 415–425.
[19] Jones B, Frasse P, Olmos E, Zegzouti H, Li Z G, Latche A, Pech J C, Bouzayen M. Down-regulation of, an auxin response factor homolog, in the tomato results in a pleiotropic phenotype including dark green and blotchy ripening fruit., 2002, 32: 603–613.
[20] Liu Y, Jiang H Y, Chen W J, Qian Y X, Ma Q, Cheng B J, Zhu S W. Genome-wide analysis of the auxin response factor () gene family in maize ()., 2011, 63: 225–234.
[21] Grennan A K. Genevestigator. Facilitating web-based gene expression analysis., 2006, 141: 1164–1166.
[22] Lescot M, Dehais P G, Marchal K, Moreau Y, Peer Y V, Rouze P, Rombauts S. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences., 2002, 30: 325–327.
[23] Galli M, Liu Q, Moss B L, Malcomber S, Li W, Gaines C, Federici S, Roshkovan J, Meeley R, Nemhauser J L, Gallavotti A. Auxin signaling modules regulate maize inflorescence architecture., 2015, 112: 13372–13377.
[24] Xing H Y, Pudake R N, Guo G G, Xing G F, Hu Z R, Zhang Y R, Sun Q X, Ni Z F. Genome-wide identification and expression profiling of auxin response factor () gene family in maize., 2011, 12: 178.
[25] Ulmasov T, Hagen G, Guilfoyle T J. Activation and repression of transcription by auxin response factors., 1999, 96: 5844–5849.
[26] Matthes M S, Best N B, Robil J M, Malcomber S, Gallavotti A, Mcsteen P. Auxin evodevo: conservation and diversification of genes regulating auxin biosynthesis, transport, and signaling., 2019, 12: 298–320.
[27] Liu Z, Miao L, Huo R, Song X, Johnson C, Kong L, Sundaresan V, Yu X L. ARF2-ARF4 and ARF5 are essential for female and male gametophyte development in., 2018, 59: 179–189.
[28] Hardtke C S, Berleth T. Thegeneencodes a transcription factor mediating embryo axis formation and vascular development., 1998, 17: 1405–1411.
[29] Wenzel C L, Schuetz M, Yu Q, Mattsson J. Dynamics ofandexpression during leaf vein pattern formation in., 2007, 49: 387–398.
[30] Wu M F, Tian Q, Reed J W. Arabidopsis microRNA167 controls patterns ofandexpression, and regulates both female and male reproduction., 2006, 133: 4211–4218.
[31] 王垒, 陈劲枫, 娄丽娜, 娄群峰. 黄瓜ARF家族序列特征及部分成员在果实发育早期的表达分析. 园艺学报, 2011, 4: 717–724. Wang L, Chen J F, Lou L N, Lou Q F. Sequence characteristics ofgene family of cucumber and an expression analysis of some members during early development of fruits., 2011, 4: 717–724 (in Chinese with English abstract).
[32] Galli M, Khakhar A, Lu Z, Chen Z, Sen S, Joshi T, Nemhauser J L, Schmitz R J, Gallavotti A. The DNA binding landscape of the maizefamily., 2018, 9: 4526.
[33] 王佳伟, 蔡文娟, 王凌健, 毛颖波, 陈晓亚. 生长素响应因子和控制拟南芥根冠发育. 见: 中国遗传学会植物遗传与基因组学专业委员会2005年学术研讨会论文摘要集, 2005. p 15.Wang J W, Cai W J, Wang L J, Mao Y B, Chen X Y. Auxin response factorsandcontrol root crown development in. In: Genetics Society of China in plant Genetics and Genomics Professional Committee Conference Abstract Set, 2005. p 15 (in Chinses).
[34] Ha C V, Le D T, Nishiyama R, Watanabe Y, Sulieman S, Tran U T, Mochida K, Dong N V, Yamaguchi-Shinozaki K, Shinozaki K, Tran L S. The auxin response factor transcription factor family in soybean: genome-wide identification and expression analyses during development and water stress., 2013, 20: 511–524.
A study of expression pattern of auxin response factor family genes in maize (L.)
LI Wen-Lan, LI Wen-Cai, SUN Qi, YU Yan-Li, ZHAO Meng, LU Shou-Ping, LI Yan-Jiao, and MENG Zhao-Dong*
Maize Research Institute, Shandong Academy of Agricultural Sciences / National Engineering Laboratory of Wheat and Maize / Key Laboratory of Biology and Genetic Improvement of Maize in Northern Yellow-Huai Rivers Plain, Ministry of Agriculture, Jinan 250100, Shandong, China
Auxin response factors (ARFs) are important transcription factors which control the expression of target genes by binding specifically to auxin response elements, and are involved in a series of developmental processes in plant species. In maize genome, dozens ofgenes are encoded, however, there is little known on their expression patterns. In this study, the analysis on the expression level ofgenes in diverse tissues and organs revealed that expression level of 32genes were higher in reproductive organs than that in vegetative organs, except,, andconstitutively expressed. The predicted results of-acting elements showed that the promoter regions of 28genes harbored the-regulatory elements related to abiotic stresses. Real-time quantitative PCR results indicated that expression of severalgenes showed a response to cold, heat, and osmotic stresses, respectively. The results highlighted the importance offamily genes in reproductive growth and abiotic stress response, and provided useful information for the comprehensive analysis of the biological function ofgenes in maize.
auxin response factor; expression; abiotic stress; maize (L.)
10.3724/SP.J.1006.2021.03043
本研究由山东省自然科学基金项目(ZR2019BC107)资助。
This study was supported by the Natural Science Foundation of Shandong Province (ZR2019BC107).
孟昭东, E-mail: mengzhd@126.com
E-mail: liwenlantutu@126.com
2020-07-07;
2020-11-13;
2020-12-15.
URL: https://kns.cnki.net/kcms/detail/11.1809.s.20201214.1748.004.html
——植物学教材质疑(六)

