花生巢式群体的脂肪含量遗传分析
黄冰艳 孙子淇 刘 华 房元瑾 石 磊 苗利娟 张毛宁 张忠信 徐 静 张梦圆 董文召 张新友,*
花生巢式群体的脂肪含量遗传分析
黄冰艳1,**孙子淇1,**刘 华1房元瑾1石 磊1苗利娟1张毛宁1张忠信1徐 静1张梦圆2董文召1张新友1,*
1河南省农业科学院河南省作物分子育种研究院/ 郑州大学研究生研究培训基地 / 农业农村部黄淮海油料作物重点实验室 / 河南省油料作物遗传改良重点实验室, 河南郑州 450002;2河南科技大学农学院, 河南洛阳 471023
巢式群体可以利用多个亲本解析复杂性状的遗传机制。本研究利用1个共同亲本与6个基础亲本所配置巢式组合F2:3家系的种子脂肪含量数据, 分析了花生脂肪含量的遗传模型, 旨在探明不同的基础亲本组合中脂肪含量性状的遗传差异, 为制定脂肪含量遗传改良的亲本选配和后代选择策略提供依据。6个组合的共同亲本为高脂肪含量的普通型大果品种豫花15号, 其他6个基础亲本为不同脂肪含量和不同植物学类型的品种。结果表明, 在不同杂交组合中脂肪含量的遗传模式有所不同, 6个组合分别符合无主基因模型、1对主基因加性显性模型和2对主基因等显性模型3种遗传模式。各种遗传效应的估计值也各不相同, 主基因遗传力从32%到80%, 说明不同杂交组合中, 控制脂肪含量的基因位点差异及其重组和分离方式不同。高脂肪含量双亲杂交后代的高脂肪含量个体较多, 但主基因遗传力较低, 不宜在早代实施表型选择; 双亲脂肪含量差异较大的后代脂肪含量变异幅度更大, 能够选择到不同脂肪含量的类型。本研究也表明, 巢式组合具有较丰富的脂肪含量变异类型, 揭示出脂肪含量性状遗传的复杂性和多基因调控的特点, 为较全面地了解脂肪含量的遗传提供了基础。该巢式群体也将有助于进一步开展脂肪含量的QTL定位研究。
花生(L.); 巢式群体; 脂肪含量; F2:3家系; 遗传模型
花生广泛种植于非洲、亚洲和美洲的100多个国家, 2018年世界花生种植面积达到2800万公顷, 总产4500余万吨[1]。花生是中国、印度、阿根廷、塞内加尔等国家植物油、蛋白营养和农民收入的主要来源, 也是美国、澳大利亚等国家的重要食品加工原料。我国是世界第一花生生产和消费大国, 花生油是深受城乡居民喜爱的优质烹调用油, 我国的花生50%以上用于榨油, 花生生产对保障我国食用油安全具有重要意义。
不同的花生品种其种子脂肪含量具有较大差异, 在我国的花生种质资源中, 花生籽仁的含油量变异幅度为45%~60%。花生品种按照植物学特征分为普通型、龙生型、珍珠豆型、多粒型和中间型5种类型, 花生的脂肪含量与品种类型相关, 也受种植环境和气候的影响, 提高花生品种的脂肪含量及单位面积的脂肪产量, 是我国花生育种的重要目标。
我国目前推广的主要花生品种脂肪含量平均为51.4%, 略高于种质资源的平均数[2]。脂肪含量高于55%的高油花生品种多为中粒型品种, 主要通过常规的杂交育种技术选育而成, 存在育种周期长、性状预见性差、选择效率低等突出问题。因此, 深入阐明花生脂肪含量遗传的分子机制, 能够为脂肪含量的品质育种提供亲本选配和后代选择的依据, 并且对于进一步开发与脂肪含量关联的分子标记或者调控基因位点, 构建分子设计育种体系具有重要意义。
近年来, 我国学者利用主基因加多基因遗传模型对花生的脂肪含量性状开展了遗传分析。禹山林等[3]研究结果显示, 该性状受2对加性-显性-上位性主基因+加性-显性多基因控制, 多基因遗传力为72.55%。陈四龙等[4]研究结果表明, 有些组合符合1对加性-显性主基因+加性-显性-上位性基因控制, 主基因遗传力为45%~47%, 多基因遗传力为18%~22%; 有些组合多基因遗传力为66%。Zhang等[5]和刘华等[6-7]的研究也得出多基因效应比较大的结论。这些研究初步探明了花生脂肪含量的主要遗传模式, 但是, 由于所用亲本材料及试验环境不同, 难以相互比较分析。本研究利用高脂肪含量品种为共同亲本, 构建了包括6个不同杂交组合的巢式群体, 通过对巢式群体F2:3世代种子脂肪含量的分析, 揭示花生脂肪含量的遗传模式, 旨在为较全面理解花生的脂肪含量性状遗传和进一步的分子机制解析提供基础。
1 材料与方法
1.1 群体材料
以豫花15号为共同亲本的巢式群体包括6个亲本组合(表1)。豫花15号是我国大面积推广的高产高油大果花生品种, 由河南省农业科学院选育, 相继通过了河南省、安徽省、北京市和国家品种审定委员会审定, 品种类型为中间型, 脂肪含量高达56%以上。另外6个基础亲本分别为远杂9102、中花6号、粤油20、四粒红、伏花生和NC94022, 分别来自我国的黄淮海地区、长江流域、华南、东北、华东等主要花生产区和美国。其中, 远杂9102、中花6号、粤油20为珍珠豆型高产品种, 分别为河南、湖北和广东选育, 脂肪含量55%左右; NC94022为美国普通型小果(兰娜型)品种, 脂肪含量约为54%; 四粒红为多粒型品种, 脂肪含量约为53%。
1.2 田间种植
2018年春季, 经过分子鉴定为真杂种的F1种子及其亲本按照组合种植于河南省农业科学院原阳试验基地; 2019年春季, 种植F2种子及其亲本, 田间按照常规管理, 秋季正常成熟后按照单株收获、晾晒荚果。

表1 巢式杂交群体信息
A: 远杂9102; B: 中花6号; C: 豫花15号; E: 粤油20; F: 四粒红; G: 伏花生; H: NC94022。
A: Yuanza 9102;.B: Zhonghua 6; C: Yuhua 15; E: Yueyou 20; F: Silihong; G: Fuhuasheng; H: NC94022.
1.3 脂肪含量测定
从F2单株上挑选成熟荚果, 剥取饱满籽粒, 利用DA7200型近红外分析仪(瑞典, Perten)测定脂肪含量, 样本容量为10粒, 取3次重复平均数。
1.4 模型分析
采用王建康等[8-9]提出的植物数量性状遗传模型主基因加多基因F2世代分析方法, 以AIC (Akaike’s Information Criterion)值较小的遗传模型为备选模型; 对备选模型进行适合性测验, 包括均匀性检验(12、22、32)、Smirnov检验(2)和Kolmogorov检验(D), 统计量均不显著或者显著个数最少的模型为最适模型。按入选的遗传模型估计相应的遗传参数, 包括基因的各种效应值、方差及遗传力等。
2 结果与分析
2.1 不同杂交组合F2脂肪含量的群体分布特征
从6个组合的脂肪含量分布图可以看出, F2脂肪含量呈现连续分布特征, 6个组合的脂肪含量变幅分别为52.31%~63.87%、51.11%~61.68%、49.14%~ 58.82%、47.55%~58.87%、47.04%~56.41%、48.88%~ 59.53%, 并且分别表现出低于低亲和高于高亲的超亲现象(图1)。
远杂9102×豫花15号、中花6号×豫花15号、豫花15号×粤油20三个组合的父母本脂肪含量差异不大, 但F2表现出2种不同的脂肪分布特点, 远杂9102×豫花15号组合F2表现出高于高亲的个体数及极端高的个体数均较多, 而且没有脂肪含量低于52%的个体; 中花6号×豫花15号组合和豫花15号×粤油20组合的F2的脂肪分布则表现偏向低脂肪含量亲本方向, 低于低亲的个体数和变异范围均多于高于高亲的个体数。
豫花15号×四粒红、豫花15号×伏花生、豫花15×NC94022三个组合的亲本脂肪含量差异较大, 其F2脂肪含量分布均偏向低脂肪含量亲本方向, 低于低亲的个体数显著多于高于高亲的个体数, 尤其是豫花15号×伏花生组合, 没有个体的脂肪含量高于高亲品种, 但豫花15号×NC94022组合有一定数量的F2个体脂肪含量高于高亲品种。
2.2 脂肪含量遗传模型的建立
根据主基因加多基因模型分别对各组合进行最大似然值和AIC值估计, 根据组合将多个拟合模型的AIC值按照从小到大排列(表2), 分别为无主基因(0 Major Gene, 0MG)、1对主基因(1MG)、2对主基因(2MG)的加性(A)、加性显性(AD)、完全显性(CD)、负向完全显性(NCD)、等加性(EA)、等显性(EAD)等模型。
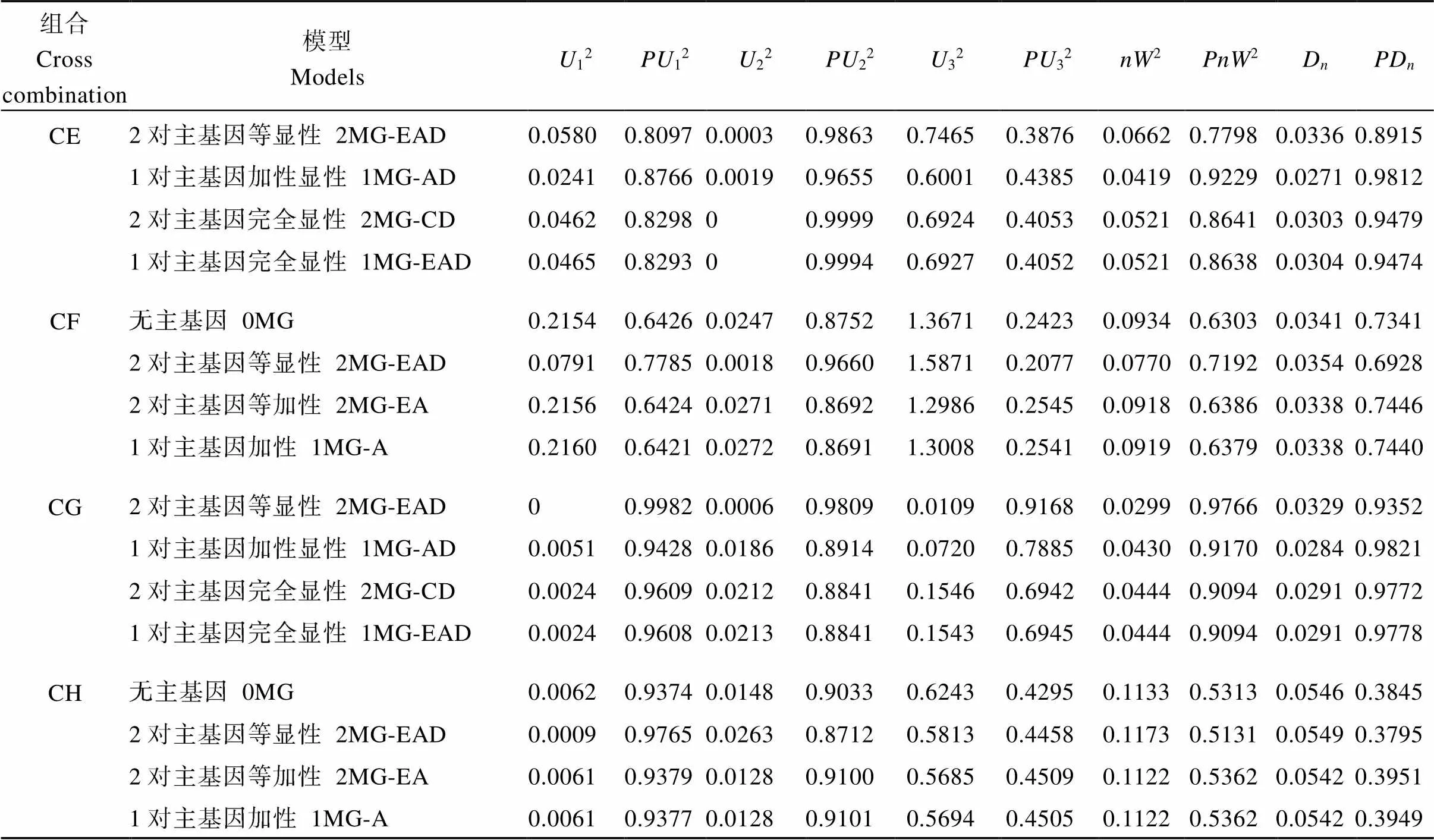
各个组合的各个遗传模型的适合性检验见表3,12、22、32、2、D五个特征参数的值均大于0.05, 未达到显著水平。说明模型的拟合度均具有统计学意义。
2.3 基于不同模型的遗传参数估计
根据AIC值最小的原则选择最适合的模型, 6个组合分别符合1对主基因加性显性模型(1MG-AD), 无主基因(0MG)、2对主基因等显性模型(2MG-EAD) 3类遗传模式, 主基因遗传力32%~80% (表4)。远杂9102和豫花15号的杂交组合表现1对主基因的加性显性遗传模式, 但主基因遗传力仅为32%; 豫花15号与中花6号、粤油20、伏花生3个杂交组合均表现2对主基因的等显性模型, 主基因遗传力为47%~80%; 豫花15号与四粒红、NC94022等2个杂交组合表现出无主基因的遗传模式。表明不同遗传背景的品种的脂肪含量调控基因的差异。
3 讨论
我国油料作物脂肪含量的遗传研究经历了双列杂交配合力分析[10-14]、双亲群体的多世代联合分析主基因加多基因遗传模型[3,5-7,15-16]、双亲群体的QTL定位等多种数量遗传学研究方法的发展[17-21]。但迄今为止, 定位到的脂肪含量QTL由于易受环境影响或者定位区间过大, 尚未成功应用于育种实践。借鉴人类利用全基因组关联分析(genome-wide association study, GWAS)的成功经验, 自然群体的GWAS分析也在油料作物的脂肪含量关联位点方面得到应用[22-23]。GWAS具有等位基因多样性丰富的优点, 分子标记密度也大大提高, 但是存在难以定位稀有等位基因的缺陷, 而且自然群体的结构和分层现象严重干扰性状关联位点的精确解析[24]。
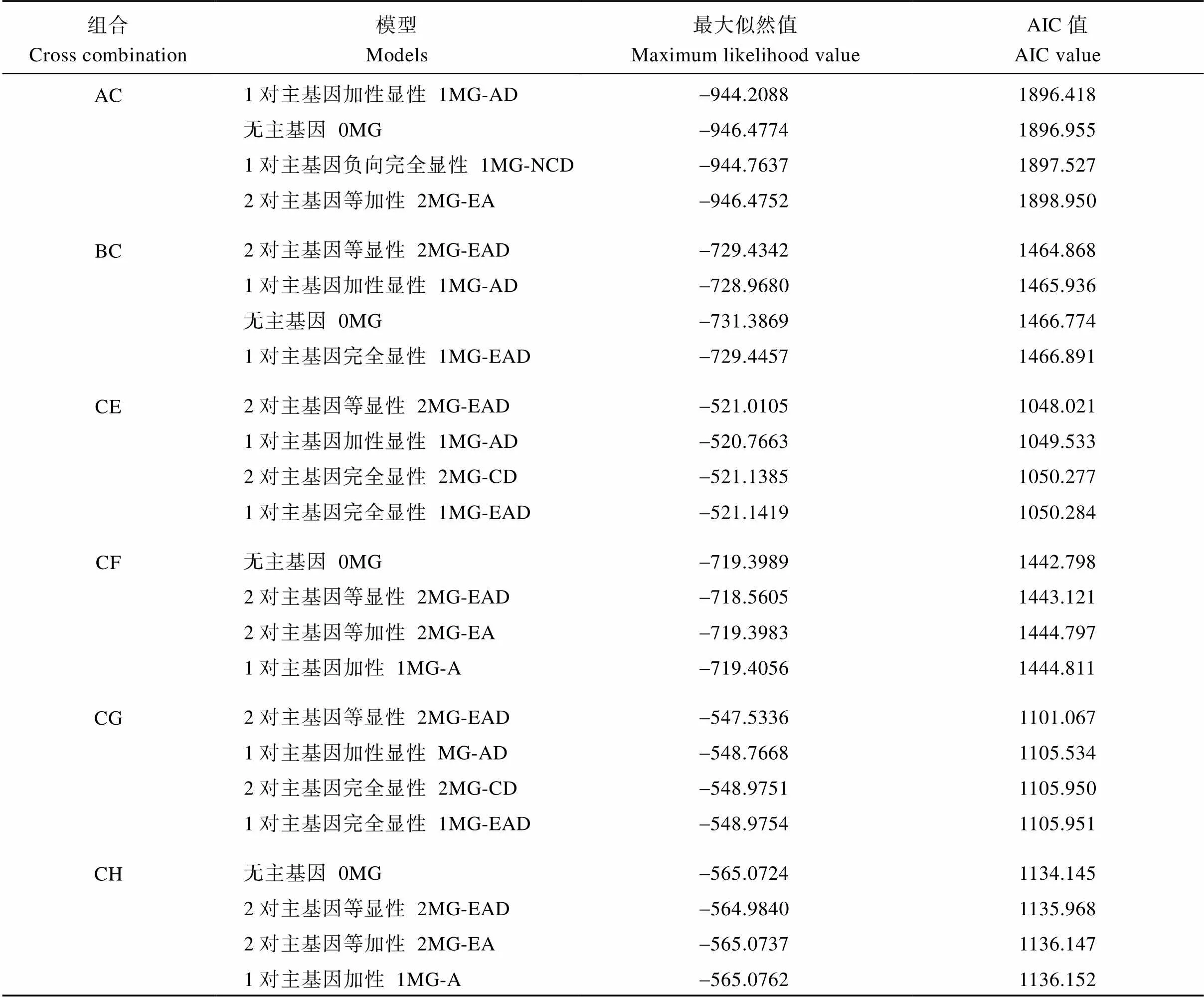
表2 脂肪含量遗传模式分析
A: 远杂9102; B: 中花6号; C: 豫花15号; E: 粤油20; F: 四粒红; G: 伏花生; H: NC94022。
A: Yuanza 9102;.B: Zhonghua 6; C: Yuhua 15; E: Yueyou 20; F: Silihong; G: Fuhuasheng; H: NC94022. MG: major gene; AD: additive and dominant; NCD: negative complete dominant; EA: equal additive; EAD: equal dominant. AIC: Akaike’s Information Criterion.
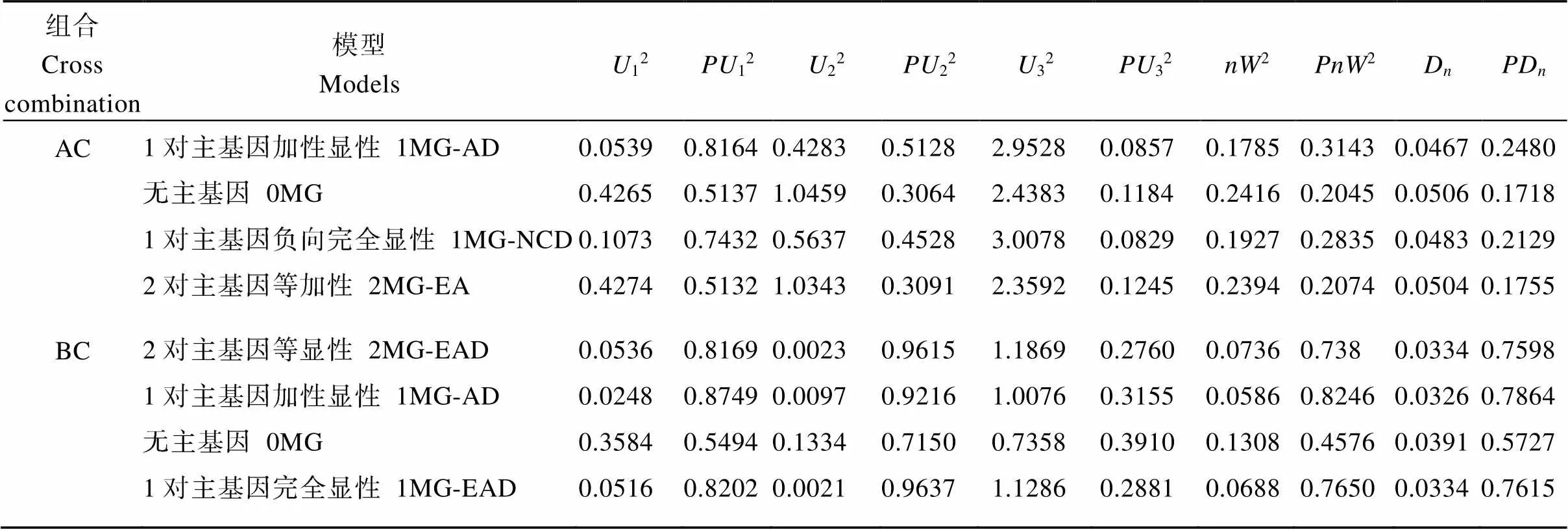
表3 遗传模式的适合性测验
(续表3)
A: 远杂9102; B: 中花6号; C: 豫花15号; E: 粤油20; F: 四粒红; G: 伏花生; H: NC94022。12、22、32、2、D分别为均匀性检验、Smirnov-Kolmogorov检验的特征参数。
A: Yuanza 9102; B: Zhonghua 6; C: Yuhua 15; E: Yueyou 20; F: Silihong; G: Fuhuasheng; H: NC94022.12,22,32,2,Dare parameters of Utility test, Smirnov test and Kolmogorov test. MG: major gene; AD: additive and dominant; NCD: negative complete dominant; EA: equal additive; EAD: equal dominant.
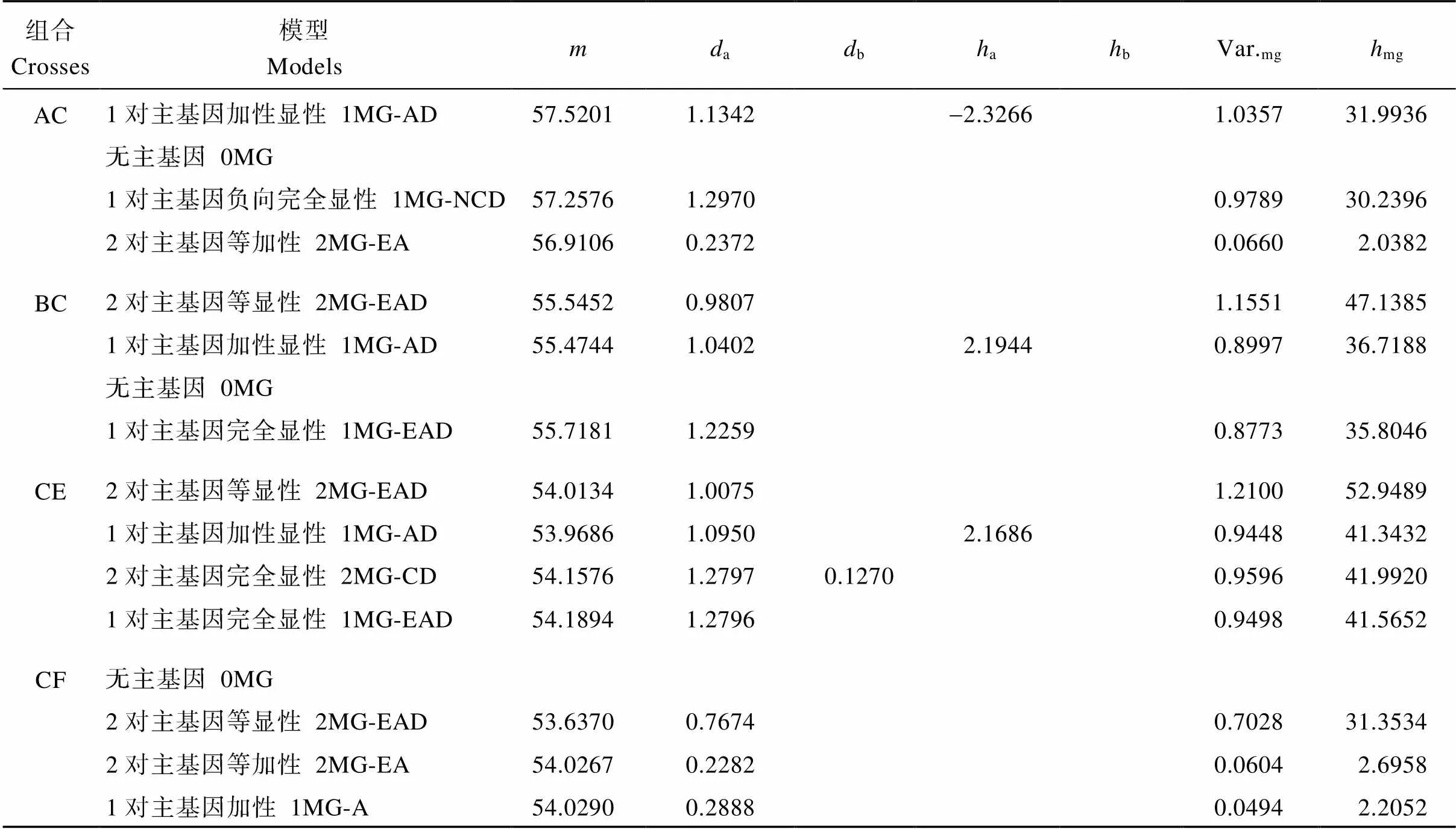
表4 脂肪含量遗传参数估计
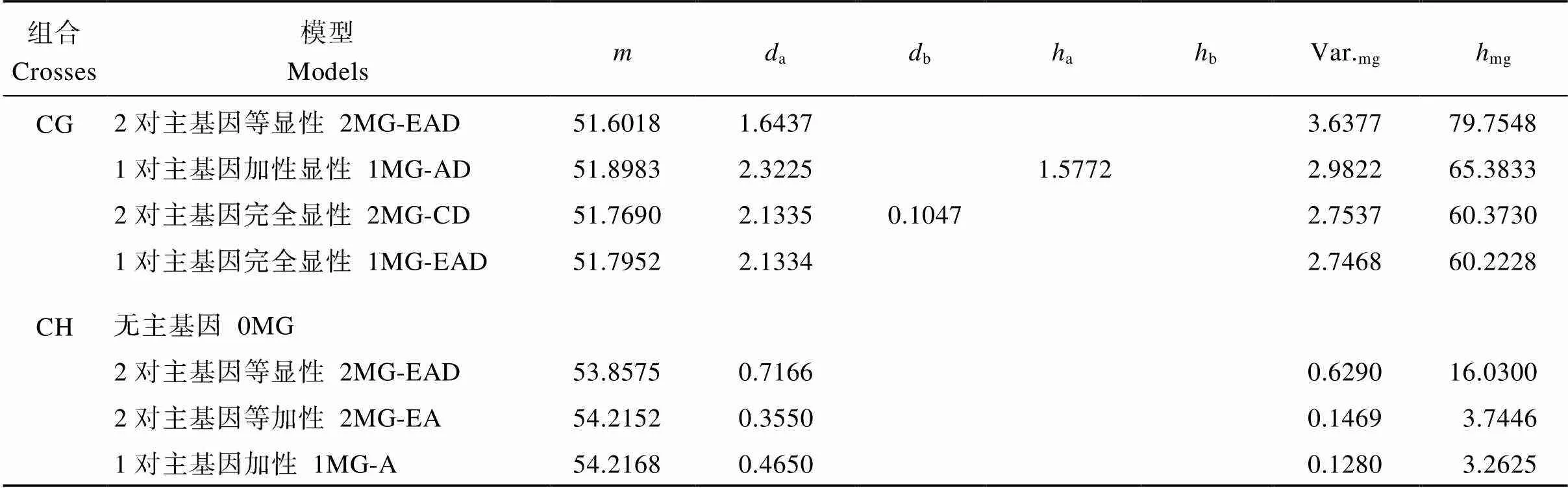
(续表4)
A: 远杂9102; B: 中花6号; C: 豫花15号; E: 粤油20; F: 四粒红; G: 伏花生; H: NC94022。: 群体平均数;a: 第1对主基因加性效应;b: 第2对主基因加性效应;a: 第1对主基因显性效应;b: 第2对主基因显性效应; Var.mg: 主基因方差;mg: 主基因遗传力。
A: Yuanza 9102; B: Zhonghua 6; C: Yuhua 15; E: Yueyou 20; F: Silihong; G: Fuhuasheng; H: NC94022. m:mean of population;a: additive effect of the first major gene;b: additive effect of the second major gene;a: dominant effect of the first major gene;b: dominant effect of the second major gene; Var.mg: major gene variance;mg: heritability of major gene. MG: major gene; AD: additive and dominant; NCD: negative complete dominant; EA: equal additive; EAD: equal dominant.
巢式关联作图群体(nested association mapping, NAM)弥补了上述2种群体缺点, 近年来逐渐成为农作物数量性状基因定位的研究热点。NAM群体综合考虑了稀有基因频率和复等位基因等问题, 图谱分辨率得到显著提高[25-26]。Bouchet等[27]利用高粱的NAM群体进行开花期和株高的QTL定位表明, QTL定位效率比关联分析提高了3倍。其他作物利用NAM群体成功解析了复杂性状的关联基因, 例如大麦[28]、大豆[29-32]、玉米[33-34]、油菜[35]、小麦[36-37]。Marla等[38]利用NAM群体解析获得了5~10个高粱耐冷QTL, 表型解释率达到20%~41%。基于花生基因组研究的快速进展, Gangurde等[39]利用2个NAM群体解析了花生百果重和百仁重的QTL, 分别获得8个和13个相关QTL。
本研究构建了6个具有共同亲本的巢式群体并利用F2:3开展了花生脂肪含量的遗传模式分析, 结果表明, 6个组合的脂肪含量遗传模型表现出多样性,分别属于无主基因、1对主基因加性显性模型、2对主基因等显性模型3种模式, 各种效应值也各不相同, 主基因遗传力从32%到80%。这些结果能够为脂肪含量遗传改良工作中亲本选配和后代选择提供参考, 高脂肪含量双亲杂交后代中高脂肪含量个体较多, 但主基因遗传力较低, 不宜在早代实施表型选择; 双亲脂肪含量差异较大的杂交后代中脂肪含量变异幅度更大, 能够选择到不同脂肪含量的类型, 但选择出脂肪含量超高亲后代的概率很低, 根据杂交组合的不同, 遗传力变幅也较大。另外, 本研究显示了巢式群体在揭示花生脂肪含量遗传复杂性和创制丰富变异类型方面的应用潜力, 同时也为进一步利用该群体的高世代NAM开展联合QTL定位和脂肪含量的遗传解析等奠定了材料基础。
4 结论
本研究利用巢式群体揭示出花生脂肪含量的遗传表现出多种模式, 受到多对主基因调控, 分别具有加性和显性等遗传效应。巢式群体包括了共同的杂交亲本和更多样性的基础亲本, 遗传变异类型丰富, 具有更加全面地解析重要农艺性状遗传调控机制的潜力。
[1] FAOSTAT, 2020. FAO. http://www.fao.org/faostat/en/#home.
[2] 廖伯寿, 雷永, 王圣玉, 李栋, 黄家权, 姜慧芳, 任小平. 花生重组近交系群体的遗传变异与高油种质的创新. 作物学报, 2008, 34: 999–1004. Liao B S, Lei Y, Wang S Y, Li D, Huang J Q, Jiang H F, Ren X P. Genetic diversity of peanut RILs and enhancement for high oil genotypes., 2008, 34: 999–1004 (in Chinese with English abstract).
[3] 禹山林, 杨庆利, 潘丽娟, 薄文娜. 花生种子含油量的遗传分析. 植物遗传资源学报, 2009, 10: 453–456. Yu S L, Yang Q L, Pan L J, Bo W N. Genetic analysis for oil content of peanut seeds., 2009, 10: 453–456 (in Chinese with English abstract).
[4] 陈四龙, 李玉荣, 程增书, 廖伯寿, 雷永, 刘吉生. 花生含油量杂种优势表现及主基因+多基因遗传效应分析. 中国农业科学, 2009, 42: 3048–3057. Chen S L, Li Y R, Cheng Z S, Liao B S, Lei Y, Liu J S. Heterosis and genetic analysis of oil content in peanut using mixed model of major gene and polygene., 2009, 42: 3048–3057 (in Chinese with English abstract).
[5] Zhang X, Zhu S, Han S, Xu J, Liu H, Tang F, Dong W, Zang X, Zhang Z X. Inheritance of fat and fatty acid compositions in peanut (L.)., 2011, 12: 943–946.
[6] 刘华, 张新友, 崔党群, 汤丰收, 董文召, 韩锁义, 徐静, 臧秀旺, 张忠信. 花生蛋白质和脂肪含量的主基因+多基因遗传分析. 江苏农业科学, 2011, 39(2): 127–130. Liu H, Zhang X Y, Cui D X, Tang F S, Dong W Z, Han S Y, Xu J, Zang X W, Zhang Z X. Genetic analysis of protein and fat content using major gene plus polygene methods in peanut (L.)., 2011, 39(2): 127–130 (in Chinese with English abstract).
[7] 刘华, 秦利, 张新友, 韩锁义, 杜培, 张忠信, 孙子淇, 齐飞艳, 董文召, 黄冰艳. 基于花生种间杂交遗传群体的脂肪及脂肪酸含量的遗传模型分析. 中国油料作物学报, 2016, 38: 172–178. Liu H, Qin L, Zhang X Y, Han S Y, Du P, Zhang Z X, Sun Z Q, Qi F Y, Dong W Z, Huang B Y. Genetic models of peanut fat and fatty acid compositions based on interspecific RIL population., 2016, 38: 172–178 (in Chinese with English abstract).
[8] 王建康, 盖钧镒. 利用杂种F2世代鉴定数量性状主基因-多基因混合遗传模型并估计其遗传效应. 遗传学报, 1997, 24: 432–440. Wang J K, Gai J Y. Identification of major gene and polygene mixed inheritance model and estimation of genetic parameters of a quantitative trait from F2progeny., 1997, 24: 432–440 (in Chinese with English abstract).
[9] Meng L, Li H, Zhang L, Wang J. QTL IciMapping: integrated software for genetic linkage map construction and quantitative trait locus mapping in bi-parental populations., 2015, 3: 269–283.
[10] 甘信民, 曹玉良, 魏家祥, 崔务峰, 顾淑媛, 刘法生. 花生数量性状配合力的研究. 中国油料作物学报, 1981, (3): 33–45. Gan S M, Cao Y L, Wei J X, Cui W F, Gu S Y, Liu F S. Combining ability analysis of quatitative traits in peanut., 1981, (3): 33–45 (in Chinese with English abstract).
[11] Isleib T G, Pattee H E, Giesbrecht F G. Oil, sugar, and starch characteristics in peanut breeding lines selected for low and high oil content and their combining ability.2004, 52: 3165−3168.
[12] 徐宜民, 甘信民, 曹玉良, 顾淑媛, 刘法生. 花生主要营养品质性状和农艺性状配合力的研究. 中国农业科学, 1995, 28(2): 15–23. Xu Y M, Gan X M, Cao Y L, Gu S Y, Liu F S. studies on combining ability of major nutritional quality characters and agronomic characters in peanut., 1995, 28(2): 15–23 (in Chinese with English abstract) .
[13] 梁炫强, 郑广柔, 向荣英, 黎秀英. 珍珠豆型花生产量和含油率性状配合力分析. 花生科技, 1991, (3): 11–14. Liang X Q, Zheng G R, Xiang R Y, Li X Y. Combining ability analysis of yield and oil content of Spanish type peanut., 1991, (3): 11–14 (in Chinese with English abstract).
[14] 梁炫强, 郑广柔, 向荣英, 黎秀英. 珍珠豆型花生产量和含油率遗传特性的双列分析. 中国油料, 1992, (1): 19–23. Liang X Q, Zheng G R, Xiang R Y, Li X Y. A diallel analysis of yield and oil content of spanish type peanut., 1992, (1): 19–23 (in Chinese with English abstract).
[15] 付三雄, 戚存扣. 甘蓝型油菜含油量的主基因+多基因遗传分析. 江苏农业学报, 2009, 25: 731–736. Fu S X, Qi C K. Major gene plus polygene inheritance of oil content inL., 2009, 25: 731–736 (in Chinese with English abstract).
[16] Zhang S F, Ma C Z, Zhu J C, Wang J P, Wen Y C, Fu T D. Genetic analysis of oil content inL. using mixed model of major gene and polygene., 2006, 2: 171–180.
[17] 金梦阳, 李加纳, 付福友, 张正圣, 张学昆, 刘列钊. 甘蓝型油菜含油量及皮壳率的QTL分析. 中国农业科学, 2007, 40: 677–684. Jin M Y, Li J N, Fu F Y, Zhang Z S, Zhang X K, Liu L Z. QTL analysis of oil and hull content inL., 2007, 40: 677–684 (in Chinese with English abstract).
[18] 张洁夫, 戚存扣, 浦惠明, 陈松, 陈锋, 高建芹, 陈新军, 顾慧, 傅寿仲. 甘蓝型油菜含油量的遗传与QTL定位. 作物学报, 2007, 33: 1495–1501. Zhang J F, Qi C K, Pu H M, Chen S, Chen F , Gao J Q, Chen X J, Gu H, Fu S Z. Inheritance and QTL identification of oil content in rapeseed (L.)., 2007, 33: 1495–1501 (in Chinese with English abstract).
[19] 张新友, 韩锁义, 徐静, 严玫, 刘华, 汤丰收, 董文召, 黄冰艳. 花生主要品质性状的QTLs定位分析. 中国油料作物学报, 2012, 34: 311–315. Zhang X Y, Han S Y, Xu J, Yan M, Liu H, Tang F S, Dong W Z, Huang B Y. Identification of QTLs for important quality traits in cultivated peanut (L.)., 2012, 34: 311–315 (in Chinese with English abstract).
[20] 黄莉, 赵新燕, 张文华, 樊志明, 任小平, 廖伯寿, 姜慧芳, 陈玉宁. 利用RIL群体和自然群体检测与花生含油量相关的SSR标记. 作物学报, 2011, 37: 1967–1974. Huang L, Zhao X Y, Zhang W H, Fan Z M, Ren X P, Liao B S, Jiang H F, Chen Y N. Identification of SSR markers linked to oil content in peanut (L.) through RIL population and natural population., 2011, 37: 1967–1974 (in Chinese with English abstract).
[21] Pandey M K, Wang M L, Qiao L X, Feng S P, Khera P, Wang H, Tonnis B, Barkley N A, Wang J P, Holbrook C C, Culbreath A K, Varshney R K, Guo B Z. Identification of QTLs associated with oil content and mapping FAD2 genes and their relative contribution to oil quality in peanut (L.)., 2014, 15: 133.
[22] 魏大勇, 崔艺馨, 梅家琴, 汤青林, 李加纳, 钱伟. 油菜种子含油量GWAS分析及位点整合系统构建. 作物学报, 2018, 44: 1311–1319. Wei D Y, Cui Y X, Mei J Q, Tang Q L, Li J N, Qian W. Genome-wide association study on seed oil content in rapeseed and construction of integration system for oil content loci., 2018, 44: 1311–1319 (in Chinese with English abstract).
[23] Sun F M, Liu J, Hua W, Sun X C, Wang X F, Wang H Z. Identification of stable QTLs for seed oil content by combined linkage and association mapping in, 2016, 252: 388–399.
[24] Yu J, Holland J B, McMullen M D, Buckler E S. Genetic design and statistical power of nested association mapping in maize., 2008, 178: 539–551.
[25] Li H, Bradbury P, Ersoz E, Buckler E S, Wang J. Joint QTL linkage mapping for multiple-cross mating design sharing one common parent., 2011, 6: e17573.
[26] Xavier A, Xu S, Muir W M, Rainey K M. NAM: association studies in multiple populations., 2015, 31: 3862–3864.
[27] Bouchet S, Olatoye M O, Marla SR, Perumal R, Tesso T, Yu J M, Tuinstra M, Morris G P. Increased power to dissect adaptive traits in global sorghum diversity using a nested association mapping population., 2017, 206: 573–585.
[28] Schnaithmann F, Kopahnke D, Pillen K. A first step toward the development of a barley NAM population and its utilization to detect QTLs conferring leaf rust seedling resistance., 2014, 127: 1513–1525.
[29] Xavier A, Jarquin D, Howard R, Ramasubramanian V, Specht J E, Graef G L, Beavis W D, Diers B W, Song Q, Cregan P B, Nelson R, Mian R, Shannon J G, McHale L, Wang D, Schapaugh W, Lorenz A J, Xu S, Muir W M., Rainey K M. Genome-wide analysis of grain yield stability and environmental interactions in a multiparental soybean population.:, 2018, 8: 519–529.
[30] Khan M A, Tong F, Wang W, He J, Zhao T, Gai J. Analysis of QTL-allele system conferring drought tolerance at seedling stage in a nested association mapping population of soybean [(L.) Merr.] using a novel GWAS procedure., 2018, 248: 947–962.
[31] Chen Q, Yang C J, York A M, Xue W, Daskalska L L, DeValk C A, Krueger K W, Lawton S B, Spiegelberg B G, Schnell J M, Neumeyer M A, Perry J S, Peterson A C, Kim B, Bergstrom L, Yang L, Barber I C, Tian F, Doebley J F. TeoNAM: a nested association mapping population for domestication and agronomic trait analysis in maize., 2019,213:1065–1078.
[32] Beche E, Gillman J D, Song Q, Nelson R, Beissinger T, Decker J, Shannon G, Scaboo A M. Nested association mapping of important agronomic traits in three interspecific soybean populations., 2020, 133: 1039–1054.
[33] Garin V, Wimmer V, Mezmouk S, Malosetti M, Eeuwijk F. How do the type of QTL effect and the form of the residual term influence QTL detection in multi-parent populations? A case study in the maize EU-NAM population., 2017, 130: 1753–1764.
[34] Monir M M, Zhu J. Dominance and epistasis interactions revealed as important variants for leaf traits of maize NAM population., 2018, 9: 627.
[35] Hu J, Guo C, Wang B, Ye J, Liu M, Wu Z, Xiao Y, Zhang Q, Li H, King G J, Liu K. Genetic properties of a nested association mapping population constructed with semi-winter and spring oilseed rapes.,2018, 9: 1740.
[36] Jordan K W, Wang S, He F, Chao S, Lun Y, Paux E, Sourdille P, Sherman J, Akhunova A, Blake N K, Pumphrey M O, Glover K, Dubcovsky J, Talbert L, Akhunov E D. The genetic architecture of genome-wide recombination rate variation in allopolyploid wheat revealed by nested association mapping., 2018, 95: 1039–1054.
[37] Kidane Y G, Gesesse C A. Hailemariam B N, Desta E A, Mengistu D K, Fadda C, Pè M E, Dell’Acqua M. A large nested association mapping population for breeding and quantitative trait locus mapping in Ethiopian durum wheat., 2019, 17: 1380–1393.
[38] Marla S R., Burow G, Chopra R, Hayes C, Olatoye M O, Felderhoff T, Hu Z, Raymundo R, Perumal R, and Morris G P. Genetic architecture of chilling tolerance in sorghum dissected with a nested association mapping population.:, 2019, 9: 4045–4057.
[39] Gangurde S S, Wang H, Yaduru S, Pandey M K, Fountain J C, Chu Y, Isleib T, Holbrook C C, Xavier A, Culbreath A K, Ozias-Akins P, Varshney R K, Guo B. Nested-association mapping (NAM)-based genetic dissection uncovers candidate genes for seed and pod weights in peanut ()., 2010, 18: 1457–1471.
Genetic analysis of fat content based on nested populations in peanut (L.)
HUANG Bing-Yan1,**, SUN Zi-Qi1,**, LIU Hua1, FANG Yuan-Jin1, SHI Lei1, MIAO Li-Juan1, ZHANG Mao-Ning1, ZHANG Zhong-Xin1, XU Jing1, ZHANG Meng-Yuan2, DONG Wen-Zhao1, and ZHANG Xin-You1,*
1Henan Academy of Crops Molecular Breeding, Henan Academy of Agricultural Sciences / Graduate R & T Base of Zhengzhou University / Key Laboratory of Oil Crops in Huang-Huai-Hai Plans, Ministry of Agriculture and Rural Affairs / Henan Provincial Key Laboratory for Oil Crops Improvement, Zhengzhou 450002, Henan, China;2College of Agriculture, Henan University of Science and Technology, Luoyang 471023, Henan, China
Nested populations can be used to dissect the heredity of complex traits. The genetic models of fat content of F2:3families in nested combinations with one common parent and six founder parents were analyzed, aiming to detect the genetic differences among the founders and to provide bases for breeding strategy for fat content improvement in peanut kernels. The common parent was Yuhua 15, an irregular-type variety with high fat content, and the other six founder parents were different botanical varieties with different fat contents. The results showed that the genetic model of fat content traits was different in different combinations. Six crosses were in accordance with three genetic patterns, including none major gene model, one major gene model with additive and dominant effect, and two major genes model with equal additive and dominant effect. The estimated values of various genetic effects were also different. The heritability of the main genes ranged from 32% to 80%, indicating that the gene loci controlling the fat content and their segregation patterns were different in different F2:3populations. There were more individuals with high fat content in the offspring from combinations with both parents of high fat content. However, the heritability was low and phenotypic selections for fat content were not suggested in the early generations in such combinations. The offspring from combinations with parents of significantly different fat content had a larger variation range in fat content, and phenotypes with variable fat content were available. In this study, the large variances in the nested populations demonstrated the genetic complexity of fat content and the characteristics of multi major gene regulation. These results provide a comprehensive base for understanding the genetics and regulation of fat content, and the nested populations will be helpful in further QTL detection of fat content in peanut.
peanut (L.); nested populations; fat content; F2:3family; inheritance model
10.3724/SP.J.1006.2021.04138
本研究由国家现代农业产业技术体系建设专项(CARS-13), 河南省现代农业产业技术体系建设专项(2016-05)和河南省重大科技专项(201300111000)资助。
This study was supported by the China Agricultural Research System (CARS-13), the Henan Agricultural Research System (2016-05), and the Key Scientific and Technological Project of Henan Province (201300111000).
张新友, E-mail: haasz@126.com
**同等贡献(Contributed equally to this work)
E-mail: huangbingyan@aliyun.com
2020-06-24;
2020-12-01;
2020-12-30.
URL:https://kns.cnki.net/kcms/detail/11.1809.S.20201230.1540.004.html

