小麦株高QTL Qph.nau-5B的效应评价
韩玉洲 张 勇 杨 阳 顾正中 吴 科 谢 全,* 孔忠新,* 贾海燕 马正强
研究简报
小麦株高QTL的效应评价
韩玉洲1,**张 勇1,**杨 阳1顾正中2吴 科3谢 全1,*孔忠新1,*贾海燕1马正强1
1南京农业大学作物遗传与种质创新国家重点实验室, 江苏南京 210095;2淮安市农业科学研究院, 江苏淮安 223001;3泰安市农业科学研究院, 山东泰安 271000
株高直接影响小麦的产量潜力, 也是植株抗倒伏性的重要组成部分。目前虽有大量株高相关QTL被鉴定到, 但大多QTL的遗传效应仍不清楚。本研究前期利用小麦品种群体, 通过关联分析鉴定到一个小麦株高主效QTL。为了评价该QTL的效应, 通过分子标记辅助选择分别构建了以南大2419、吉春1016和郑麦9023为供体亲本, 中优9507为背景的3种等位变异的近等基因系, 背景回复率均高于93%。在7个独立的试验环境中, 所有近等基因系的株高较轮回亲本均显著降低, 平均降幅为11.1 cm (10.3%)。不同等位变异效应强弱不同, 其中来源于吉春1016和郑麦9023的等位变异平均降秆效应相似(12.4 cm), 显著大于南大2419的等位变异(8.6 cm), 但各等位变异相对降秆效应大小受环境影响。此外,对单株穗数、穗长、千粒重等农艺性状无明显负效应。本研究结果表明具有重要的育种价值, 可为小麦的株型分子设计育种提供基因资源。
小麦; 株高; QTL;; 近等基因系; 分子标记辅助选择
小麦(L.)是世界上最重要的粮食作物之一, 当前种植面积达2.2亿公顷, 总产量约7亿吨, 为全球25亿人提供20%的能量(http://www.fao.org/ faostat/)。株高是小麦的重要农艺性状, 既影响小麦的产量潜力, 又与植株的抗倒伏性、抗病性(如赤霉病)等密切相关。在我国小麦品种更替进程中, 株高由120 cm左右降至目前的70~80 cm, 高产和稳产能力大幅度提高[1]。因此, 研究株高的遗传决定机制对于小麦品种改良至关重要。
小麦株高属于多基因控制的复杂数量性状, 受加性、上位性及环境互作效应共同调控, 而加性效应占主导地位, 具有较高的遗传力[1-4]。鉴定株高基因, 发掘与基因紧密连锁的分子标记, 是推动株高分子设计育种的重要前提。在先前研究中, 通过连锁作图、关联分析、同源克隆和突变体鉴定等方法, 已鉴定了约29个小麦矮秆基因()[5-9]和167个QTL[3,9-18]。在已定名的矮秆基因中, 10个基因对外施赤霉素不敏感, 包括(=)、(=)、(=)、(=)、(=)、(=)、(=)、(=)、、等; 其余19个基因对外施赤霉素敏感, 包括‒、‒、、和等[5-9]。尽管已鉴定的矮秆基因/QTL较多, 但这些基因/QTL的克隆进展缓慢, 主要集中在位点[19-24]。在生产上, 仅、、、、、等基因有较多的应用, 这些基因的遗传效应研究也相对较深入[1]。
起源于日本小麦品种‘农林10号’的矮秆基因和直接引发了20世纪50至60年代的“绿色革命”。该基因可以显著提高小麦的抗倒伏性和高肥耐受力, 使单位面积粒数、收获指数和单产大幅度提高[25-26]。目前, 世界上绝大多数的小麦矮秆/半矮秆品种携带和/或[27-28]。然而实践经验表明,和也会产生诸多不利影响, 如缩短胚芽鞘长度, 降低出苗率、幼苗活力和氮肥利用率等; 在干旱、高温等逆境种植条件下,和对产量的负效应尤其明显[28-31]。源于我国西藏小麦品种“大拇指矮(Tom Thumb)”, 降秆效应较强(约50%), 可提高穗粒数和收获指数, 抗穗发芽, 在特定环境下具有增产效应, 但该基因降低籽粒大小和蛋白含量[26,32]。来自日本矮秆品种赤小麦(Akakomugi)[33], 降秆效应仅为和的一半, 但对胚芽鞘抑制效应小, 是和的替代性矮源[34]。目前在我国小麦品种中的分布频率达56%, 高于(44%)和(29%)[34-42]。与太谷核不育基因()紧密连锁, 用二者选育的矮败小麦是杂交育种的重要材料[43]。可降秆约40%, 表现为晚熟、粒小[44-45]。由此可见, 在生产上应用的矮秆基因仍然较少, 且各有优缺点, 进一步发掘、评价、利用新的矮秆基因具有重要的意义。
在前期研究中, 利用255份小麦品种材料进行了全基因组关联分析, 在5B染色体上鉴定到一个小麦矮秆QTL, 定位于标记--区间内, 最高可解释42%的表型变异, 效应显著[46]。此外, 该QTL较为稳定, 在2007、2008、2009、2011和2017年的田间试验中均被重复检测到。该QTL在其他材料中也有报道, 包括我国山东高产半矮秆品种鲁麦14[3]、西藏半野生小麦材料Zang 1817[17]、英国半矮秆品种Avalon和Charger[18]、瑞士半矮秆品种Forno[16]、以及国际玉米小麦改良中心(CIMMYT)半矮秆高产品种Seri M82[12]等。本研究以携带不同矮秆等位变异的南大2419、吉春1016和郑麦9023为供体亲本, 携带高秆等位变异的中优9507为轮回亲本, 构建了不同等位变异的近等基因系, 在多环境中进一步评价了该QTL的降秆效应及其对农艺性状的影响, 以明确的育种利用价值, 为小麦株高遗传改良提供基因资源。
1 材料与方法
1.1 植物材料
中优9507是北部冬麦区高秆小麦品种, 不含、、、、、、、、等常见矮秆基因[10,35,42,47-48]。前期关联分析结果显示该品种含有的高秆等位变异, 本研究中用作近等基因系构建的轮回亲本。南大2419 (长江中下游冬麦区, 基因型为)[6,49]、郑麦9023 (黄淮冬麦区, 基因型为)[35,48-49]和吉春1016 (东北春麦区, 基因型为)[48]含有的矮秆等位变异, 且QTL区间单倍型互不相同, 用作不同等位变异近等基因系构建的供体亲本。
1.2 回交和分子标记辅助选择
以南大2419、吉春1016和郑麦9023为父本, 中优9507为母本进行杂交, 然后以中优9507为轮回亲本进行回交。在每一回交世代, 使用连锁分子标记--进行前景选择。在BC4F2群体, 利用上述分子标记筛选位点纯合的单株。期间利用温室进行加代, 光周期和温度设置为: 16 h/25℃ (白天, 光强为600~700 μmol m–2s–1) + 8 h/18℃ (夜晚)。对于中选单株, 利用分子标记检测其背景回复率(recipient genome composition, RGC)。所用分子标记来自: (1) Xue等[50]发表的小麦高密度遗传图谱(选择均匀分布于小麦基因组的分子标记); (2) 已发表的、、、、、、、、、、、、、等常见矮秆基因的连锁/功能分子标记[10,35,40,42,51]。
1.3 DNA提取和PCR分析
采用Ma和Sorrells[52]的方法提取DNA。取幼嫩叶片, 液氮冰冻、研磨, 加入适量SDS提取液, 震荡混匀, 65℃恒温水浴30 min, 冷却至室温后加入1.5倍体积的氯仿/异戊醇(24∶1), 置于摇床上萃取15 min, 离心, 吸取上清液, 加入1.5倍体积、–20℃预冷的无水乙醇, 析出絮状沉淀后离心, 弃上清。利用70%乙醇洗涤2次, 风干后加适量TE溶解DNA, 原液置于–20℃储藏待用。
PCR反应体系为12.5 µL, 包含10~20 ng的DNA模板, 1 mmol L–110× PCR buffer (含Mg2+), 0.1 mmol L–1dNTPs, 0.2 µmol L–1引物, 1 UDNA聚合酶。PCR反应程序为: 94℃预变性3 min; 94℃变性30 s, 55~65℃退火40 s (退火温度因引物而异), 72℃延伸40 s, 36个循环; 最后72℃延伸5 min。将PCR扩增产物在8%的非变性聚丙烯酰胺凝胶(PAGE)上进行电泳, 硝酸银染色, 后置于凝胶成像仪上进行观察和照相。
1.4 田间试验与表型调查
近等基因系及轮回亲本于2017、2018和2019年种植在江苏省淮安市农业科学院(分别简称为2017HA、2018HA和2019HA), 2018和2019年种植于江苏省南京市江浦试验站(2018JP)和江苏省农业科学院六合试验站(2019LH), 2017年和2018年种植于山东省泰安市农业科学院(2017TA和2018TA)。所有试验均按完全随机区组设计, 每个环境设置2个重复, 每个株系种植4行, 每行播种30粒, 行长1.5 m, 行宽25 cm。田间根据需要进行浇水、施肥、除草、病虫害防治等。
在小麦成熟期调查各近等基因系和轮回亲本的农艺性状, 包括株高、单株穗数、穗长和千粒重。其中, 株高、单株穗数、穗长调查基于小区中部10个单株, 排除边际效应。成熟后收获小区中部麦穗, 晾晒后手工脱粒。对于每个小区材料随机选取1000粒种子, 置于37℃烘箱烘干至恒重, 记录千粒重。
1.5 统计分析
对于所获表型数据, 以基因型(株系)、环境(年份×地点)及基因型与环境互作作为变异因子, 采用一般线性模型(general linear model, GLM)进行方差分析。基因型间表型差异显著性分析采用最小显著差数法(LSD)。方差分析和多重比较使用Minitab V17统计软件。
2 结果与分析
2.1 Qph.nau-5B等位变异近等基因系的选育
通过连续四代的回交和分子标记辅助选择, 获得了中优9507为背景, 携带南大2419、吉春1016和郑麦9023等位变异的BC4F1植株。为了获得各等位变异的近等基因系, 利用分子标记、和分别筛选了大小为122、176和205个BC4F2单株的群体, 获得了32、45和50个在位点纯合的单株。
为明确回交后代轮回亲本遗传组成的恢复程度, 利用233个分布于全基因组的分子标记(含基因连锁/功能标记)进行了多态筛选, 得到92、90和124个分别能在南大2419/中优9507、吉春1016/中优9507、郑麦9023/中优9507间显示多态的标记。利用这些多态性标记计算了上述纯合单株的背景回复率, 结果表明, 所获纯合株系的背景回复率均高于85%。从每组合中选择背景回复率最高的单株作为下一步分析的近等基因系(表1)。
2.2 Qph.nau-5B等位变异对株高的影响
2017—2019年, 在7个不同试验点评价了等位变异对株高的效应。从方差分析结果可以看出, 不仅基因型显著影响株高(< 0.0001), 种植环境(年份×地点)对株高也有显著影响(< 0.0001) (表2)。尽管基因型与环境互作达到显著水平, 但其对株高的影响远小于基因型的效应(表2)。
从7个环境的平均株高来看, 与轮回亲本中优9507相比, 所有3种等位变异近等基因系的株高均显著降低, 平均降幅为11.1 cm (10.3%) (图1-A)。在的3种等位变异之间, 来自吉春1016的和郑麦9023的降秆效应相似, 分别为12.2 cm (11.3%)、12.6 cm (11.7%); 二者的降秆效应大于南大2419的等位变异(8.6 cm或8.0%), 差异达极显著水平(图1-A)。单环境株高分析显示,的3种等位变异在不同环境中的相对降秆效应并不一致(图1-B~H)。对于2017TA、2018TA和2019HA,的降秆效应最强, 平均为17.2%; 对于2017HA和2018HA,的降秆效应最强, 平均为14.0%; 对于2018JP和2019LH, 3种等位变异的降秆效应无显著差异, 平均为11.8%。
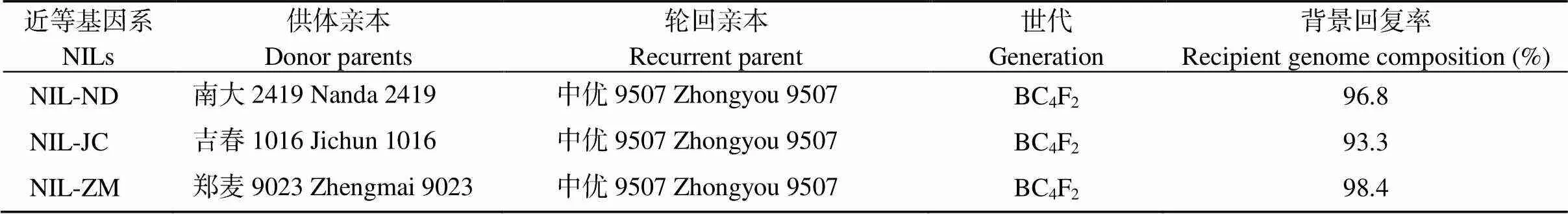
表1 Qph.nau-5B近等基因系的亲本、世代与背景回复率

表2 不同变异因子对株高的影响
A: 7个环境株高平均表现; B~H: 单环境株高表现; ZY: 轮回亲本中优9507。柱形图上方不同小写字母表示在= 0.05水平上差异显著。
A: average plant height under seven environments; B–H: plant height in individual environments; ZY: the recurrent parent Zhongyou 9507. Different lowercases on the bars indicate significant differences at= 0.05.
2.3 Qph.nau-5B对其他农艺性状的影响
为探究是否影响其他农艺性状, 对各等位变异近等基因系的单株穗数、穗长和千粒重也进行了调查分析(表3~表5)。结果表明, 仅NIL-ZM在2017TA的单株穗数低于轮回亲本中优9507, 其他各近等基因系在所有环境中均与轮回亲本无显著差异(表3)。对于穗长, NIL-JC在所测的7个环境中穗长增加, 在2017HA、2018HA和2019LH环境中其差异达显著水平; 在所有环境中, NIL-ND和NIL-ZM的穗长与轮回亲本并无显著差异(表4)。千粒重分析显示, 近等基因系与轮回亲本在75%的环境中无显著差异, 在其余环境中近等基因系的千粒重或增或降, 变化不具有一致性(表5)。
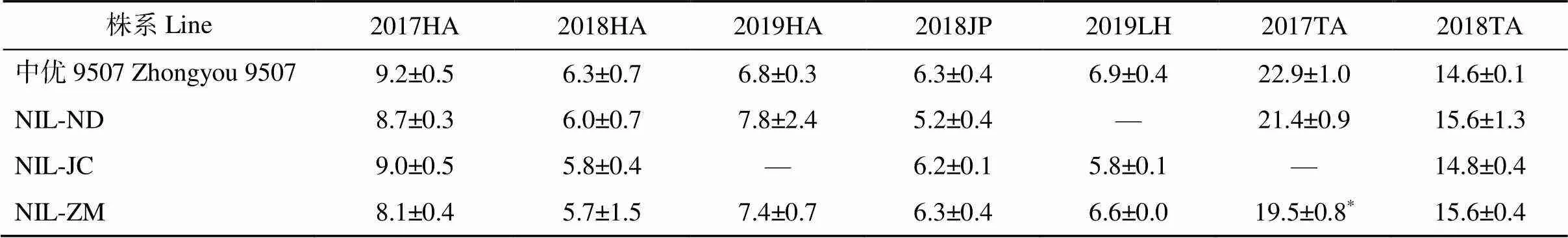
表3 Qph.nau-5B不同等位变异近等基因系及其轮回亲本的单株穗数
*表示近等基因系与轮回亲本相比在= 0.05水平上差异显著。“—”表示数据缺失。
*indicates significant differences at= 0.05 between the NILs and the recurrent parent. “—” indicates missing data.
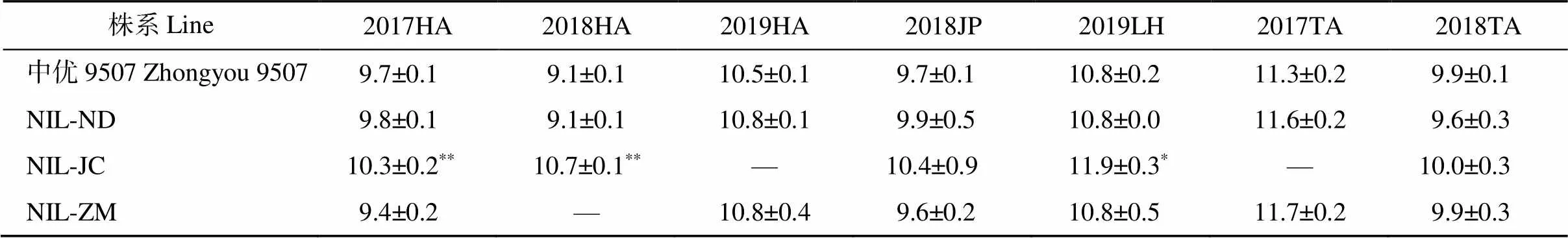
表4 Qph.nau-5B不同等位变异近等基因系及其轮回亲本的穗长
各穗长数值单位为厘米(cm);*和**分别表示近等基因系与轮回亲本相比在= 0.05、= 0.01水平上差异显著。“—”表示数据缺失。
The values of spike length are shown in centimeter (cm);*and**indicate significant differences at= 0.05 and= 0.01 between the NILs and the recurrent parent, respectively. “—” indicates missing data.
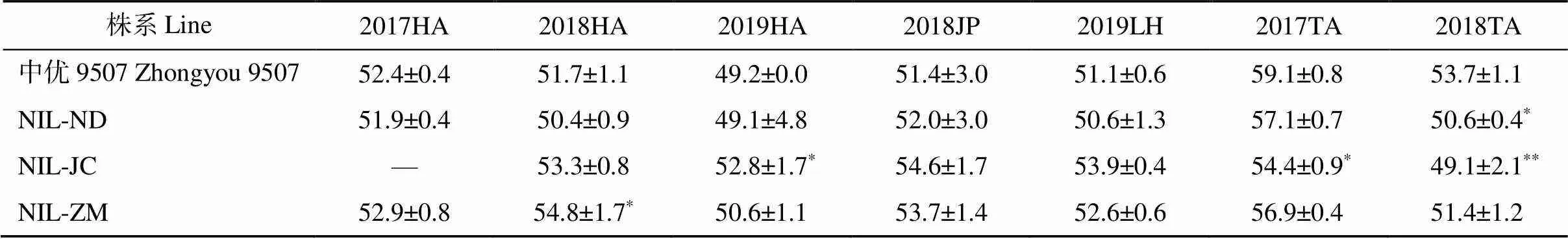
表5 Qph.nau-5B不同等位变异近等基因系及其轮回亲本的千粒重
各千粒重数值单位为克(g);*和**分别表示近等基因系与轮回亲本相比在= 0.05、= 0.01水平上差异显著。“—”表示数据缺失。
The values of 1000-grain weight are shown in gram (g);*and**indicate significant differences at= 0.05 and= 0.01 between the NILs and the recurrent parent, respectively. “—” indicates missing data.
3 讨论
是先前通过关联分析鉴定到的一个小麦株高QTL。本研究通过回交转育和分子标记辅助选择的方法, 进一步将南大2419、吉春1016和郑麦9023携带的不同矮秆等位变异导入中优9507, 选育了各自的近等基因系。田间试验表明, 轮回亲本中优9507在不同环境中的株高变异较大, 在泰安、淮安、南京试验点的株高均值为106、108、115 cm; 相比之下, 该品种在北部冬麦区株高为90 cm左右[42,53]。这可能是受到该品种自身的光周期敏感基因(已检测含和)、未知矮秆基因及其互作的调控。近等基因系分析发现,平均降秆强度为11.1 cm (10.3%), 与生产上广泛应用的类似(11 cm/12%)[1,34,54-55], 弱于(18 cm/ 18%)[1,25,34,54-55]、(24 cm/23%)[1,25,34,54-55]、(50 cm/46%)[1,44-45]、(57 cm/55%)[1,24]和(63 cm/55%)[1,25]。对单株穗数、穗长、千粒重等农艺性状无明显负效应。其中, NIL-JC的千粒重在2019HA较轮回亲本显著提高, 而在2017TA和2018TA则显著降低, 这主要是由于该株系在灌浆期对干热风敏感。在后期研究中, 需继续开展多环境田间试验, 综合评价对产量、生育时期、幼苗活力等其他农艺性状的影响。
具有3种主要的矮秆等位变异,和平均降秆效应相似, 大于的效应。除外, 其他矮秆基因也具有丰富的等位变异, 且各等位变异间的效应也不尽相同。如位点至少含有14种等位变异(-)[6,49], 而的降秆效应是的3倍[1,25,34,54-55]。在位点已鉴定到9种等位变异(-)[6],和的降秆效应相差2倍以上[1,24-25,34,54-55]。因此, 充分发掘每一矮秆基因的等位变异,鉴定不同等位变异对株高和其他农艺性状的影响, 这对于有效利用现有矮秆基因资源具有重要的意义。本研究同时发现,不同等位变异相对降秆效应大小因环境而异。在2017年和2018年淮安试验点,的降秆效应最强, 而2019年淮安点则以的降秆效应最强, 这可能与2019年淮安的气候变化有关。历史气象数据分析显示, 2017、2018年淮安点在小麦生长季内的月平均气温、降雨量和日照时数相似; 在2019年, 月日照时数保持不变, 但月平均气温(15.7℃)和降雨量(60.6 mm)较前2年分别提高24%、104% (http://data.cma.cn/)。另外, 在2017、2018年泰安试验点的降秆效应最强, 而在2018、2019年南京2个试验点中3种等位变异的降秆效应相似。因此, 在育种中利用等位变异时应考虑QTL与环境的互作效应。
分子标记辅助选择和回交转育技术是将特定基因/QTL导入品种, 实现特定性状遗传改良的重要方法。本研究利用连锁分子标记、和, 对每一回交世代F1单株进行基因型检测, 获得的杂合单株继续回交直至BC4F1, 再从BC4F2群体中筛选纯合株系进行田间试验。期间通过温室加代加快世代进程。实践表明, 利用本方案可在2~3年内完成近等基因系选育工作, 与传统育种方法相比其效率相对较高。本研究结果也证实了分子标记、和的有效性, 为的分子育种利用创造了条件。该方案仍可进一步优化, 包括: (1) 对每一世代的中选单株进行背景回复率检测, 选择回复率最高的单株进行回交, 加快背景回复进程, 由此可尝试在BC3F2甚至更早世代中获得近等基因系; (2) 采用“Speed breeding”(快速育种)技术[56]进一步提高年均加代次数, 预期可在2年内获得近等基因系。
小麦株高属于复杂的数量性状, 受多基因共同调控。尽管本研究所获得的近等基因系背景回复率达93%以上, 但由于小麦基因组庞大(约16 Gb), 在近等基因系遗传背景中仍含有较多的供体亲本的遗传物质。这些背景遗传物质是否包含其他株高基因或与互作的基因, 目前仍不得而知。为准确评价该QTL的效应, 需通过增加回交次数, 或逐代挑选背景回复率最高的单株继续回交等方法, 进一步提高背景回复率; 同时, 应采用更多的分子标记检测背景遗传组成, 如广泛应用的iSelect 90K SNP芯片[57]。对于每一组合, 可选育多个独立的近等基因系对目标性状进行考察, 提高QTL效应评价的准确性。另一方面,定位区间仍然较大, 含有的基因仍然较多, 在近等基因系选育过程中, 这些基因被同时导入轮回亲本。除功能基因外, 该区间的其他基因也可能影响株高或其他农艺性状。这是利用近等基因系对小麦QTL进行效应评价所面临的共同难题[58-59]。为此, 应利用重组体进一步精细定位QTL, 从而采用更紧密连锁的分子标记选育近等基因系, 减少连锁累赘。
[1] 李杏普, 兰素缺, 李孟军. 小麦矮秆基因. 北京: 中国农业出版社, 2009. pp 1–3. Li X P, Lan S Q, Li M J. Wheat Dwarfing Genes. Beijing: China Agriculture Press, 2009. pp 1–3 (in Chinese).
[2] Wang Z, Wu X, Ren Q, Chang X, Li R, Jing R. QTL mapping for developmental behavior of plant height in wheat (L.)., 2010, 174: 447–458.
[3] Wu X, Wang Z, Chang X, Jing R. Genetic dissection of the developmental behaviours of plant height in wheat under diverse water regimes., 2010, 61: 2923–2937.
[4] Cui F, Li J, Ding A, Zhao C, Wang L, Wang X, Li S, Bao Y, Li X, Feng D, Kong L, Wang H. Conditional QTL mapping for plant height with respect to the length of the spike and internode in two mapping populations of wheat., 2011, 122: 1517–1536.
[5] Yang T, Zhang X, Liu H, Wang Z. Chromosomal arm location of a dominant dwarfing genein common wheat variety- XN0004., 1993, 21: 13–17.
[6] McIntosh R A, Yamazaki Y, Dubcovsky J, Rogers J, Morris C, Appels R, Xia X C. Catalogue of gene symbols for wheat. In: Ogihara Y, Takumi S, Handa H, eds. Proceedings of the 12thInternational Wheat Genetics Symposium. Yokohama, Japan, 2013. pp 8–13.
[7] Chen S, Gao R, Wang H, Wen M, Xiao J, Bian N, Zhang R, Hu W, Cheng S, Bie T, Wang X. Characterization of a novel reduced height gene () regulating panicle morphology and plant architecture in bread wheat., 2015, 203: 583–594.
[8] Tian X, Wen W, Xie L, Fu L, Xu D, Fu C, Wang D, Chen X, Xia X, Chen Q, He Z, Cao S. Molecular mapping of reduced plant height genein bread wheat., 2017, 8: 1379–1379.
[9] Mo Y, Vanzetti L S, Hale I, Spagnolo E J, Guidobaldi F, Al-Oboudi J, Odle N, Pearce S, Helguera M, Dubcovsky J. Identification and characterization of, a locus on chromosome arm 6AS affecting wheat plant height, heading time, and spike development., 2018, 131: 2021–2035.
[10] Ellis M H, Rebetzke G J, Azanza F, Richards R A, Spielmeyer W. Molecular mapping of gibberellin-responsive dwarfing genes in bread wheat., 2005, 111: 423–430.
[11] Hai L, Guo H, Wagner C, Xiao S, Friedt W. Genomic regions for yield and yield parameters in Chinese winter wheat (L.) genotypes tested under varying environments correspond to QTL in widely different wheat materials., 2008, 175: 226–232.
[12] McIntyre C L, Mathews K L, Rattey A, Chapman S C, Drenth J, Ghaderi M, Reynolds M, Shorter R. Molecular detection of genomic regions associated with grain yield and yield-related components in an elite bread wheat cross evaluated under irrigated and rainfed conditions., 2010, 120: 527–541.
[13] Wüerschum T, Langer S M, Longin C F H. Genetic control of plant height in European winter wheat cultivars., 2015, 128: 865–874.
[14] Chai L, Chen Z, Bian R, Zhai H, Cheng X, Peng H, Yao Y, Hu Z, Xin M, Guo W, Sun Q, Zhao A, Ni Z. Dissection of two quantitative trait loci with pleiotropic effects on plant height and spike length linked in coupling phase on the short arm of chromosome 2D of common wheat (L.)., 2018, 131: 2621–2637.
[15] Zhou C, Xiong H, Li Y, Guo H, Xie Y, Zhao L, Gu J, Zhao S, Ding Y, Song X, Liu L. Genetic analysis and QTL mapping of a novel reduced height gene in common wheat (L.)., 2020, 19: 1721–1730.
[16] Schnurbusch T, Paillard S, Fossati D, Messmer M, Schachermayr G, Winzeler M, Keller B. Detection of QTLs for Stagonospora glume blotch resistance in Swiss winter wheat., 2003, 107: 1226–1234.
[17] Liu G, Jia L, Lu L, Qin D, Zhang J, Guan P, Ni Z, Yao Y, Sun Q, Peng H. Mapping QTLs of yield-related traits using RIL population derived from common wheat and Tibetan semi-wild wheat., 2014, 127: 2415–2432.
[18] Griffiths S, Simmonds J, Leverington M, Wang Y, Fish L, Sayers L, Alibert L, Orford S, Wingen L, Snape J. Meta-QTL analysis of the genetic control of crop height in elite European winter wheat germplasm., 2012, 29: 159–171.
[19] Peng J R, Richards D E, Hartley N M, Murphy G P, Devos K M, Flintham J E, Beales J, Fish L J, Worland A J, Pelica F, Sudhakar D, Christou P, Snape J W, Gale M D, Harberd N P. ‘Green revolution’ genes encode mutant gibberellin response modulators., 1999, 400: 256–261.
[20] Wen W, Deng Q, Jia H, Wei L, Wei J, Wan H, Yang L, Cao W, Ma Z. Sequence variations of the partially dominant DELLA genein wheat and their functional impacts., 2013, 64: 3299–3312.
[21] Bazhenov M S, Divashuk M G, Amagai Y, Watanabe N, Karlov G I. Isolation of the dwarfing() gene from wheat and the development of an allele-specific PCR marker., 2015, 35: 213.
[22] Li A, Yang W, Guo X, Liu D, Sun J, Zhang A. Isolation of a gibberellin-insensitive dwarfing gene,, and development of an allele-specific PCR marker., 2012, 30: 1443–1451.
[23] Pearce S, Saville R, Vaughan S P, Chandler P M, Wilhelm E P, Sparks C A, Al-Kaff N, Korolev A, Boulton M I, Phillips A L, Hedden P, Nicholson P, Thomas S G. Molecular characterization ofdwarfing genes in hexaploid wheat., 2011, 157: 1820–1831.
[24] Li Y, Xiao J, Wu J, Duan J, Liu Y, Ye X, Zhang X, Guo X, Gu Y, Zhang L, Jia J, Kong X. A tandem segmental duplication (TSD) in green revolution generegion underlies plant height variation., 2012, 196: 282–291.
[25] Fischer R A, Quail K J. The effect of major dwarfing genes on yield potential in spring wheats., 1990, 46: 51–56.
[26] Flintham J E, Borner A, Worland A J, Gale M D. Optimizing wheat grain yield: effects of(gibberellin-insensitive) dwarfing genes., 1997, 128: 11–25.
[27] Evans L T. Feeding the Ten Billion: Plants and Population Growth. Cambridge, UK: Cambridge University Press, 1998. pp 20–137.
[28] Yan J, Zhang S. Effects of dwarfing genes on water use efficiency of bread wheat., 2017, 4: 126–134.
[29] Fick G N, Qualset C O. Seedling emergence, coleoptile length, and plant height relationships in crosses of dwarf and standard-height wheats., 1976, 25: 679–684.
[30] Allan R E. Agronomic comparisons betweenandsemidwarf genes in winter wheat., 1989, 29: 1103–1108.
[31] Li S, Tian Y, Wu K, Ye Y, Yu J, Zhang J, Liu Q, Hu M, Li H, Tong Y, Harberd N P, Fu X. Modulating plant growth-metabolism coordination for sustainable agriculture., 2018, 560: 595–600.
[32] Flintham J E, Gale M D. The Tom Thumb dwarfing genein wheat: 2. Effects on height, yield and grain quality., 1983, 66: 249–256.
[33] 贾继增, 丁寿康, 李月华, 张辉. 中国小麦的主要矮秆基因及矮源的研究. 中国农业科学, 1992, 25(1): 1–5. Jia J Z, Ding S K, Li Y H, Zhang H. Studies of main dwarf genes and dwarf resources on Chinese wheat., 1992, 25(1): 1–5 (in Chinese with English abstract).
[34] Tang N, Jiang Y, He B, Hu Y. The effects of dwarfing genes (,, and) with different sensitivity to GA3on the coleoptile length and plant height of wheat., 2009, 8: 1028–1038.
[35] 周阳, 何中虎, 张改生, 夏兰琴, 陈新民, 张立平, 陈锋. 用微卫星标记鉴定中国小麦品种中矮秆基因的分布. 作物学报, 2003, 29: 810–814.Zhou Y, He Z H, Zhang G S, Xia L Q, Chen X M, Zhang L P, Chen F.dwarf gene distribution in Chinese wheats identified by microsatellite marker., 2003, 29: 810–814 (in Chinese with English abstract).
[36] 王玉叶, 张海萍, 来得娥, 赵秋霞, 常成, 马传喜. 257 份小麦品种资源中矮秆基因的分子检测. 安徽农业大学学报, 2013, 40: 860–866.Wang Y Y, Zhang H P, Lai D E, Zhao Q X, Chang C, Ma C X. Detection of dwarf genes in 257 wheat variety resources using molecular markers., 40: 860–866 (in Chinese with English abstract).
[37] 马东钦, 王晓伟, 许兰杰, 朱有朋, 詹克慧, 王冬梅. 黄淮麦区部分小麦种质资源中矮秆基因的分布. 河南农业大学学报, 2009, 43(2): 118–125.Ma D Q, Wang X W, Xu L J, Zhu Y P, Zhan K H, Wang D M. Distribution of dwarfing genes derived from some wheat germplasms in Huang-Huai wheat area., 2009, 43(2): 118–125 (in Chinese with English abstract).
[38] Zhang X, Yang S, Zhou Y, He Z, Xia X. Distribution of the,andreduced height genes in autumn- sown Chinese wheats detected by molecular markers., 2006, 152: 109–116.
[39] 张德强, 宋晓朋, 冯洁, 马文洁, 武炳瑾, 张传量, 崔紫霞, 冯毅, 孙道杰. 黄淮麦区小麦品种矮秆基因、和的检测及其对农艺性状的影响. 麦类作物学报, 2016, 36: 975–981.Zhang D Q, Song X P, Feng J, Ma W J, Wu B J, Zhang C L, Cui Z X, Feng Y, Sun D J. Detection of dwarf genes,andin Huang-Huai Valley winter wheat areas and their influences on agronomic characteristics., 2016, 36: 975–981 (in Chinese with English abstract).
[40] 周晓变, 赵磊, 陈建辉, 阳霞, 王永彦, 张香粉, 闫雪芳, 董中东, 崔党群, 陈锋. 黄淮麦区小麦种质资源矮秆基因分布及其与农艺性状的关系. 麦类作物学报, 2017, 37: 997–1007.Zhou X B, Zhao L, Chen J H, Yang X, Wang Y Y, Zhang X F, Yan X F, Dong Z D, Cui D Q, Chen F. Distribution of dwarf genes and their association with agronomic traits in bread wheat from the Yellow and Huai wheat region., 2017, 37: 997–1007 (in Chinese with English abstract).
[41] 周强, 袁中伟, 欧俊梅, 任勇, 杜小英, 陶军, 李生荣, 刘登才. 四川小麦主要矮秆基因的分子鉴定. 麦类作物学报, 2015, 35: 1624–1630.Zhou Q, Yuan Z W, Ou J M, Ren Y, Du X Y, Tao J, Li S R, Liu D C. Molecular identification of the main dwarfing genes in wheat varieties in Sichuan., 2015, 35: 1624–1630 (in Chinese with English abstract).
[42] 杨松杰. 我国小麦品种(系)矮秆基因的分子检测. 新疆农业大学硕士学位论文, 新疆乌鲁木齐, 2004.Yang S J. Molecular Detection of Dwarfing Genes in Chinese Bread Wheat. MS Thesis of Xinjiang Agricultural University, Urumqi, Xinjiang, China,2004 (in Chinese with English abstract).
[43] 刘秉华, 杨丽, 王山荭. 小麦4D染色体上基因、和着丝点的连锁关系图. 国外农学——麦类作物, 1995, (5): 36–38.Liu B H, Yang L, Wang S H. Linkage map of the genes,and centromere on chromosome 4D of wheat.:, 1995, (5): 36–38 (in Chinese).
[44] Chen L, Phillips A L, Condon A G, Parry M A J, Hu Y. GA-responsive dwarfing geneaffects the developmental and agronomic traits in common bread wheat., 2013, 8: e62285.
[45] Worland A J, Sayers E J, Börner A. The genetics and breeding potential of, a dominant dwarfing gene in wheat., 1994, 113: 187–196.
[46] 万洪深. 小麦骨干亲本南大2419产量相关基因组区段的定位及其等位变异的效应. 南京农业大学博士学位论文, 江苏南京, 2013.Wan H S. Yield-related Genomic Regions of Founder Wheat Parent Nanda 2419 and the Effects of Their Allelic Variations. PhD Dissertation of Nanjing Agricultural University, Nanjing, Jiangsu, China, 2013 (in Chinese with English abstract).
[47] Wu Y, Li M, He Z, Dreisigacker S, Wen W, Jin H, Zhai S, Li F, Gao F, Liu J, Wang R, Zhang P, Wan Y, Cao S, Xia X.
Development and validation of high-throughput and low-cost STARP assays for genes underpinning economically important traits in wheat., 2020, 133: 2431–2450.
[48] Zhao J, Wang Z, Liu H, Zhao J, Li T, Hou J, Zhang X, Hao C. Global status of 47 major wheat loci controlling yield, quality, adaptation and stress resistance selected over the last century., 2019, 19: 5.
[49] Li A, Yang W, Lou X, Liu D, Sun J, Guo X, Wang J, Li Y, Zhan K, Ling H, Zhang A. Novel natural allelic variations at theloci in wheat., 2013, 55: 1026–1037.
[50] Xue S, Zhang Z, Lin F, Kong Z, Cao Y, Li C, Yi H, Mei M, Zhu H, Wu J, Xu H, Zhao D, Tian D, Zhang C, Ma Z. A high-density intervarietal map of the wheat genome enriched with markers derived from expressed sequence tags., 2008, 117: 181–189.
[51] Haque M A, Martinek P, Watanabe N, Kuboyama T. Genetic mapping of gibberellic acid-sensitive genes for semi-dwarfism in durum wheat., 2011, 39: 171–178.
[52] Ma Z, Sorrells M E. Genetic analysis of fertility restoration in wheat using restriction fragment length polymorphisms., 1995, 35: 1137–1143.
[53] 赵广才, 何中虎, 田奇卓, 刘利华, 李振华, 张文彪, 张全良. 农艺措施对中优9507小麦蛋白组分和加工品质的调节效应. 作物学报, 2003, 29: 408–412.Zhao G C, He Z H, Tian Q Z, Liu L H, Li Z H, Zhang W B, Zhang Q L. Regulating effect of the treatment of agronomic practice on protein component and bread making quality in Zhongyou 9507 wheat., 2003, 29: 408–412 (in Chinese with English abstract).
[54] 冯洁, 许小宛, 李小东, 张传量, 崔紫霞, 冯毅, 孙道杰. 黄淮麦区小麦品种和CIMMYT材料的矮秆基因型及其对株高和胚芽鞘的影响. 麦类作物学报, 2018, 38: 668–673.Feng J, Xu X W, Li X D, Zhang C L, Cui Z X, Feng Y, Sun D J. Dwarf genotype of wheat from Huang-Huai River wheat area and CIMMYT and their effects on plant height and coleoptile length., 2018, 38: 668–673 (in Chinese with English abstract).
[55] 许琦, 杨娜, 柴永峰, 杨淑巧, 赵智勇, 裴蕾, 郭文治, 刘跃鹏. 中国小麦主要矮秆基因的分布及其对株高的影响. 西北农业学报, 2014, 23(5): 59–64.Xu Q, Yang N, Chai Y F, Yang S Q, Zhao Z Y, Pei L, Guo W Z, Liu Y P. Distribution and impact on plant height of major wheat dwarfing genes in China., 2014, 23(5): 59–64 (in Chinese with English abstract).
[56] Watson A, Ghosh S, Williams M J, Cuddy W S, Simmonds J, Rey M, Asyraf Md Hatta M, Hinchliffe A, Steed A, Reynolds D, Adamski N M, Breakspear A, Korolev A, Rayner T, Dixon L E, Riaz A, Martin W, Ryan M, Edwards D, Batley J, Raman H, Carter J, Rogers C, Domoney C, Moore G, Harwood W, Nicholson P, Dieters M J, DeLacy I H, Zhou J, Uauy C, Boden S A, Park R F, Wulff B B H, Hickey L T. Speed breeding is a powerful tool to accelerate crop research and breeding., 2018, 4: 23–29.
[57] Wang S, Wong D, Forrest K, Allen A, Chao S, Huang B E, Maccaferri M, Salvi S, Milner S G, Cattivelli L, Mastrangelo A M, Whan A, Stephen S, Barker G, Wieseke R, Plieske J, Lillemo M, Mather D, Appels R, Dolferus R, Brown-Guedira G, Korol A, Akhunova A R, Feuillet C, Salse J, Morgante M, Pozniak C, Luo M, Dvorak J, Morell M, Dubcovsky J, Ganal M, Tuberosa R, Lawley C, Mikoulitch I, Cavanagh C, Edwards K J, Hayden M, Akhunov E. Characterization of polyploid wheat genomic diversity using a high-density 90 000 single nucleotide polymorphism array., 2014, 12: 787–796.
[58] Röder M S, Huang X Q, Börner A. Fine mapping of the region on wheat chromosome 7D controlling grain weight., 2008, 8: 79–86.
[59] Wu X, Cheng R, Xue S, Kong Z, Wan H, Li G, Huang Y, Jia H, Jia J, Zhang L, Ma Z. Precise mapping of a quantitative trait locus interval for spike length and grain weight in bread wheat (L.)., 2014, 33: 129–138.
Effect evaluation of QTLcontrolling plant height in wheat
HAN Yu-Zhou1,**, ZHANG Yong1,**, YANG Yang1, GU Zheng-Zhong2, WU Ke3, XIE Quan1,*, KONG Zhong-Xin1,*, JIA Hai-Yan1, and MA Zheng-Qiang1
1State Key Laboratory of Crop Genetics and Germplasm Enhancement, Nanjing Agricultural University, Nanjing 210095, Jiangsu, China;2Huai’an Academy of Agricultural Sciences, Huai’an 223001, Jiangsu, China;3Tai’an Academy of Agricultural Sciences, Tai’an 271000, Shandong, China
Plant height affects directly the yield potential of wheat, and constitutes an important component of plant resistance to lodging. Although a large number of QTLs for plant height were detected, most of them have not been evaluated for their genetic effects yet. In the previous study, a major QTLcontrolling wheat plant heightwas identified through association mapping in wheat variety collection. To evaluate the dwarfing effect of this QTL, three near-isogenic lines (NILs) with different alleles ofwere developed using marker-assisted selection with Nanda 2419, Jichun 1016, and Zhengmai 9023 as donor and Zhongyou 9507 as receptorThe recipient genome compositions of these NILs were higher than 93%. Seven independent field trials were conducted and revealed that, compared with the recurrent parent, all NILs indicated a significant decrease in plant height (11.1 cm or 10.3% on average). Three alleles ofshowed different degrees of dwarfing effects. The alleles coming from Jichun 1016 and Zhengmai 9023 displayed a similar effect on plant height (12.4 cm) in all environments, stronger than that of Nanda 2419 (8.6 cm). However, the relative dwarfism effects of different alleles were affected by different environments. Further analysis elucidated that this QTL had little detrimental influence on other agronomical traits such as spike number per plant, spike length and 1000-grain weight. These results suggested the breeding value ofthat would be utilized for molecular design breeding of plant architecture in wheat.
wheat (); plant height; QTL;; near-isogenic lines (NILs); marker-assisted selection
10.3724/SP.J.1006.2021.01053
本研究由国家重点研发计划项目(2016YFD0100402), 国家自然科学基金项目(31801354, 31871620)和江苏省“双创计划”资助。
This study was supported by the National Key Research and Development Program of China (2016YFD0100402), the National Natural Science Foundation of China (31801354, 31871620), and the Innovation and Entrepreneurship Talents Program of Jiangsu.
谢全, E-mail: quanxie@njau.edu.cn; 孔忠新, E-mail: zhxkong@njau.edu.cn
**同等贡献(Contributed equally to this work)
韩玉洲, E-mail: 2016101115@njau.edu.cn; 张勇, E-mail: 2017101102@njau.edu.cn
2020-06-27;
2020-12-01;
2020-12-31.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20201231.0927.002.html

