SARS-CoV-2刺突蛋白基因密码子偏好性与进化分析
陈 渏,杨雅丽, 陈 旭, 王 杰, 周嘉裕, 廖 海
(西南交通大学 生命科学与工程学院,四川 成都 610031)
新型冠状病毒(SARS-CoV-2)是继重症急性呼吸综合征冠状病毒(SARS-CoV)与中东呼吸综合征冠状病毒(MERS-CoV)之后出现的又一种高传染性冠状病毒。SARS-CoV-2的基因组中至少含有6个开放阅读框,分别编码S-蛋白(spike protein)、M膜蛋白、E包膜蛋白、N核衣壳蛋白以及多种复制转录酶。其中,S-蛋白能够与人体细胞表面的血管紧张素转换酶2(ACE2)发生特异性相互作用,决定了SARS-CoV-2入侵宿主的亲和能力及组织特异性[1-2]。由于ACE2广泛分布于人类呼吸道、心脏、肾脏、睾丸与胃肠道,推测SARS-CoV-2的作用靶器官可能多样化[3-5]。相较于SARS-CoV,SARS-CoV-2传染性更强,推测其与S-蛋白和ACE2的亲和力更高[6-7]。比较SARS-CoV-2与SARS-CoV的S-蛋白,发现有27处氨基酸残基发生了改变,其中有6个氨基酸位于受体结合区(Receptor binding domain,RBD)。这些改变有可能赋予新型冠状病毒的S-蛋白独特的三维结构特性,进而影响(或增强)其与ACE2 的相互作用。与此同时,由于S-蛋白在侵染过程中的核心地位,其成为预防及治疗新型冠状病毒的单抗或疫苗开发的重要靶点。例如,Yang等[8]获得了靶向RBD(位于S-蛋白的319~545位氨基酸残基)的重组疫苗,能够有效抑制RBD与ACE2的结合,从而阻断SARS-Cov-2假病毒和SARS-Cov-2活病毒的感染。Zhu等[9]利用携带S-蛋白基因的腺病毒载体,注射入人体后,诱导产生S-蛋白抗体。为了更好地开展S-蛋白的结构与功能研究,筛选更有效的SARS-CoV-2疫苗与抗体,急需建立S-蛋白的高效表达体系,确保科研人员获得足量的S-蛋白。然而,不同生物常常具有不同的密码子偏好性,往往倾向于使用一种或几种特定的同义密码子。分析密码子偏好性,从中筛选最优密码子,对于提高重组蛋白质的表达效率是不可或缺的。此外,密码子的偏好性分析对揭示物种间或某一物种家族间的基因进化规律也具有指导价值[10]。本研究从NCBI中收集包括SARS-CoV-2在内的71条(51条SARS-CoV-2,20条其他类型的冠状病毒)S-蛋白的CDS序列,对其开展密码子偏好性分析,最后进行了初步的密码子优化与表达验证,旨在为S-蛋白基因的重组表达筛选最适表达系统,并从密码子偏好性的角度,对SARS-CoV-2开展系统进化分析。
1 材料与方法
1.1 材料
数据来源 S-蛋白基因来源于GenBank(http://www.ncbi.nlm.nih.gov/genbank/),登录序号见表1。大肠埃希菌(Escherichiacoli)、酵母菌(Saccharomycescerevisiae)等模式生物的基因组密码子偏好性数据来源于密码子使用数据库(Codon Usage Database)(http://www.kazusa.or.jp/codon/)。分析51条SARS-CoV-2病毒的S-蛋白基因序列,发现其核苷酸序列高度相似(大于99%),因此选取1条具有代表性的序列(如2019新冠病毒MN908947.3)开展后续分析。
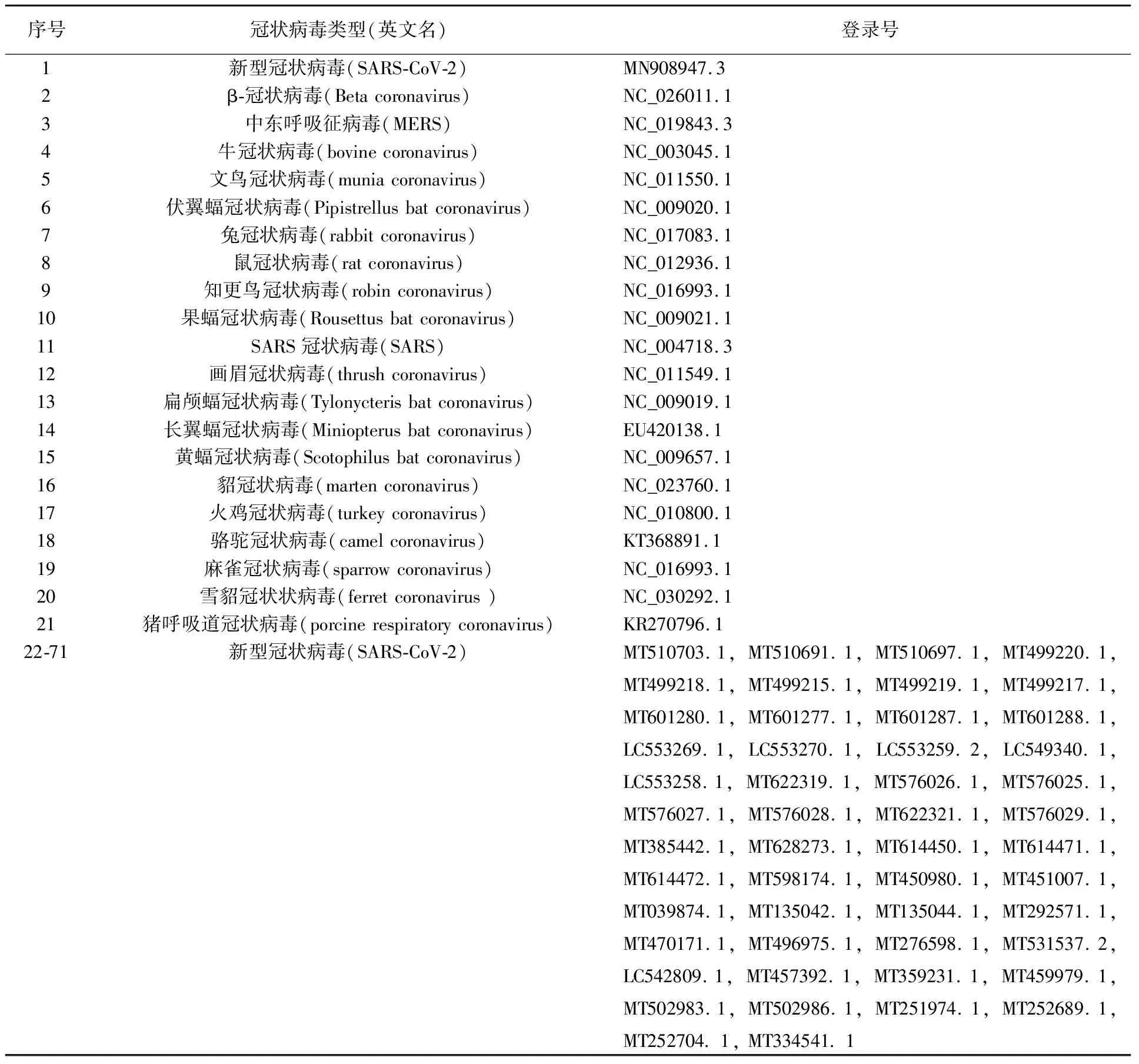
表1 冠状病毒S-蛋白基因序列编号
1.2 方法
1.2.1 S-蛋白基因的密码子偏好性分析 采用 CodonW 软件和EMBOSS在线程序对冠状病毒S-蛋白的RSCU、有效密码子数(Effective Number of Codon, ENc)、密码子 G/C 含量 GC、密码子第三位 G/C 含量 GC3s、密码子适应指数(Codon adaptation index,CAI)等密码子偏好性参数进行了统计分析。RSCU被定义为在编码氨基酸的若干同义密码子中,某一特定密码子的相对使用概率。若RSCU 为1,则表明该密码子无偏好性;若其值大于1,则表明其相对使用频率较高;若其值小于1,则说明其相对使用频率较低[11-12]。ENc 值能够反映密码子家族中同义密码子非均衡使用的偏好程度,其值介于 20~61 之间,越接近于 20,说明偏好性越强。用各个冠状病毒基因的ENc及GC3s值构建散点图和ENc-GC3s期望曲线(ENc=2+GC3s+29/[GC3s2+(1-GC3s)2]),各点与期望曲线的相对位置可以反映出密码子偏好性的形成是由于碱基突变还是自然选择[13-15]。若某一基因的密码子偏好性受碱基突变影响较大时,其 ENc-GC3s 点将分布于期望曲线附近;若其受自然选择影响较大时,则会分布在偏离期望曲线较远的位置[16-18]。
1.2.2 S-蛋白基因的系统进化树及基于RSCU值的相关聚类分析 通过MEGA软件,采用邻接法(Neighbor-joining)构建冠状病毒S-蛋白基因的系统进化树,重复次数设为1 000。利用SPSS软件组间联结法对各基因的RSCU值进行系统聚类分析。
1.2.3 适合S-蛋白基因表达的外源宿主 利用Codon W计算大肠埃希菌、酵母、λ噬菌体等模式生物基因组的密码子使用频率,并利用SPSS软件与SARS-CoV-2的S-蛋白基因的密码子使用频率进行比较,确定其合适的外源表达系统和遗传转化受体。
1.2.4 新冠病毒S-蛋白的RBD区域的原核表达 参考大肠埃希菌基因组的密码子偏好性,对S-蛋白的RBD(Arg128至Pro398,共271个氨基酸残基)区域进行密码子优化,同时在RBD区域的5′-端引入EcoR Ⅰ酶切位点,3′-端引入TAA终止密码与XhoⅠ酶切位点,由南京集思慧远生物科技有限公司完成该RBD片段全合成。合成的RBD片段与pGEX-4T-1载体经EcoR Ⅰ与XhoⅠ双酶切、连接形成pGEX-4T-1-RBD重组载体。提取pGEX-4T-1-RBD重组载体,转化大肠埃希菌Arctic-Express,37 ℃、200 r/min培养至菌液OD值0.6~0.7,加入IPTG 使其终浓度为0.5 mmol/L,11 ℃、160 r/min诱导表达12 h。表达完成后,培养物在室温条件下,10 000 r/min,10 min离心2次,弃上清,用 PBS重悬菌体沉淀。重悬液进行超声波破碎后,分别取上清液(10 μL)与沉淀液加入上样缓冲液重悬。最后采用12% SDS-PAGE和考马斯亮蓝染色检测。
2 结果与分析
2.1 冠状病毒的S-蛋白基因同义密码子相对使用度分析
基于21条冠状病毒S-蛋白基因的RSCU值制作热图(图1)。结果显示有28个密码子具有偏好性,分别为UUU、UUA、UUG、UCU、UCA、UAU、UAA、UGU、CUU、CCU、CCA、CAU、CAA、CGU、AUU、ACU、ACA、AAU、AAA、AGU、AGA、AGG、GUU、GCU、GCA、GAU、GAA和GGU。其中RSCU≥2的有CCU、GUU与GGU,最优密码子为UAA,其RSCU值为3.00,表明冠状病毒倾向于UAA作为终止密码子。RSCU≤0.5的密码子有17个,分别为UUC、UCG、CUA、CCC、CAC、CGC、CGG、ACC、ACG、AGC、GCC、GCG、GGG、UGA、UAG、CGA和CCG,表明以上17个密码子使用频率较低。
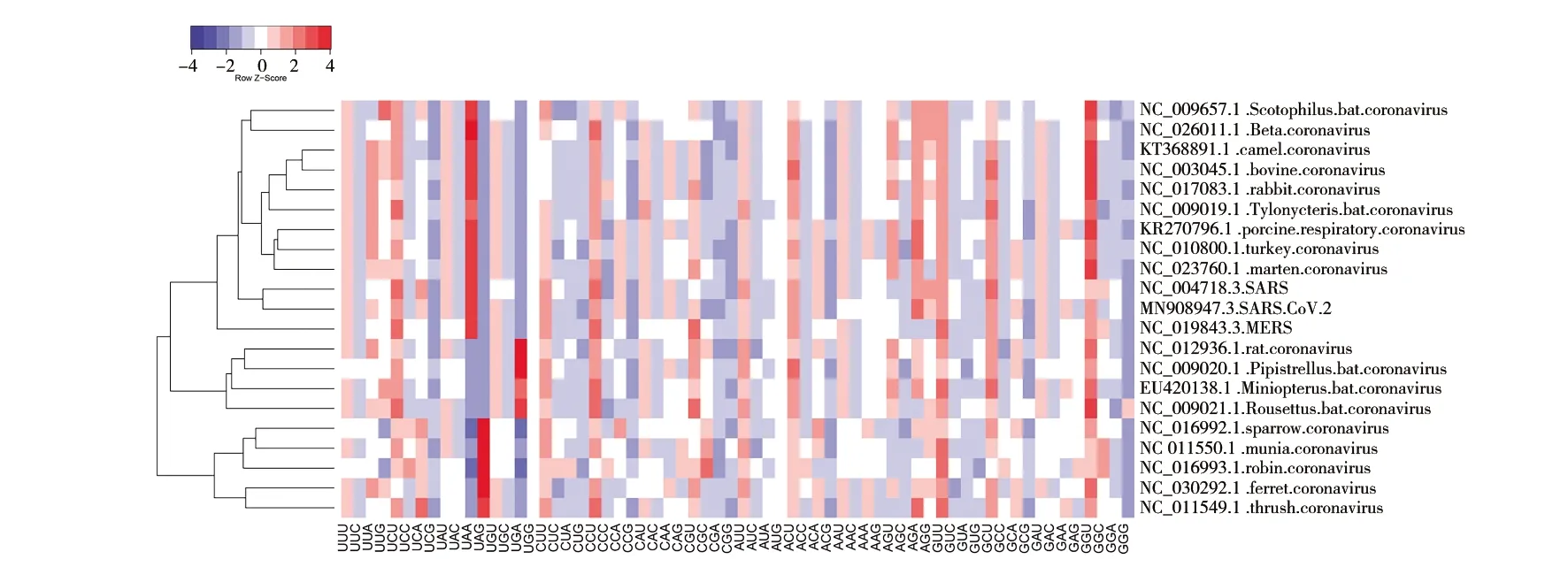
图1 冠状病毒S-蛋白基因的RSCU热图
2.2 冠状病毒S-蛋白基因偏好性相关参数比较分析
由表2可知,大部分冠状病毒的ENc值低于50,其中扁颅蝠冠状病毒的ENc值最低,仅为40.97;仅有β、文鸟、知更鸟、麻雀和伏翼蝠冠状病毒的ENc值高于50,表明冠状病毒S-蛋白基因的密码子偏好性较弱。不同冠状病毒的CAI值相差不大,均处在0.2~0.3之间,说明冠状病毒的S-蛋白基因的表达强度偏低。除知更鸟冠状病毒GC含量46.6%,GC3s值47.6%较高外,其他冠状病毒GC含量介于36.0%~42.3%之间,GC3s值介于22.0%~37.6%之间,表明冠状病毒S-蛋白基因对碱基A和T有更强的偏好性。

表2 S-蛋白基因偏好性相关参数
2.3 SARS-CoV-2密码子偏好性分析
相较于冠状病毒的密码子偏好性整体分析结果,SARS-CoV-2具有偏好性的密码子有28个,偏好性极强(RSCU>2)的密码子有7个(增加了UCU、UAA、CUU、AGA与GCU),最优密码子也为UAA。SARS-CoV-2的GC、GC3s、CAI与ENc值等密码子偏好性参数与SARS的S-蛋白较为接近。其GC含量与GC3s值分别为37.3%与25.1%,CAI与ENc值分别为0.226与44.15,表明SARS-CoV-2的S-蛋白基因偏向于A/U编码,且密码子偏好性较弱。
2.4 ENc-plot图分析
ENc-plot图(图2)显示,除知更鸟冠状病毒S-蛋白基因远离期望曲线,其余冠状病毒的S-蛋白基因均分布于期望曲线附近,表明冠状病毒的S-蛋白基因密码子偏好性的形成主要受到碱基突变的影响,自然选择的影响较小。进一步分析发现,SARS-CoV-2和SARS不仅位于期望曲线较近,且紧密靠近,表明两者密码子偏好性形成具有高度相似性。
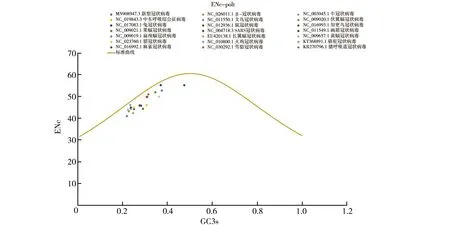
图2 冠状病毒S-蛋白基因的ENc-plot分析
2.5 基于CDS序列的系统进化树和基于RSCU值的聚类
通过邻接法构建了冠状病毒S-蛋白基因的系统进化树,该进化树具有较高的自展值,可信度较高,能够用于分析冠状病毒S-蛋白基因的系统进化关系(图3A)。21种不同来源的冠状病毒聚类为二簇,第一簇由哺乳动物为宿主的冠状病毒组成,第二簇包括了鸟类与部分以哺乳动物为宿主的冠状病毒。MERS、SARS与SARS-CoV-2聚类在第一簇中较近的位置,其中SARS-CoV-2与SARS在系统进化树中靠得最近,表明SARS-CoV-2与SARS具有最近的亲缘关系,而与MERS亲缘关系稍远。
在基于RSCU值的SPSS聚类(图3B)中,21条冠状病毒S-蛋白基因被聚为两类。其中,第一类由画眉等4种鸟类冠状病毒和雪貂冠状病毒组成,第二类由SARS-CoV-2等13种哺乳动物冠状病毒和火鸡冠状病毒组成,相较于CDS序列,基于RSCU的聚类更适合用于冠状病毒的系统发育分析。两种聚类结果中,SARS-CoV-2、SARS与MERS表现出相似的聚类结果,表明SARS-CoV-2与SARS的亲缘关系最近,可能来源于同一个祖先冠状病毒,这为 SARS-CoV-2的系统发育与溯源提供了参考。
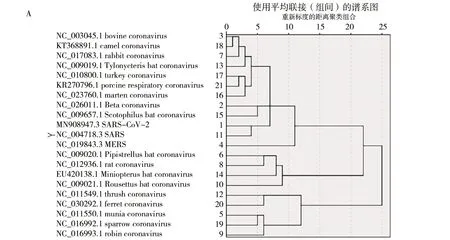
图3 冠状病毒S-蛋白基因RSCU值聚类和系统进化树分析结果
2.6 SARS-CoV-2的S-蛋白基因与不同模式生物的密码子偏好性比较
比较不同模式生物基因组与SARS-CoV-2的S-蛋白基因的密码子使用频率,将比值<0.5和>2的密码子视为使用频率差异较大的密码子。结果显示,SARS-CoV-2的S-蛋白基因密码子与大肠埃希菌、噬菌体与酿酒酵母相比,使用频率差异较大的密码子数分别为28、27与15,表明酿酒酵母更适合作为新冠病毒S-蛋白基因的外源表达载体。
2.7 RBD区域的原核表达
由于SARS-CoV-2的S-蛋白与大肠埃希菌有较多的差异密码子,若以大肠埃希菌为表达宿主,需要进行密码子优化。根据本研究的分析结果,对S-蛋白的RBD区域密码子完成了优化、改造及全基因合成,成功构建了pGEX-4T-1-RBD重组载体。pGEX-4T-1载体带有GST标签,因此重组蛋白的理论分子量为57.89 kDa。由图4可知,相较于未诱导的大肠埃希菌,含有pGEX-4T-1-RBD的重组菌经IPTG诱导后,产生了一条较明显的蛋白条带(57 kDa),与预期分子量一致,表明RBD片段获得了成功诱导表达。
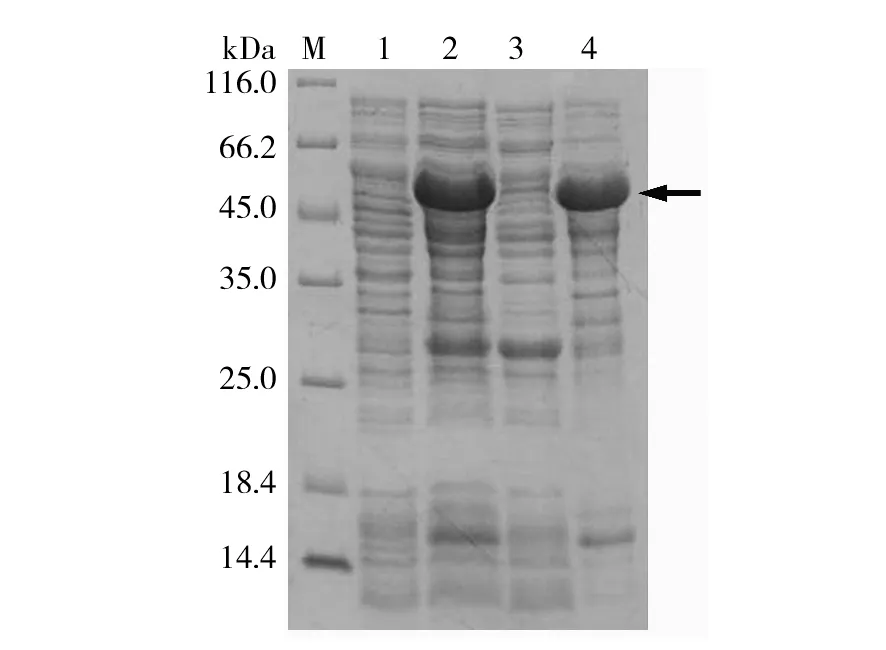
图4 RBD区域表达的SDS-PAGE
3 讨 论
密码子作为基因密码的基本单位和进化单位,对于其偏好特点的分析与计算需要依赖生物信息学的发展。本研究使用一些常用的生物信息软件围绕密码子使用模式,对51条SARS-CoV-2的S-蛋白基因密码子偏好性及其与以往报道的20条其他物种的冠状病毒S-蛋白基因的进化关系进行分析。结果发现,冠状病毒S-蛋白基因中有17个非优势密码子,这些密码子即成为未来密码子改造的重点区域。ENc值是评判密码子偏好强弱的通用指标,本研究发现,冠状病毒S-蛋白基因的ENc值在44左右,密码子偏好性总体一般。这种现象可能与冠状病毒的多宿主性有关,以往报道多种哺乳动物及鸟类为其宿主,因此低偏好性可能有利于冠状病毒的跨物种传播。绝大部分冠状病毒的GC含量介于36.0%~42.3%之间,GC3s值介于22.0%~37.6%之间,表明冠状病毒S-蛋白基因对碱基A和T有更强的偏好性,相似结果也出现在28个优势密码子与全基因组碱基组成中[19]。
SARS-CoV-2与SARS具有相近的密码子偏好性参数,这不仅体现在两者具有相近的GC含量、GC3s、CAI与ENc值,并且在ENc-plot曲线上也位于相近位置。更有趣的是,在基于RSCU值的聚类树及基于CDS序列的系统进化树中,SARS-CoV-2与SARS均紧密聚类,该结果不仅表明二者的亲缘关系最近,也表明二者可能起源于某一种祖先冠状病毒。由于SARS-CoV-2与SARS的亲缘关系最近,因此推测二者可能拥有同一类中间宿主,即蝙蝠。Zhou等[20]基于冠状病毒全基因组序列的系统进化树,也证实SARS-CoV-2与SARS具有最近的亲缘关系,这与本研究结果相同。然而,陈嘉源等[21]基于冠状病毒Nankai基因CDS序列的系统进化树,却发现SARS-CoV-2与 SARS 冠状病毒相距较远,究其原因可能有两点:①在进化过程中,冠状病毒的不同基因或基因组不同区域受到了不同的选择压力;②不同病毒在进化过程中,出现了基因的平行转移。
根据本研究的分析结果,对S-蛋白的RBD区域的密码子完成了优化与改造,并在E.coli中成功实现了高效表达,验证了分析结果。下一步将对RBD的表达条件进行优化筛选,以提高RBD的可溶性表达。

