革兰氏染色细菌显微图像深度学习分类与计数
董宇波,王蕊,赵慧娟,张书景
前言
细菌的分类和计数在生物工程、环境检测和医药学等领域都有十分重要的意义。传统的细菌计数方法有平板计数法[1-3],这是一种间接培养计数法,由于单个细菌难以观测,因此将分散的细菌在培养基上培养形成菌落,将菌落个数作为计数标准,这种方法需要花时间培养细菌,结果不稳定。流式细胞术是一种通过仪器分析细胞而完成计数的方法[4-6],被荧光标记过的细菌以一定的速度流过流式细胞仪测定区域,荧光反应的信号强度能够检测细菌的组成和含量;然而流式细胞仪价格昂贵,对单个细菌的检测准确,对混合细菌分类效果不好。最大可能数(MPN)计数方法通过概率统计来估计细菌浓度[7-8]。显微镜计数是一种常用的方法,显微镜可以放大细菌或染色细菌,以便于人们观察,对于单个视野中细菌分布不均匀且数量众多的显微镜计数工作量巨大,过程繁琐,并且极易出错[9-11]。
针对以上问题,本研究提出一种基于深度学习的显微图像细菌计数方法,该方法能够将实际任务图像中的革兰氏阳性杆菌、革兰氏阴性杆菌、革兰氏阳性球菌和革兰氏阴性球菌进行分类识别和计数,图1显示了4 种不同类型细菌的显微图像。该算法过程流程模拟人眼观察,对4种细菌的分类计数过程为:“分割”→“分类器训练”→“识别”→“分类计数”。首先采用U-Net“渐进式分割”方法去除背景,分割出细菌部分;然后将分割后的细菌部分分别投入经过训练后的ResNet50 模型和VGG19 模型进行识别和计数。

图1 4种革兰氏染色菌形态差异Fig.1 Morphological differences among four kinds of Gram-stained bacteria
1 渐进分割
图像分割是将图像分成具有独特特征的特定区域,也是后续图像分析的预处理和关键准备[12]。由于显微图像中的细菌尺寸小且存在黏连和边缘模糊的情况,以往的分割方法难以很好地将背景剔除。U-Net[13-14]在医学图像分割方面有很好的表现,在2015年ISBI 细胞追踪比赛中取得了优秀的表现。U-Net网络结构如图2所示,它是一个编码器-解码器结构,左半边为编码器,与传统的分类网络结构类似,是“下采样阶段”,右半边为解码器,是“上采样阶段”,中间的灰色箭头为跳跃连接,将浅层的特征与深层的特征拼接,浅层通常可以抓取图像一些简单的特征,比如边界、颜色,深层的特征是高维特征,经过训练后记忆在网络参数中。

图2 U-Net模型结构Fig.2 The U-Net model structure
1.1 训练U-Net
细菌显微图像和医学图像有很多相同的特点,例如图像颜色种类少、背景单调、噪声高、分辨率低;不同的是细菌显微图像中微小细菌的数量十分巨大。因此,首先将一幅细菌显微图像分割成若干个30×30 像素的子块,然后选取部分子块作标记,对U-Net 模型进行训练。每个子块进行分割之后,将它们拼接起来作全局分割掩膜。掩膜为一个二值图像,白色为细菌部分,黑色为背景部分,将掩膜与原图相乘即可得到分割后的细菌。图3显示了U-Net分割后的掩膜,图4显示了子块合成掩膜的过程。

图3 U-Net子块分割掩膜Fig.3 The mask of sub-blocks segmented by U-Net

图4 合成全局分割掩膜过程Fig.4 The synthesis process of the whole mask segmented by U-Net
本文中使用的U-Net 网络模型包括19 次卷积运算和4 次转置卷积运算,4 次上采样和4 次下采样,最终输出由Simoid 函数激活。先将图像分解成30×30像素的子块,从中选择50 个作标签,用于U-Net 模型的训练。该模型经过500次迭代,取得了良好的分割效果。分割掩膜很难通过一次性阈值得到,较高的阈值会导致部分细菌的丢失,而较低的阈值则会导致细菌黏连不能完全分割。因此,本研究采用渐进分割,通过至少两次调整阈值,可以完成几乎所有细菌轮廓分割。
1.2 渐进分割
由U-Net模型生成的掩膜的像素分布如图5所示。通过观察U-Net模型生成的掩膜的像素分布,选择中点处约0.500 5作为首次分割阈值,去除大部分背景。再选首次阈值和最大值之间中点处约0.500 75作为二次分割阈值。
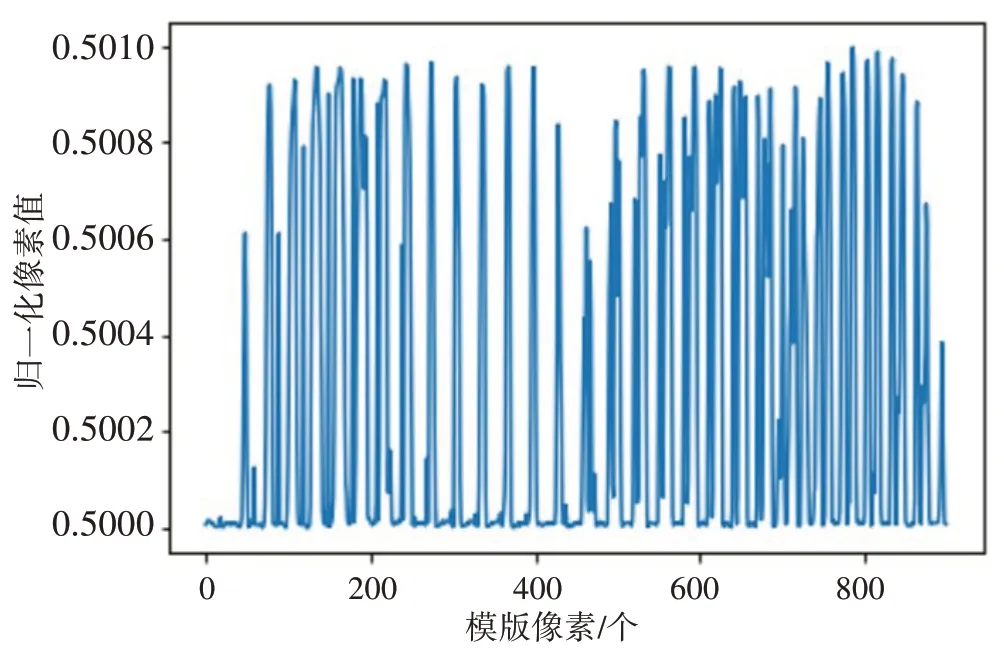
图5 掩膜像素灰度值及分布情况Fig.5 The pixel value and grey value of mask generated by U-Net and their distribution
1.2.1 首次分割在得到图3所示的首次全局分割掩膜后,我们通过分水岭算法找到连通域[15]。这些连通域中有的包含多个细菌,通过简单比较连通域与最大单个细菌面积,挑选出包含多个细菌的连通域进行二次分割。
1.2.2 二次分割选择更高的阈值为U-Net 输出二值掩膜,称之为“主要部分”。在得到更加细致的分割掩膜的同时,也丢失了部分包含细菌的部分。为了找回其余部分的细菌,我们将首次分割结果进行开运算,将得到的结果和首次分割结果相减得到的结果称之为“残差部分”,经过开运算后得到的“残差部分”可以减少两部分相减造成的边缘轮廓和毛刺。然后再次分别搜索“主要部分”和“残差部分”中的连通域,这两部分的连通域几乎覆盖了所有且独立的细菌位置。图6显示了二次分割的过程和分割后的细菌位置定位,图7显示了黏连严重的细菌经过渐进分割后得到的结果。

图6 渐进分割过程Fig.6 The progressive segmentation

图7 黏连细菌分割结果Fig.7 Segmentation of adhesive bacteria
2 分类和计数
为了比较结果的准确性,我们选择了两种深度卷积神经网络进行分类和计数。
2.1 深度卷积神经网络
VGGNet模型[16]:VGGNet使用3×3卷积核和3×3池化核,通过不断深化网络结构来提高性能。堆叠式小卷积核优于大卷积核,因为多非线性层可以增加网络深度以保证更复杂模式的学习,而且代价相对较小(参数较少)。
ResNet 模型[17]:当神经网络层数增多时,由于存在梯度消失问题,训练困难。ResNet 模型主要使用3×3的卷积核。模型的核心内容是残差,如图8所示,其学习特性表示为H(x)。当输入为x时,残差是F(x)=H(x)-x,那么原始学习特性应该为:H(x) =F(x) +x,残差的学习结构称为跳跃连接,可以从网络的一层激活,然后快速反馈到另一层甚至更深层的神经网络。这也使卷积层能够根据输入特征学习新的特征,从而获得更好的性能。
2.2 数据源

图8 ResNet 模型Fig.8 The ResNet model
实验中的显微图像为滨州医学院公共卫生与管理学院实验室采集的革兰氏染色菌显微图像,并对用以U-Net 模型、ResNet50 模型和VGG19 模型所使用分割后的训练子图进行标注。标签将革兰氏染色细菌分为阳性杆菌、阳性球菌、阴性杆菌和阴性球菌4类。最后,总共有600个细菌图像被标记为训练集,100 个细菌图像被标记为测试集。然后,通过水平翻转、垂直翻转、水平垂直翻转和转置等操作,将训练集扩大到3 000个,测试集扩大到500个。
2.3 训练
分别使用ResNet50 模型和VGG19 模型进行训练。为了防止过拟合,当训练集的准确度小于95%时,提前停止对测试集参数进行保存。经过100次迭代,最终选取在训练集和测试集均有最高准确率时的模型参数,VGG19 模型在测试集中的准确率达到88%,ResNet50 模型的准确率达到90%,两者在训练集中的准确率均达到95%。
2.4 识别和计数
由于细菌特征较少,我们将渐进分割后得到的细菌放在白色背景的矩阵中,以此减少噪声干扰,将细菌输入VGGNet模型和ResNet模型进行识别和计数。
3 实验结果
我们对多张革兰氏染色细菌显微图像进行实验,并分别记录两种深度学习模型识别计数的结果。图9显示了VGGNet模型和ResNet模型的结果。表1和表2分别列出了阳性及阴性菌和杆菌及球菌的混淆矩阵[18]。混淆矩阵对应行代表实际情况,列代表深度学习判定的情况。实验结果表明,VGGNet 模型和ResNet模型在阴性细菌和阳性细菌的分类中均有较好的表现。然而,在杆菌和球菌的分类上,ResNet模型优于VGGNet模型。另外,ResNet比VGGNet平均少花9%的时间,在图9所展示的3 组图像结果中ResNet 和VGGNet 的计算时间分别为178.1、111.4、95.9 s和185.4、137.4、100.2 s。

图9 识别分类结果(显微镜放大倍数为1 000倍)Fig.9 Classification results(The microscope magnifies 1 000 times)

表1 阳性菌及阴性菌混淆矩阵Tab.1 Positive-negative Confusion matrix
4 结语
使用深度学习算法进行细菌分类和计数,可以节省时间和人力,本文中的图片经比较,人眼计数与ResNet 计数结果误差率不超过5%。与此同时,单幅图片人工计数需要数个小时,而使用ResNet 模型计数仅需要几分钟。一些分辨率低的黏连细菌目前仍旧难以分割,同样也难以进行人工识别。但随着深度学习模型的能力越来越强,计算机视觉将实现低分辨率重建和更精确的分割。因此,细菌计数在未来将更加依赖于先进的人工智能技术。
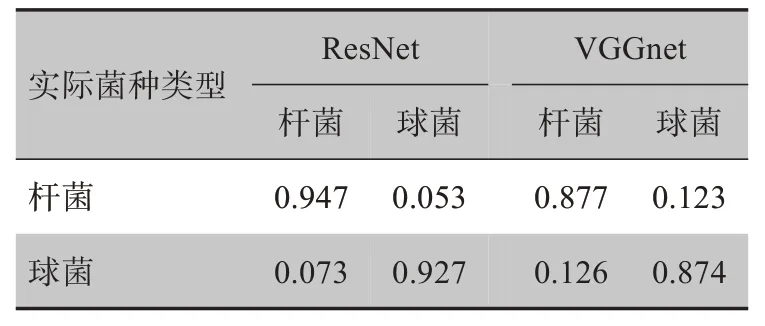
表2 杆菌及球菌混淆矩阵Tab.2 Bacilli and cocci Confusion matrix

