单域抗体T7噬菌体展示文库构建与鉴定
徐 海,王 健,郭长明,董洪燕,邓碧华,侯继波,*
(1.江苏农牧科技职业学院 江苏省兽用生物制药高技术研究重点实验室,江苏 泰州 225300; 2.江苏省农业科学院 动物免疫工程研究所,江苏 南京 210014)
1985年Smith首次利用丝状噬菌体表达外源基因,开启了噬菌体展示技术的研究[1-2]。将外源DNA片段插入噬菌体基因组中,使外源基因与噬菌体衣壳蛋白基因融合表达,所编码的外源蛋白被展示在噬菌体的表面,其最大优势在于将基因型与表型进行统一。噬菌体展示文库的构建与应用进一步扩展了该技术的应用范围,例如蛋白质之间的互作[3-4]、配体与受体的筛选[5-6]、生物标记[7]、抗原表位发掘[8-9]、疫苗和药物研制[10-12]等。用于构建文库的噬菌体包括丝状噬菌体、T7噬菌体、T4噬菌体、λ噬菌体与MS噬菌体[13]等,不同噬菌体展示拷贝数、多肽长度、展示末端,以及氨基酸偏好等方面有差异。目前,以氨基端展示的丝状噬菌体和羧基端展示的T7噬菌体应用较为广泛,可以展示随机氨基酸多肽文库,也可构建抗体可变区文库。
1993年Hamers-Casterman等[14]发现骆驼血清中同时存在2种结构免疫球蛋白:2条重链和2条轻链组成的传统4链抗体;天然缺失2条轻链,仅有2条重链的特殊结构抗体。后续研究发现,驼科动物、鲨鱼和一些软骨鱼类的体内也存在类似的抗体分子[15]。克隆该特殊抗体的重链可变区即可为单域抗体(Variable domain of heavy chain antibody, VHH),由于VHH的相对分子质量约为15 ku,仅为常规抗体的十分之一,因此也被称为纳米抗体。纳米抗体结构稳定具有较高的亲和力和抗原结合特性,相对分子质量小容易穿透组织屏障,易于生产制备和进行基因工程改造,凭借其独特的优势成为替代单克隆抗体的理想选择[16]。近年来,通过构建纳米抗体文库进行生物淘选已有广泛的报道,涌现出许多新的研究成果,随着分子生物学的进步、纳米抗体进一步的完善,必将产生更加深远的影响[17-18]。
由于展示文库的序列多样性、拷贝数直接关系到生物淘选的得率,构建高品质的展示文库一直是研究人员不断的追求。本研究在常规噬菌体展示文库构建方法基础上,通过扩大羊驼血样来源、优化VHH基因扩增引物、噬菌体载体的选择,以及多次建库等方式,构建高品质的羊驼源天然纳米抗体T7噬菌体展示文库,为后续文库筛选工作的顺利开展提供保障。
1 材料与方法
1.1 实验动物与主要试剂
健康成年羊驼6只,雌雄各半,分别饲养于扬州、泰州动物园,未经过主动免疫。外周血淋巴细胞分离液购自索莱宝公司;总RNA提取试剂盒、反转录试剂盒、Taq酶、T4连接酶、T载体、限制性内切酶等分子生物学试剂购自大连宝生物公司;T7 select 415-1b克隆试剂盒购自Merck公司;其他试剂均为进口或国产分析纯。
1.2 引物设计
根据文献报道[19-20]使用的引物以与GenBank公布的Lamapacos单链抗体序列进行优化调整,设计2对引物进行套式PCR扩增。其中F1位于抗体编码的前导序列保守区,R1位于抗体CH2结构域保守区,该对引物可扩增出大小约1 000 bp的VH-CH1-CH2和700 bp的VHH-CH2片段;F2和R2-1、R2-2用于从VHH-CH2片段中扩增约400 bp的VHH片段,如图1所示。T7 select up和T7 select down扩增T7噬菌体载体多克隆位点上下游片段,用于检测外源基因的插入。文库构建与鉴定引物由擎科生物公司合成,如表1所示。
1.3 淋巴细胞总RNA提取与cDNA合成
颈静脉采集6只羊驼抗凝血各5 mL,淋巴细胞分离液分离,具体操作步骤参照说明书进行。提取总RNA,Nanodrop测定含量。以总RNA为模板,分别以Random 6和Oligo dT2种引物进行反转录成cDNA。反应程序为:40 ℃ 40 min;70 ℃ 15 min;12 ℃终止,-80 ℃保存备用。
1.4 VHH基因扩增
以cDNA为模板,F1、R1为引物,PCR扩增羊驼重链抗体可变区编码基因,反应程序为:94 ℃ 3 min;94 ℃ 30 s;52 ℃ 30 s;72 ℃ 1 min,30个循环;72 ℃ 10 min,切胶回收约700 bp片段。以回收的片段为模板,再以F2、R2-1和R2-2为引物,反应程序为:94 ℃ 3 min;94 ℃ 30 s;50 ℃ 30 s;72 ℃ 30 s,30个循环;72 ℃ 10 min,PCR扩增约400 bp的VHH基因,胶回收目的条带,Nanodrop定量,-80 ℃保存备用。
1.5 质粒文库构建与鉴定

图1 VHH基因扩增与噬菌体表面展示示意图Fig.1 Schematic of VHH gene PCR amplification and phage surface display

表1 文库构建与鉴定引物
将回收的VHH基因以摩尔比3∶1与pMD18-T simple连接,转化大肠埃希菌DH5α感受态细胞。转化产物涂布在含有Amp+/IPTG/X-gal LB固体培养基的培养皿,37 ℃过夜。计算菌落总数和蓝色菌落数,计算质粒文库的容量和重组率。质粒库容量=菌落总数×转化产物量;重组率=(菌落总数-蓝色菌落数)/菌落总数×100%。剔除蓝色菌落,收集培养皿中所有菌落,LB液体培养基重悬菌落,提取质粒,EcoRⅠ、HindⅢ双酶切鉴定,回收400 bp目的片段。
1.6 T7噬菌体展示文库构建
将质粒文库中酶切回收的VHH片段与同酶处理的T7 selectEcoRⅠ/HindⅢ载体以摩尔比1∶1.5连接,取5 μL连接产物与25 μL包装蛋白混合,25 ℃反应2 h,反应体系中加入270 μL LB液体培养基终止反应,即为拯救的噬菌体文库。取5 μL包装产物10倍梯度稀释并与200 μL过夜培养的T7噬菌体宿主细菌BLT5403混合,经双层琼脂夹心法测定噬菌体滴度,计算文库容量;挑取单噬斑,PCR检测目的基因插入情况,计算重组率。噬菌体库容量=噬斑数×稀释倍数×300;重组率=PCR阳性噬斑数/检测噬斑总数×100%。随机挑选20个噬斑PCR阳性产物进行序列测定,采用DNAStar序列分析软件对测序结果进行比对分析。
1.7 T7噬菌体展示文库的鉴定
将剩余的295 μL包装产物转接100 mL对数生长期BLT5403,37 ℃摇床培养3 h至宿主菌完全裂解,PEG-NaCl沉淀法回收噬菌体,双层琼脂夹心法测定文库滴度,Western-blot检测噬菌体表面展示的纳米抗体,通过扬州大学测试中心扫描电镜观察重组噬菌体形态。以MOI=1∶100转接回收的噬菌体至新的BLT5403培养液,连续传5代,噬斑PCR再次检测重组率。
2 结果与分析
2.1 VHH基因扩增与质粒文库构建
从6份羊驼抗凝血中分离淋巴细胞,提取总RNA,反转录成cDNA作为模板,用F1、R1引物扩增重链抗体可变区,分别得到1 000和700 bp两条带(图2-A)。回收700 bp条带作为模板,用F2、R2-1/R2-2引物扩增得到约400 bp的VHH基因(图2-B)。将VHH基因插入T载体,构建质粒文库,提质粒经EcoRⅠ和HindⅢ双酶切,获得与预计大小相符的条带,说明质粒文库构建成功(图2-C)。经过10次重复构建,质粒文库的容量达到4.05×108CFU (colony-forming units),见表2。

A、B,套式PCR扩增羊驼重链抗体可变区;M,DL2000 marker;1-6,6份羊驼样品。C,酶切鉴定VHH质粒文库;M,DL5000 marker;1,VHH质粒文库双酶切鉴定。A, B, Heavy chain antibody variable region amplification by nested PCR; M, DL2000 marker; 1-6, Six alpaca samples. C: Identification of VHH plasmid library; M, DL5000 marker; 1, Enzyme digestion of VHH plasmid library.图2 VHH基因扩增与质粒文库鉴定Fig.2 PCR amplification of VHH gene and enzyme digestion of plasmid library
2.2 T7噬菌体展示文库构建
将VHH基因插入T7噬菌体p10B基因下游的多克隆位点,通过包装蛋白包装拯救重组噬菌体。测定每一次包装产物的噬菌体滴度,均能达到108PFU (plaque forming unit),混合5次拯救产物即为最终T7噬菌体展示文库,库容达到2.73×109PFU,满足文库筛选的要求,结果见表2。
2.3 T7噬菌体展示文库的鉴定
用双层琼脂夹心法测定文库滴度,随机挑取噬菌体噬斑,用多克隆位点下游引物检测VHH基因插入情况,成功插入外源基因的噬斑PCR可扩增出约600 bp条带而未插入外源基因的噬斑可扩增出200 bp条带。从图3-A可以看出,VHH基因成功插入T7噬菌体基因组,重组率高达100%。将噬菌体展示文库盲传5代,噬斑形态大小不均一(结果未列出),挑选10个正常大小噬斑(图3-B,1-10)、10个偏大噬斑(图3-B,11-20)进行PCR检测,其中4个噬斑无VHH基因插入,该结构说明构建的噬菌体展示文库中传代过程中会出现目的基因丢失,重复传代的文库不能用于亲和筛选。
T7 select 415-1b噬菌体侵染BL21大肠埃希菌,以415个拷贝形式展示50个氨基酸以内的外源多肽,本研究通过更换BLT5403宿主,以p10B、p10B-VHH两种蛋白组装噬菌体头部,首次尝试对150个氨基酸大小的纳米抗体进行展示,故对纳米抗体的表达与重组噬菌体颗粒结构做进一步鉴定。利用p10B蛋白氨基端的Tag标签进行Western-blot检测,从图4可以看出,成功检测到p10B、p10B-VHH两种蛋白,纳米抗体在噬菌体表面成功展示,但在p10B和p10B-VHH之间出现2条杂带,暗示VHH基因在表达时存在提前终止现象。电镜检测显示,T7噬菌体展示文库样品中观察到完整的重组噬菌体颗粒(图5-B)与对照T7 select 415-1b结构相似,这一结果说明,通过2种结构蛋白的组合可以组装出完整的噬菌体颗粒,进而突破外源蛋白长度限制,提高噬菌体的展示能力。
2.4 VHH纳米抗体序列分析
随机挑取20个阳性噬斑PCR产物进行序列测定。通过与GenBank报道的羊驼源纳米抗体序列进行同源性比对分析,结果显示,构建的T7噬菌体文库展示的均为羊驼抗体重链区序列。氨基酸多序列比对可见明显的纳米抗体特征区域,其中骨架区(FR1-4)氨基酸序列较为保守,而互补决定区(CD1-3)氨基酸序列差异较大,20个克隆的CD3区各不相同,间接说明构建的文库具有较高序列多样性,见图6。
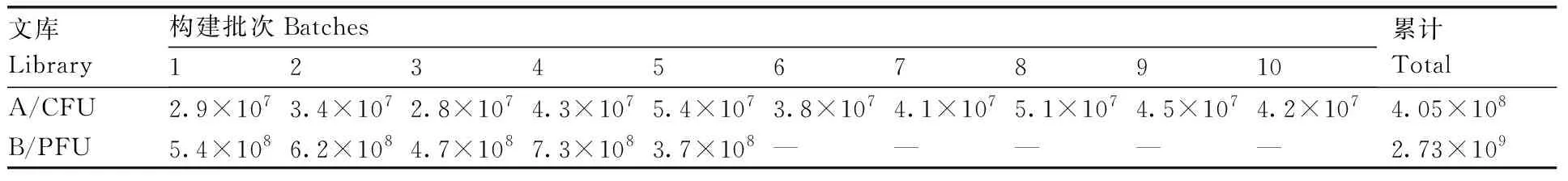
表2 质粒文库(A)与噬菌体文库(B)库容

A,第1代文库重组率测定;M,DL2000 marker;1-20,20个噬菌体噬斑PCR检测;B,第5代文库重组率测定;M,DL2000 marker;1-20,20个噬菌体噬斑PCR检测。A, The recombination rate of the first generation of library; M, DL2000 marker; 1-20, PCR test of 20 unique phage clones; B, The recombination rate of the fifth generation of library; M, DL2000 marker; 1-20, PCR test of 20 unique phage clones.图3 噬斑PCR检测噬菌体展示文库重组率Fig.3 Identification of recombinant phage by plaque PCR testing

M,预染蛋白Marker;1,T7 select 415-1b 噬菌体;2,第1代T7噬菌体展示文库;3,第5代T7噬菌体展示文库。M, Protein marker; 1, T7 select 415-1b phage; 2, The first generation of T7 phage display library; 3, The fifth generation of T7 phage display library.图4 T7噬菌体衣壳蛋白Western-blot鉴定Fig.4 Western-blot of T7 phage capsid protein

A,T7 select 415-1b噬菌体;B,T7噬菌体纳米抗体展示文库。A, T7 select 415-1b phage; B, T7 phage displayed nano-antibody library.图5 电镜检测噬菌体形态Fig.5 Electron microscopy observation of T7 phages
3 讨论
噬菌体展示抗体文库是亲和筛选的重要工具,可分为免疫文库和天然非免疫文库[21]。对于天然非免疫文库,序列多样性是衡量文库质量的重要指标。构建的天然非免疫库中应避免重复序列的出现,以保证库容量全部为有效容量,这样可以从文库中筛选到更多种类的抗体。为此,本研究通过比对已报道的羊驼抗体序列,选择抗体前导区、CH2区的保守序列设计引物,确保PCR扩增的特异性;同时比对VHH基因并在引物设计时引入兼并碱基,以提高扩增产物序列多样性。另外,考虑到动物个体差异,本研究采集6只羊驼外周血分离淋巴细胞用于VHH基因的扩增,并经多次质粒文库和噬菌体文库的构建,进一步提高序列的多样性。随机挑取20份阳性噬斑PCR产物进行测序分析(图6),所测得序列的CDR3区各不相同,说明所采取的策略达到预期效果。

图6 VHH纳米抗体随机克隆氨基酸多序列分析Fig.6 Nanobody amino acid sequence analysis
除了提高纳米抗体序列多样性,其在噬菌体表面的展示拷贝数亦至关重要。T7 select 415-1b载体能够展示415拷贝50个氨基酸多肽,T7 select 10-3b载体能够展示5~15拷贝1 200个氨基酸的蛋白质[22]。羊驼纳米抗体是150个氨基酸的小分子蛋白,如何选择合适的噬菌体载体实现纳米抗体高效展示关系到后期文库筛选亲和力的强弱。本研究选择T7 select 415-1b载体,借用BLT5403宿主表达的p10B蛋白与重组噬菌体表达p10B融合蛋白共同完成噬菌体衣壳的组装,成功拯救出完整的重组噬菌体颗粒,实现高丰度展示文库的构建。既突破了T7 select 415-1b载体50个氨基酸的长度限制,又比T7 select 10-3b载体展示拷贝数大幅提高。从Western-blot检查结果(图4)可以看出,宿主表达的p10B蛋白与重组噬菌体表达的p10B-VHH融合蛋白WB条带显示强度相当,间接说明采用该模式可以大幅提高纳米抗体表面展示拷贝数,但每个重组噬菌体展示的确切拷贝数目前尚无法测定。
由于首次采用T7 select 415-1b载体展示羊驼纳米抗体,所构建文库的传代稳定与文库中重组噬菌体的形态特性有待检测。将文库盲传5代,双层琼脂夹心法测定噬菌体滴度发现,噬斑大小不均一,夹杂成斑快、直径大的噬斑,PCR检测显示(图3-B)部分噬菌体已丢失VHH基因,在无外源基因负载的情况下,生长性能得到改善导致噬斑变大。因此,仅低代次噬菌体文库可用于亲和筛选的研究。T7噬菌体表面高拷贝展示纳米抗体分子,分子间的空间位阻可能影响噬菌体头部的组装,电镜观察显示,文库中的重组噬菌体(图5-B)与供体噬菌体T7 select 415-1b(图5-A)表现相同的形态特征,说明通过p10B与p10B-VHH两种蛋白能够实现噬菌体头部的正确组装,但二者的比例可能由噬菌体根据空间位阻情况自行调整。
噬菌体展示技术建立以来,构建噬菌体抗体展示文库进行亲和筛选的研究已有诸多报道[23-24]。本研究旨在前期研究的基础上,进一步提高羊驼天然纳米抗体噬菌体展示文库的序列多样性与表面展示拷贝数,构建高品质文库进而应用于亲和筛选的研究工作。

