基于图像融合的乳腺肿瘤感兴趣区域边缘识别*
曾果,刘彦荣,王力,许方彧,蒋烈夫,靳玉川
(1.南阳市第二人民医院医学影像中心,南阳 473000;2.南阳医学高等专科学校,南阳 473000;3.河北医科大学,石家庄 050017)
1 引 言
乳腺肿瘤在临床检测中具有隐匿性,不易被识别[1-3]。目前使用计算机辅助检测乳腺肿块是预防乳腺肿瘤的有效方法。而针对乳腺肿瘤的感兴趣区域边缘检测和识别作为检测的第一步,其准确率直接影响乳腺肿瘤的检测和诊断[4-6]。随着医疗水平的提高,相关学者发现通过核磁共振成像技术以及CT技术等均可以检测患者的乳腺情况。在对乳腺图像的识别中多使用ROI识别检测技术,该技术在识别中常采用单一仪器检测的图像,而单一图像对乳腺区域的病理反馈不足,易影响识别的准确率[7-9]。
2 基于图像融合的乳腺肿瘤感兴趣区域边缘识别方法设计
本研究设计的图像融合乳腺肿瘤感兴趣区域边缘识别方法流程,见图1。
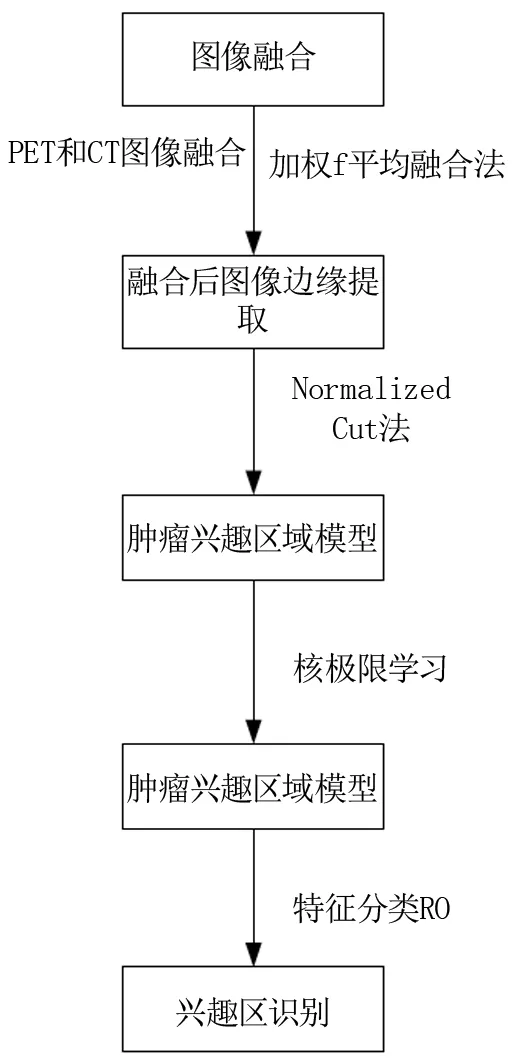
图1 图像融合的乳腺肿瘤感兴趣区域边缘识别流程图
2.1 加权平均图像融合方法
在对乳腺肿瘤的检查中,正电子发射型计算机断层显像技术(positron emission computed tomography, PET)和CT检测对比其他技术获得的图像更清晰[10-12]。因此,本研究以PET和CT图像为例,通过两个图像的融合得到更准确的病变图像。假设将两幅源图像分别定义为F1和F2,字母R表示融合的图像,通过加权平均融合后F1和F2图像可表示为:
R(m,n)=w1F1(m,n)+w2F2(m,n)
(1)
其中,m代表图像像素行号,n代表图像像素的列号。在图像F1中的加权系数设为w1,图像F2中的加权系数为w2,当w1+w2=1且w1=w2=0.5时,表示平均融合,可以使用两幅图像的相关系数来确定公式中的权值,即:
(2)
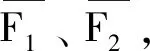
(3)
通过结合两幅源图像中的像素灰度信息,可以有效减少源图像中的噪音,提高信噪比。
2.2 Normalized Cut法自动提取边缘
对多个源图像融合后,采用一致相干斑噪的各向异性扩散技术进行去噪和边缘增强,同时将带有权重领域灰度信息的Normalized Cut自动分割,把待分割图像存在的像素点组成像素点集V,像素点集的组建是要保证集内像素点的相似度为像素点间边上的权重E,并得到无向带权图G=(V,E)。假设获得的诊断图像尺寸为N×N,那么诊断图像中像素点相似度的矩阵可以表示为W∈N2×N2,而在图像像素点集V中分为两个不相交的部分,设为A和B,满足A∪B=V,A和B之间的权重设为Wcut(A,B),当Wcut(A,B)值取最小时,即可得到图像分为二的最优分割。为避免分割后出现个别孤立点的情况,使用Normalized Cut算法予以克服。A和B两个点集合中各节点权值的总和分别为assoc(A,V)和assoc(B,V)。同时,将权重与各节点权值的总和的比值相加得出WNcut(A,B),获得图像上的正规化最小分割,即:
(4)
得到图像通过A到B之间的最优分割值WNcut(A,B),对WNcut(A,B)的求解为NP(non-deterministic polynomial,NP)完全问题,定义x作为|V|维的指示向量,当xi=1时,说明定义下的像素节点属于A,而当xi=-1时则说明节点属于B,其中节点i在像素点中的连接度为di=∑jW(i,j),j为与i相邻的节点,将图像中的对角阵设为D,并满足D(i,i)=di,设k=∑xi>0di/∑idi,则WNcut的表达可转为:
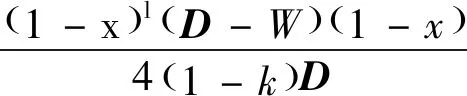
(5)
其中,l代表图像分割系数,即可完成最优分割的求解。
2.3 建立肿瘤兴趣训练模型
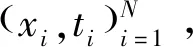
(6)
同时每组特征均通过独立基分类。核极限与神经算法不同,优化的目标是训练中的误差和权重的范数,并保证数值在最小范围内,即:
min∑‖β·h(xi)-ti‖2,min‖β‖
(7)
其中,h(xi)代表隐含层输出矢量,β则代表输出权重。在利用约束优化问题时,可以将优化目标设为:
(8)
其中,ξi代表训练样本xi所对应的输出节点的训练误差向量,c则代表训练误差最小化以及边缘距离最大化之间的权衡正则化参数。给定一个新样本,利用ELM(extremelearningmachine,ELM)的乳腺肿瘤感兴趣区域分类给出的公式得出:
(9)
其中,g作为构成d组的特征感兴趣区域分类的向量,H代表隐藏层下结构区域内的肿瘤兴趣率。不同特征差异属于非齐次状态,具有不同概率密度函数。采用Mercer条件,将ELM中的公式以核矩阵形式表现出来:
ΩELM=HHt:ΩELM(i,j)=h(xi)·h(xj)=K(xi,xj)
(10)
可以推导出KELM(extremelearningmachinewithkernel,KELM)中的输出函数,见式(11)。
(11)
通过核函数KLF(kernel function, KLF)来计算K(x,xi),同时计算核函数ELM时,无需给定隐藏层节点数目和巡展最优数目,以减少时间复杂度。通过给定训练样本集{(xi,ti)|xi∈RN,ti∈RM,i=1,2,...,N}和得到的核函数K(x,xi),计算输出为:
(12)
在计算过程中,可直接使用K(xi,xj)来替代特征映射函数的表现。临床上由于各组特征和结果的相关程度不同,因此,模型应确定每组的特征权重因子。
2.4 特征分类ROI提取识别
在上述模型中,选择特征的子块(x,xN)计算多个特征,并进行归一元化。所有特征取值均除以其最大值,将所有的特征取值分布在0到1区间内。图像中位置特征的取值均可归一元到[0,1]之间,并与权重系数ω相乘。每个子块的特征组成一个9维向量,输入到分类器中,提取图像融合后的图像ROI。将分割后的肿瘤边缘和医生手动提取的图像边缘对比,最终的诊断结果表示为:
F=max(W·F)
=max([ω1,ω2]·[f1,f2])
(13)
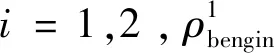
3 实验论证分析
为验证本研究方法的可行性,使用某医院病理图像资料库中乳腺肿瘤患者的图像资料并取得患者同意,对比文献[2]、[4]、[5]及本研究方法进行肿瘤感兴趣区域边缘识别的效果,图像资料包括CT及RET资料,共785张。本研究图像构成见表1。
3.1 实验过程
针对乳腺肿瘤的感兴趣区域边缘识别,以结果方差和敏感性作为评判标准,在实验中使用十折交叉验证法来测试其准确性,将乳腺肿瘤患者作为一个图像集,并分层不同交叉的子集,每次实验均从子集中选取训练集以及识别集,再对上述四种方法进行训练,并于训练后实施肿瘤感兴趣区域边缘识别。为保证实验的可靠性,每十次实验为一组,取50组实验结果的平均值。
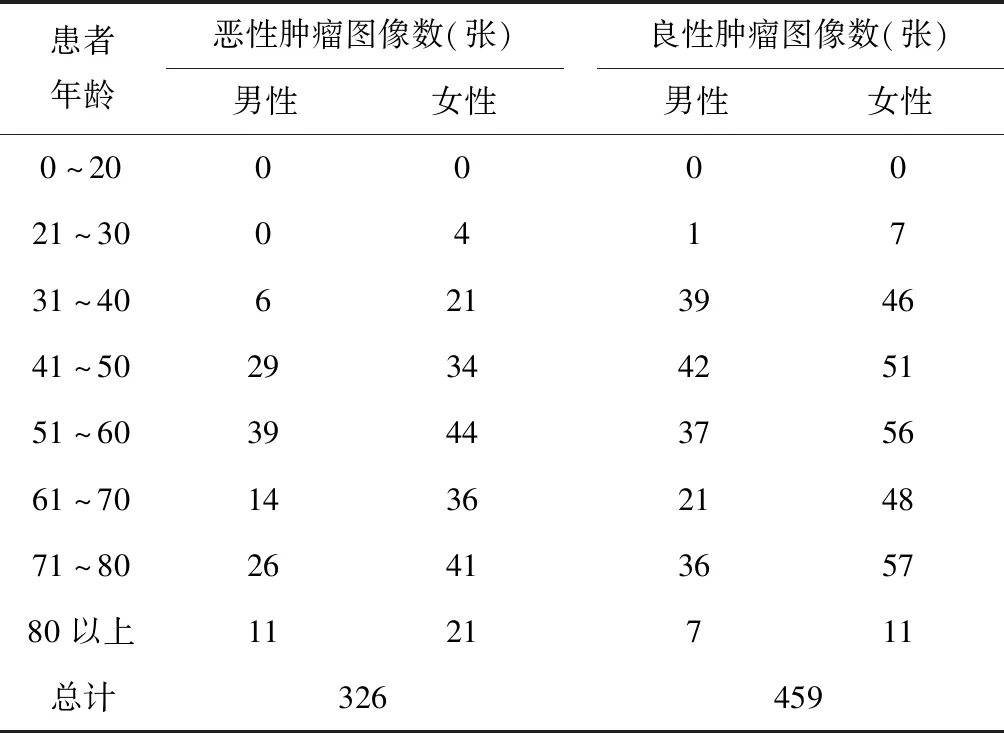
表1 本研究乳腺肿瘤病理图像构成
3.2 实验结果
图像中的肿瘤包括良性肿瘤和恶性肿瘤。识别过程中,由于肿瘤类型会对肿瘤感兴趣区域的识别造成影响,因此,对四种方法的分类准确率进行对比,结果见表2。

表2 四种方法在实验结果中的指标比较
方法一、二、三、四分别为本研究方法、文献[2]、文献[4]和文献[5]的方法。由表2可知,本研究方法在分类正确率和恶性、良性肿瘤的敏感性指标上,以及恶性、良性肿瘤的预测指标上均优于其它方法。恶性肿瘤边感兴趣区域边缘识别结果见表3。

表3 恶性乳腺肿瘤感兴趣边缘识别结果
由表3可知,本研究方法相比其他方法准确率更高,本研究算法和方法二的运算准确率标准方差均为E-05数量级,说明该算法的稳定性更好,同时单次识别中波动较小,可行度较高,对乳腺良性肿瘤感兴趣边缘识别结果,见表4。
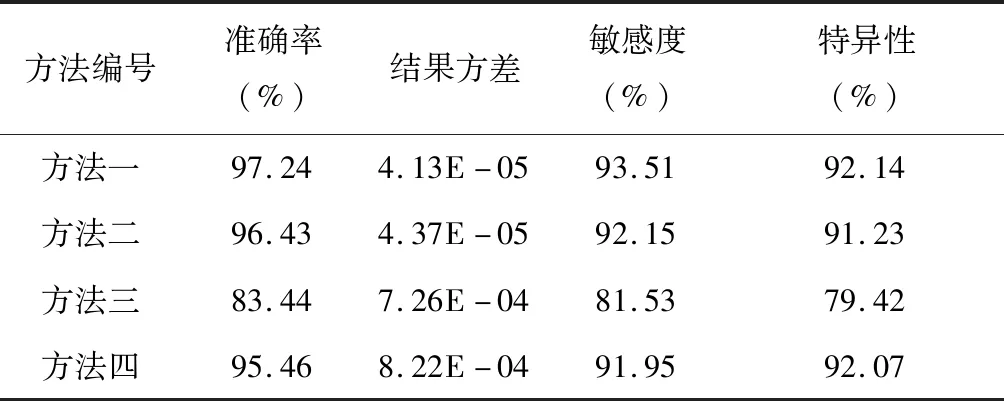
表4 良性乳腺肿瘤感兴趣边缘识别结果
由表3、表4可知,方法一识别良性乳腺肿瘤的准确率、敏感度以及特异性较识别恶性乳腺肿瘤均有所降低,说明良性肿瘤的感兴趣区域更加复杂,识别时较为困难。方法四虽对良性肿瘤感兴趣的识别性能优于对恶性肿瘤感兴趣区域的识别,但仍低于本研究方法的识别性能,表明本研究方法的识别准确性更高,具有可行性。
4 结论
本研究通过使用图像融合技术,融合多设备的图像结果,提高乳腺肿瘤感兴趣区域边缘识别时图像的稳定性,实验结果表明,本研究方法的准确率更高。但由于使用图像融合技术,在建立肿瘤感兴趣模型时,需要考虑更多的权重数值,增加了计算量。因此,在今后的研究中仍需进一步完善。

