雨生红球藻caleosin基因的克隆、序列分析及表达特性
朱 琴,张宏江,张春辉,崔红利,薛金爱,李润植
(山西农业大学农学院分子农业与生物能源研究所,太谷 030801)
雨生红球藻(Haematococcus pluvialis)是一种单细胞真核绿藻,在分类学上属于绿藻门(Chlorophyta)、红球藻属(Haematococcus)[1]。雨生红球藻细胞在适宜环境条件下为含少量虾青素的绿色游动形态,逆境条件下(如高光、高温、高盐)转变为富含虾青素的红色不动细胞[2-3]。虾青素(astaxanthin)是一种红色酮式类胡萝卜素,其抗氧化活性远大于类胡萝卜素和天然维生素E,被誉为“超级抗氧化剂”[4-5]。雨生红球藻是目前已知的虾青素含量最高的物种,在胁迫条件下虾青素含量最高可达到细胞干重的6%[6],因此被认为是提取天然虾青素的理想材料[7]。
雨生红球藻虾青素主要是以酯化形式(虾青素单酯和双酯)存在,存储于油体中[8]。油体又称脂滴或脂质体,是高等植物种子、花粉、裸子植物种子、真菌和藻类中储存中性脂质的细胞器[9-10]。油体主要分为三酰甘油(triacylglycerols, TAG)内层,磷脂单分子(phospholipids, PL)外层及镶嵌其中的油体结合蛋白。目前在不同种子的油体中已鉴定出3类完整的油体结合蛋白,即油蛋白(oleosin)、钙蛋白(caleosin)和甾体蛋白(steroleosin)[11],它们广泛存在于高等植物和藻类的油体中[12]。
油体结合caleosin和oleosin的结构相似,都具有稳定油体的功能[13],形成差异的原因主要是这2种蛋白的3个结构域的长度不同:caleosin和oleosin分别有85%和55%的残基在其N末端和C末端结构域中突出,并覆盖于油体表面,其余15%和45%的残基位于中央疏水结构域[14]。据推测,当使用相同量的三酰甘油时,等量的caleosin比oleosin能覆盖更多的油体表面积,可见caleosin似乎是比oleosin更有效(或经济)的结构蛋白[15]。caleosin在植物体中具有重要的生物学功能(如参与膜融合和脂肪体融合等过程)[16],然而,与其他高等植物芍药[16]、蓖麻[17]、油菜[18]等相比,藻类中油体结合蛋白的生理和功能特性仍缺乏研究,雨生红球藻中还未见caleosin基因的相关报道。
本研究通过对雨生红球藻caleosin(HaeClo)的基因进行分子克隆与表达分析,旨在揭示HaeClo蛋白的生物学功能,为HaeClo基因在植物基因工程、生物技术及生物化学的应用上提供科学依据,进而为通过遗传改良HaeClo蛋白来提高雨生红球藻虾青素的含量奠定基础。
1 材料与方法
1.1 材料及培养条件
本试验所用藻种为雨生红球藻,现存于山西农业大学分子农业与生物能源研究所。将雨生红球藻接种于MCM(modified Chalmers medium)培养基上,于(22.5±1.0)℃、光照条件下静置培养,光照强度为1 300 lx,光 /暗周期为12 h/12 h,且每 8 h摇匀1 次。本试验所用受体菌株为大肠杆菌(Escherichia coli)BL21(DE3),载体质粒为pET-28a(+),均保存于山西农业大学分子农业与生物能源研究所。
1.2 试验方法
1.2.1 总RNA的提取
取对数期生长的雨生红球藻作为试验样品,通过Takara公司的试剂盒,用TRizol法提取总核糖核酸(ribonucleic acid, RNA),操作步骤详见说明书。所提RNA必须于-80℃冰箱中保存以避免降解。琼脂糖凝胶电泳检测后测其浓度和纯度[19]。
1.2.2 cDNA模板制备和cDNA末端快速扩增技术(RACE)模板制备
本试验用Takara公司的反转录试剂盒(Prime-ScriptTMRT Reagent Kit with Gdna Eraser)合成 cDNA模板,用Clontech公司的试剂盒(SMARTer TM RACE cDNA Ampli-cation Kit)制备cDNA末端快速扩增技术(rapid-amplification of cDNA ends, RACE)模板,以上操作步骤详见试剂盒说明书[20]。
1.2.3 同源克隆及RACE扩增
从NCBI GenBank中查找出与HaeClo基因序列亲缘关系较近的4种绿藻:盘藻(Gonium pectoral)、莱茵衣藻(Chlamydomonas reinhardtii)、团藻(Volvox carteri f. nagariensis)和小球藻(Chlorellavulgaris)。序列比对获得高度保守的氨基酸序列,用CODEHOP软件设计HaeClo的同源克隆引物(F1,R1和F2,R2)。以制备的cDNA为模板结合设计的引物,按照TaKaRa LA Taq®扩增体系进行聚合酶链式反应(polymerase chain reaction,PCR),从而获得HaeClo的同源克隆片段。1.2%的琼脂糖凝胶电泳检测后回收目的条带用于后续试验。在此基础上设计5'和3'端引物(5'RACE R3,R4和3'RACE F3,F4),以制备的RACE cDNA、5'和3'端引物为模板进行PCR扩增。将扩增产物进行1%琼脂糖凝胶电泳检测后通过DNAStar软件拼接,得到HaeClo基因cDNA序列全长,并据此设计表达框引物(F5,R5)。以上设计的引物信息均由上海生工生物工程公司合成,详见表1。
1.3 生物信息学分析
HaeClo的生物信息学分析需要用到大量在线软件和本地软件,现将本试验分析所用软件名称、用途及网址收集整合至表2。

表1 文中用到的引物信息Tab. 1 Primer information used in the article
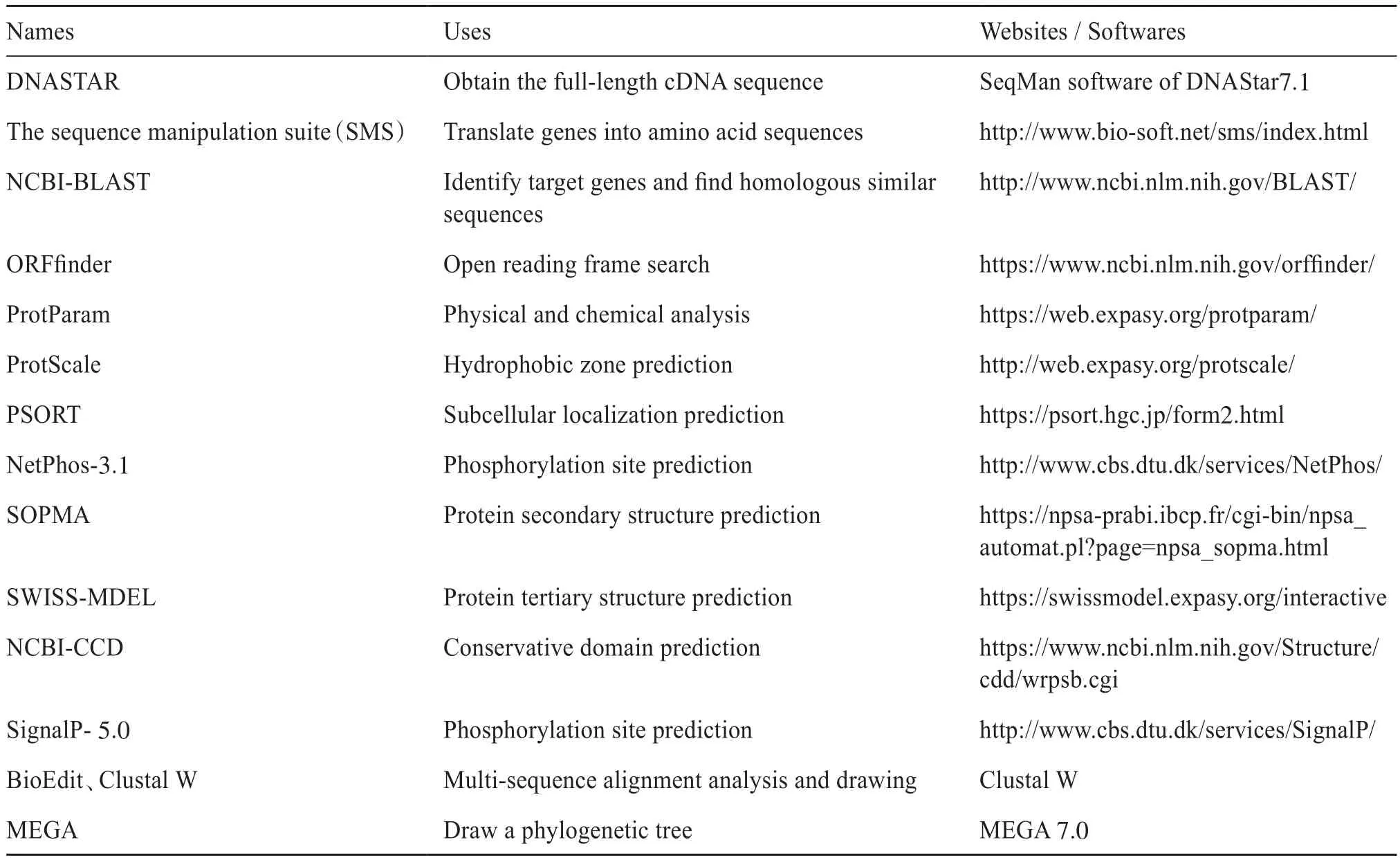
表2 生物信息学分析资源Tab. 2 Bioinformatics analysis resources
1.4 HaeClo基因在胁迫条件下的表达分析
取对数期生长的雨生红球藻作为试验样品,混匀后均分为6组,每组设3个平行,根据给予不同的胁迫处理分为以下试验组别:正常光全氮组、高白光全氮组、高蓝光全氮组、1/4氮组、高白光+1/4氮组及高蓝光+1/4氮组,分别记为CK、HLW、HLB、1/4N、HLW+1/4N及HLB+1/4N。全氮及1/4氮处理根据MCM培养基中(NH4)MO7O24·4H2O和KNO3的浓度换算配制,高白光和高蓝光光照强度为3 000 lx。在处理第0、1、2、3、4天时分别收集雨生红球藻细胞样品制备cDNA模板,然后进行荧光定量PCR分析HaeClo基因的表达。
1.5 HaeClo蛋白的原核表达和十二烷基硫酸钠聚丙烯酰胺凝胶电泳(SDS-PAGE)分析
为了研究HaeClo蛋白的特性,本研究设计引物并进行ORF的PCR扩增,获得了克隆载体,抽提质粒后将其与表达载体质粒pET-28a(+)分别进行双酶切,切胶回收后的HaeCloORF片段和pET-28a(+)载体片段连接构成重组质粒pET-28a(+)-HaeClo,将重组质粒转化到大肠杆菌感受态中诱导原核表达,以上操作步骤参考于晓娜[21]的方法。在28℃下用0.5 mmol/L的异丙基-β-D-硫代半乳糖苷(isopropyl-beta-D-thiogalactopyranoside, IPTG)诱导转化重组质粒的大肠杆菌BL21菌株,同时以相同条件处理的pET-28a(+)空载体做对照,分别在12 h和24 h取样进行十二烷基硫酸钠聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis, SDS-PAGE)检测。
2 结果与分析
2.1 总RNA的提取与HaeClo基因克隆
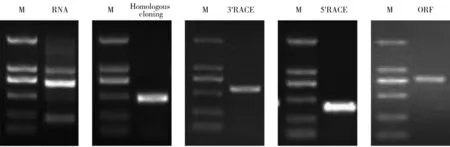
图1 雨生红球藻总RNA的提取及HaeClo基因克隆电泳图Fig. 1 Extraction of total RNA from H. pluvialis and electrophoresis of HaeClo gene cloningM :Marker DL2000。M: Marker DL2000.
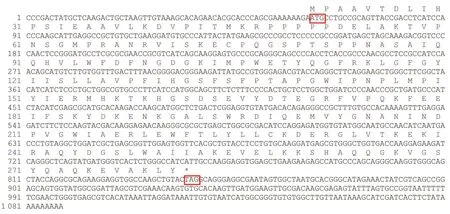
图2 雨生红球藻中HaeClo的核苷酸序列和氨基酸序列Fig. 2 Nucleotide sequences and amino acid sequences of HaeClo in H. pluvialis红色框标出的“ATG”代表起始密码子,“TAG”代表终止密码子。The “ATG” marked in the red box represents the initiation codon, and the “TAG” represents the termination codon.
如图1中泳道所示,琼脂糖凝胶电泳检测到RNA未发生明显降解,其浓度和纯度均在适宜范围内,质量较好,可进行后续试验。雨生红球藻总RNA反转录后得到cDNA模板,结合设计的引物进行PCR扩增后获得HaeClo的同源克隆片段,以制备的RACE cDNA和5'、3'端引物为模板进行PCR扩增,将获得的雨生红球藻5'端、中间片段和3'端序列拼接起来得到HaeClo基因的cDNA全长序列(NCBI注册号:MT612719)。分析表明(图2),HaeClo的cDNA序列全长为1 088 bp,共编码262个氨基酸,编码区从62~847共786 bp,其中5'-非翻译区(5'-untranslation region, 5'-UTR)和3'-非翻译区(3'-UTR)的长度分别为61 bp和241 bp,还带有一个poly(A)尾巴。通过BLAST在线对HaeClo的氨基酸序列进行同源比对分析,结果表明,其与盘藻和莱茵衣藻来源的caleosin(NCBI检索号见表3)相似性分别达到66%和61%。
2.2 HaeClo蛋白的理化性质分析
ProtParam软件分析结果表明,雨生红球藻中Hae-Clo蛋白的分子式为C1345H2067N351O378S9,理论等电点为7.73,属于碱性蛋白。编码该蛋白的氨基酸共有20种,其中含量较高的氨基酸为脯氨酸(Pro,8.8%)、赖氨酸(Lys,8.0%),而半胱氨酸(Cys,0.8%)和组氨酸(His,2.3%)的含量较低。带正电荷的氨基酸残基(Arg+Lys)与带负电荷的氨基酸残基(Asp+Glu)总数分别为31和30个。该蛋白的消光系数为53 525,吸光系数为1.815,平均亲水系数为-0.413,不稳定性指数(II)为47.61,脂肪酸系数为77.74,因此我们将该蛋白质归类为不稳定蛋白。
ProtScale软件分析结果显示:HaeClo蛋白亲水性/疏水性分析最高值为2.556,位于第105和第106位氨基酸,最低值为-2.744,位于第167位氨基酸;该蛋白在中间区域存在一个明显的疏水区,有较强的疏水性,在N端和C端氨基酸残基区域都表现出较强的亲水性。这表明HaeClo蛋白是一个两末端区域为亲水性、中间区域为疏水性的蛋白。
使用PSORT软件对HaeClo蛋白的亚细胞定位进行预测,结果显示,HaeClo蛋白定位于细胞质内,为胞内蛋白。NetPhos-3.1软件预测到HaeClo蛋白的丝氨酸(Ser)最多,有29个,在C端分布比较紧密。苏氨酸(Thr)的磷酸化位点有8个,酪氨酸(Tyr)的磷酸化位点最少,为4个。
2.3 HaeClo蛋白的高级结构分析
SOPMA软件预测HaeClo蛋白二级结构结果显示(图3a),HaeClo蛋白中无规则卷曲(图3a中紫色线条区域)所占蛋白质的比例最高(42.15%),α-螺旋(蓝色线条区域)所占蛋白质比例次之(41.38%),此外,延伸链(红色线条区域)和β-折叠(绿色线条区域)分别占10.34%和6.13%,由此可推测,HaeClo蛋白为混合型蛋白。图3b为HaeClo蛋白的三级结构预测结果。NCBI-CDD软件预测HaeClo蛋白在122~291位存在一个结构域,属于典型的caleosin家族。SignalP 5.0软件信号肽预测结果表明,编码HaeClo蛋白的氨基酸序列中没有信号肽剪切位点,由此可知,HaeClo为非分泌蛋白。
2.4 HaeClo蛋白的多序列比对分析
为了更好地研究HaeClo,本研究通过Clustal W和BioEdit软件对HaeClo进行了多序列比对分析,结果显示(图4),目标序列HaeClo(用红色标出)与从团藻(Volvocales)、小球藻(Chlorella)、拟南芥(Arabidopsis thaliana)、水稻(Oryza sativaL.)及芝麻(Sesamum indicum)等中分离的caleosin有相似的结构,参照Pasaribu等[22]的研究结果,将caleosin结构从上到下分隔标出区域,分别为N末端亲水性钙结合结构域、中央疏水性油体锚定结构域和C末端亲水性磷酸化结构域。以上相关序列信息在表3中显示。

表3 caleosin序列物种名称及其登录号Tab. 3 Caleosin sequence species name and their GenBank accession numbers

图3 雨生红球藻中HaeClo蛋白二级结构(a)和三级结构(b)的预测Fig. 3 The secondary structure (a) and tertiary structure (b) prediction of HaeClo in H. pluvialis
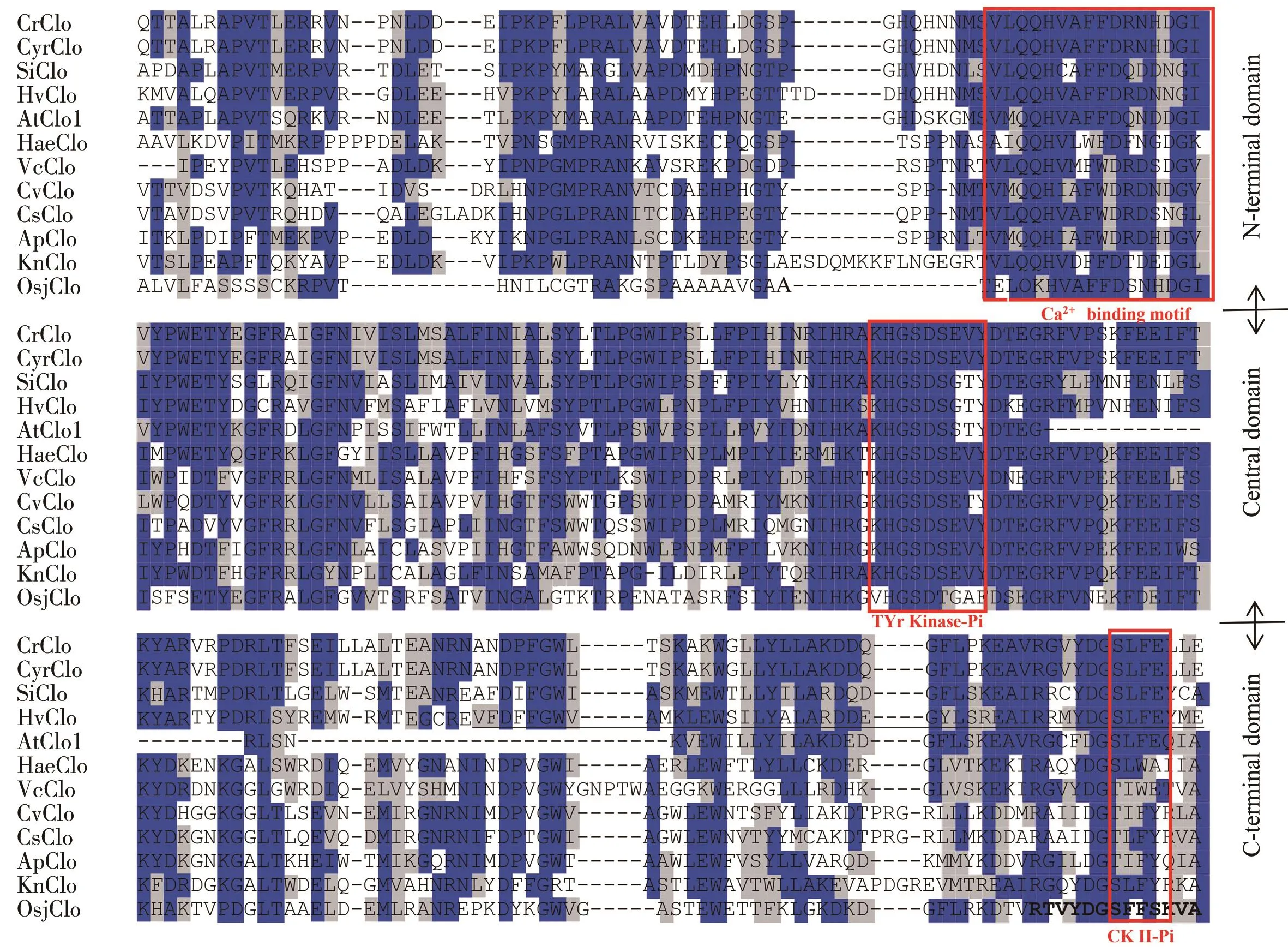
图4 雨生红球藻HaeClo与其他来源caleosin的部分序列比对Fig. 4 Partial sequence alignment of HaeClo in H. pluvialis with caleosins from other species钙结合基序、脯氨酸结基序和酪蛋白激酶II磷酸化位点的片段用红色方框标出,名称在底部标出。The fragments of calcium binding motif, proline knot motif and casein kinase II phosphorylation site are marked with red boxes and the names are marked at the bottom.
2.5 HaeClo蛋白的系统进化分析
我们从NCBI数据库搜集到20个物种的caleosin序列(包括高等植物和藻类)并进行了系统进化分析(MEGA-7软件构建进化树),以便更好地研究HaeClo与其他来源caleosin的进化关系。分析结果显示(图5),HaeClo与团藻、莱茵衣藻和盘藻等藻类来源的caleosin明显聚为一支,高等植物类caleosin另聚为一支,两支最终汇在一起。由此可见,HaeClo与其他藻类来源的caleosin亲缘关系比高等植物类的caleosin亲缘关系更近一些,追溯根源,它们可能具有共同的祖先和相似的功能。以上相关序列信息(包括物种名称、简写及其登录号)在表3中显示。
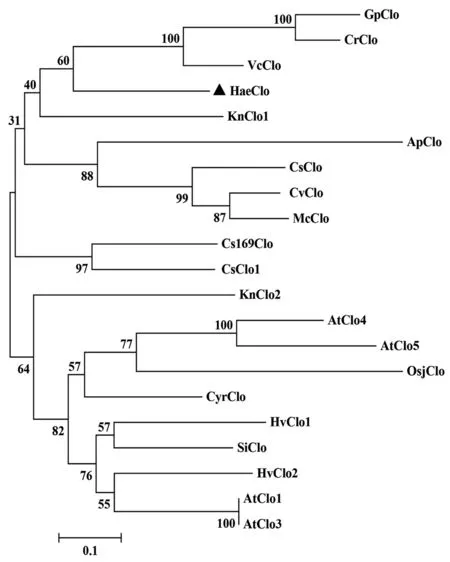
图5 雨生红球藻HaeClo的系统进化树分析Fig. 5 Phylogenetic analysis of HaeClo in H. pluviali
2.6 HaeClo基因在胁迫条件下的表达分析
本文检测了HaeClo基因在正常培养和其他不同胁迫条件下的表达谱(图6a),结果显示,在6种不同胁迫条件下,0 ~ 3 d时HaeClo基因的表达量逐渐上升,第3天时表达量均达到最高值,4 d后HaeClo基因的表达量开始下降。在处理第3天时,HLB+1/4N组HaeClo的表达量达峰值,比CK组的表达量提高了2.4倍,HLW+1/4N组HaeClo的表达量次之,HLB组HaeClo的表达量高于HLW组、1/4N组和CK组。取表达量最高的第3天进行半定量PCR分析检测(图6b),结果与图6a表达一致。
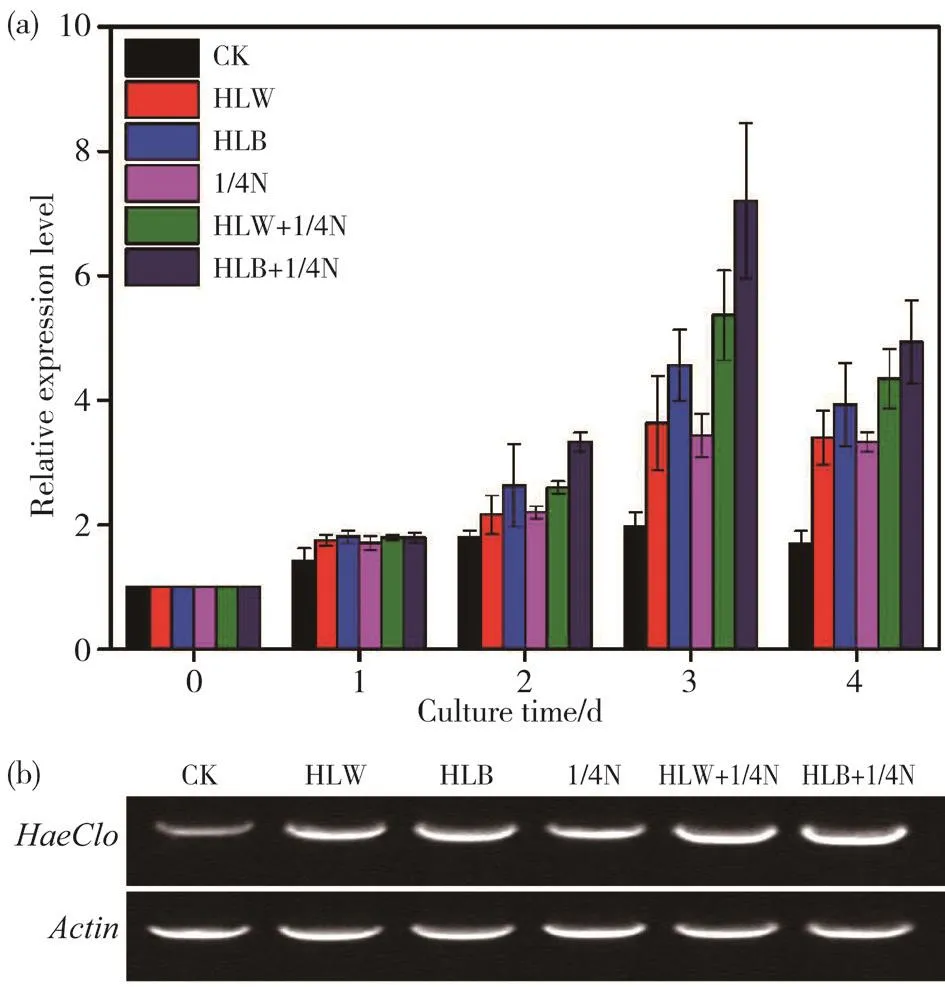
图6 雨生红球藻HaeClo基因在不同处理下的表达分析Fig. 6 Expression analysis under different treatments of HaeClo gene in H. pluviali(a)荧光定量PCR;(b)半定量PCR(a) Quantitative reverse transcription and polymerase chain reaction(qRT-PCR); (b) Semi-quantitative reverse transcription and polymerase chain reaction (sqRT-PCR)
2.7 HaeClo蛋白的原核表达和SDS-PAGE分析
由图7分析结果可以看出,与pET-28a(+)空载体相比较(泳道1、2),pET-28a(+)-HaeClo在29.5 kD的位置出现了新条带(泳道3、4),大小均与预测蛋白大小相吻合,初步证明了pET-28a(+)-HaeClo在BL21菌株中表达成功。而比较不同的IPTG诱导时间可以看出,诱导24 h的蛋白表达量较诱导12 h的蛋白表达量有明显提高。
3 讨论
TAG是由内质网上脂肪酸合成相关酶催化合成的,caleosin合成后被运送到内质网上与已经合成的TAG结合形成复合体(即油体的前体),复合体经过多次融合后脱离内质网形成油体[23]。由此可见,caleosin是油体合成必不可少的物质。本研究通过克隆获得雨生红球藻caleosin基因,并对其编码蛋白的理化特征及功能进行了分析,结果表明,HaeClo的cDNA序列全长为1 088 bp,共编码262个氨基酸,含量最高的是脯氨酸(Pro),其中5'端和3'端长度分别为61 bp和241 bp,带有一个poly(A)尾巴。HaeClo的氨基酸序列与盘藻和莱茵衣藻来源的caleosin相似性分别达到66%和61%,暗示该基因在雨生红球藻中可能编码caleosin蛋白,是油体合成必不可少的物质。

图7 雨生红球藻HaeClo原核表达和纯化SDS-PAGE分析Fig. 7 SDS-PAGE analysis of prokaryotic expression and purification of HaeClo in H. pluvialiM :蛋白质Marker;1~2 :pET-28a(+)空载诱导12 h、24 h ;3~4 :pET-28a(+)-HaeClo诱导12 h、24 h ;5 :镍柱亲和纯化后的目的蛋白。箭头代表目的蛋白。M: Protein Marker; 1~2: pET-28a(+) no-load induction for 12 h, 24 h;3~4: pET-28a(+)-HaeClo induction for 12 h, 24 h; 5: Puri fied target protein after af finity puri fication on nickel column. Arrow represents the target protein.
丁勇等[24]的研究表明,caleosin的N末端亲水性钙结合结构域含有能结合1个钙离子的EF-hand模体,这可能与钙离子调控的信号转导途径有关。中间疏水性锚定结构域中的脯氨酸-结模体与α-螺旋域相邻,一方面能在caleosin与油体及双层内质网膜(endoplasmic reticulum, ER)的靶向过程中发挥作用,另一方面可以增加caleosin蛋白的疏水性,从而增加种子油体的稳定性[25]。HaeClo属于典型的caleosin家族,其C末端结构域含有3个酪蛋白激酶II(casein kinase II, CKII)磷酸化位点,位于第142、144和163位氨基酸,可能参与钙结合和翻译后修饰(如二硫键和部分丝氨酸磷酸化),并在油体成熟及动员中传递信号,这与Purkrtova等[26]、丁勇等[24]的研究结果一致。多序列比对及系统进化分析验证了HaeClo与其他来源的caleosin具有共同的祖先和相似的功能,进一步暗示了该基因在雨生红球藻中可能编码caleosin蛋白,从而增加油体的稳定性。
caleosin的特殊结构保持了油体之间的相互独立和稳定,其中间疏水区域插入到油体内部,C末端和N末端留在油体表面,像许多树杈将油体表面紧密包裹起来,形成一道屏障来保护油体[17],从而使油体对高温、强酸、强碱等有了一定的耐受能力[27]。在干旱[28]、低温[29]、盐胁迫[30]等逆境胁迫条件下,caleosin基因上调表达,同时植物合成大量油体。也有研究表明,caleosin基因可以调控脂肪酸的积累[17]。目前caleosin基因的克隆在植物中研究较多,在藻类中研究较少,且雨生红球藻中caleosin的相关研究分析尚未见报道。本研究中,高白光、高蓝光和缺氮等胁迫均有利于雨生红球藻caleosin基因的上调表达,其中高蓝光培养的效果大于高白光和缺氮培养,多因素组合胁迫(高蓝/白光+1/4氮)效果大于单因素胁迫,这与翟映雪[31]、Katsuda等[32]研究中caleosin基因上调表达的同时植物大量合成油体的结果一致。Hae-Clo蛋白的原核表达和SDS-PAGE分析结果表明,目的蛋白在大肠杆菌中成功表达,可以进行下一步的功能研究。
本研究首次从雨生红球藻中克隆获得编码Hae-Clo的cDNA序列并进行了生物信息学分析,同时对高光和缺氮等不同胁迫条件下的雨生红球藻进行了半定量PCR和荧光定量PCR分析,进一步研究了HaeClo基因在雨生红球藻中的表达,为HaeClo基因在植物基因工程、生物技术及生物化学上的应用提供了科学依据,进而为探究雨生红球藻中HaeClo调控雨生红球藻油脂含量、虾青素含量以及虾青素在油体储存的相关机制等方面奠定了基础。

