香樟转录组基因密码子偏好性分析
梁晓静 朱昌叁 李开祥 安家成 王鹏良


摘 要: 為了解香樟基因密码子偏好性,该文以NCBI网站中香樟转录组数据为材料,利用生物信息学手段评价转录组数据质量,选取高质量数据的转录组,去除低质量序列,组装转录组,预测基因结构,再利用自编perl脚本提取以AUG开头的基因序列37 Mb序列34 931个基因,进一步利用CodonW分析基因密码子偏好性。结果表明:GC含量的变化范围为0.273~0.742,均值为0.452;ENC的范围为26.29~61.00,均值为52.76;CAI的范围为0.064~0.401,均值为0.199;RSCU值大于1的密码子数目为27个,其中以U或A结尾的有22个;中性分析表明,小部分基因在对角线上,大多数基因偏离对角线;ENC-plot分析表明小部分基因在标准曲线上,大多数基因偏离标准曲线。上述研究结果表明,香樟基因的密码子偏好性比较弱,密码子常以A/U结尾;突变和选择两者都在密码子偏好中起作用,而选择作用更大;最终确定了GUU、CAG、GAA、UCU、GCU、GGU为最优密码子,通过对目标基因密码子的校正,提高表达效率,从而为利用基因工程技术改良香樟重要性状奠定了基础。
关键词: 香樟, 转录组, 基因, 密码子, 偏好性
中图分类号: Q945.4
文献标识码: A
文章编号: 1000-3142(2021)12-2077-07
收稿日期: 2020-07-22
基金项目: 自治区主席科技资金项目(1517-06);第八批广西特聘专家专项;广西林科院基本科研业务费项目(林科201806号);广西重点实验室开放课题(19-B-04-01) [Supported by the Scientific Program of Chairman in Guangxi (1517-06); the Eighth Batch Guangxi Special Expert Program; Basic Research Fund of Guangxi Forestry Research Institute (LinKe 201806); Open Project of Guangxi Key Laboratory (19-B-04-01)]。
作者简介: 梁晓静(1983-),硕士,高级工程师,主要从事经济林栽培育种研究,(E-mail)liangxj2013@126.com。
通信作者: 王鹏良,博士,教授,硕士研究生导师,主要从事植物遗传育种研究,(Email)pengliang_wang@163.com。
Codon bias of transcriptomic genes in Cinnamomum camphora
LIANG Xiaojing1, ZHU Changsan1, LI Kaixiang1, AN Jiacheng1, WANG Pengliang2*
( 1. Guangxi Forestry Research Institute, Guangxi Key Laboratory for Cultivation and Utilization of Special Non-Timber Forest Crops, Engineering and Technology Research Center for Anise and Cinnamon of State Forestry Administration, Guangxi Engineering and Technology Research Center for Woody Spices, Nanning 530002, China; 2. Beibu Gulf University, Qinzhou 535011, Guangxi, China )
Abstract: In order to understand the genes codon bias of transcriptome in Cinnamomum camphora, all transcriptome data of C. camphora were downloaded, screened against their quality. The screened data were assembled and annotated in gene structure after low-quality reads were deleted. Then 37 Mb gene sequences initiated with AUG were extracted by perl script with 34 931 genes. The codon bias was analyzed using CodonW software. The results were as follows: GC content ranged from 0.273 to 0.742 with the average of 0.452; ENC ranged from 26.29 to 61.00 with the average of 52.76; CAI ranged from 0.064 to 0.401 with the average of 0.199; There were 27 genes whose RSCU were greater than 1, of which 22 genes ended with U or A; Neutral plot analysis indicated that a great many genes were not on the line and ENC-plot showed the similar results. The above results elucidate that the gene codon bias of transcriptome was weak, and most ended with U or A; Selection played more important role than mutation in codon bias; Finally, GUU, CAG, GAA, UCU, GCU and GGU were taken as the optimal codons. This will provide a solid foundation for improving traits of C. camphora via the codon revision of the targeted genes.
Key words: Cinnamomum camphora, transcriptome, genes, codon, bias
香樟(Cinnamomum camphora)是樟科樟属的一种常绿大乔木,为我国国家二级保护植物,广泛分布于我国南方及西南各省(中国科学院中国植物志编辑委员会,1982)。香樟的木材是良好的建筑用材,其根、枝叶、木材和种子均可提取樟脑和樟油,用途十分广泛,具有很高的应用价值。
香樟群体及个体间均存在较大遗传变异。根据樟油化学成分差异,香樟可分为本樟、芳樟和油樟(中国科学院中国植物志编辑委员会,1982);从外部表型上看,材用和油用樟树木材构造存在差异(王军锋等,2019),不同化学型油用樟树叶片解剖结构和抗旱性也存在差异(王坤等,2019),从内部遗传物质上看,等位基因也存在较大差异(刘爽,2019)。丰富的遗传变异为香樟的定向遗传改良提供了良好的资源。遗传改良通常利用杂交等传统的方法开展,然而这些方法存在周期长、成本高、盲目性大等不足。而分子育种能有效克服这些缺点成为现代育种的重要手段。
尽管香樟的分子研究起步较迟,但已有一些报道。Liu et al. (2015)利用同源克隆的方法克隆了香樟CcBBM,并进行了转化验证;Chen et al. (2018)利用转录组对香樟萜类生物合成基因开展了鉴定分析;另外,研究人员对香樟不同化学成分合作途径的关键基因CcHMGRs(郑汉等,2020a)、CcPMK(郑汉等,2020b)及NBS-LRR类抗病基因家族开展克隆和部分功能分析的工作(郑永杰等,2018);同时施雪萍(2009)和陈甘明(2014)先后建立了香樟体细胞再生体系并成功将Barnase、PaFT、CBF3和DREB1等基因转入香樟,为香樟不同成分的合成、抗逆机理探索和遗传改良奠定基础。基因工程育种是分子育种的重要方法之一,具有周期短、效率高、目标明确等优点(王关林和方宏筠,2014)。密码子指导氨基酸的合成(Krebs et al., 2018)。不同的密码子对基因的表达和功能产生较大影响(Hershberg & Petrov, 2008; Zhou et al., 2016)。不同物种密码子偏好性存在较大差异(陆育生等,2018),了解香樟基因的密码子偏好性,对利用基因工程技术培育香樟新品种具有十分重要的作用。因此,本文分析香樟转录组基因密码子偏好性,确定最优密码子,从而为利用基因工程改良香樟奠定坚实基础。
1 材料与方法
1.1 材料
从NCBI网站中下载6个SRR6436155、SRR6436156、SRR8316438、SRR6436010、SRR6436011、SRR6374671香樟转录组测序的结果。本文利用FastQC检测转录组的序列质量,筛选数据质量较高的转录组为材料,再利用Trimmomatic修剪去除低质量的序列和接头。在此基础上利用Trinity对转录组进行组装,利用TransDecoder预测基因结构。提取以AUG开头的基因用于本文的后续分析。
1.2 数据分析
1.2.1 密码子偏好参数估计 以所选的编码蛋白基因为研究对象,采用CodonW 1.4.2軟件分析密码子偏好性的参数:有效密码子数目(ENC)、密码子适应指数(CAI)、密码子偏好性指数(CBI)、最优密码子使用频率(Fop)、基因表达的蛋白质疏水性(Gravy)和芳香族氨基酸的频率(Aromo)及密码子不同位置碱基的比例(GC1、GC2和GC3分别代表第一位、第二位和第三位密码子的GC含量,GC3s表示第三位同义密码子的GC含量)。
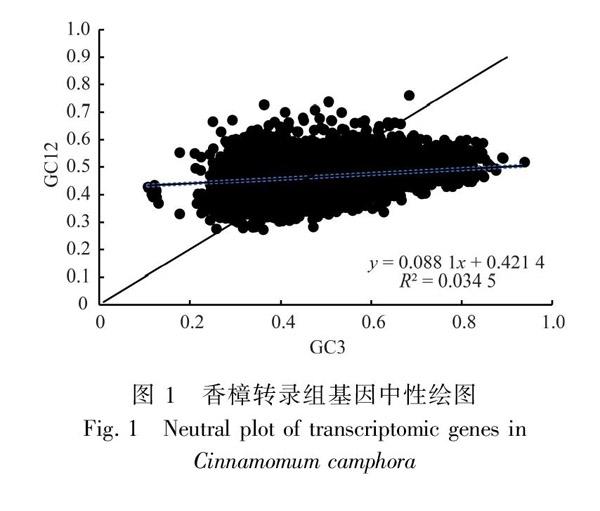
1.2.2 中性绘图分析 为了初步摸索对密码子偏好性的影响因素,中性绘图根据GC1和GC2均值记为GC12,其作为纵坐标,GC3作为横坐标,用基因散点图的方式进行作图。根据基因所在位置与对角线的关系从而判断影响基因密码子偏好性的因素。
1.2.3 ENC-plot绘图 为进一步了解造成密码子偏好的原因,本文以ENC为纵坐标,以GC3s为横坐标建立坐标系,根据基因坐落的位置作散点图。再利用方程添加其理论标准曲线ENCexp=2+GC3s+29GC3s2+(1-GC3s)2 ,并计算ENCRatio=ENCexp-ENCobsENCexp。根据散点图及ENC比值的结果推断造成密码子偏好的原因。
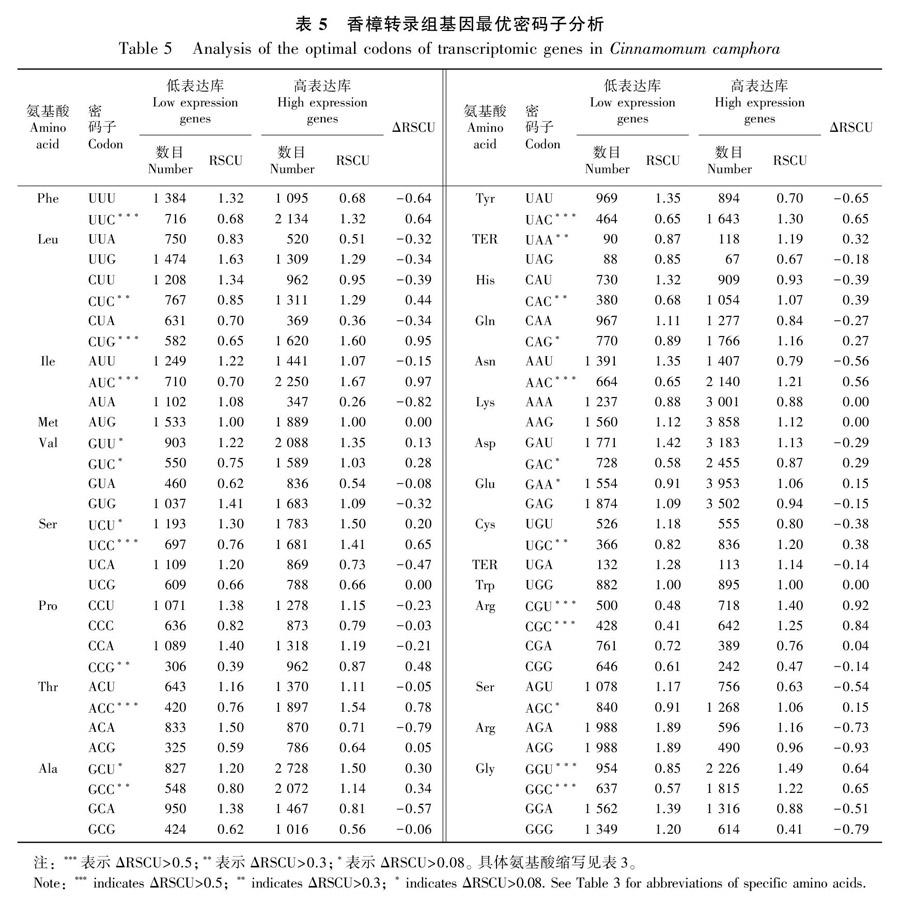
1.2.4 最佳密码子确定 为了确定最优密码子,本文以CAI密码子偏好性参数为标准对参试基因进行排序,分别从高低两端取总数的1%建立高表达库和低表达库,并以高表达库的RSCU与低表达库的对应的RSCU的差值为ΔRSCU,以ΔRSCU大于0.08且RSCU大于1的密码子为最优密码子。
2 结果与分析
2.1 香樟转录组选取
为了准确分析香樟密码子偏好性,本文从NCBI中下载了6个香樟转录组测序的结果。但由于许多转录组没有明确注明转录组的组织,或者对转录测序质量比较差的结果进行剔除,最后只剩下SRR6436155一个转录组用作本文的实验数据。该转录组数据量为9.7 G。利用Trinity组装转录组后得到305 Mb序列,利用cd-hit-est命令以默认参数进一步延伸得到229 Mb。在此基础上,利用TransDecoder将转录组序列与Swissprot和pfam-A数据库同源比对后得到54 Mb数据。利用自编perl脚本提取以AUG开头的37 Mb基因序列用于后续分析。
2.2 密码子偏好性参数
本文利用CodonW软件(https://sourceforge.net/projects/codonw/)计算所有参试基因密码子偏好性参数。由表1可知,密码子不同位置碱基的GC含量差异较大,第一位、第二位和第三位密码子GC含量的变化范围分别为0.245~0.913、0.179~0.858和0.108~0.939,均值分别为0.511、0.408和0.434;总GC含量的变化范围为0.273~0.742,均值为0.452; CAI的范围为0.064~0.401,均值为0.199;CBI的范围为-0.513~0.364,均值为-0.056;Fop的范围为0.153~0.634,均值为0.384;Gravy的范围为-2.747~1.436,均值为-0.259;其氨基酸长度变化范围86~476 5,均值为366;Aromo的变化范围为0.000~0.324,均值为0.085。ENC的范围为26.29~61.00,均值为52.76,在34 931基因中,有80个基因的ENC小于35(Jiang et al., 2008),其余基因的ENC均大于35,这说明香樟转录组中只有极少数基因偏好性较强,绝大多数基因的偏好性较弱,甚至有些基因没有密码子偏好性。
密码子偏好性参数相关性分析结果(表2)表明,GC1、GC2、GC3s与其余参数都呈极显著相关;GC3除与Gravy不相关外,与其他参数均显著相关。上述说明基因的碱基组成对密码子偏好性参数存在一定影响。基因长度(N)与基因不同位置的碱基含量极显著相关,GC1、GC2及GC3的相关系数从0.145到-0.103,说明基因越长,GC1越高,GC3越低。基因长度与CAI无显著相关;尽管基因长度与密码子偏好性参数CBI、Fop、ENC为极显著相关,但其相关系数极低,说明基因长度对密码子偏好影响不大。
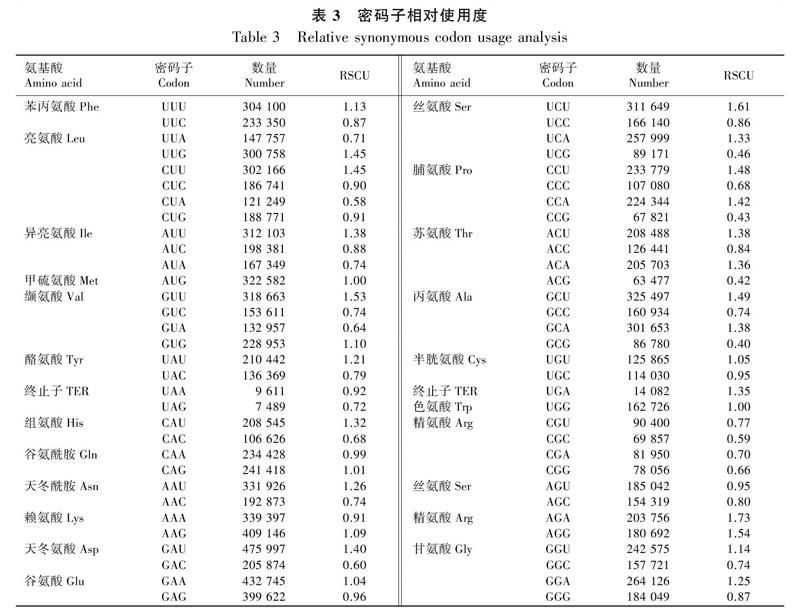
RSCU分析结果(表3)表明,RSCU值大于1的密码子数目为27个,其中以U結尾的有14个,以A结尾的有8个,以G结尾的有5个;以A或U结尾的密码子占81.48%。
2.3 中性绘图
香樟叶片转录组基因中性绘图结果(图1)表明,GC12的变化范围为0.271~0.759,GC3变化范围为0.108~0.939。图1中代表基因的点有一些落在对角线上,有更多的点偏离对角线,GC12与GC3的相关性不显著。上述结果综合表明香樟转录组基因密码子偏好性同时受到突变和选择的作用。
2.4 ENC-plot绘图
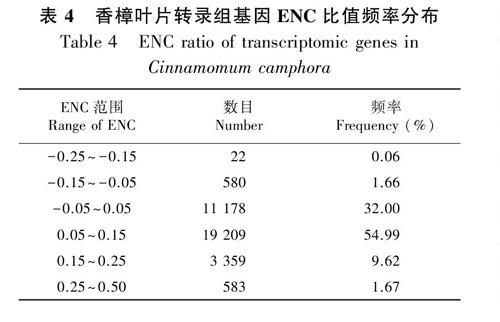
以ENC作为y轴,以GC3s作为x轴,开展ENC-plot绘图。将参试基因都布置于该坐标系,GC3s分布于0.077~0.937之间,代表基因的点有的着落在标准曲线的上方,有的着落在标准曲线的下方及另外一部分在标准曲线上(图2)。为了进一步分析基因分布的具体情况,本文分析了ENC比值的频率分布(表4),32%的基因分布于-0.05~0.05之间,54.99%的基因分布于0.05~0.15之间,1.66%分布于-0.15~-0.05之间,9.62%分布于0.15~0.25之间,1.67%分布于0.25~0.5之间。说明突变在香樟叶片转录组基因偏好性形成过程中起重要作用,而选择的作用更大。
2.5 最优密码子
按照基因密码子偏好性参数CAI数值大小进行排序,从两端分别取1%的基因分别建立高表达库和低表达库,再分别计算两个库的RSCU,进一步计算ΔRSCU。64个密码子中,有25个密码子的ΔRSCU大于0.08,其中以G结尾的有3个,以C结尾的有15个,以A结尾的有2个,以U结尾的有5个。结合表3中RSCU大于1的高频密码子与表5中标星号的高表达密码子,从而确定最优密码子。最终确定香樟叶片转录组基因中有6个最优密码子分别为GUU、CAG、GAA、UCU、GCU、GGU,其中5个密码子以A或U结尾,另外1个以G结尾。
3 讨论与结论
本文以测序质量较高转录组数据为材料研究香樟密码子偏好性。在转录组数据组装、 延伸去除冗余序列的基础上,利用TrasnsDecoder结合Swissprot数据库和pfam-A数据库的同源搜索大大提高了捕捉功能开放阅读框(opening reading framework, ORF)的敏感性和准确性,同时确实也可能存在一些操纵子和嵌合体。然而在目前没有香樟基因组的情况下,转录组基因密码子分析仍然可以提供重要参考。
64种密码子编码20种氨基酸,其中3种终止密码子,另有AUG和UGG分别只单独编码甲硫氨酸和色氨酸,其余59种密码子编码18种氨基酸(朱圣庚和徐长发,2016;Krebs et al.,2018)。18种氨基酸中每种氨基酸至少有2种密码子编码,最多的有6种密码子编码(Nelson & Cox,2017),由于不同物种或同一物种不同基因组利用不同密码子编码同一氨基酸(王鹏良等,2018,2019;赖瑞联等,2019),因此产生密码子使用的偏好性。
香樟叶绿体基因组密码子偏好性报道表明其密码子偏好性较弱(秦政等,2018),本文中密码子偏好性参数ENC均值为52.76,大于叶绿体基因组的ENC,表明香樟转录组基因密码子偏好性比叶绿体更弱,也就是说在同一密码子中选择任意密码子对翻译效率和蛋白功能影响较小。RSCU分析表明,香樟转录组基因密码子仍然偏向以A或U结尾。这与橄榄转录组密码子偏好性相似(赖瑞联等,2019)。
密码子偏好受碱基组成、选择、突变、tRNA丰度、蛋白质长度、疏水性和脂肪族氨基酸含量的影响。ENC几乎与所有的密码子参数极显著相关,相关系数均低于0.1,与GC2呈极显著相关,相关系数为-0.45;而另一密码子偏好性参数CAI与不同位置GC含量相关,与氨基酸长度相关性较小,均说明密码子偏好性与碱基组成有关,氨基酸长度相关性小。CAI与CBI及Fop的紧密相关表明基因选择高表达的密码子。中性分析和ENC-plot分析表明突变在密码子偏好形成中起作用,而选择在这个过程中起更大作用。本文筛选出6个香樟基因最优密码子分别为GUU、CAG、GAA、UCU、GCU、GGU。最优密码子的确定为利用基因工程技术改良香樟奠定了坚实基础。
目前,香樟的转录组研究、重要基因分子克隆正在迅猛开展。香樟的遗传转化体系正逐步完善。施雪萍(2009)将GFP基因转入香樟体细胞胚中,通过绿色荧光检测、PCR检测和Southern blotting检测证实了GFP基因成功导入香樟基因组中,建立了相对完善的遗传转化体系;施雪萍(2009)将Barnase和PaFT基因分别转入香樟基因组, 然而未对基因转化的表型效应进行鉴定;王长宪(2009)将沙冬青抗寒基因AmEBP1和AmGS转入香樟基因组,并通过抗寒能力测定表明,转基因植株的抗寒能力有所提高。然而施雪萍(2009)和王长宪(2009)都没有对目的基因的密码子按照物种最优密码子进行调整。Young & Purton (2016)报道了不适合的遗传密码子在一定程度上降低或阻止转化目的基因的表达。按照密码子偏好性结果对转化目的基因进行密码子的修饰能够大大促进基因表达,提高目的基因的应用效果。本研究的成果对利用基因工程技术改良香樟具有重要的指导意义。
参考文献:
CHEN CH, ZHENG YJ, ZHONG YD, et al., 2018. Transcriptome analysis and identification of genes related to terpenoid biosynthesis in Cinnamomum camphora [J]. BMC Genomics 19: 550-564.
CHEN GM, 2014. Optimization of genetic transformation system and transformation of cold resistant genes in Cinnamomum camphora L. [D]. Wuhan: Huazhong Agricultural University [陈甘明, 2014. 樟树遗传转化体系的优化及抗寒基因的转化 [D]. 武汉: 华中农业大学.]
Editorial Board Committee of Flora in China,1982. Flora Reipublicae Popularis Sinicae [M]. Beijing: Science Press. [中国科学院中国植物志编辑委员会, 1982. 中国植物志 [M]. 北京: 科学出版社.]
HERSHBERG R, PETROV DA, 2008. Selection on codon bias [J] Ann Rev Genet, 42: 287-299.
JIANG Y, DENG F, WANG HL, et al., 2008. An extensive analysis on the global codon usage pattern of baculoviruses [J] Arch Virol, 153(12): 2273-2282.
KREBS JE, GOLDSTEIN ES, KILPATRICK ST, 2018. GENE XII [M]. USA Massachusetts Burlington: Jones & Bartlett Learning.
LAI RL, FENG X, CHEN J, et al., 2019. Codon usage bias of Canarium album (Lour.) R. transcriptome and its influence factors [J]. J Nucl Agric Sci, 33(1): 31-38 [賴瑞联, 冯新, 陈瑾, 等, 2019. 橄榄转录组密码子使用偏好性及其影响因素 [J]. 核农学报, 33(1): 31-38.]
LIU QH, ZHANG CJ, CHENG YH, et al., 2016. Molecular cloning, characterization, and functional analysis of CcBBM gene from camphor tree (Cinnamomum camphora L.) [J]. Trees, 30: 1033-1043.
LIU S, 2019. Population genetic structure of Cinnamomum camphora in China [D]. Nanchang: Jiangxi Agricultural Univeristy [刘爽, 2019. 中国樟树群体遗传结构研究 [D]. 南昌: 江西农业大学.]
LU YS, PENG C, CHEN Z, et al., 2018. Analysis of codon usage bias in Clausena lansium through annotated transcriptome data [J]. Mol Plant Breed, 18(18): 5904-5913 [陆育生, 彭程, 陈喆, 等, 2018. 黄皮转录组密码子使用偏好性分析 [J]. 分子植物育种, 18(18): 5904-5913.]
NELSON DL, COX MM, 2017. Lehninger principles of biochemistry [M]. New York: W. H. Freeman and Company.
QIN Z, ZHENG YJ, GUI LJ, et al., 2018. Codon usage bias analysis of chloroplast genome of camphora tree [J]. Guihaia, 38(10): 1346-1355 [秦政, 郑永杰, 桂丽静, 等, 2018. 樟树叶绿体基因组密码子偏好性分析 [J]. 广西植物, 38(10): 1346-1355.]
SHI XP, 2009. Enhancement of somatic enbryogenesis and genetic transformation with Barnase and PaFT genes in Cinnamomum caphora L. [D]. Wuhan: Huazhong Agricultural Univeristy [施雪萍, 2009. 樟树体细胞胚再生体系的优化和转化Barnase、PaFT基因的研究 [D]. 武汉: 华中农业大学.]
WANG CX, 2009. Study on Cinnamomum camphora Presl new germplasm with cold resistance through genetic engineering [D]. Tai’an: Shandong Agricultural University [王长宪, 2009. 利用基因工程创造香樟抗寒树种新种质技术研究 [D]. 泰安: 山东农业大学.]
WANG GL, FANG HY, 2018. Plant genetic engineering [M]. Beijing: Science Press. [王关林, 方宏筠, 2014. 植物基因工程 [M]. 北京: 科学出版社.]
WANG JF, HUANG TH, AN JC, et al., 2019. A comparative study on wood structure of timber and oil-used and timber-used Cinnamomum camphora [J]. J W China For Sci, 48(6): 15-20 [王军锋, 黄腾华, 安家成, 等, 2019. 材用和油材两用人工林樟树木材构造对比研究 [J]. 西部林业科学, 48(6): 15-20.]
WANG K, AN JC, ZHU CS, et al., 2019. Relationship between leaf anatomical structure and drought resistance of different chemotypes oil-used camphor trees [J]. J S Agric, 50(11): 2525-2531. [王坤, 安家成, 朱昌叁, 等, 2019. 不同化学型油用樟树叶片解剖结构特征及其抗旱特性 [J]. 南方农业学报, 50(11): 2525-2531.]
WANG PL, WU SC, YANG LP, et al., 2019. Analysis of codon bias of chloroplast genome in Eucalyptus grandis [J]. Guihaia, 39(12): 1583-1592. [王鵬良, 吴双成, 杨利平, 等, 2019. 巨桉叶绿体基因组密码子偏好性分析 [J] . 广西植物, 39(12): 1583-1592.]
WANG PL, YANG LP, WU HY, et al., 2018. Codon bias of chloroplast genome in Camellia oleifera [J]. Guihaia, 38(2): 135-144. [王鹏良, 杨利平, 吴红英, 等, 2018. 普通油茶叶绿体基因组密码子偏好性分析 [J]. 广西植物, 38(2): 135-144.]
YOUNG R, PURTON S, 2016. Codon reassignment to facilitate genetic engineering and biocontainment in the chloroplast of Chlamydomonas reinhardtii [J]. Plant Biotechnol J, 14:1251-1260.
ZHENG H, YU MY, PU CJ, et al., 2020a. Cloning and expression analysis of 3-hydroxy-3-methylglutaryl coenzyme a reductase (CcHMGR) genes in Cinnamomum camphora (L.) Presl [J]. Acta Pharm Sin,55(1):152-159 [郑汉, 虞慕瑶, 濮春娟, 等, 2020a. 香樟3-羟基-3-甲基戊二酰辅酶A还原酶 (CcHMGRs) 基因家族的克隆及表达分析 [J]. 药学学报, 55(1): 152-159.]
ZHENG H, YU MY, PU CJ, et al., 2020b. Cloning and expression analysis of 5-phosphomevalonate kinase gene (CcPMK) in Cinnamomum camphora [J]. Chin J Chin Mat Med, 45(1): 78-84. [郑汉, 虞慕瑶, 濮春娟, 等, 2020b. 香樟甲羟戊酸-5-磷酸激酶基因 CcPMK 的克隆和表达分析 [J]. 中国中药杂志, 45(1): 78-84.]
ZHENG YJ, WU YF, LI J, et al., 2018. Identification of NBS-LRR-like disease-resistant genes in Cinnamomum camphora and clone of two CcRNL genes [J]. Biotechnol Bull, 34(2): 142-149 [郑永杰, 伍艳芳, 李江, 等, 2018. 樟树NBS-LRR类抗病基因家族分析与CcRNL基因克隆 [J]. 生物技术通报, 34(2): 142-149.]
ZHOU ZP, DANG YK, ZHOU M, et al., 2016. Codon usage is an important determinant of gene expression levels largely through its effects on transcription [J]. Proc Natl Acad Sci USA, 26: e6117-e6125.
ZHU SG, XU CF, 2016. Biochimestry [M]. 4th ed. Beijing: Science Press. [朱圣庚, 徐长发, 2016. 生物化学 [M]. 4版. 北京: 高等教育出版社.]
(责任编辑 李 莉)

