紫花苜蓿叶绿体基因组密码子偏好性分析
喻凤 韩明


摘 要: 为分析紫花苜蓿叶绿体基因组密码子偏好性的使用模式,该文以紫花苜蓿叶绿体基因组中筛选到的49条蛋白质编码序列为研究对象,利用CodonW、CUSP、CHIPS、SPSS等軟件对其密码子的使用模式和偏好性进行研究。结果表明:(1)紫花苜蓿叶绿体基因的第3位密码子的平均GC含量为26.44%,有效密码子数(ENC)在40.6~51.41之间,多数密码子的偏好性较弱。(2)相对同义密码子使用度(RSCU)分析发现,RSCU>1 的密码子数目有30个,以A、U结尾的有29个,说明了紫花苜蓿叶绿体基因组A或U出现的频率较高。(3)中性分析发现,GC3与 GC12的相关性不显著,表明密码子偏性主要受自然选择的影响;ENC-plot 分析发现一部分基因落在曲线的下方及周围,表明突变也影响了部分密码子偏性的形成。此外,有17个密码子被鉴定为紫花苜蓿叶绿体基因组的最优密码子。紫花苜蓿叶绿体基因组的密码子偏好性可能受自然选择和突变的共同作用。该研究将为紫花苜蓿叶绿体基因工程的开展和目标性状的遗传改良奠定基础。
关键词: 紫花苜蓿, 叶绿体基因组, 密码子偏好性, 最优密码子
中图分类号: Q943.2
文献标识码: A
文章编号: 1000-3142(2021)12-2069-08
收稿日期: 2020-08-11
基金项目: 贵州师范大学2017年博士科研启动项目(GZNUD [2017]16) [Supported by Doctoral Foundation of Guizhou Normal University (GZNUD [2017]16)]。
作者简介: 喻凤(1988-),博士,讲师,研究方向为植物细胞遗传学,(E-mail)yufengcaocao@163.com。
通信作者
Analysis of codon usage bias in the chloroplast genome of alfalfa (Medicago sativa)
YU Feng*, HAN Ming
( School of Life Sciences, Guizhou Normal University, Guiyang 550025, China )
Abstract: To character the codon usage patterns of chloroplast genome in alfalfa, forty-nine coding DNA sequences were analyzed using CodonW, CUSP, CHIPS and SPSS softwares. The results were as follows: (1) The average GC content at the 3rd codon position was 26.44% in the alfalfa chloroplast genome. The effective codon number (ENC) ranged from 40.6 to 51.41, indicating that codon bias was weak in the alfalfa chloroplast genome. (2) Thirty codons showed relative synonymous codon usage (RSCU) value greater than 1, including 29 codons ending with A and U, which suggested that the third codon position preferred A or U. (3) Neutrality analysis showed no significant correlation was observed between GC3 and GC12, suggesting natural selection mainly contributed to the codon usage bias. ENC-plot analysis showed that there were some genes with low ENC values lying below or near the expected curve, which indicated that mutation also affected the formation of codon usage bias. In addition, 17 codons were identified as optimal codons in alfalfa. We concluded that the codon usage bias of alfalfa is formed under effect of natural selection and mutational bias. This study will lay a good foundation for development of chloroplast genetic engineering and molecular breeding of alfalfa.
Key words: Medicago sativa, chloroplast genome, codon usage bias, optimal codons
紫花苜蓿(Medicago sativa)是一种多年生豆科牧草,由于其具有适口性好、蛋白质含量高等优点,享有“牧草之王”的美誉(杨青川和孙彦,2011)。其栽培范围广布世界各地,是一种备受关注的世界性优质牧草。李刚(2019)研究表明,以紫花苜蓿作为饲料饲养的畜禽,可以提高畜禽的生产繁殖、肉品质以及机体的免疫力。近年来,随着我国畜牧业的发展,对优质牧草的需求量也日益增加。但是,我国紫花苜蓿等牧草产业的发展相对滞后,产量不足以满足我国市场需求,仍需进口大量牧草。这使得紫花苜蓿在我国的种植面积不断增加,然而,种植过程中田间病虫害和杂草等问题却时有发生,严重时会导致大幅减产和品质下降,制约着我国苜蓿产业的发展(边佳辉等,2020)。传统的育种方法在解决这些难题时收效甚微,而基因工程育种技术的应用却成功获得了大量具有抗病虫和抗除草剂等优良品质的作物材料(杨朝国等,2010;郭三堆等,2015)。由于紫花苜蓿为四倍体植物,遗传背景复杂,与利用核基因工程相比,叶绿体基因工程则具有外源基因可以定点整合、基因拷贝数多、母系遗传等优点(程霞英,2004;周菲等,2015;Jin & Daniell, 2015)。因此,利用叶绿体基因工程引入抗病、抗虫、抗除草剂等基因到紫花苜蓿中对其进行遗传性状改良,将对畜牧业的可持续发展具有十分重要的意义。
在叶绿体基因工程育种中,有一个关键的环节就是外源基因的表达。有研究表明外源基因的表达水平会受到物种密码子偏好性的影响(Hershberg & Petrov, 2008;Plotkin & Kudla, 2011;Quax et al., 2015)。对某一种生物体的密码子偏好性使用模式进行研究,根据该生物的最优密码子来设计外源基因表达载体,可以起到优化外源基因在叶绿体基因组中表达的作用。因此,本文将分析紫花苜蓿叶绿体基因组密码子偏好性的特征及其密码子偏性的影响因素,并确定其最优密码子,为紫花苜蓿叶绿体基因工程的开展和目标性状的遗传改良奠定基础。
1 材料与方法
1.1 叶绿体基因组序列的获得
完整的紫花苜蓿叶绿体基因组下载于NCBI数据库(GenBank登录号为KU321683.1)(Tao et al., 2017),从该叶绿体基因组中共获得75条编码蛋白质的基因序列。为了更加准确分析密码子偏好性,首先剔除小于300 bp的基因编码序列,其次选择起始密码子为ATG,终止密码子为TAA、TAG、TGA的基因编码序列,最终一共获得49条序列用于后续的数据分析(杨国锋等,2015;刘慧等,2017)。
1.2 分析方法
1.2.1 相对同义密码子使用度分析 使用CodonW软件对筛选出的49条序列的相对同义密码子使用度(relative synonymous codon usage, RSCU)进行计算。当RSCU值等于1,表示该密码子使用无偏好性;RSCU值大于1,表示该密码子的使用频率比其他同义密码子高,反之亦然(Sharp & Li, 1987)。
1.2.2 中性绘图分析 为了分析有关密码子偏好性的影响因素,利用MEGA 6软件分别统计密码子3个位置的GC含量,GC1、GC2和GC3分别表示为第一位、第二位和第三位密码子的GC含量。GC3s表示同义密码子第三位的GC含量,GC12则表示第一位和第二位密码子的GC含量的平均值。中性绘图分析时,以GC3为横坐标、GC12为纵坐标进行绘图。
1.2.3 ENC-plot绘图分析 为了进一步分析碱基组成对密码子偏好性的影响,ENC-plot绘图分析以GC3s为横坐标、有效密码子数(effective number of codon,ENC)为纵坐标进行绘制散点图。绘图中的ENC标准曲线计算公式为ENC=2+GC3s+29/[GC3s2+(1-GC3s)2](Wright, 1990)。
1.2.4 奇偶偏好(parity rule 2, PR2)分析 为了评估密码子第三位碱基A与T或C与G之间的组成偏差是否对密码子偏好性产生影响,对具有4个同义密码子编码的氨基酸的各密码子第三位的碱基组成进行作图分析。计算每个基因A3/(A3+T3) 和G3/(G3+C3)后,分别以A3/(A3+T3) 和G3/(G3+C3)为纵坐标和横坐标作图,平面图中心表示A=T,同时G=C(Sueoka, 2001)。
1.2.5 最优密码子的确定 本文以ENC值为偏好性标准确定最优密码子。首先,将ENC值进行排序;然后,从ENC值最高和最低的两极各选10%的基因,分别建成高低偏性库(低ENC值对应高偏性库),并分别计算RSCU值和高低两库的ΔRSCU;最后,选取两库ΔRSCU>0.08,且RSCU>1的密码子定义为最优密码子(王鹏良等,2019)。
2 结果与分析
2.1 密码子组成分析
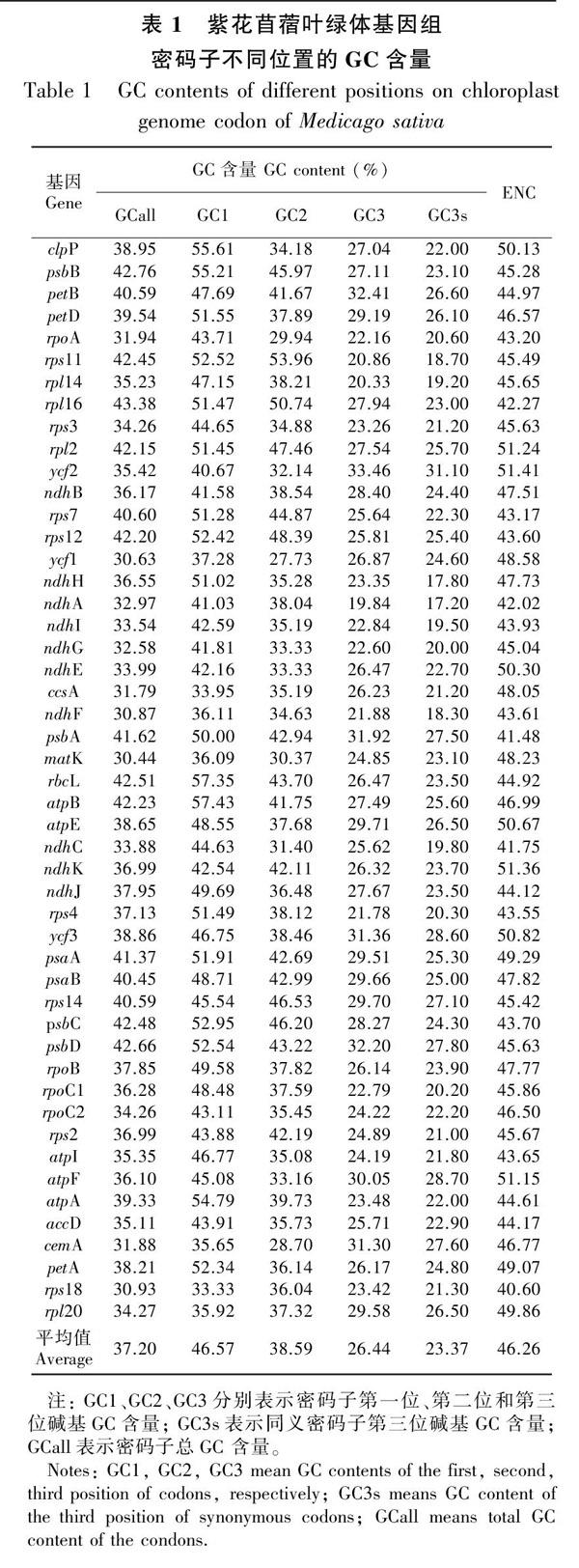
将紫花苜蓿叶绿体基因组中的49条编码序列作为一个整体进行碱基组成分析,结果表明其平均GC含量为37.20%,GC1、GC2、GC3含量分别为46.57%、38.59%和26.44%(表1)。由此可以看出密码子不同位置的碱基组成的GC分布并不均匀,其中GC1含量最高,而GC3含量最低。
有效密码子数测量的是某基因的密码子偏好程度,理论上的取值范围为21~60。当某一个基因平均地使用每一个密码子,则其有效密码子数为60;当某一个基因只使用每组同义密码子中的一个,则其有效密码子数为20。参考前人研究以ENC值为35作为偏性强弱的区分标準(Comeron & Aguadé, 1988)。本研究结果显示,紫花苜蓿叶绿体基因组ENC取值范围都在40.6以上,因此紫花苜蓿叶绿体基因密码子偏性普遍较弱(表1)。
碱基组成和有效密码子数的相关性分析结果显示:GCall和GC1、GC2、GC3的相关性都达到了极显著水平,并且第一位碱基组成和第二位碱基组成的相关性也达到了极显著水平,但两者与第三位碱基组成的相关性均未达到显著水平(表2)。表明紫花苜蓿叶绿体编码基因密码子第一位和第二位碱基组成较相似,但与第三位碱基组成却存在差异;而有效密码子数仅与GC3的相关性达到极显著水平,说明密码子第三位上的碱基组成对紫花苜蓿叶绿体编码基因的密码子偏好性影响显著。
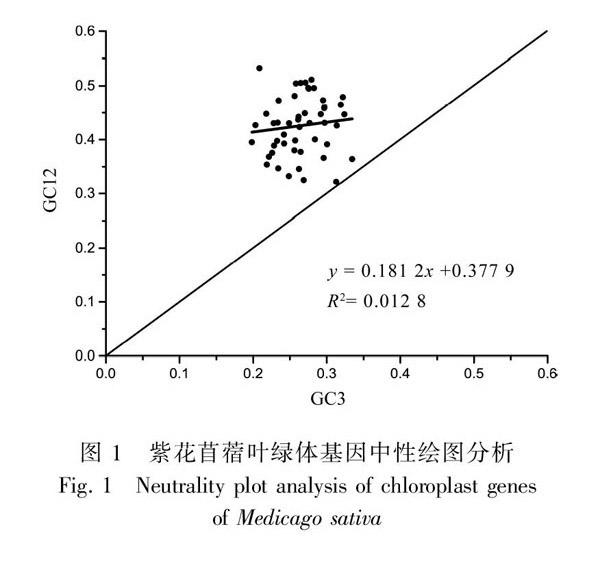
2.2 中性绘图分析
中性绘图分析结果如图1所示,由图1可知,GC12分布于0.321 8~0.532 4之间,GC3分布于0.198 4~0.334 6之间,几乎各基因的坐标点都未落在对角线上或沿着对角线分布。GC12和GC3的相关系数为0.113 3,统计结果表明两者的相关性不显著。回归曲线斜率为0.181 2,表明GC12和GC3的相关性较弱。进一步说明突变对密码子偏性形成的影响较弱,其他因素(如选择)可能对密码子的偏好性形成影响较大。
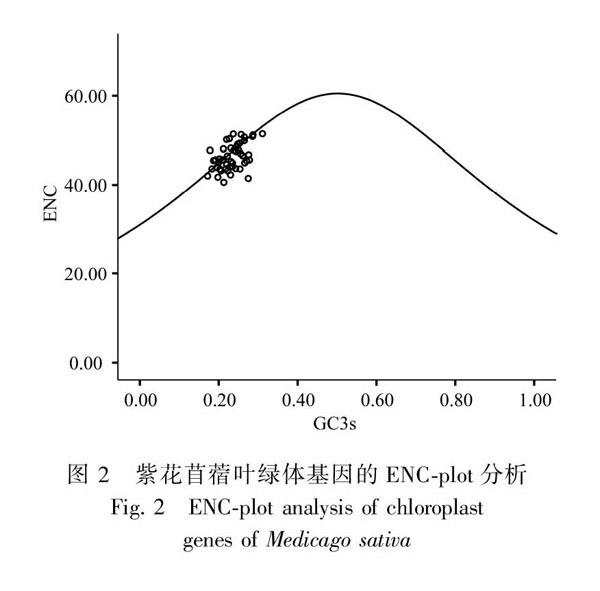
2.3 ENC-plot绘图分析
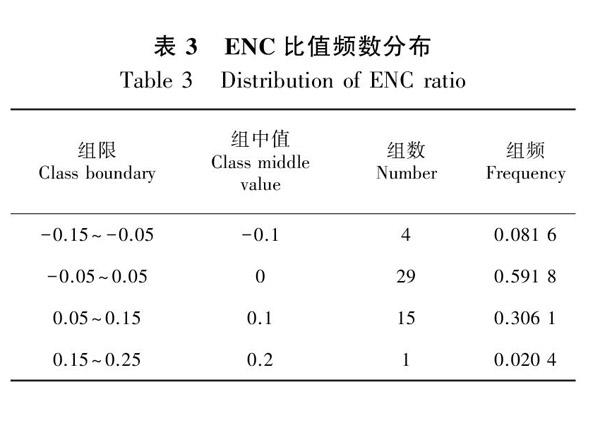
为了进一步分析碱基组成对密码子偏好性的影响, ENC-plot绘图以有效密码子数为纵坐标,GC3s为横坐标,定位所有基因的坐标系,同时添加标准曲线。结果显示大部分基因沿标准曲线分布或位于标准曲线附近(图2)。为了进一步反映差异,根据ENCexp公式求出ENC的理论值,再根据公式ENCratio=(ENCexp-ENCobs)/ENCexp求算出ENC比值。在此基础上分析所有参与研究基因的ENC频数分布(表3)。统计结果表明,大多数基因的ENC比值分布在-0.05~0.15之间,该范围内实际ENC与期望ENC值相差较小。因此,这说明突变对紫花苜蓿叶绿体基因组的部分基因密码子偏好性的形成影响较大。
2.4 PR2-plot分析
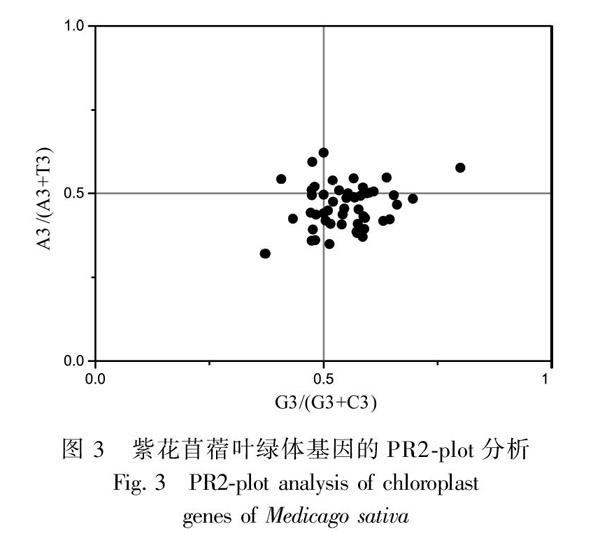
利用PR2-plot绘图分析了紫花苜蓿叶绿体基因组中部分氨基酸家族中密码子第三位碱基A与T、C与G之间的关系(图3)。结果显示,大部分基因分布在图的右下方,表明密码子第三位碱基T的使用频率高于A,G的使用频率高于C。如果密码子使用模式仅受到突变的影响,则A和T、G和C的碱基使用频率理论上应该相等。但本研究的结果显示A和T、G和C的使用存在不均衡,表明紫花苜蓿叶绿体密码子的使用模式不仅只受到突变的影响,还可能受到其他因素,如选择等的影响。
2.5 相对同义密码子使用度分析
紫花苜蓿叶绿体基因组中各氨基酸的相对同义密码子的使用度分析如表4所示,结果显示共有30个密码子的RSCU大于1。其中以U或A结尾的密码子占比为96.67%,以U结尾的为13个,以A结尾的为16个,说明紫花苜蓿偏爱使用结尾为U和A的密码子。
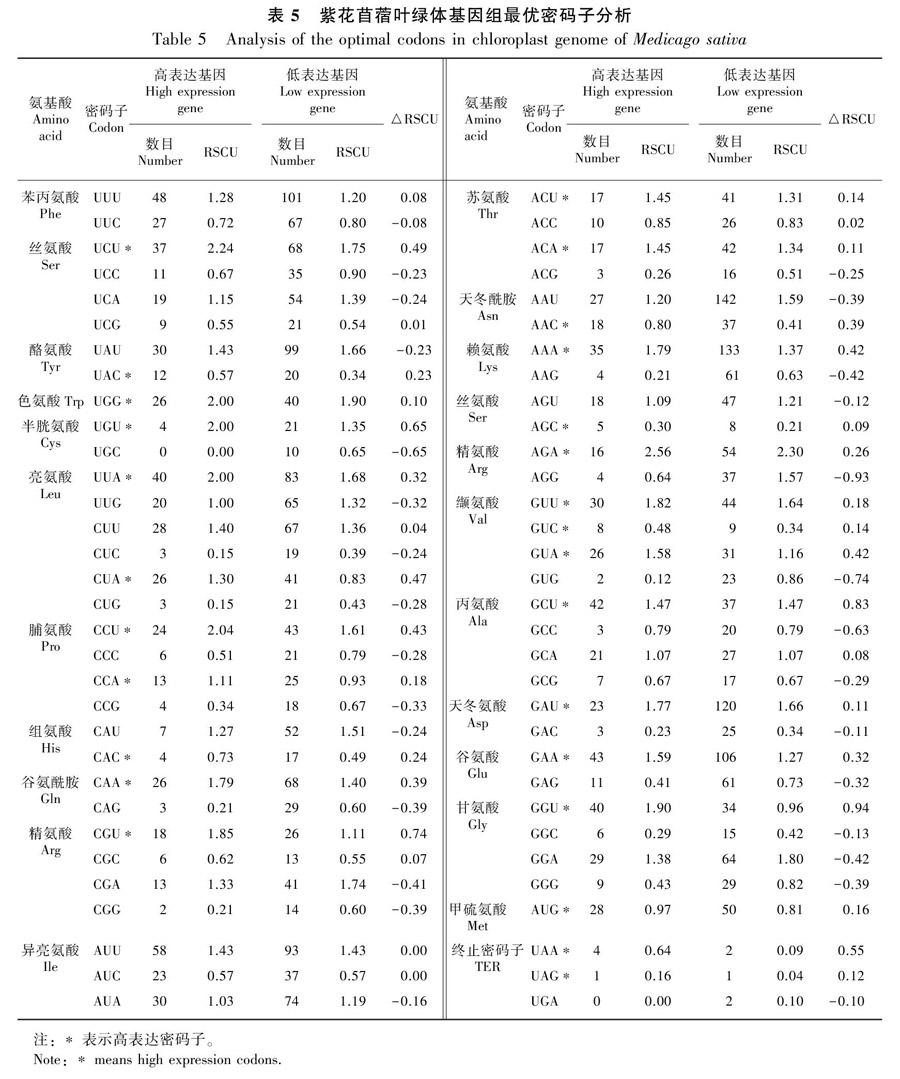
2.6 最优密码子的确定
本文根据ΔRSCU法确定最优密码子。首先将ENC值进行排序,然后从ENC值最高和最低的两极各选10%的基因,最后分别建成高低偏性库,并分别计算RSCU值和高低两库的ΔRSCU(表5)。结果发现两库ΔRSCU>0.08的一共有27个高表达密码子(表5中*标明)。选取表5中的高表达密码子和表4中的高频密码子相结合进行分析,将其共有密码子定义为最优密码子。最终确定了紫花苜蓿叶绿体基因组的最优密码子17个(UGU、UUA、CCU、CCA、CAA、CGU、ACU、ACA、AAA、AGA、GUU、GUA、GCU、GAU、GAA、GGU和UAA),在这17个最优密码子中,全以A或U结尾,此结果与GCall和GC3的高A、T含量一致。
3 讨论与结论
在长期进化过程中,特定物种为了适应自身的基因组环境,往往形成了一套具有偏好性的密码子使用模式(李平等,2011)。密码子的使用偏好性受到基因组大小、CpG岛、GC分布的位置、基因密度、基因序列长度、基因表达水平、tRNA丰度、蛋白质二级结构以及密码子变异偏好性等多种因素影响(Hanson & Coller, 2018;Paul et al., 2018)。其中,自然选择和突变是影响密码子使用偏性的两个主要因素,也是目前备受关注的两个因素(Sharp et al., 2010;Rao et al., 2011;石元豹等,2018)。由于密码子第三位碱基突变通常受到的选择压较第一位、第二位上的碱基小,因此,分析密码子第三位碱基的碱基组成对密码子的使用模式研究具有重要意义(原晓龙等,2021)。本研究表明紫花苜蓿叶绿体基因组各基因第三位密码子碱基富含A和T,与蒺藜苜蓿(Medicago truncatula)(杨国锋等,2015)、酸枣(Ziziphus jujuba var. spinosa)(胡晓艳等,2019)和蝴蝶兰(Phalaenopsis aphrodite subsp. formosana)(续晨等,2010)等植物叶绿体基因组第三位密码子使用偏性相似,表明这类植物的叶绿体基因组密码子使用模式具有高度相似性。这提示我们高等植物叶绿体基因组可能含有较高的A、T碱基含量,且密码子使用模式的偏好性在进化上存在一定的保守性。
紫花苜蓿的叶绿体基因组编码基因的ENC值都大于40.6,说明紫花苜蓿基因组存在较弱的密码子偏性。中性绘图分析结果表明紫花苜蓿叶绿体基因组密码子GC12与GC3相关性不显著,表明密码子的使用偏性可能更多受到选择的影响;而ENC-plot绘图分析表明突变是部分基因密码子偏好性形成的重要因素;PR2-plot分析的结果则进一步显示密码子的偏好性不仅受到突变的影响还受到选择的影响。因此,在长期的进化过程中,紫花苜蓿叶绿体基因组密码子的使用偏性更多地受到选择的影响,而突变也是部分基因的密码子偏性形成的重要因素。这与前人对剑麻(Agave hybrid)(金刚等,2018)、糜子(Panicum miliaceum)(刘慧等,2017)和梧桐属植物(陆奇丰等,2020)的叶绿体基因組密码子偏性影响因素分析结果相似,表明影响植物叶绿体基因组密码子偏性的因素是综合性的。但具体的影响机制还有待通过进一步的深入研究来揭示。
本研究将高表达的高频密码子定义为最优密码子,在紫花苜蓿叶绿体基因组中确定的17个最优密码子分别为UGU、UUA、CCU、CCA、CAA、CGU、ACU、ACA、AAA、AGA、GUU、GUA、GCU、
GAU、GAA、GGU和UAA。这些最优密码子都以A或U碱基结尾,这与蒺藜苜蓿(Medicago truncatula)(杨国锋等,2015)、巨桉(Eucalyptus grandis)(王鹏良等,2019)、糜子(Panicum miliaceum)(刘慧等,2017)和籽粒苋(Amaranthus hypochondriacus)(冯瑞云等,2019)等物种叶绿体基因组最优密码子分析结果一致。同时最优密码子的结果提示在紫花苜蓿的叶绿体基因工程的外源基因载体的设计时,选用这些A或U碱基结尾最优密码子或可提高外源基因的表达效率。
参考文献:
BIAN JH, LIU ZY, LI ZY, et al., 2020. Advances in alfalfa improvement using genetic engineering [J]. Pratac Sci, 37(1): 139-155. [边佳辉, 刘自扬, 李宗英, 等, 2020. 紫花苜蓿基因工程改良研究进展 [J]. 草业科学, 37(1):139-155.]
CHENG XY, 2004. Advances in the research of chloroplast genetic transformation [J]. Bull Biol, 39(3): 15-17. [程霞英, 2004. 叶绿体遗传转化的研究进展 [J]. 生物学通报, 39(3): 15-17.]
COMERON JM, MONTSERRAT A, 1998. An evaluation of measures of synonymous codon usage bias [J]. J Mol Evol, 47(3): 268-274.
FENG RY, MEI C, WANG HJ, et al., 2019. Analysis of codon usage in the chloroplast genome of grain amaranth (Amaranthus hypochondriacus L.) [J]. Chin J Grassl, 41(4): 8-15. [冯瑞云, 梅超, 王慧杰, 等, 2019. 籽粒苋叶绿体基因组密码子偏好性分析 [J]. 中国草地学报, 41(4): 8-15.]
GUO SD, WANG Y, SUN GQ, et al., 2015. Twenty years of research and application of transgenic cotton in China [J]. Sci Agric Sin, 48(17): 3372-3387. [郭三堆, 王远, 孙国清, 等, 2015. 中国转基因棉花研发应用二十年 [J]. 中国农业科学, 48(17): 3372-3387.]
HANSON G, COLLER J, 2018. Codon optimality, bias and usage in translation and mRNA decay [J]. Nat Rev Mol Cell Biol, 19(1): 20-30.
HERSHBERG R, PETROV DA, 2008. Selection on codon bias [J]. Ann Rev Genet, 42(1): 287-299.
HU XY, YU YQ, HAN YZ, et al., 2019. Codon usage bias analysis of the chloroplast genome of Ziziphus jujuba var. spinosa [J]. J For Environ, 39(6): 621-628. [胡曉艳, 许艳秋, 韩有志, 等, 2019. 酸枣叶绿体基因组密码子使用偏性分析 [J]. 森林与环境学报, 39(6): 621-628.]
JIN G, TAN X, LONG LY, et al., 2018. Characteristics of codon usage in the chloroplast protein-coding genes of Agave hybrid No. 11648 [J]. J Fujian Agric For Univ(Nat Sci Ed), 47(6): 705-710. [金刚, 谭旭, 龙凌云, 等, 2018. 剑麻叶绿体基因组编码序列密码子的使用特征 [J]. 福建农林大学学报(自然科学版), 47(6): 705-710.]
JIN S, DANIELL H, 2015. The engineered chloroplast genome just got smarter [J]. Trends in Plant Sci, 20(10): 622-640.
LI G, 2019. Research progress in Medicago sativa L. [J]. Anhui Agric Sci Bull, 25(14): 71-75. [李刚, 2019. 紫花苜蓿研究进展 [J]. 安徽农学通报, 25(14): 71-75.]
LI P, BAI YF, FENG RY, 2011. Analysis of codon bias of NAD-ME gene in Amaranthus hypochondriacus [J]. J Appl Environ Biol, 17(1): 12-17. [李平, 白云凤, 冯瑞云, 2011. 籽粒苋苹果酸酶(NAD-ME)基因密码子偏好性分析 [J]. 应用与环境生物学报, 17(1): 12-17.]
LIU H, WANG MX, YUE WJ, et al., 2017. Analysis of codon usage in the chloroplast genome of broomcorn millet (Panicum miliaceum L.) [J]. Plant Sci J, 35(3): 362-371. [刘慧, 王梦醒, 岳文杰, 等, 2017. 糜子叶绿体基因组密码子使用偏性的分析 [J]. 植物科学学报, 35(3): 362-371.]
LU QF, LUO WH, HUANG ZH, 2020.Codon usage bias of chloroplast genome from two species of Firmiana Marsili [J]. Guihaia, 40(2): 173-183. [陆奇丰, 骆文华, 黄至欢, 2020. 两种梧桐叶绿体基因组密码子使用偏性分析 [J]. 广西植物, 40(2): 173-183.]
RAO YS, WU GZ, WANG ZF, et al., 2011. Mutation bias is the driving force of codon usage in the Gallus gallus genome [J]. DNA Res, 18(6): 499-512.
PAUL P, MALAKAR AK, CHAKRABORTY S, 2018. Codon usage and amino acid usage influence genes expression level [J]. Genetica, 146(1): 53-63.
PLOTKIN JB, KUDLA G, 2011. Synonymous but not the same: the causes and consequences of codon bias [J]. Nat Rev Genet, 12(1): 32-42.
QUAX TE, CLAASSENS NJ, SOELL D, et al., 2015. Codon bias as a means to fine-tune gene expression [J]. Mol Cell, 59 (2): 149 -161.
SHARP PM, EMERY LR, ZENG K, 2010. Forces that influence the evolution of codon bias [J]. Phil Trans Roy Soc London B, 365(1544): 1203-1212.
SHARP PM, LI WH, 1987. The codon adaptation index—a measure of directional synonymous codon usage bias, and its potential applications [J]. Nucl Acid Res, 15(3): 1281-1295.
SHI YB, WANG GB, YANG XM, et al., 2018. Analysis of codon usage bias of gene factors in Ginkgo biloba WRKY family [J]. Mol Plant Breed, 17(5): 1503-1511. [石元豹, 汪贵斌, 杨晓明, 等, 2018. 银杏WRKY家族基因密碼子使用偏向性分析 [J]. 分子植物育种, 17(5): 1503-1511.]
SUEOKA N, 2001. Near homogeneity of PR2-bias fingerprints in the human genome and their implications in phylogenetic analyses [J]. J Mol Evol, 53(4-5): 469-476.
TAO XL, MA LC, ZHANG ZS, et al., 2017. Characterization of the complete chloroplast genome of alfalfa (Medicago sativa) (Leguminosae) [J]. Gene Reports, 6: 67-73.
WANG PL, WU SC, YANG LP, et al., 2019. Analysis of codon bias of chloroplast genome in Eucalyptus grandis [J]. Guihaia, 39(12): 1583-1592. [王鹏良, 吴双成, 杨利平, 等, 2019. 巨桉叶绿体基因组密码子偏好性分析 [J]. 广西植物, 39(12): 1583-1592.]
WRIGHT F, 1990. The‘effective number of codons’ used in a gene [J]. Gene, 87(1): 23-29.
XU C, BEN AL, CAI XN, 2010. Analysis of synonymous codon usage in chloroplast genome of Phalaenopsis aphrodite subsp. formosana [J]. Mol Plant Breed, 8(5): 945-950. [续晨, 贲爱玲, 蔡晓宁, 2010. 蝴蝶兰叶绿体基因组密码子使用的相关分析 [J]. 分子植物育种, 8(5): 945-950.]
YANG CG, YANG FF, ZHANG DF, 2010. Methods of screening transgenic maize with herbicide glufosinate resistance [J]. Crops, 269(6): 32-35. [杨朝国, 杨方方, 张登峰, 2010. 抗除草剂草丁膦转基因玉米后代筛选方法的研究 [J]. 作物杂志, 269(6): 32-35.]
YANG GF, SU KL, ZHAO YR, et al., 2015. Analysis of codon usage in the chloroplast genome of Medicago truncatula [J]. Acta Pratac Sin, 24(12): 171-179. [杨国锋, 苏昆龙, 赵怡然, 等, 2015. 蒺藜苜蓿叶绿体密码子偏好性分析 [J]. 草业学报, 24(12): 171-179.]
YANG QC, SUN Y, 2011. The history, current situation and development of alfalfa breeding in China [J]. Chin J Grassl, 33(6): 95-101. [杨青川, 孙彦, 2011. 中国苜蓿育种的历史、现状与发展趋势 [J]. 中国草地学报, 33(6): 95-101.]
YUAN XL, LI YQ, ZHANG JF, et al., 2021. Analysis of codon usage bias in the chloroplast genome of Dalbergia odorifera [J]. Guihaia, 41(4): 622-630. [原晓龙, 李云琴, 张劲峰, 等, 2021. 降香黄檀叶绿体基因组密码子偏好性分析 [J]. 广西植物, 41(4): 622-630.]
ZHOU F, LU SZ, GAO L, et al., 2015. Plastid genome engineering: novel optimization strategies and applications [J]. Hereditas, 37(8): 777-792. [周菲, 路史展, 高亮, 等, 2015. 植物质体基因工程:新的优化策略及应用 [J]. 遗传, 37(8): 777-792.]
(责任编辑 周翠鸣)

