DNA密码学研究进展与应用
董校生, 王 雷, 王 燕*, 薛志东**
(华中科技大学 a.软件学院; b.生命学院, 湖北 武汉 430074)
计算机技术的快速发展为人们带来便利的同时也带来了一些新的问题,人们对于信息的保护意识日益增强,信息安全的重要性显著提高.密码技术,作为信息安全的核心技术之一,于公元前5世纪便以密码器械“天书”的形式应用于战场.历经2 000多年的发展与应用,密码技术早已渗透到各个领域.数学难题的计算复杂度是传统密码学的安全性的保障,一旦攻击者具备了破解数学难题的计算能力,就能破解传统密码学的密钥.在计算资源更易获得的今天,传统密码学的信息安全性受到了严峻的挑战,密码学也开始寻求新的发展.
1994年美国加州大学的Adelman首次利用DNA计算方法解决了一种NP完全问题,实现了DNA计算[1].利用Waston-Crick互补配对原则对信息进行编码,DNA计算的目的是通过DNA链间的生化反应实现困难数学问题的求解.DNA计算首先会建立一个运算对象与DNA链的映射,并利用各种特定的生物酶生成各种数据池,再通过DNA链的可控的生化反应过程得到原始问题的全部可能解.最后,根据原始问题的约束条件设计过滤步骤(如凝胶电泳)得到可行解,并利用DNA探针等技术读取运算结果[2].
DNA计算拥有独特的数据存储和计算机制,不仅可以解决传统的困难数学问题,也可应用于密码学领域,DNA密码学也就应运而生.DNA密码学利用DNA分子本身所具有的高并行性和高存储密度等特点,借助现代分子生物技术,可实现加密、隐写、签名认证等功能[3].本文拟对基于DNA分子的加密、信息隐藏及应用的研究进展予以综述,并给出研究展望.本文涉及到的概念如图1所示.
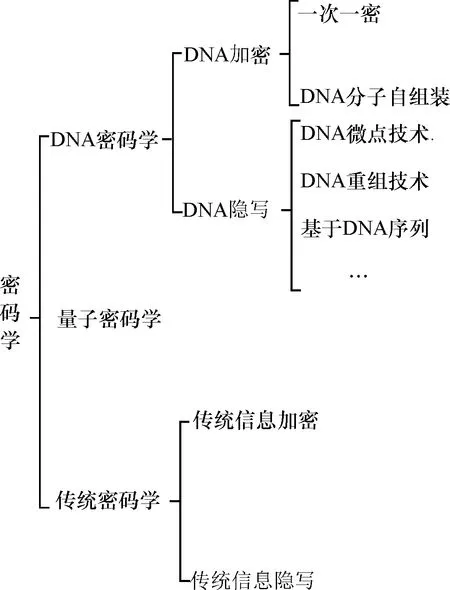
图1 概念框架图Fig.1 The conceptual framework
目前密码学可以从加密的方式分为传统密码学、量子密码学与DNA密码学;又可以从加密的目的分为信息加密和信息隐藏.基于DNA分子的信息加密可以采用“一次一密”的方式以及结合DNA分子自组装技术的方式实现,基于DNA分子的信息隐藏可以采用DNA微点、DNA重组和基于DNA序列等技术或方式实现.
1 基于DNA分子的加密方法
自1994年Adleman[1]利用DNA计算解决NP完全问题之后,由DNA计算衍生出的DNA密码学[4]无论是在理论上还是在实践中都取得了飞速的发展.1999年,Gehani首次以DNA链为载体提出了两种“一次一密”的加密方案,分别为替代法和异或法[5].2000年,Leier等[6]基于DNA二进制链设计了两种加密方案,第一种加密方案实现了快速的加密和解密,第二种方案针对图形进行设计.2003年,Chen[7]构建了一种基于碳纳米管的消息转换和基于DNA链的密码体系.2007年,Lu等[8]利用微阵列技术、DNA芯片技术以及DNA杂交技术设计了一种对称密钥DNA加密系统DNASC.2009年,崔光照等[9]进一步提出了一种采用PCR扩增技术的加密方案,该方案以PCR扩增的引物和DNA编码方式作为密钥,并对明文进行传统加密方式的预处理以防止外部对引物的攻击.2010年,Lai等[10]又在DNASC的基础上进一步提出了一种非对称DNA加密和签名系统DNA-PKC.2013年,Legoff等[11]将DNA微粒子技术与热缩片结合,构建了一种微粒子阵列加密模型.2014年,王延峰等[12]总结了基于核酸的信息安全的研究现状,并从长远的角度出发,对我国今后在该领域的发展提出了全面的建设性的意见.2015年,梁超等[13]对DNA密码学的研究现状作了综述,并分析了一些不足及提出了对DNA加密研究的展望.2020年,Grass等[14]通过读取人类基因组和DNA合成技术设计了一种DNA加密存储系统,使得有价值的信息能够安全地存储在合成DNA中,并受到个性化密钥的保护.
1.1 基于“一次一密”机制的加密方法
“一次一密”机制的安全性主要基于密钥的随机生成和不重复使用,即每次加密所使用的密钥通过随机算法生成,且每次加密所使用的密钥仅使用一次,随机密钥的长度需与明文长度一致[15].“一次一密”机制生成的密码本一般非常庞大,而DNA分子相较传统信息媒介的超高存储密度特性使得其非常适合“一次一密”机制[16].
1999年,Gehani等设计了映射替代法和DNA芯片异或法两种基于DNA分子的加密方案,实现了分子层面的“一次一密”的加密机制[5].
映射替代法首先定义一张明文——密文映射表,即“一次一密”密码本,每一个固定长度的DNA明文序列单元都对应一个DNA密文序列.“一次一密”密码本DNA序列如图2所示,其中密码本的重复单元由映射表中一个明文序列集的序列Pi、Pi在映射表中对应的密文序列Ci和作为“终止子”的聚合酶序列等三部分组成.加密时,把明文中所有字母的互补序列作为PCR扩增引物进行扩增得到后续的的互补序列,并且扩增会在终止子处终止,也就是说密文序列通过PCR扩增技术得到.
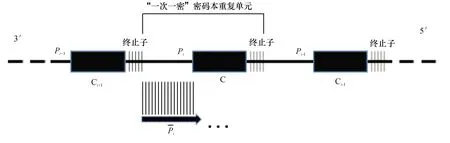
图2 “一次一密”密码本DNA序列Fig.2 DNA sequences of one-time-pad codebook
异或法的目的是从分子层面实现明文序列和密钥序列的按位异或运算,往往会利用DNA分子技术、荧光标记技术等生物分子技术进行实现.图3为一种利用DNA瓦片进行异或运算的过程,DNA瓦片是DNA计算中用于DNA分子自组装的最常用的一种DNA分子结构,它是一簇具有分支结构的DNA交叉分子,每个分子都具有粘性末端.
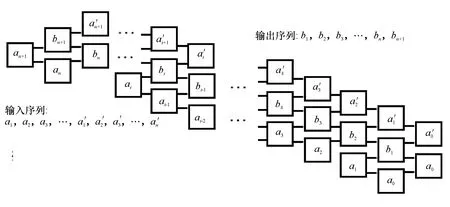
图3 利用DNA瓦片进行异或运算示意图Fig.3 XOR computation by the use of DNA tiles
2003年,Chen[7]提出了基于DNA计算的并行性的“一次一密”的加密系统,该系统利用PCR扩增技术在分子层面实现了二进制数的模2加法运算,并结合“一次一密”密码本对消息序列进行加密.DNA密码学的研究也一直与DNA计算领域的发展联系在一起,结合DNA计算中DNA分子自组装技术的DNA加密系统的相关研究也是DNA密码学领域的研究热点.
1.2 结合DNA分子自组装的增强加密方法
DNA自组装技术是DNA分子在DNA分子间非共价键的作用下,自发组装形成较复杂的有序DNA分子结构的过程[17].在DNA自组装模型计算过程中,设计满足系统特定功能的DNA分子自组装结构是系统设计中最重要的一步.
2014年,Yang等[18]提出一种基于DNA自组装技术的“一次一密”加密系统,系统流程如图4所示.该系统首先设计了一种可以从分子层面实现异或运算的DNA自组装结构,实现异或运算的操作是DNA链的替换,然后通过这个DNA自组装结构对二进制的明文序列与随机生成的密钥序列进行逐位异或运算得到二进制的密文序列,随机密钥序列长度与明文序列一致.密文序列通过安全信道发送至接收方,接收方使用与加密过程中相同的DNA自组装结构并结合随机密钥序列进行解密,解密同样通过异或运算也就是通过是DNA链的替换实现.系统设计的DNA自组装结构中利用了荧光淬灭技术,使得加密得到的密文序列实际是一组荧光信号,根据荧光信号的强度高低划分‘0’和‘1’,系统通过荧光检测的方式验证了该加密系统的可行性.
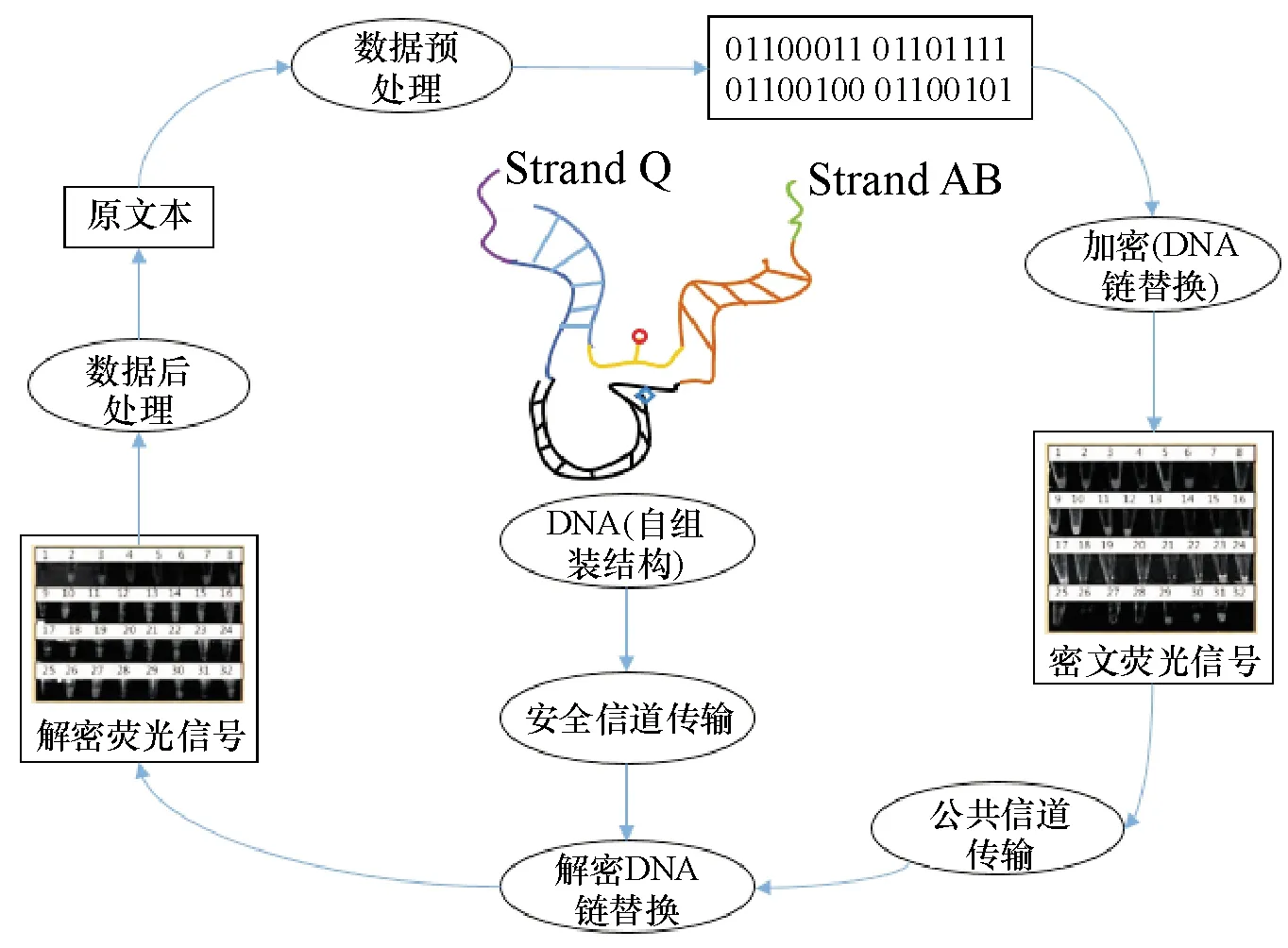
图4 “一次一密”加密系统的荧光检测流程图Fig.4 Fluorescence detection flowchart of one-time-pad encryption system
DNA折纸术是Rothemund[19]于2006年首次提出的一种全新的DNA自组装方法.DNA折纸术,顾名思义,就是将DNA链折叠形成一个预期的图案,实际是利用DNA分子的自组装特性和碱基互补配对规则,将天然DNA长链(骨架链)按照一定方式进行折叠,并用短链(钉子链)加以固定,构造出预期图案的DNA链结构[19].DNA折纸术的前期设计阶段可以通过计算机全自动组装得到若干短链,实验阶段是将长链和短链在一定条件下进行反应,二者就会自发地结合形成预先设计的图案结构.
2019年,樊春海院士的团队[20]设计了一种基于DNA折纸术的分子加密系统.在针对文本信息的加密系统中,加密过程并未涉及到DNA折纸术的具体过程,首先将文本信息按一定规则编码成3×3的点阵排列,并根据点阵排列的各斑点位置信息合成对应数量的短链,短链进行生物素化标记,生物素化后外部不可见,然后将骨架链与生物素化后的短链杂交得到最终的密文链.解密过程首先就要利用DNA折纸术得到预期形状,通过骨架链折叠方式生成所需的钉子链,然后与骨架链进行物理折叠形成与点阵排列相同的形状,其中生物素化位点的位置与加密过程中的点阵排列斑点位置一致,通过链霉亲和素识别出各图案所代表的文本信息,最终解密得到原始文本信息.
该加密系统中通过生物素——亲和素作用机制实现了信息隐写,整个加密系统是加密和隐写的结合.系统的算法复杂度主要来自于骨架链的折叠过程,系统所采用的骨架链为7 249个核苷酸的M13病毒,通过计算理论密钥长度可达702位,远超经典AES算法的256位.系统可通过设计不同的DNA折纸形状实现对不同的加密信息类型的加密.
2 基于DNA分子的信息隐藏方法
信息隐藏的过程是将秘密信息隐藏在载体信息中得到载密信息,载密信息允许在公开的信道中进行传输,其目的是保证秘密信息不被第三方所发现或读取.近年来,人们对个人隐私、知识产权信息的保护和身份信息的认证等方面有了更高的需求,信息隐藏技术也得到了人们更多的关注.基于DNA分子的信息隐藏技术也被称为DNA隐写术,与传统的以多媒体为载体的信息隐藏方法相比,DNA隐写术不仅能够隐藏秘密信息,还能对生物学领域的知识产权实施保护.对基于DNA分子的信息隐藏方法研究较多的,最早有Clelland等[2]于1999年提出的基于DNA微点技术的信息隐藏方法.2000年,Leier等[6]提出了一种基于DNA二进制链的信息隐藏方法,该方法不需要附加的映射关系编码表,无需DNA测序,解密更加迅速.2007年,崔光照等[16]以质粒为载体提出了一种基于重组技术的信息隐藏方法.2010年,Shiu等[21]提出了插入法、互补配对法和替代法这3种基于DNA序列的可逆信息隐藏方法,有效提升了算法的鲁棒性和容量.2012年,Guo等[22]通过建立一个两个秘密信息位和互补规则之间的映射关系表,有效地将两个秘密信息位替换为一个字符,在很大程度上提高了隐写方案的信息嵌入率.2013年,Mitras等[23]提出了一种在DNA序列中隐藏秘密信息的方法,使用RSA算法对秘密消息进行加密后,该秘密消息隐藏在使用互补字符已知的DNA序列中,DNA参考序列可以从不同的DNA数据库(如EBI、NCBI)中选择.2018年,Mohammed等[24]提出了一种结合神经网络的基于DNA分子的隐写方法,将所有密码子最后一个碱基的最低有效位LSB列表作为神经网络模型的输入,训练得到加密信息.
2.1 基于DNA微点技术
DNA微点技术是将包含大量DNA链的溶液喷到信纸上制成DNA微点,每个微点都包含所有的DNA链.基于DNA微点技术的信息隐藏的思路就是把明文DNA链和大量无关DNA链混合制成DNA微点进行传输,从而实现隐藏明文DNA链的目的[12].
1999年,Clelland等[2]采用DNA微点技术将一段文本信息“June 6 Invasion: Normandy”进行了DNA隐写,并成功从微点中恢复出原始文本信息.这一实验也开创了基于DNA分子的信息隐藏技术的先河.实验第一步是定义一种字符——碱基的映射关系表,例如,碱基“CGA”就对应字符“A”、“CCA”对应字符“B”,然后把明文信息根据该映射关系表转换成明文DNA序列,并在明文DNA链的尾部加上一段引物序列进行标记.第二步选取一条与明文DNA链无关的人类DNA链,利用超声波技术对其进行破碎得到大量与明文DNA链物理结构相似的冗余DNA链,将这些冗余DNA链与明文DNA链混合并喷到信纸上形成无色的DNA微点,然后将含有无色的DNA微点的信纸通过可公开的方式发送到接收方.这两步的操作成功实现了基于DNA微点技术实现信息隐写的实验目的.第三步是接收方的解密操作,首先从信件中提取出DNA混合溶液,利用第一步中的引物序列采取PCR扩增技术放大得到明文DNA链,然后利用第一步中定义的字符——碱基映射关系表对提取出的明文DNA链解码得到原始明文信息.
该信息隐藏方案的安全性基于三个方面:第一,DNA微点具有无色无味的特点;第二,明文DNA链与数以万计的物理结构相似的冗余DNA链混合在一起,若没有引物序列而直接提取明文DNA链无异于大海捞针;第三,即使攻击者成功提取出了明文DNA链,下一步要把DNA链解码成字符的形式,还需要方案所定义的字符——碱基映射关系表.
从以上分析可以发现,该信息隐藏方案的密钥是第一步中定义的字符——碱基映射关系表以及引物序列.引物序列的安全性就显得尤为重要,一旦引物序列被攻击者获悉,那么,方案中实现DNA隐写的前两步DNA分子生物操作就基本失去了意义,但论文中并未对这一点提出进一步的分析和改进.2006年,卢明欣等[25]针对论文中的这一安全性问题进行了分析,并设计出一种改进的信息隐藏方案.该方案提出了一种保密增强的算法,该算法以引物和编码方式为密钥或者用传统加密技术对要隐藏的信息预先进行加密,以防止利用关键词作为引物序列进行攻击.该论文的方案同样采用的是DNA微点技术,但以目前的DNA微点技术水平很难保证信息的安全传递,信息的传输安全性还需要结合其它方法保障,这一点也还需要进一步的研究.
2.2 基于DNA重组技术
DNA重组技术是一种人为改变生物性状表达的DNA体外操作技术,在体外将供体的基因与载体进行拼接重组,然后转入到受体体内,就可以人为使受体表达出转入基因的性状.由于DNA重组技术自身具有天然的加密和解密过程,有一些密码学学者开始利用DNA重组技术设计加密系统.
2007年,崔光照等[16]就基于DNA重组技术提出了一种新型的信息隐藏方案.首先,系统设计了明文信息(字符)——碱基的映射关系表,根据该映射关系表把明文编码成明文DNA序列后将明文DNA序列嵌入到参考DNA序列中得到假DNA序列,这一步就实现了对明文DNA序列的隐藏;然后将假DNA序列重组到作为载体的质粒中并转入到作为受体的细菌中,这一步就是DNA重组的过程;最后将受体细菌与大量无关细菌混合后发送到接收方,这一步是为了对明文序列进行进一步隐藏.该信息隐藏方案的密钥为选择性标记、生物酶以及参考序列等.该信息隐藏方案的过程及实验流程如图5所示.
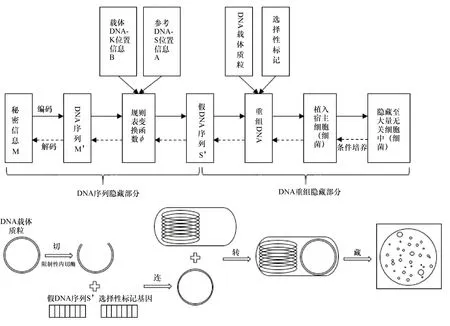
图5 基于DNA重组技术的信息隐藏方法流程及实验流程Fig.5 Information hiding method process and experimental process based on DNA recombination technology
实验结果表明,这种基于DNA重组技术的加密方法隐藏和提取明文信息的效率很高,并且该方法鲁棒性较高,抵御外部攻击的能力较强.
3 DNA密码学的应用
3.1 结合图像加密的混合加密
前文中提到的DNA密码学方法都是基于文本信息设计的加密系统,对图像信息的加密效率较低.近年来,将DNA加密的方法结合已有的图像加密算法实现更高效、安全的图像加密系统,成为图像加密领域新的研究方向.
2013年,周士华[26]以DNA序列和DNA自组装为手段设计出新型的图像加密系统,系统利用DNA瓦片构建自组装模型,实现了基于DNA自组装技术的“一次一密”图像加密系统.混沌映射是图像加密领域中生成密钥空间最为常用的一种方法.早期基于混沌的图像加密算法都是基于一维的混沌映射,但一维的混沌映射生成的密钥空间较小,算法安全强度不够高.为增强基于混沌的图像加密算法的强度,学者开始结合DNA密码学的方法进行加密系统设计.比如,2012年,Babaei[27]利用DNA计算和混沌理论设计并验证了一种在文本与图像上加密效果优于AES算法的加密算法;2015年,张健等[28]采用DNA互补编码和混沌索引相结合的方法,提出一种数字图像加密技术;2019年,胡春杰等[29]提出一种基于多混沌映射与DNA编码规则的彩色图像加密算法.2020年,Akkasaligar等[30]提出了一种采用DNA密码学和双重超混沌映射技术来为数字医学图像提供更高的安全性的图像加密系统.
可以发现,近年的相关文献[31]开始减少DNA加密方法中的生物操作,有脱离生物技术仅采用DNA编码规则设计加密方法的趋势,这种脱离了生物技术的DNA加密算法也被称为伪DNA加密[32].例如,Popli[33-34]在2018年和2019年先后提出的结合自然启发式算法如花粉算法和遗传算法的DNA加密算法,这种方式从算法复杂度上提高了加密算法的安全性,但无法从生物层面提供安全性.这种趋势的原因可能是基于DNA分子生物技术的加密方法的研究受生物技术发展限制而发展相对缓慢.
3.2 签名认证技术的新发展
由于DNA分子本身具有的高存储密度、高并行性以及物理存在形式等特点,使得DNA密码学在签名认证技术上具有传统密码学无法比拟的优势.DNA认证技术的原理是生物体DNA序列的特殊性以及较近亲缘关系个体之间DNA序列存在相似性[15].
2000年的悉尼奥运会,加拿大的DNA Technology公司将Clelland等[2]于1999年所提出的基于DNA微点技术的信息隐藏方法成功应用于商标认证中.该公司利用该方法实现了采用含有特定DNA信息的墨水对奥运会中所有的商品进行标记的目的,鉴定商标真伪只需用扫描仪识别商标中的DNA信息即可[35].这种DNA商标的制作成本相较于普通商标甚至更低,同时它被攻击者复制的难度也更高.DNA水印技术作为一种DNA加密技术与数字水印技术的集成技术[36],具有难以被定位或篡改,不易受到环境影响的特点,为签名认证技术带来了新的发展方向.2007年,Heider等[37]提出一种可以植入活体的DNA水印,并解决了DNA水印植入活体后可能发生的突变问题,从而可以通过识别DNA水印来认证用户身份或版权信息.2015年,美国纽约州立大学奥尔巴尼分校研发了一种合成DNA签名认证技术,人们可以利用该技术为自己的作品添加DNA标记,保护自己的知识版权.
3.3 数据传输、存储的应用
DNA密码学作为一种新的加密方式,在数据的安全传输和存储领域也有相应发展[38].2010年,Singh等[39]提出了一种基于DNA密码学保证移动网络中的信息安全传输的方法.2014年,Sasikumar等[40]设计了一种基于DNA加密技术和量子加密技术的数据传输加密系统,量子加密的核心是量子密钥交换,并通过实验验证了该系统为数据传输提供了更高的安全性.2018年,邹红霞等[41]设计了一种基于联盟链和DNA编码的局域网加密方法,加密结构采用基于多中心的联盟链拓扑结构,并设计了基于混沌映射和DNA编码的加密方法,提高了局域网中数据传输的保密性和完整性.DNA加密技术在近年来发展火热的云计算领域也有一定的应用[42],为云环境中的数据存储提供新的安全保障.2018年,Sukumaran等[43]提出了一种用于保护云环境中数据存储安全的基于DNA的加密算法,该算法通过使用索引和DNA隐写术技术以及二进制编码规则来实现成本效益和安全性.
4 与传统密码学、量子密码学的对比
目前密码学的三大分支为传统密码学、量子密码学以及DNA密码学[44].这三大分支的各方面的对比如表1所示.

表1 三大密码学对比表Table 1 Comparison of three major cryptography
从发展历史来看,传统密码学可以追溯到2000年前甚至更早的凯撒密码,目前所有实用密码如DES、RSA算法都视为传统密码学的范畴.量子密码学在二十世纪七十年代应运而生,其理论基础充足完善,但难以实施,目前还无法普及.DNA密码学只有二十多年的历史,作为一门生物学与密码学的交叉学科,其衍生自DNA计算,但DNA计算的研究成果并不能直接应用在DNA密码学,DNA计算目前也未形成完备的理论体系,一种DNA计算模型只适用某一类问题,不具有通用性.因此,目前DNA密码学缺乏完善的理论体系支撑,且其应用耗资较高,尚处于起步阶段.
从安全性来看,传统密码学只能实现计算安全性,也就是说,理论上具有无限计算能力的对手可以破解传统的加密算法.量子密码学基于量子不可克隆原理与海森堡不确定性原理,在量子加密通信过程中,若有攻击者想要破解密钥,他的任何攻击行为都会改变密钥,也就是说从理论上量子密码学是无法被破解的.DNA密码学的安全依据,是一些人类暂时不明白的机理,但可以使用的生物学难题的高破解难度,即DNA密码学可以同时提供生物安全性和计算安全性.
从应用方向来看,传统密码学通过有线、无线信道或光纤传输数据,通过磁性介质储存数据,在数据传输和存储上较为方便,可以实现公钥、私钥加密以及数字签名、身份认证等功能.量子密码学必须在量子通道上实现,且存储也很不方便,有较大局限性,比如无法实现公钥加密和数字签名功能,它的主要优势是在实时通信上的应用,即量子通信.由于DNA分子本身的高并行性和高存储密度,并且DNA密码学对实时性要求不高,DNA密码学在安全数据存储、身份验证、数字签名和隐写术等加密功能中具有特殊优势.
5 结论及展望
DNA密码学衍生自DNA计算,但DNA计算目前也未形成完备的理论体系,因此,目前DNA密码学同样也缺乏完善的知识、方法和理论体系支撑,且其实现基于DNA分子生物技术,其发展自然会受到DNA分子生物技术的制约,以目前的生物技术水平来看,其实现难度相较传统密码学较大,且应用耗资较高,因此,DNA密码学的研究尚处于起步阶段.
近年来DNA密码学领域的研究进展相对缓慢,基于DNA分子生物技术的加密方法研究较为少见,更多的是关于DNA密码学在图像加密、数据传输安全上应用的研究,但这些研究实际上脱离了DNA分子生物技术,仅是在研究中结合了DNA编码的方式,也就不具备生物安全性.这样的发展形势也是现代生物技术的发展速度相对信息技术的发展速度较为缓慢这一现状的体现.
但DNA密码学作为生命科学与密码学的交叉学科,本身就是对基于数学难题的传统密码学的有益补充,其所具有的生物安全性是任何传统密码学系统都不具备的.DNA分子具有超高存储密度、超低能量消耗以及超高并行运算能力等特性,其超高的开发潜力在等待着科研人员的探索.这个探索过程需要生命科学、密码学和信息科学等多领域的科研人员的精诚合作与共同努力.相信随着科研人员的进一步研究,生物技术的进一步发展,DNA密码学一定能在信息安全领域展示出自己巨大的潜力和能量.

