新型冠状病毒刺突蛋白及其受体的结构与功能
江明金,温金华,潘德城,周 健,吕燕妮,魏筱华
(南昌大学第一附属医院药学部,江西 南昌 330006)
目前,全球大流行的新型冠状病毒感染疾病(corona virus disease 2019,COVID-19),其病原体经分离鉴定为新型冠状病毒,命名为“严重急性呼吸综合征冠状病毒2(severe acute respiratory syndrome coronavirus 2,SARS-CoV-2)”[1-4]。根据世界卫生组织最新情况报告,截至2020年5月12日,全球累计报告-COVID-19确诊病例数超400万例,死亡超28万例,涉及200多个国家和地区。COVID-19疫情已成为全球各国共同面临的公共卫生安全挑战。尽管SARS-CoV-2与SARS相关冠状病毒具有遗传相似性,但其传播方式及所致疾病特点有显著差异,SARS-CoV-2的传染源、致病机制、传染性及毒性仍不清楚。
冠状病毒是具有囊膜的单股正链核糖核酸(ribonucleic acid,RNA)病毒,基因组大小为27~32 kb,是目前已知RNA病毒中基因组最大的病毒[5]。冠状病毒分为α、β、γ、δ 4个属,其中β属冠状病毒又分为A、B、C、D 4个亚群[6]。SARS-CoV-2是目前发现的第7种可感染人类的冠状病毒,与SARS-CoV同属β属冠状病毒B亚群(Tab 1)[7]。SARS-CoV-2传染性强、传播速度快,刺突蛋白(spike protein,S蛋白)的裂解活化对病毒入侵能力以及毒力起到关键作用[8-9]。血管紧张素转化酶2(angiotensin-converting enzyme 2,ACE2)已被证实是SARS-CoV-2感染宿主细胞的功能性受体[2, 8, 10-12]。尽管SARS-CoV-2 S蛋白[13]、ACE2[14]及S蛋白受体结合结构域(receptor binding domain,RBD)-ACE2复合物[14-15]的三维晶体结构逐渐被解析,但对其入侵机制仍所知甚少,处于起步阶段。本文主要针对SARS-CoV-2 S蛋白及其与ACE2相互作用的现阶段研究进展进行综述,旨在阐明SARS-CoV-2 S蛋白、ACE2及RBD-ACE2复合物的结构与功能,为进一步研究提供参考。
1 SARS-CoV-2 S蛋白的结构与功能
1.1 SARS-CoV-2 S蛋白的组成及三维结构SARS-CoV-2 S蛋白是一个I型三聚体跨膜糖蛋白,分子量约180 ku,由1273个氨基酸残基组成,包含22个N-糖基化位点[8-9]。S蛋白伸出囊膜呈棒-球状,在病毒粒子表面形成酷似“王冠”的结构,冠状病毒因此而得名。S蛋白由N端的S1(球状)和C端的S2(棒状)2个部分组成。其中S1包含2个相对独立的结构域:N-和C-端结构域(N/C-terminal domain,N/CTD)(Fig 1)[16]。RBD、受体结合基序(receptor binding motif,RBM)分别是S蛋白与宿主细胞受体直接结合的结构域及结构域中最核心的结合区段,SARS-CoV-2、SARS-CoV RBD及RBM都位于CTD中[9, 16-17]。S2包含连接区、融合肽(fusion peptide,FP)、2个保守的七肽重复区(heptad repeat region,HR)HR1和HR2、跨膜区及胞内区(Fig 1)[16]。冠状病毒主要依靠S蛋白完成与宿主细胞受体结合以及膜融合过程。S1特异性地识别和结合宿主细胞表面受体;S2将整个S蛋白固定到病毒外膜,介导病毒外膜与细胞膜的膜融合和病毒遗传物质的进入;相比S2氨基酸序列的相对保守,S1的变异性更大,即使是同一病毒的不同毒株也存在差异[16]。
Wrapp等[13]通过冷冻电镜(cryo-electron microscopy,Cryo-EM)技术,成功构建出分辨率为3.5埃的SARS-CoV-2 S蛋白胞外域三聚体融合前构象的首个三维结构图,发现SARS-CoV-2较SARS-CoV的S蛋白RBD更偏向三聚体中心部位,其3个RBD有1个向上螺旋突出使S蛋白形成易与宿主表面受体结合的空间构象(Fig 2)。Walls等[9]也构建了1个融合前稳定的S蛋白胞外域的三聚体三维结构,观察到结构域B(即CTD)的多种构象,包括1个单一打开的结构域B的三聚体和3个关闭的结构域B的封闭三聚体。与SARS-CoV、中东呼吸综合征冠状病毒(Middle East respiratory syndrome coronavirus,MERS-CoV)S蛋白结构相类似,SARS-CoV-2 S蛋白RBD在三聚体的顶点处打开,引起三聚体的多种构象变化,这些结构变化对于这3种病毒的受体结合是必需的,并导致膜融合构象变化的启动[9]。这3种病毒的RBD均可发生“铰链”样的构象运动,当其向上时,S蛋白与宿主受体结合;向下时,与受体结合的部位被隐藏,即向上结合,向下隐藏。对于其他4种感染人类的冠状病毒,目前仅检测到封闭的S蛋白三聚体。
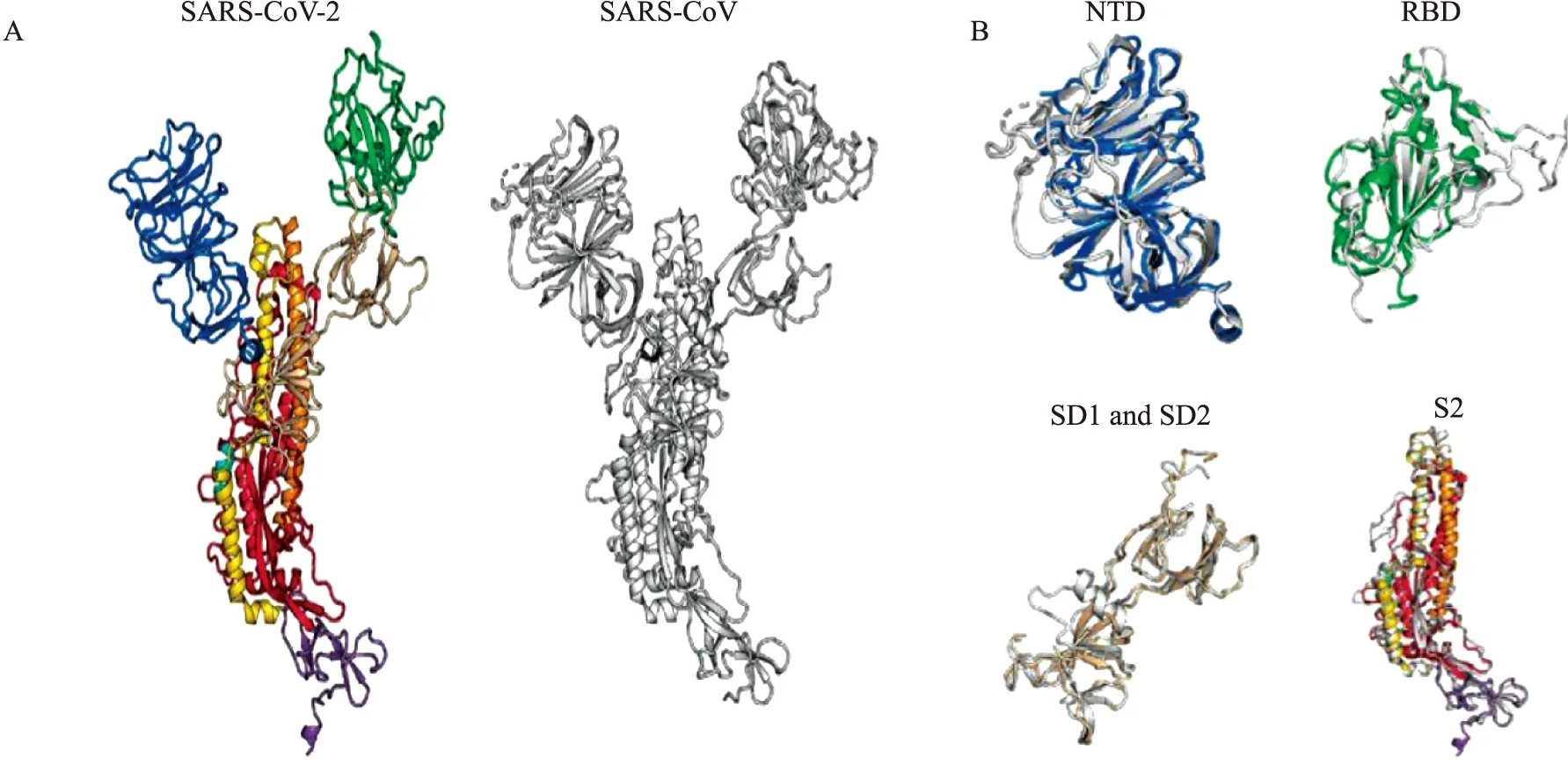
Fig 2 Structural comparison between SARS-CoV-2 and SARS-CoV S proteins[13]A: The monomers of SARS-CoV-2 and SARS-CoV S proteins are shown in ribbons; B: The following structural domains from SARS-CoV-2 S protein have been aligned to their counterparts from SARS-CoV S protein; NTD (top left), RBD (top right), SD1 and SD2 (bottom left) and S2 (bottom right). SARS-CoV-2: colour ribbons, SARS-CoV: white ribbons.
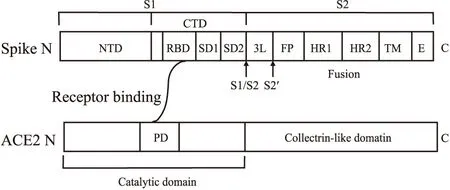
Fig 1 The schematic diagram of SARS-CoV-2 spike protein and its receptor ACE2Spike protein contains two subunits, S1 and S2; NTD: N-terminal domain; CTD: C-terminal domain; RBD: receptor binding domain; SD1: subdomain 1; SD2: subdomain 2; L: linker region; FP: fusion peptide; HR1: heptad repeat region 1; HR2: heptad repeat region 2; TM: transmembrane region; E: intracellular region; S1/S2 and S2′ represent two cleavage sites; PD: peptidase domain.
1.2 SARS-CoV-2和SARS-CoV的S蛋白结构比较整体看,SARS-CoV-2和SARS-CoV的S蛋白结构相似度很高,有超过959个对应的Cα原子的均方根偏差值为3.8埃,序列相似性87.2%(Tab 1)[13, 18]。二者RBD有174个对应的Cα原子的均方根偏差为1.2埃,序列相似性82%(Tab 1),RBM也高度相似(均方根偏差为1.3埃)[13, 15]。二者S2部分结构保守,有超过417个对应的Cα原子的均方根偏差值为1.2埃,序列相似性达88%(Tab 1)[9]。SARS-CoV-2 RBD位于333~527位氨基酸,其核心区由包含短连接螺旋和环(loop)的扭曲的4个反平行的β-折叠片层(β1、β2、β3和β6)组成;RBM位于438~506位氨基酸,其在核心区的β3-与β6-折叠之间,由短的β4-和β5-折叠、α4-和α5-螺旋以及loop组成,包含SARS-CoV-2与ACE2结合的大部分氨基酸残基[15]。SARS-CoV-2 RBD共发现9个半胱氨酸残基,其中6个形成3对二硫键:核心区2对(Cys336-Cys361、Cys379-Cys432)用来固定β-折叠结构,另1对(Cys480-Cys488)连接RBM远端的loop[15]。SARS-CoV RBM有13~14个氨基酸区域形成一个独特的loop结构,该结构通过2个半胱氨酸残基之间的二硫键稳定。与SARS-CoV比较,尽管SARS-CoV-2 RBM的loop结构的氨基酸序列有很大不同,但这2个半胱氨酸残基是保守的[19]。
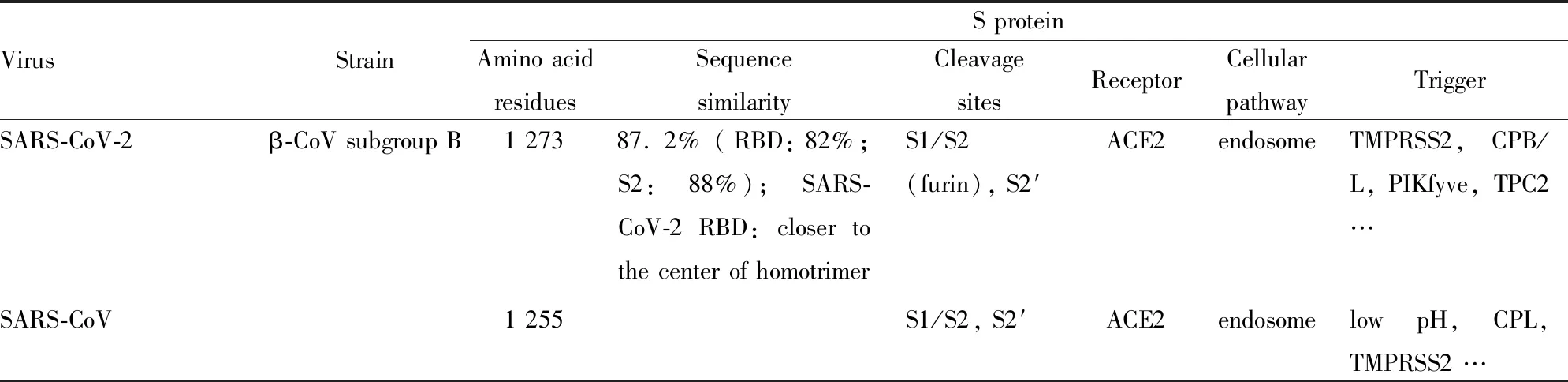
Tab 1 Comparison of structure and function of SARS-CoV-2 and SARS-CoV S proteins
1.3 SARS-CoV-2 S蛋白具有弗林蛋白酶(furin)识别位点冠状病毒含S1/S2和S2′两个酶切位点,宿主蛋白酶通过在这两个位点剪切以激活S蛋白是影响病毒宿主嗜性和致病性的重要因素[5]。多个研究发现[9, 13, 19-22],与其他SARS相关冠状病毒相比,SARS-CoV-2 S蛋白在S1和S2交界处(S1/S2位点)有1个独特的4个氨基酸残基插入(681-PRRA-684,或核苷酸位置为23619~23632),形成了1个“RRAR”多碱性furin酶切位点。这个特征在多种禽流感和人类流感病毒中常见,但在冠状病毒中少见[13]。SARS-CoV-2 S蛋白同时具备furin(R685)和胰蛋白酶(R815)酶切位点,分别对应SARS-CoV的胰蛋白酶切位点(R667和R797)[20]。酶切效率预测显示,SARS-CoV-2比SARS-CoV在S1/S2位点有更高的剪切效率[21]。相对于SARS-CoV,furin的广泛表达可能参与扩大SARS-CoV-2的宿主嗜性,增加其传播性,甚至改变其致病性。同时,蛋白三维结构预测显示,S1/S2位点4个插入的氨基酸残基在SARS-CoV-2 S蛋白表面形成一个延伸的loop,该loop结构易于被跨膜丝氨酸蛋白酶2(transmembrane protease serine 2,TMPRSS2)识别[21]。
1.4 SARS-CoV-2 S蛋白表面的糖基化修饰及功能冠状病毒S蛋白被从三聚体表面伸出的非均相N-连接聚糖密集地修饰,这些低聚糖参与S蛋白折叠,影响宿主蛋白酶的启动,还可能调节抗体识别[5]。SARS-CoV-2和SARS-CoV S蛋白分别包含22和23个N-连接糖基化位点。SARS-CoV-2 S蛋白的22个N-连接糖基化位点有20个在SARS-CoV中保守,其中包含S1部分13个中的9个及S2部分仅有的9个[9]。Shajahan等[23]通过高分辨率质谱技术对SARS-CoV-2糖基化修饰进行表征,在22个N-连接糖基化位点观察到17个N-连接聚糖。与其他SARS相关冠状病毒相比,SARS-CoV-2 S蛋白表面的furin酶切位点附近有3个独特的O-连接聚糖位点(S673、T678和S686)[24]。此外,Shajahan等[23]在SARS-CoV-2 S蛋白RBD区域内观察到2个O-连接聚糖修饰(T323和S325)。尽管O-连接聚糖结构的潜在功能尚不清楚,但其可产生1个“粘蛋白样结构域”,以保护SARS-CoV-2 S蛋白上的某些潜在表位或关键氨基酸残基[24]。
2 ACE2及RBD-ACE2复合物的结构与功能
2.1 ACE2是SARS-CoV-2感染宿主细胞的功能性受体金属肽酶ACE2是具有单一胞外催化结构域的I型跨膜糖蛋白[25]。人ACE2蛋白分子量约120 ku,由805个氨基酸残基组成,广泛分布于肾脏、肺脏、心血管及胃肠道系统,在调节心、肾功能及控制血压中起关键作用[25]。ACE2由1个N端催化结构域和1个C端Collectrin样结构域组成(Fig 1)。先前SARS-CoV已被证实通过结合ACE2感染人体细胞[26]。SARS-CoV的S蛋白三聚体通过识别ACE2催化域的疏水区位点与ACE2结合,随后细胞内化作用导致病毒颗粒内吞,继而造成SARS-CoV感染[26]。最近多个研究[2, 8, 10-12]表明,ACE2也是SARS-CoV-2感染宿主细胞的功能性受体,并且宿主蛋白酶如TMPRSS2可能参与入侵过程。Zhou等[2]使用来自人、中华菊头蝠、果子狸、猪及小鼠表达或不表达ACE2的HeLa细胞进行病毒感染研究,发现SARS-CoV-2能够利用除小鼠外所有其他物种的ACE2作为进入细胞的受体。Hoffmann等[10]使用了一种SARS-CoV-2不易感的BHK-21细胞进行研究,发现只有在ACE2表达的情况下,SARS-CoV-2才能有效入侵细胞,表明ACE2是SARS-CoV-2入侵细胞需结合的受体。进一步研究发现,TMPRSS2和内体组织蛋白酶B/L能协同激活SARS-CoV-2 S蛋白,引发病毒受体结合和膜融合,且TMPRSS2的作用更为关键[10]。
2.2 RBD-ACE2复合物的结构及功能Yan等[14]通过Cryo-EM技术解析了人ACE2全长蛋白与中性氨基酸转运载体B0AT1(ACE2-B0AT1)复合物的三维结构,发现ACE2-B0AT1以二聚体形式存在,随着ACE2肽酶结构域(peptidase domain,PD)的移位而呈现开放和关闭2种构象,PD的爪状结构提供了冠状病毒S蛋白的直接结合位点。结构建模显示,ACE2-B0AT1二聚体可同时和2个S蛋白三聚体结合,并且无论开放还是关闭状态下,S蛋白都位于ACE2-B0AT1的外侧。他们又进一步解析了RBD-ACE2-B0AT1三元复合物的三维结构,发现SARS-CoV-2 S蛋白RBD与ACE2 PD主要通过极性残基相互作用介导识别,这与SARS-CoV相似[14]。与ACE2-B0AT1不同,仅观察到RBD-ACE2-B0AT1关闭的构象。RBD一段延长的loop区域像一座桥跨越在ACE2的拱形α1螺旋上,桥的两端主要附着在α1的N-(称为右侧)和C-(称为左侧)终端,α1的中间段参与增强两极残基的相互作用。在左侧,RBD的Gln498、Thr500、Asn501与ACE2的Tyr41、Gln42、Lys353、Arg357形成氢键网络;在中间,RBD的Lys417、Tyr453分别与ACE2的Asp30、His34相互作用;在右侧,RBD的Gln474与ACE2的Gln24氢键结合,而Phe486则通过范德华力与ACE2的Met82相互作用。比较SARS-CoV-2、SARS-CoV与ACE2结合界面,发现了大量的序列变化和构象差异,其中最显著的变化是位于SARS-CoV RBD中间位置的Val404在SARS-CoV-2中被替换为Lys417,这可能改变RBD与ACE2的亲和力。Lan等[15]利用X射线衍射技术对SARS-CoV-2 RBD-ACE2复合物晶体结构进行解析,发现RBD-ACE2组成包括RBD的Cys336-Glu516位残基、PD的Ser19-Asp615位残基及63个溶剂分子。SARS-CoV-2 RBM的凹面型外表面容纳ACE2的N端螺旋结构,形成一个主要的连接界面。SARS-CoV RBD-ACE2界面共发现16个RBD残基与20个ACE2残基连接,而SARS-CoV-2 RBD-ACE2界面有18个RBD残基和20个ACE2残基连接,二者20个ACE2残基中有17个一致并且大部分位于N端螺旋结构上。二者界面的1个突出和共同的特征是亲水相互作用网络,SARS-CoV-2 RBD-ACE2界面有17个氢键和1个盐桥,SARS-CoV RBD-ACE2界面有12个氢键和2个盐桥;另1个共同特征是多个酪氨酸残基参与形成与极性羟基的氢键相互作用,这些包括来自SARS-CoV-2 RBD的Tyr449、489、495、505及来自SARS-CoV RBD的Tyr436、475、491。在二者RBM与ACE2相互作用的14个共有残基中有8个残基相同;6个不同的残基中,SARS-CoV的Tyr442、Leu472、Asn479和Thr487已被证实是参与ACE2结合的关键残基。另一研究显示,参与SARS-CoV RBD与ACE2结合的6个关键氨基酸残基(Tyr442、Leu472、Asn479、Asp480、Thr487、Tyr4911)在SARS-CoV-2中均被替换,这可能改变病毒与ACE2的结合亲和力[24]。Wrapp等[13]利用表面等离子共振技术对S蛋白胞外域与ACE2的亲和力进行分析,发现SARS-CoV-2 S蛋白与ACE2的亲和力约是SARS-CoV的10~20倍,推测这种高亲和力可能是SARS-CoV-2比SARS-CoV更容易人传人的原因之一。
3 SARS-CoV-2的入胞途径
冠状病毒入侵宿主细胞的过程主要包括受体结合、S蛋白裂解、膜融合三个步骤,它们相互协调,遵循一定的时空模式。在整个过程中,一些相关的宿主细胞蛋白酶如furin、TMPRSS-2、组织蛋白酶或胰蛋白酶等可通过不同途径对S蛋白或受体进行酶切,促进病毒的入侵。冠状病毒RBD对宿主细胞膜表面受体进行识别并与之结合后,促使细胞膜内陷,导致病毒颗粒被内吞,随后S1和S2分子间的结合力减弱,或者S蛋白被宿主蛋白酶剪切成S1和S2,S2从融合前构象转变为融合后构象,S2上的疏水FP插入细胞的内体膜,HR1和HR2反向折叠形成六螺旋束,使得病毒囊膜与内体膜相互靠拢发生膜融合,进而释放核衣壳或病毒基因组到细胞质中[27]。
SARS-CoV感染宿主细胞时,首先与细胞膜表面ACE2结合,随后通过内吞途径进入内体,内体的低pH环境和组织蛋白酶L、TMPRSS2等相关宿主蛋白酶可裂解活化S蛋白引发病毒囊膜与内体膜膜融合(Tab 1)[28-29]。此外,SARS-CoV S蛋白在胞外可被胰蛋白酶、嗜热菌蛋白酶、弹性蛋白酶等活化,促进合胞体的形成。与SARS-CoV一致,SARS-CoV-2可通过内吞途径进入细胞(Tab 1)。Ou等[8]发现SARS-CoV-2主要通过内吞作用进入稳定表达ACE2的人胚胎肾细胞HEK 293细胞,磷脂酰肌醇3-磷酸5-激酶、双孔通道2蛋白和组织蛋白酶L对于病毒进入细胞至关重要。然而,SARS-CoV-2 S蛋白可以非依赖外源性蛋白酶而触发合胞体,提示在没有外源性蛋白酶启动或激活的情况下,受体结合可触发SARS-CoV-2 S蛋白,这与SARS-CoV不同[8]。研究还发现SARS-CoV-2 S蛋白的稳定性比SARS-CoV的S蛋白低,提示SARS-CoV-2 S蛋白可通过降低其热稳定性来降低其能量屏障,这可能有助于提高SARS-CoV-2的传播效率[8]。
4 小结与展望
作为冠状病毒重要的结构蛋白,S蛋白是位于病毒囊膜表面的同源三聚体蛋白,介导受体结合和膜融合过程,是决定病毒宿主嗜性、入侵能力和毒力的关键因素以及药物研发的重要靶点。S蛋白的三维结构解析揭示,SARS-CoV-2 S蛋白三聚体存在多种不同的构象状态,其自发地采取封闭和开放的构象对于受体的结合和膜融合构象变化的启动至关重要。这与具有高致病性的SARS-CoV、MERS-CoV的S蛋白结构类似,而在其他4种可感染人类的低致病性冠状病毒中仅观察到S蛋白的封闭构象。ACE2是SARS-CoV-2感染宿主细胞的功能性受体,TMPRSS2等相关宿主蛋白酶可能参与激活S蛋白,引发病毒入侵细胞。RBD-ACE2复合物的三维结构解析揭示,SARS-CoV-2和SARS-CoV的RBD-ACE2结合界面在包埋区域、相互作用残基及亲水作用网络等方面有很大相似性,但表面静电势存在差异,6个关键氨基酸残基的突变可能导致病毒与ACE2结合亲和力的改变。SARS-CoV-2 S蛋白中独特furin酶切位点的插入以及RBD与ACE2更高的结合亲和力可能是导致其比SARS-CoV传染性更强的重要原因。尽管SARS-CoV-2可通过内吞途径进入细胞,但其具体的入侵过程和机制仍需进一步研究阐明。总之,SARS-CoV-2 S蛋白、ACE2及RBD-ACE2复合物结构的解析与功能的阐明将为抗SARS-CoV-2的入侵机制和抗病毒药物的设计提供有力的线索。
(致谢:本文在南昌大学第一附属医院临床药理研究室完成,感谢全体实验室人员对文章相关工作的指导和建议!)

