中国玉米骨干亲本黄早四杂种优势形成的遗传基础解析
李永祥,李春辉,杨俊品,杨华,程伟东,汪黎明,李凤艳,李会勇,王延波,李淑华,扈光辉0,刘成,黎裕,王天宇
(1中国农业科学院作物科学研究所,北京 100081;2四川省农业科学院作物研究所,成都 610066;3重庆市农业科学院玉米研究所,重庆 401329;4广西农业科学院玉米研究所,南宁 530007;5山东省农业科学院玉米研究所,济南 250100;6西北农林科技大学农学院,陕西杨凌 712100;7河南省农业科学院粮食作物研究所,郑州 450002;8辽宁省农业科学院玉米研究所,沈阳 110161;9吉林省农业科学院玉米研究所,吉林公主岭 136100;10黑龙江省农业科学院玉米研究所,哈尔滨 150086;11新疆农业科学院粮食作物研究所,乌鲁木齐 830000)
0 引言
【研究意义】杂种优势是指双亲杂交产生的后代在一种或多种性状上表现优于双亲的现象。玉米是利用杂种优势最为有效的作物之一,基于杂种优势群进行群内改良、群间组配是实现玉米高产的重要育种途径[1-2]。在中国玉米长期育种实践中,以骨干亲本黄早四为代表的黄改系杂种优势群的形成和广泛利用,极大地推动了中国玉米生产的发展。因此,开展黄早四杂种优势形成的遗传基础研究,对指导玉米骨干亲本高效利用和高产育种具有重要理论研究与生产利用价值。【前人研究进展】玉米育种的历史可追溯到 19世纪中后期,该时期主要通过混合选择的方法进行品种改良。在大量的育种实践中,人们发现,玉米品种间杂交种的产量比亲本品种具有明显的增产作用。SHULL[3]在 1908年首次提出了选配自交系间杂交组合的思路和方法,随后杂种优势的利用在玉米品种选育中逐步成为主流途径。例如,1934年美国玉米种植面积中杂交种仅占0.4%,但到1956年在全美已普及玉米杂交种。在中国,玉米杂种优势的利用开始于20世纪30年代,新中国成立后,得到迅速发展,并在育种实践中,逐步形成了中国的玉米杂种优势类群和杂种优势利用模式[1-2]。在玉米杂交育种的前期,国内地方品种是自交系培育的重要种质资源。其中,以地方品种四平头选育出自交系塘四平头,之后中国玉米育种家以塘四平头天然杂株(或以塘四平头为母本的杂种分离后代)培育出高配合力自交系黄早四[4]。以黄早四为基础材料,又进一步选育出444、吉853、K12、Lx9801、昌7-2等一批优良衍生自交系,利用这些衍生系组配出近百个推广面积在4 000万hm2以上的优良玉米杂交品种[5]。在这个过程中,黄早四以其配合力高、株型紧凑、花粉量大、灌浆速率快、广适性强等优点被广泛利用,成为中国玉米育种的骨干亲本,并以该自交系为代表,形成了黄改系杂种优势群,该杂种优势群种质与改良瑞德等外来种质具有很强的杂种优势,是中国玉米品种选育中的主要杂种优势利用模式之一。近年来,随着分子生物学研究手段的发展,利用分子标记对骨干亲本黄早四形成的遗传基础已经开展了一些工作。如借助MaizeSNP50高密度基因型芯片,对 42个黄早四优良衍生系的遗传构成进行分析,在全基因组范围内鉴定到15个来自黄早四的保守染色体区段(被60%以上的衍生系所保留),认为这些染色体区段可能对黄改系杂种优势群的形成具有重要作用[6]。其中,位于第4染色体的Bin4.05区段,可被84%的黄早四衍生系所保留,在该染色体区段还可同时定位到百粒重、灌浆速率等主效 QTL位点[7-9]。同时,随着黄早四参考基因组的公布,更多的黄改系类群保守区段得到了检测[10]。为全面解析玉米骨干亲本黄早四形成的遗传基础,还以黄早四为共同亲本,用 11个来自不同杂种优势群的代表性自交系为其他亲本,构建了一个包含近 2 000个重组自交系(recombination inbred lines,RILs)的巢式关联分析群体(nested association mapping population,NAM)。利用该巢式关联分析群体(以下简称为“中国玉米NAM群体”或“NAM群体”),先后开展了玉米产量[7]、雄穗[11]、株型[12]、花期[13]及抗旱性[14]等重要性状的遗传解析,定位到一批产量及重要农艺性状的主效位点,为基于分子标记的玉米骨干亲本黄早四高效利用和改良提供了大量的遗传定位信息。【本研究切入点】迄今为止,在杂种优势利用方面,关于玉米骨干亲本黄早四形成的遗传基础研究还鲜有报道,因此,有必要利用测交群体,在杂交组合状态下,评估骨干亲本黄早四对产量及重要农艺性状的贡献,解析其杂种优势形成的遗传基础。【拟解决的关键问题】本研究以前期工作中构建的基于黄早四背景的NAM群体为试验材料,同时与改良瑞德×黄改系杂种优势利用模式的代表性自交系昌7-2和郑58进行测交,获得该NAM 群体的双重测交群体,并在全国不同玉米主产区开展多环境表型鉴定。在此基础上,进行杂种优势利用下的产量及重要农艺性状遗传位点定位和遗传效应评估,解析黄早四杂种优势形成的遗传基础,以期为中国玉米骨干亲本高效利用和高产育种提供基础信息和重要依据。
1 材料与方法
1.1 试验材料与试验设计
在前期工作中,以中国玉米黄改系杂种优势群骨干亲本黄早四为共同亲本,以11个来自不同杂种优势群的代表性自交系(K12、掖 478、郑 58、获白、齐319、威风322、旅28、黄野四3、多229、Pa405和Mo17)为其他亲本,构建了由11个RIL群体(包含1 972个RILs)组成的中国玉米NAM群体[11]。为了开展基于杂种优势利用的玉米骨干亲本黄早四遗传基础研究,该NAM群体所包含的家系分别与改良瑞德×黄改系杂种优势利用模式的代表性自交系郑 58和昌7-2作为测验种进行杂交测配,获得了该NAM群体的郑 58测交群体(ZH58-TCs)测交群体和昌 7-2(CH7-TCs)。
测交群体的表型鉴定于2013年在中国4个玉米主产区的10个试验点同时展开,包括西南玉米区(广西来宾、重庆潼南和四川成都)、黄淮海夏播区(河南原阳、山东齐河和陕西杨凌)、东北春播区(辽宁沈阳、吉林公主岭和黑龙江哈尔滨)、西北春播区(新疆乌鲁木齐)。在不同环境下对11个RIL测交群体分别进行随机排布,采取2行小区种植,小区行长3 m,行距0.6 m,密度60 000株/hm2。测交群体的鉴定涉及10个环境,可用多个环境间的最优无偏估计(best linear unbiased prediction,BLUP)对表型进行准确估计,因此,试验点内不设重复[15]。田间试验过程中田间施肥、灌溉、防虫与除草等与当地大田生产管理一致。
1.2 表型鉴定与数据采集
记载小区50%植株吐丝的日期,并根据播种日期,计算播种至吐丝之间的天数,作为各小区的吐丝期(days to silking,DTS)。吐丝后 15 d,调查各小区株高,从小区第1行的第3株开始,连续调查5株,取平均值,作为小区株高(plant height,PH,cm)。成熟后,收获小区内全部植株的有效果穗(30粒以上),并从中取5个代表性果穗,用于统计穗行数和行粒数,取平均值,作为各小区的穗行数(kernel row number,KRN)和行粒数(kernel number per row,KNPR)。完全风干后,5个代表性果穗混合脱粒,称重后平均,获得单穗产量(ear yield,g)。数取100个籽粒进行称重,3次重复,平均后获得百粒重(kernel weight per hundred,KWPH,g)。小区其余果穗混合脱粒、称重,并与5个代表性果穗产量进行合并,获得小区产量(plot yield,kg)。
1.3 表型数据的统计分析
利用QTL IciMapping、SAS和R软件进行数据的统计与分析。利用QTL IciMapping的ANOVA模块,计算各性状广义遗传力(h2)[16]。利用SAS的“PROC MIXED”模块,计算多环境下各测交组合的产量及重要农艺性状的最优线性无偏估计(BLUP值),用于后续的分析。利用R软件进行性状之间的相关性分析,并绘制散点图、频数分布图、热图和密度分布图。
1.4 QTL 分析
在前期工作中,利用GBS测序[17]完成了中国玉米NAM群体的基因型鉴定,并构建了包含4 932个标记的高密度遗传图谱,标记间平均距离为0.34 cM[18]。基于该遗传图谱和各性状 BLUP值,利用 SAS的“PROC GLMSELECT”模块[19],分别进行昌7-2和郑 58测交群体的联合逐步回归(Joint stepwise regression)分析[14],完成QTL定位(显著性水平为P<0.001)。为了对NAM群体与其测交群体的定位结果进行比较,利用中国玉米NAM群体的多环境表型数据[7,11-13],还开展了 NAM 群体对应性状的 Joint stepwise regression分析和QTL定位。最后,利用SAS软件的“PROC GLM”模块,通过“Population*Marker”的交互效应分析(Population代表NAM群体的11个家系组合,Marker代表各QTL的最显著分子标记),进行了QTL的复等位遗传效应分析,用于QTL位点优良等位变异的鉴定和挖掘。
2 结果
2.1 中国玉米NAM测交群体的表型分析
从测交群体的遗传构成来看,测交群体中50%的遗传成分来自测验种(郑58或昌7-2),其余50%的一半又来自NAM群体的共同亲本黄早四,因此,NAM测交群体为遗传成分一致性相对较高的群体。测交群体的表型也很好地反映了这一情况(表1)。与昌7-2测交群体相比,郑58测交群体普遍表现出花期较早、植株较矮、穗行数较少、百粒重高等测验种郑58所具有的表型特点。与改良瑞德×黄改系杂优利用模式一致,NAM 群体(含有 50%的黄早四遗传成分,作为黄改系材料)与改良瑞德代表自交系郑 58测交群体(改良瑞德×黄改系,为强优势测交群体)表现出了更高的小区产量,显著高于昌7-2测交群体(黄改系×黄改系,为弱优势测交群体)(P<7.84×10-140)。遗传力计算结果表明,2个 NAM测交群体在单穗产量和小区产量上都表现出了相对较低的遗传力,而花期、株高和穗行数的遗传力相对较高。其中,郑 58测交群体的株高遗传力达到了0.92,这与育种实践中郑58测配组合稳定的株高表现一致。郑58测交群体的吐丝期遗传力为0.80,高于昌7-2测交群体的0.76,这与昌7-2具有热带血缘而存在一定的光周期反应相符合。同时,对测交群体内各个RIL测交组合的变异系数(coefficient of variation,CV)进行计算发现,在2个测交群体中,遗传力较高的吐丝期都表现出了最小的变异系数,而低遗传力的小区产量在测交组合之间表现出了最大的变异系数(意味着丰富的小区产量表型变异)。该结果在一定程度上反映了杂种优势利用模式下不同性状遗传结构的不同,而丰富的表型变异也为NAM测交群体产量性状主效位点的挖掘提供了保证。
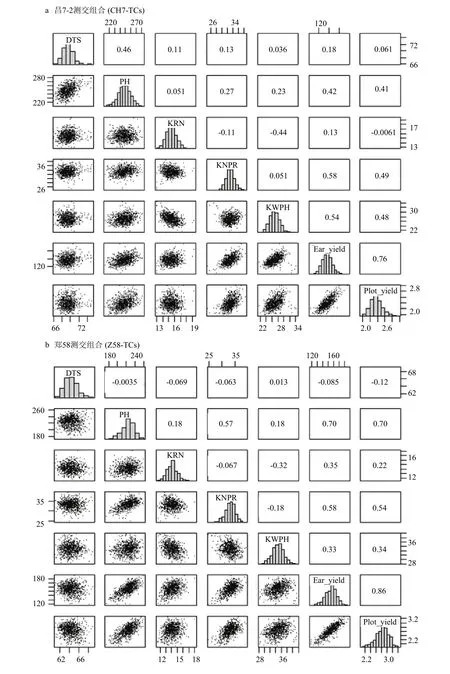
图1 NAM测交群体的产量及重要农艺性状间的相关性分析Fig. 1 Correlation analysis of yield and important agronomic traits for NAM test-cross populations
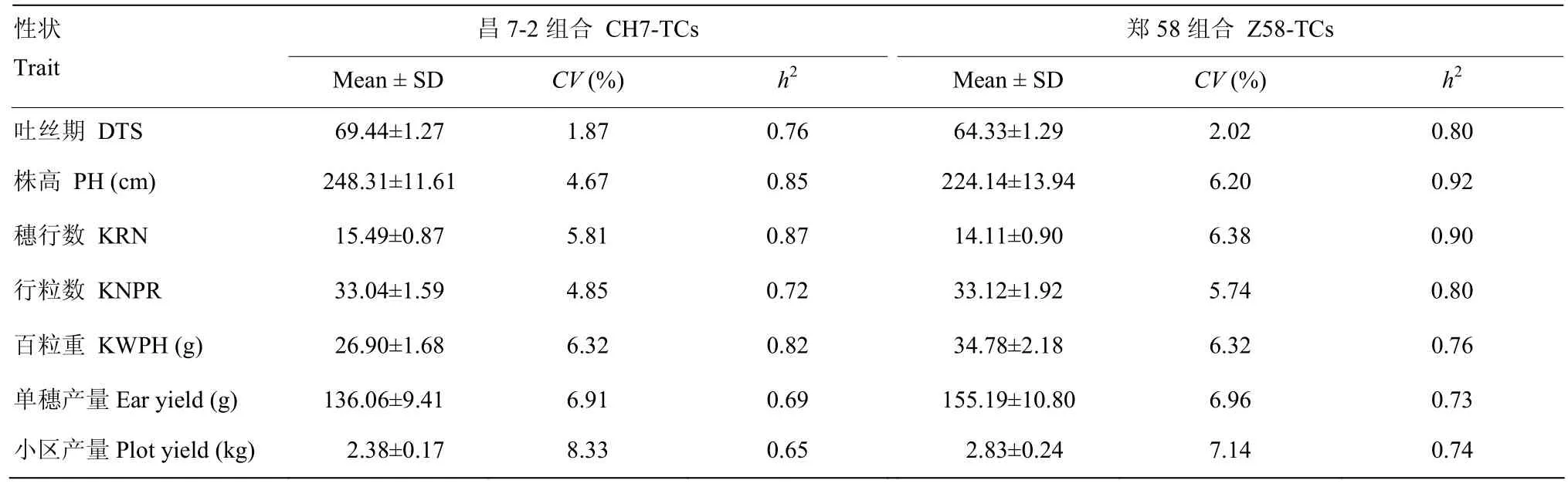
表1 中国玉米NAM测交群体的产量及重要农艺性状统计分析Table 1 Statics of yield and important agronomic traits for Chinese maize NAM test-cross populations
对测交群体产量及重要农艺性状间的相关性进行分析(图1),发现2个测交群体的单穗产量都与小区产量表现出了最高的正相关关系。对郑58测交群体来说,株高与小区产量的相关系数达0.70,远高于昌7-2测交群体的0.41。可见,在郑58测交群体中,高大测交组合更容易获得较高小区产量。同时发现,郑58测交群体的穗行数与小区产量表现出一定程度的正相关关系(r=0.22),但在昌 7-2测交群体中相关性很低。郑58为少穗行自交系(11.84行),当NAM家系无法对郑58的穗行数进行有效互补时,较低的穗行数将影响小区产量。与之相反,昌7-2本身具有较高的穗行数(14.70行),其测交组合整体表现出较高的穗行数,从而降低了小区产量对穗行数的依赖。其他性状表现出了相似的现象,例如,郑58测交群体百粒重与小区产量的相关系数为0.34,而昌7-2测交群体高达0.48。郑58为大粒自交系(30.11 g),其百粒重远高于昌7-2(21.28 g)。然而,在2个NAM测交群体中都没有检测到吐丝期与小区产量之间的显著相关关系。表明在相对一致的遗传背景下,花期早晚可能不是杂交组合获得高产的必要条件。
为了研究NAM群体家系自身与其对应测交群体之间的表型关系,开展了各性状在不同类型群体之间的相关性分析(图 2)。结果发现,具有较高遗传力的性状在NAM群体及其2个测交群体之间可表现出较高的正相关。如,吐丝期、株高和穗行数在3个不同群体之间的相关系数都超过了 0.50。但百粒重在NAM 群体及其郑 58测交群体之间的相关系数为0.39,低于昌 7-2测交群体的 0.51。同样,NAM群体与其郑58测交群体之间在单穗产量上的相关系数仅为 0.14,但与昌 7-2测交群体的相关系数达到了0.26。表明在杂种优势利用模式下,来自不同杂种优势群的测验种对NAM群体测交组合的田间表现具有不同影响。
2.2 NAM及其测交群体的产量和重要农艺性状QTL定位分析
利用Joint stepwise regression模型,分别进行了NAM及其测交群体的产量和重要农艺性状QTL定位分析。在P<0.001的显著水平下,NAM及其昌7-2和郑58测交群体,分别定位到112个、75个和72个QTL位点(表2)。这些位点可分别解释对应群体各性状的表型变异为39.27%—68.52%、52.47%—64.80%和58.37%—79.07%。整体来说,NAM群体QTL对各性状的表型解释率较低,平均为52.68%;郑58测交群体QTL对各性状的表型变异解释率最高,平均达65.25%;昌7-2测交群体居中,平均为60.66%。从单个性状考虑,高遗传力性状QTL的表型解释率相对较高,如吐丝期、株高和穗行数,这些性状在 3个群体中的表型变异解释率均超过 55%,其中,郑58测交群体穗行数的表型变异解释率达到了79.07%。NAM群体的单穗产量、单株产量,及测交群体的单穗产量和小区产量QTL解释的表型变异率较低。其中,NAM群体单穗产量的表型变异解释率仅为 39.27%;NAM 群体单株产量同样较低,为42.17%。NAM 群体单穗产量和单株产量 QTL较低的表型变异解释率,可能与NAM群体本身较低的生活力和环境适应性不佳有关。
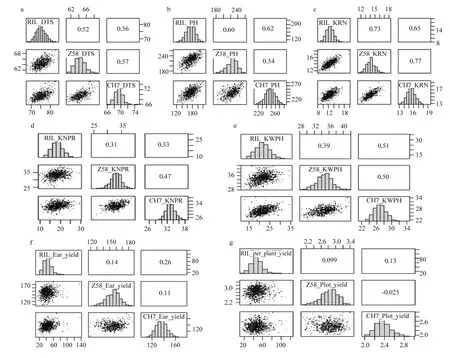
图2 产量及重要农艺性状在NAM及其测交群体间的相关性分析Fig. 2 Correlation analysis of NAM and its test-cross populations for yield and important agronomic traits
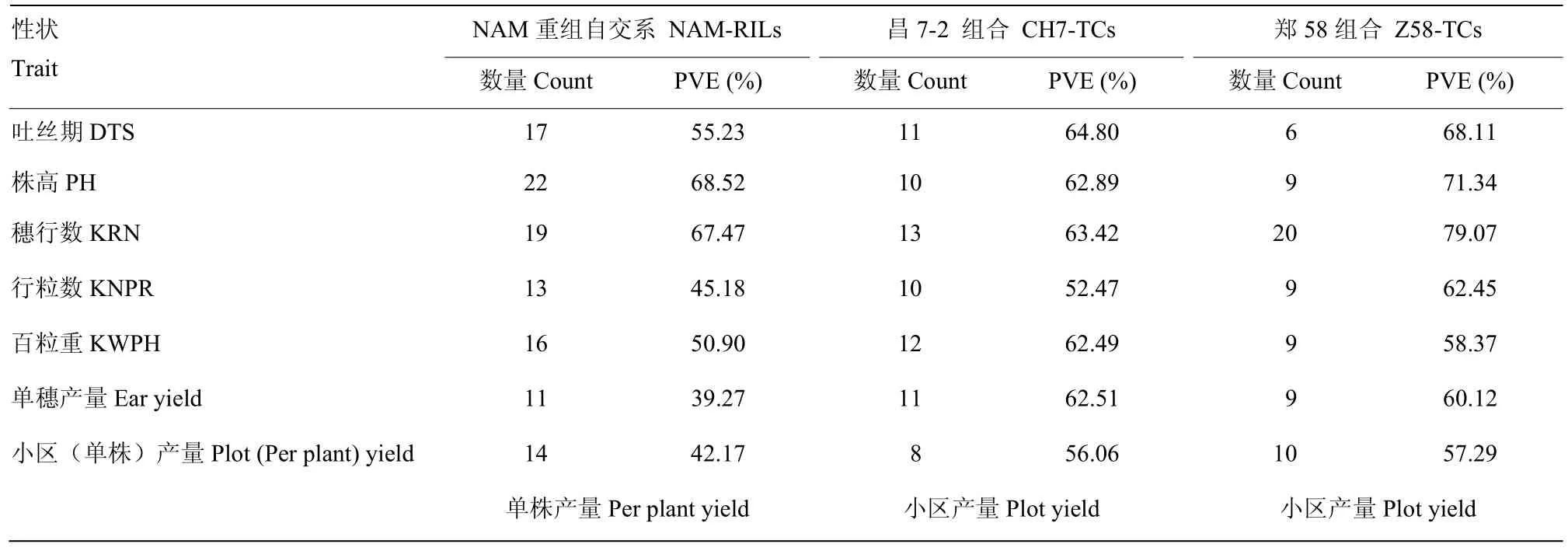
表2 中国NAM及其测交群体产量及重要农艺性状QTL定位分析Table 2 QTL analysis of yield and important agronomic traits for NAM and its test-cross populations
从定位到的QTL数目来看,NAM群体定位到的株高相关QTL位点最多,为22个。在3个群体中都定位到了较多的穗行数QTL。与昌7-2测交群体(13个QTL)相比,郑58测交群体定位到了更多的穗行数QTL(20个QTL),这可能与郑58本身穗行数较少有关(来自郑58的增加穗行数的等位变异较少),导致郑 58测交群体表现出更加广泛的穗行数表型变异,从而提高了穗行数QTL位点的检测效率。除去穗行数,郑58测交群体在各性状上尽管检测到了数目相对较少的QTL,但都表现出了相对较高的表型变异解释率。以上结果表明,郑58测交群体在基因组范围可能拥有更多的显性位点或部分显性位点,从而掩盖了NAM群体RILs对应位点等位变异的表达。
对NAM及其测交群体产量和重要农艺性状QTL的染色体分布进行比较分析(图3),发现昌 7-2和郑58测交群体定位到的QTL中,全部性状分别只有20个(占总数的27%)和18个(占总数的25%)与NAM群体定位到的QTL重叠或邻近,具体分别为:吐丝期5个和4个,株高4个和3个,穗行数3个和4个,行粒数4个和3个,百粒重均为2个,单穗产量都为1个,小区产量与单株产量都为1个。结果表明,在自交系群体与测交群体之间互相进行分子预测的准确性将会较差。同时,也发现了一些可同时被NAM 群体和测交群体定位的一致性遗传区段,这些区段不受杂种优势群和遗传背景影响,包括:2个吐丝期染色体片段(第3染色体的154.66—159.38 Mb、第8染色体的124.36—132.19 Mb)、2个株高相关染色体片段(第3染色体的158.89—165.45 Mb、第8染色体的115.92—133.28 Mb)、2个穗行数相关染色体片段(第4染色体的188.06—199.34 Mb、第5染色体的13.30—17.12 Mb)、1个行粒数相关染色体片段(第7染色体的142.99—150.89 Mb)、1个百粒重相关染色体片段(第7染色体的15.69—23.21 Mb)、1个单穗产量相关染色体片段(第6染色体的34.60—80.64 Mb)。另外,还定位到了一些可同时影响多个性状的“一因多效”染色体区段,例如,所有群体的全部考察性状都可在位于第3染色体154.66—162.19 Mb的区段内定位到QTL,2个测交群体都可在该区段定位到单穗产量和小区产量的主效QTL,但NAM群体在该区段没有定位到相关的QTL,表明该遗传区段内含有重要的玉米重要性状相关基因,且可能与杂种优势的形成具有紧密的联系。另外,位于第 8染色体的115.92—133.28 Mb区段也是一个影响多个性状的染色体片段,在该区段内,NAM 群体及其测交群体可同时检测到吐丝期和株高的相关QTL,该区段还显著影响百粒重、单穗产量和小区产量等重要性状。
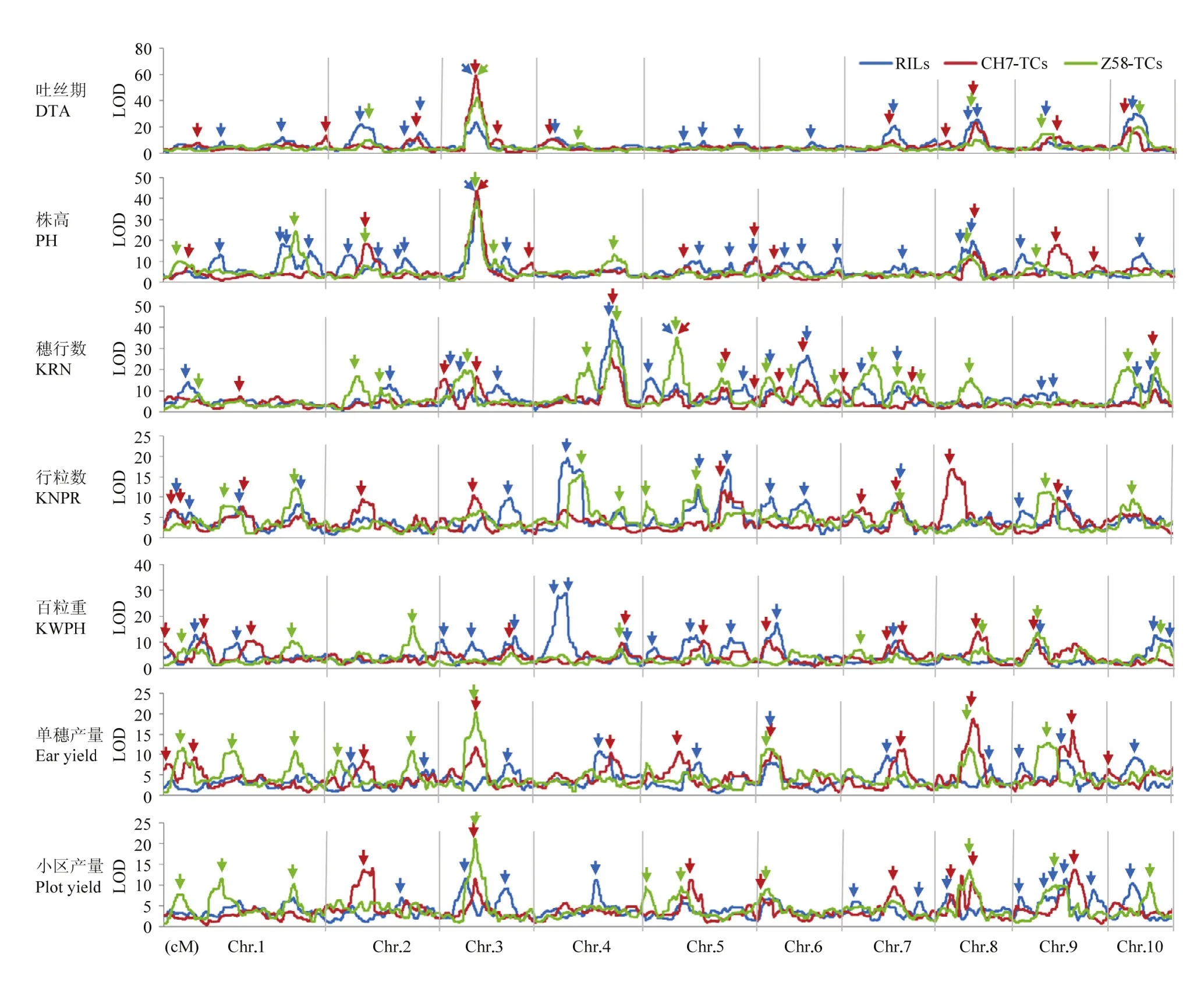
图3 NAM及其测交群体的产量及重要农艺性状相关QTL的染色体分布Fig. 3 Distributions of yield and important agronomic traits related QTL for NAM and its test-cross populations
2.3 产量QTL位点遗传效应的复等位分析
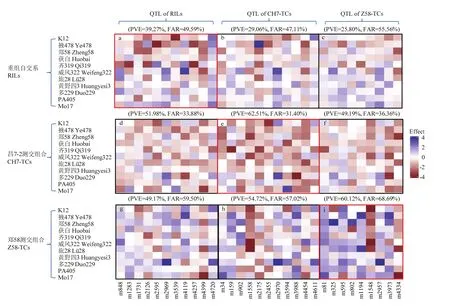
图4 NAM及其测交群体单穗产量相关QTL遗传效应的复等位分析Fig. 4 Multiple-allelic effects of ear yield related QTL for NAM and its test-cross populations
复等位分析对基因位点优良单倍型的挖掘具有重要意义。对于 NAM 群体中的每一个家系组合(RIL群体),各位点的等位变异可分为黄早四和非黄早四,这样就可对黄早四及其他NAM亲本等位变异的遗传效应进行评估。本研究中,针对单穗产量开展了NAM及其测交群体QTL在各个RIL群体中的复等位遗传效应分析(图 4)。结果发现,基于NAM群体自身定位到的单穗产量QTL位点,在11个RIL群体中,49.59%的增产等位变异来自共同亲本黄早四(蓝色为增效)。黄早四等位变异在各RIL群体中的效应值甚至效应方向都有所不同,表现出明显的复等位变异特点。值得注意的是,位于第 10染色体的主效 QTL(m4720),来自黄早四的等位变异在全部11个家系组合中全部表现为增效(图4-a),该位点在郑58测交群体中尽管没有被检测到,但来自黄早四的等位变异同样在郑 58测交后的全部家系组合中表现为增效(图 4-g)。进一步分析发现,在黄早四与 K12、掖478、郑58的RIL群体中,黄早四的等位变异大都表现为减效,但在威风322、多229、Mo17家系组合中,黄早四的等位变异大都表现为增效(图4-a)。对于昌7-2测交群体定位到的单穗产量QTL,在11个家系组合中,只有 31.40%的增产等位变异来自黄早四(图4-e)。分析其原因,可能是由于昌7-2相对于其亲本黄早四富集了更多的优良等位变异,其测交后代遮盖了黄早四等位变异表现为增效的基因位点,从而使一些来自黄早四的不利等位变异位点得到了更多检测。对于郑58测交群体定位到的单穗产量QTL位点,在11个家系组合中,高达 68.69%的增产等位变异来自共同亲本黄早四(图4-i)。可以看出,与郑58测交后,来自黄早四的等位变异在多数位点上表现为增效。但在郑58测交群体定位到的9个单穗产量QTL中,其中2个QTL(m1548和m4334)在绝大多数组合中,来自黄早四的等位变异表现为减效,表明黄早四中仍存在着不利等位变异,且在杂种优势利用方面仍存在继续改良的空间。例如,作为黄早四的改良系K12在3个单穗产量QTL上的表现优于黄早四(m325、m1548和m4334);Mo17在2个单穗产量QTL上(m595和m4334)的等位变异优于黄早四(图4-i)。
利用NAM群体的郑58测交组合,分别定位到9个单穗产量和10个小区产量QTL,分布在13个染色体区段(表3)。遗传效应分析发现,在郑58测交群体中,单穗产量和小区产量分别有7个和8个QTL的增效等位变异来自黄早四,表明在强优势测交组合中(郑58测交群体),来自黄早四的等位变异对高产具有重要遗传贡献。而这些黄早四表现为增效等位变异的QTL,对解释黄早四杂种优势形成的遗传基础将具有重要的生物学意义。
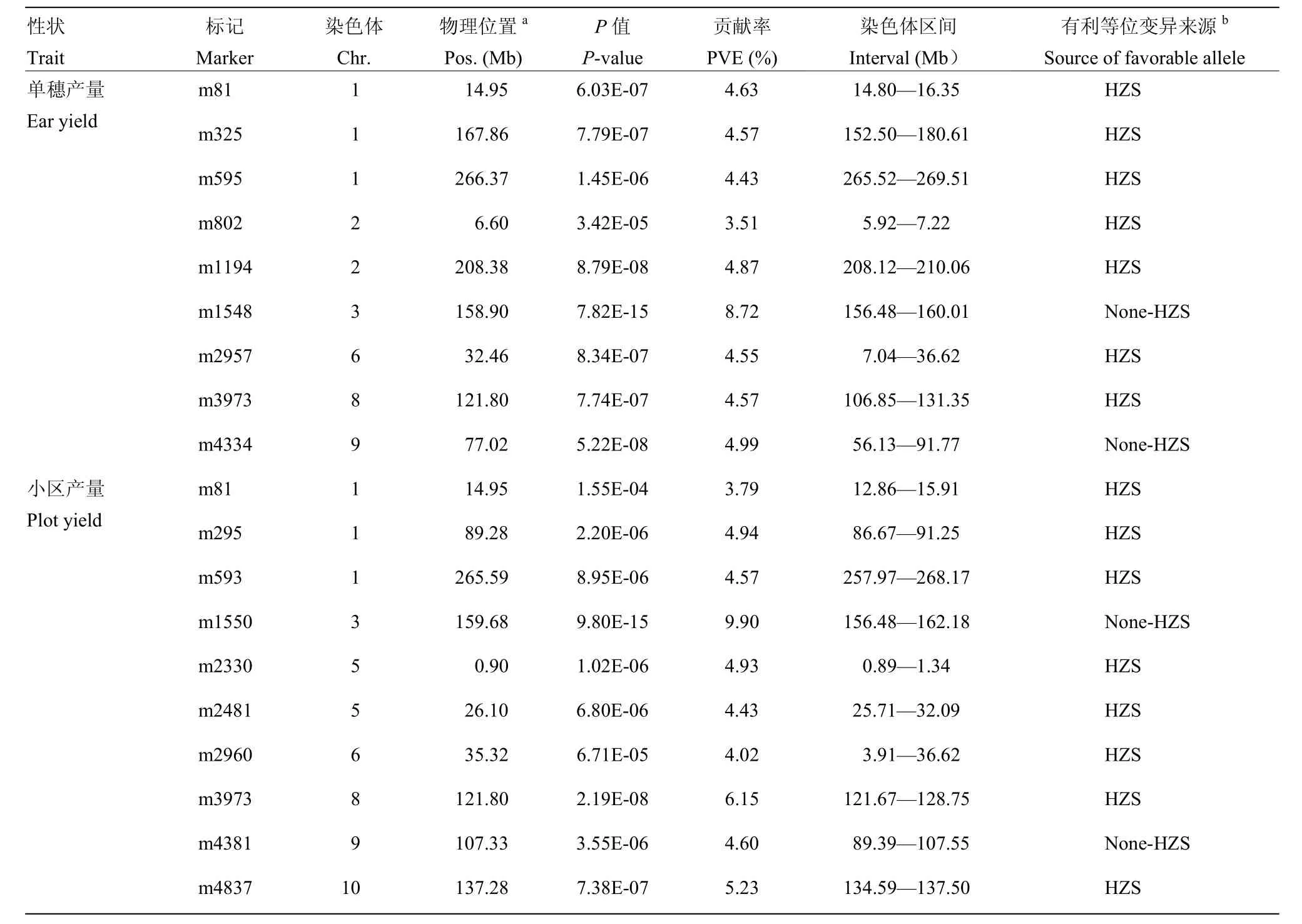
表3 郑58测交群体单穗产量和小区产量QTL定位结果Table 3 QTL related to ear yield and plot yield for test-cross population of Zheng 58
2.4 产量及重要农艺性状QTL遗传区域重组率分析
在杂种优势利用模式下,杂种优势相关位点将倾向于分布在低重组率的遗传区域,原因是这些区域重组率低,有害突变位点难以被重组出去[20]。为此,对NAM及其测交群体QTL区域的染色体重组率进行了分析(图5)。结果发现,郑58测交群体(强优势杂交组合)定位 QTL分布区域的重组率(平均为 0.75 cM/Mb)显著低于NAM群体(平均为1.36 cM/Mb,P<2.59×10-2)和昌7-2测交群体(弱优势杂交组合)(平均为1.91 cM/Mb,P<2.02×10-2)定位QTL分布区域的重组率,但NAM群体定位QTL分布区域重组率与昌7-2测交群体无显著差异(P<0.14)。
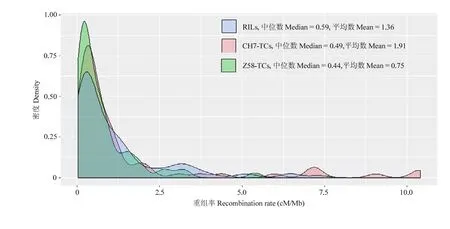
图5 NAM及其测交群体产量和重要农艺性状QTL区域的重组率比较分析Fig. 5 Analysis of recombination rates within QTL regions for NAM and its test-cross populations
3 讨论
3.1 NAM群体在玉米骨干亲本形成遗传基础研究中的利用价值
巢式关联分析群体(NAM)是通过选用具有丰富遗传多样性的多个亲本构建的遗传定位群体,相对双亲定位群体而言,最大优点是可大幅提高群体的遗传异质性,从而提高群体构建过程中可检测交换事件的检出频率,进而提高遗传连锁图谱精度和QTL检出效率[21]。同时,利用 NAM群体还可实现主效位点遗传效应的复等位分析,这对优良单倍型的挖掘极为有利[15]。另外,NAM 群体的构建有一个共同亲本,利用NAM群体进行遗传定位,可实现共同亲本遗传基础的深度解析。美国康奈尔大学以玉米参考基因组测序自交系B73为共同亲本与其他25个多样性自交系为供体亲本,构建了一个包含5 000个RILs的NAM群体[21],利用该NAM群体先后开展了玉米花期[15]、花序[19]、叶片结构[22]、小斑病[23]、大斑病[24]和株型[25]等多个复杂性状的遗传结构研究,获得了大量的主效位点和候选基因,推动了玉米复杂数量性状的遗传基础研究。在中国,为深入研究玉米骨干亲本黄早四形成的遗传基础,从2005年开始,笔者所在课题组着手构建了一个以黄早四为共同亲本,11个来自不同杂种优势群的代表性自交系为其他亲本,包含近2 000个RILs的中国玉米NAM群体。利用该NAM群体,笔者先后开展了玉米产量[7]、雄穗[11]、株型[12]等产量及重要农艺性状的遗传解析。近期,在引进美国玉米NAM群体的基础上,整合中美2个玉米NAM群体(总计7 000个RILs),开展了玉米花期性状[13]和雄穗[26]的遗传结构分析。
玉米是杂种优势利用最成功的作物,针对其杂种优势形成开展遗传基础研究,对玉米高产育种意义重大。本研究构建了中国玉米NAM群体与经典杂种优势利用模式改良瑞德×黄改系代表自交系郑 58与昌7-2的双重测交群体,进行了该NAM群体测交组合的多环境表型鉴定,开展了基于杂种优势利用的玉米产量和重要农艺性状遗传定位。结果发现,利用测交群体定位到的QTL与NAM群体自身的定位结果一致性仅为四分之一,这意味着基于自交系定位到的主效位点将难以有效应用于玉米杂交育种[27],该结果与MÔRO等[28]的研究报道一致。在此条件下,基于NAM测交群体,尤其是强优势测验种的测交群体(如郑58)的研究,对解析骨干亲本黄早四杂种优势形成的遗传基础将更具利用价值。本研究表明,在郑58测交群体中定位到的产量QTL中,大多数位点的增产等位变异来自黄早四(图4-i和表3)。在强优势测验种郑58遗传背景下,来自黄早四的等位变异对测交组合的高产形成具有重要遗传贡献,更重要的是这些遗传区段可能与杂种优势密切相关。另外,利用NAM的群体结构优势,开展主效位点遗传效应的复等位分析,可获得各位点在不同遗传背景下的遗传效应和方向,这将为骨干亲本优良等位变异的保留和不利等位变异的定向改良提供重要的信息指导。综上可以看出,NAM群体试验设计在解析骨干亲本形成遗传基础方面具有重要利用价值。
3.2 玉米骨干亲本黄早四杂种优势形成的遗传基础分析
杂种优势形成的机理在遗传学研究领域一直存在较多的争论。针对杂种优势的遗传基础问题,人们开展了大量研究,并提出了不同的遗传模型,这些模型都有一定支撑或依据。现分子生物学技术手段的兴起,尤其是基因组学研究技术的大量应用,为解析杂种优势形成遗传机理提供了很多新的手段与证据。在水稻中,利用大规模遗传群体的高密度基因型数据,开展了水稻产量性状杂种优势遗传结构研究,认为来自不同亲本的具有显性效应的优良等位变异累加可能是水稻产量杂种优势形成的遗传基础[29]。进一步研究发现,在多个父本遗传背景下,一些来自母本的遗传位点能够解释很大比例的产量性状表型变异,并推测杂合位点的部分显性遗传效应在水稻产量杂种优势中具有重要作用[30]。在玉米中,利用 12个玉米自交系的深度基因组测序数据,发现有害突变在基因组范围内广泛存,且这些有害突变主要分布在低重组率染色体区段;同时发现,基因组范围内优良等位变异对有害等位变异的不完全显性对杂交组合的表型变异和杂种优势具有重要作用[20]。这些研究为了解杂种优势利用及杂种优势群形成提供了重要信息。
郑 58是中国玉米改良瑞德杂种优势群的代表性优良自交系,该系植株清秀、长势健壮、适应性广、自身产量高。更为可贵的是,郑58一般配合力高,且其测交组合普遍能传承该系的优良特点。因此,可以认为郑 58在绝大多数基因位点上拥有优良的等位变异,这些优良等位变异以显性或部分显性的方式存在,在其测交组合中可对其他亲本的等位变异进行互补,从而使测交后代更多表现出郑58表型。但玉米基因组中存在大量的有害突变,在郑58基因组中不可避免地存在一些有害突变位点,且由于有害突变位点一般位于染色体低重组区,导致很难通过重组将不利等位变异淘汰出去。在此条件下,如果存在较郑58更加优良的单倍型,在显性或部分显性的基因作用方式下,测交组合中来自其他亲本的优良单倍型将对郑 58基因组中的有害突变进行有效互补,从而使测配组合表现出更高的生长活力(高产的第一关联要素)和产量。而那些能够在较多基因位点上互补郑 58基因组有害突变的材料,将会与郑58产生较强的杂种优势。如果能够较多互补具有相似基因组结构特点的一批自交系中(如以郑58为代表的改良瑞德)存在的有害突变,那么这些自交系及其类似的种质材料(如以黄早四为代表的黄改系)将会形成一个杂种优势类群,并与前面的类群形成有效的杂种优势利用模式(如改良瑞德×黄改系)。
根据上面的假定,黄早四作为玉米黄改系优势群的骨干亲本,理论上应该将能够与改良瑞德优势群的代表性自交系郑58在产量QTL位点上形成有效的互补,并且能够提供更多的优良等位变异。本研究结果表明,郑58测交群体单穗产量QTL位点在11个RIL群体中的优良等位变异确实主要来自黄早四(图4-i),其中2个QTL(m81和m194)在全部11个RIL群体中的优良等位变异全部来自黄早四,表明黄早四在这些基因位点上能够很好的互补郑 58基因组中的不利单倍型。同时,郑58测交群体QTL也确实主要分布于较低重组率的染色体区域(图 5),符合有杂种优势相关位点的染色体分布特点。以上结果表明,利用郑58测交群体定位到的产量相关QTL对强优势组合郑58×黄早四杂种优势的形成具有重要的遗传贡献。基于产量性状(单穗产量和小区产量),利用郑 58测交群体在基因组中确定了13个重要的染色体区段,这些染色体区段对解析黄早四杂种优势形成的遗传基础,及指导骨干亲本遗传改良具有重要意义,而其真正的基因组遗传变异,将在完成中国玉米NAM群体亲本深度重测序工作之后进行深入的探讨。
4 结论
在杂种优势利用模式下,强优势杂交组合的产量表现受家系自身产量的影响较小。与NAM群体相比,测交群体中检测到的QTL较少,但能解释更多的表型变异。测交群体定位到的QTL中,只有四分之一左右的位点与NAM群体定位结果重叠或相邻。郑58测交群体单穗产量QTL位点在11个RIL群体中,多数增产等位变异来自黄早四。在郑58测交群体中,共鉴定到13个重要的产量相关染色体区段,来自黄早四的等位变异在其中的11个表现为增产,表明这些染色体区段对玉米骨干亲本黄早四杂种优势的形成具有重要作用。

