基于不同数据库来源数据的胃癌长链非编码RNA预后预测模型构建
周新童,党胜春
(1.苏州大学附属张家港医院 普通外科,江苏 张家港 215600;2.江苏大学附属医院 普通外科,江苏 镇江 212001)
胃癌是全球五大恶性肿瘤之一,其病死率在所有癌症中居第2位[1]。中国是胃癌的高发病区,发病患者数大约占全世界的一半[2]。由于其发病隐匿,病情进展较快,5年生存率25%~30%[3]。随着近几年诊断及治疗水平的进步,早期患者的生存率已经得到明显提高,但进展期胃癌患者生存率依旧很低[4-6]。因此,建立良好的诊断和预后筛选评估体系,对胃癌的诊治尤为重要。
在过去的几十年里,人们在胃癌的分子机制研究方面取得了非常大的突破[7-9]。然而,目前尚无能用于胃癌治疗及预后评估的分子标志物。长链非编码RNA(long non-coding RNA,lncRNA)是一类长度超过200个核苷酸的RNA,其本身并不具备编码蛋白质的功能[10]。近年来研究显示,lncRNA在基因组中广泛分布,参与调节染色质修饰及基因表达等重要生理过程[11-13]。lncRNA在肿瘤的早期诊断和预后评估方面的价值也逐渐的被挖掘出来[14-16]。
近几年来,越来越多的人尝试利用公共数据库,比如从癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库中获得大样本数据进行研究[17-19]。然而,单一的TCGA数据库使用存在着一些问题,比如正常对照样本的匮乏(TCGA中仅有32例胃癌样本存在癌旁对照信息)。此外,笔者发现,基因型-组织表达数据库(Genotype-Tissue Expression,GTEx)中保存有大量可作为正常对照的样本信息。
本研究拟将TCGA数据库中胃癌患者和GTEx数据库中正常人胃黏膜的转录组数据联合起来,构建一个由10种lncRNA组成的模型(10-lncRNA预后模型),作为一种新的判断胃癌患者预后状态的指标。随后,通过基因表达汇编数据库(Gene Expression Omnibus,GEO),引入一个独立的数据集(GSE62254),验证该预后模型的稳健性[20]。
1 资料与方法
1.1 数据下载
从TCGA官方网站(https://portal.gdc.cancer.gov/repository)下载获取胃癌的转录组测序数据集,其中胃癌患者的样本375例,对照组样本32例,同时下载相应的临床资料。在进行预后分析及研究风险值(Risk Score)与临床资料的关系时,剔除临床信息不全的样本,共剩余317例。从GTEx官方网站(https://gtexportal.org)下载获取正常人的胃黏膜样本转录组测序数据,共计194例。在筛选差异表达lncRNA时,将32例对照组样本以及 194例正常胃黏膜样本合并,作为对照组与375例癌组织样本进行差异分析。从GEO官方网站(https://ncbi.nlm.nih.gov/geo)下载获取GSE62254数据集,共计300例,同获取该数据集的临床样本文件。
1.2 数据处理
将下载好的TCGA和GTEx的转录组数据合并,取与GSE62254共有的LncRNA,共计1250个。最终获得的TCGA+GTEx矩阵有1250行×601列(lncRNA×样本名),此矩阵将作为建模组用于后续构建预后模型;GEO矩阵有1250行×300列,此矩阵将作为验证组用于对模型进行验证。
1.3 筛选差异表达lncRNA
首先通过R软件中的edgeR包对数据进行标准化,并使用sva包的combat函数对数据进行批次校正,之后使用经验贝叶斯估计,计算癌组织和对照组样本的差异显著性。通过Benjamini &Hochberg方法对P值进行校正,筛选出差异倍数(fold change,FC)>4,且校正后的P值(false discovery rate,FDR)<0.01的基因。
1.4 构建预后模型
将上述筛选出的lncRNA与TCGA 下载的生存数据进行合并,首先采用单因素Cox回归进行生存分析,筛选出P值<0.05的lncRNA。随后进行多因素Cox回归分析,分析方法为向前-向后法,通过赤池信息量准则(Akaike information criterion,AIC),选出AIC值最小,即最优化的预后模型[21-22],并得到模型中各lncRNA的比例系数β,以此计算风险值(risk score),计算公式为:Risk Score=β1X1+β2X2+…+βnXn,其中β表示各lncRNA相关系数,X表示lncRNA表达量。
根据计算出来的各个样本风险值,以中位数为界把胃癌患者分为高风险及低风险组,并且用R软件中的pheatmap包将风险值可视化。使用survivalROC包绘制时间依赖性ROC曲线并计算曲线下面积(area under curve,AUC)。
1.5 统计学处理
本研究所使用的数据处理统计软件为R软件(版本3.4.0)和SPSS(版本19.0),各项分析均为双侧检验,所有统计检验结果的P<0.05被认为具有统计学意义。通过单因素及多因素Cox回归分析,筛选与胃癌预后独立相关的危险因素。预后风险值以中位数(四分位数间距)[M(IQR)]表示,其与临床的相关性分析使用Wilcoxon秩和检验进行两组间比较,多组间比较选用Kruskal-Wallis检验。生存分析结果通过Kaplan-Meier曲线来展示,差异显著性采用Log-rank法检验。
2 结 果
2.1 胃癌lncRNA表达矩阵的建立、差异分析
合并后的实验组(TCGA+GTEx)矩阵进行差异分析,共筛选出288个差异表达lncRNA(FDR< 0.01,|Log2FC|>2),其中236个上调表达,52个下调表达,绘制火山图,见图1。
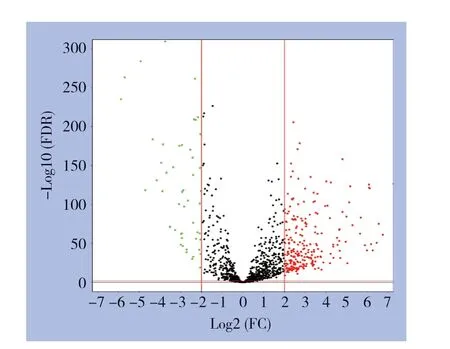
图1 建模组(TCGA+GTEx)矩阵中的差异表达lncRNA(红色代表上调lncRNA,绿色代表下调lncRNA)Figure1 The differentially expressed lncRNAs in modeling cohort (TCGA+GTEx) matrix (the red nodes presenting the up-regulated lncRNAs,and the green nodes indicating down-regulated lncRNAs)
2.2 预后模型的构建
首先对上述的lncRNA进一步筛选,使用单因素Cox回归分析,确定与胃癌患者的总体生存相关的28个lncRNA(均P<0.05),用于预后模型的构建。通过多因素Cox回归分析来构建模型,当纳入的lncRNA个数为10个时,模型具有最小的AIC值:1 451.36,此时模型的拟合程度最优。这10个lncRNA分别是MEG3(P=0.039)、DNAJC9-AS1(P=0.01)、ACTA2-AS1(P=0.122)、C15orf54(P=0.024)、LINC01210(P<0.01)、OVAAL(P=0.078)、POU6F2-AS2(P=0.07)、ERICH3-AS1(P<0.001)、LINC00326(P=0.084)、LINC01526(P=0.078)。最终预后模型:风险值=(MEG3表达值×0.174)+(DNAJC9-AS1表达值×(-0.212))+(ACTA2-AS1表达值×0.112)+(C15orf54表达值×0.140)+(LINC01210表达值×(-0.178))+(OVAAL表达值×0.104)+(POU6F2-AS2表达值×0.076)+(ERICH3-AS1表达值×0.269)+(LINC00326表达值×0.098)+(LINC01526表达值×0.162)。在下文验证组(GSE62254)中同样依据此公式计算风险值。
2.3 预后模型的风险值评估
根据公式所计算出预后风险值的中位数,将样本分为高及低风险组,并对结果进行可视化展示(图2),同时,绘制高及低风险组的Kaplan-Meier曲线,并进行Log-rank检验,分析显示,高风险组的总体生存率明显低于低风险组,高及低风险组5年总体生存率差异存在统计学意义[(18.6±6.2)% vs.(57.6±6.4)%,P<0.001](图3A)。另外,高风险组无病生存率明显低于低风险组,高及低风险组5年无病生存率差异有统计学意义[(34.0±8.8)% vs.(61.4±6.6)%,P<0.01](图3B)。绘制时间依赖性ROC曲线(图4),结果显示,该预后模型的AUC(0.700)明显大于性别(0.542)、年龄(0.586)、T分期(0.564)、N分期(0.576)、M分期(0.532)、TNM分期(0.606)、分化程度(0.563)等指标,提示该预后模型对于评估胃癌患者的预后有一定准确性。
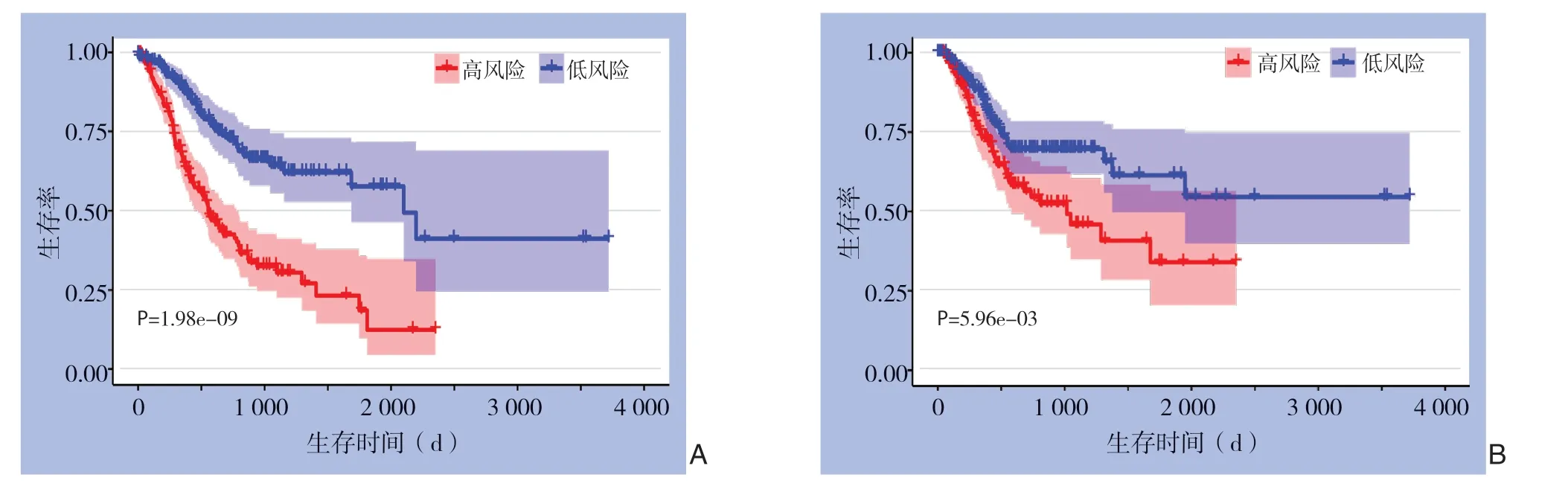
图3 建模组中高及低风险组患者Kaplan-Meier 曲线 A:总体生存曲线;B:无病生存率的曲线Figure3 Kaplan-Meier curves for patients in high-risk and low-risk groups of modeling cohort A:Overall survival curves;B:Diseasefree survival curves
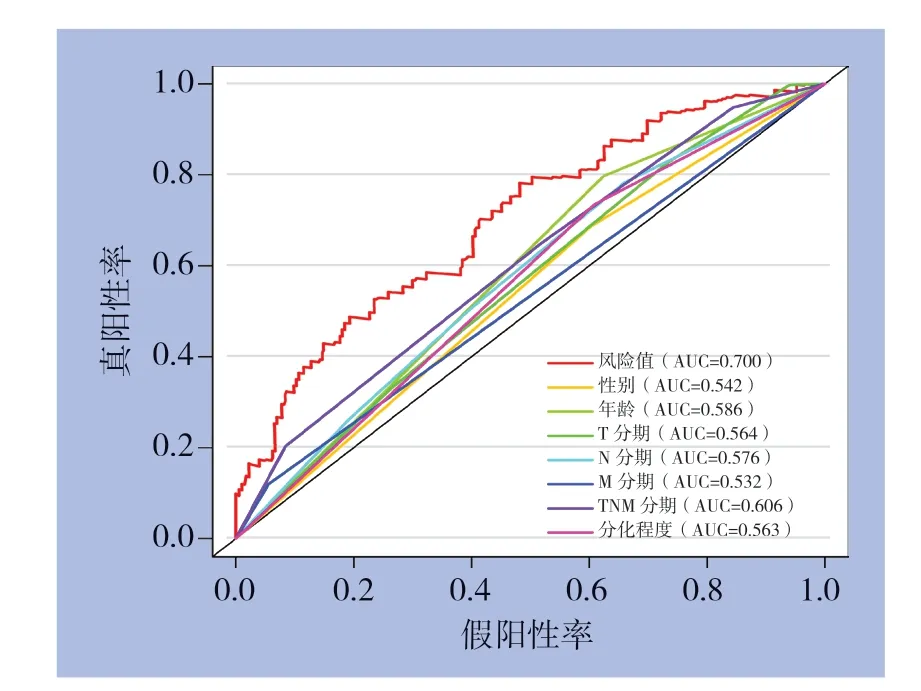
图4 各种临床指标以及预后风险值的时间依赖性ROC曲线以及AUC值Figure4 Time-dependent ROC curves and AUC values for various clinical characteristics and risk score
2.4 筛选影响胃癌生存预后的独立危险因素
通过单因素Cox回归分析寻找与胃癌患者生存预后有关的因素,并绘制森林图,结果显示年龄较高(>60岁)、肿瘤T分期较晚、N分期较晚、TNM分期较晚、风险值较高的患者预后较差(均P<0.05)(图5A)。随后进一步将上述阳性指标纳入多因素Cox回归分析,结果显示年龄和风险值是胃癌患者的独立危险因素(均P<0.001)(图5B)。
2.5 预后模型与临床因素的关系
分析显示,预后风险值与胃癌患者的T分期、肿瘤分化程度明显有关(均P<0.05),而与性别、年龄、N分期、M分期、TNM分期之间无明显关系(均P>0.05)(表1)。
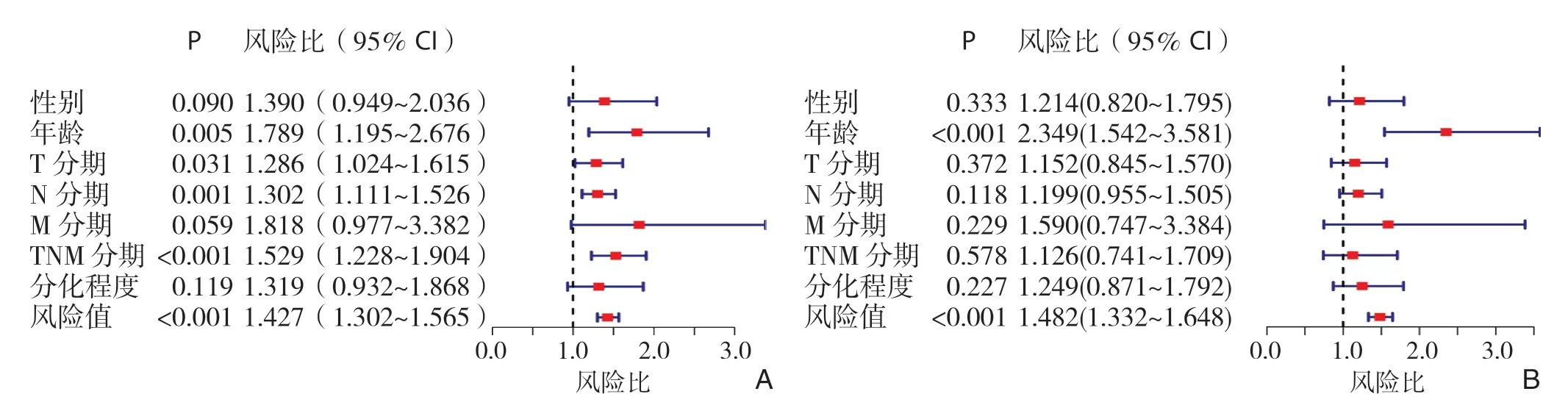
图5 各临床指标以及预后风险值对总体生存时间影响的森林图 A:单因素Cox回归分析;B:多因素Cox回归分析Figure5 Forest plots of the impact of clinical characteristics and risk score on overall survival time A:Univariate Cox regression analysis;B:Multivariate Cox regression analysis
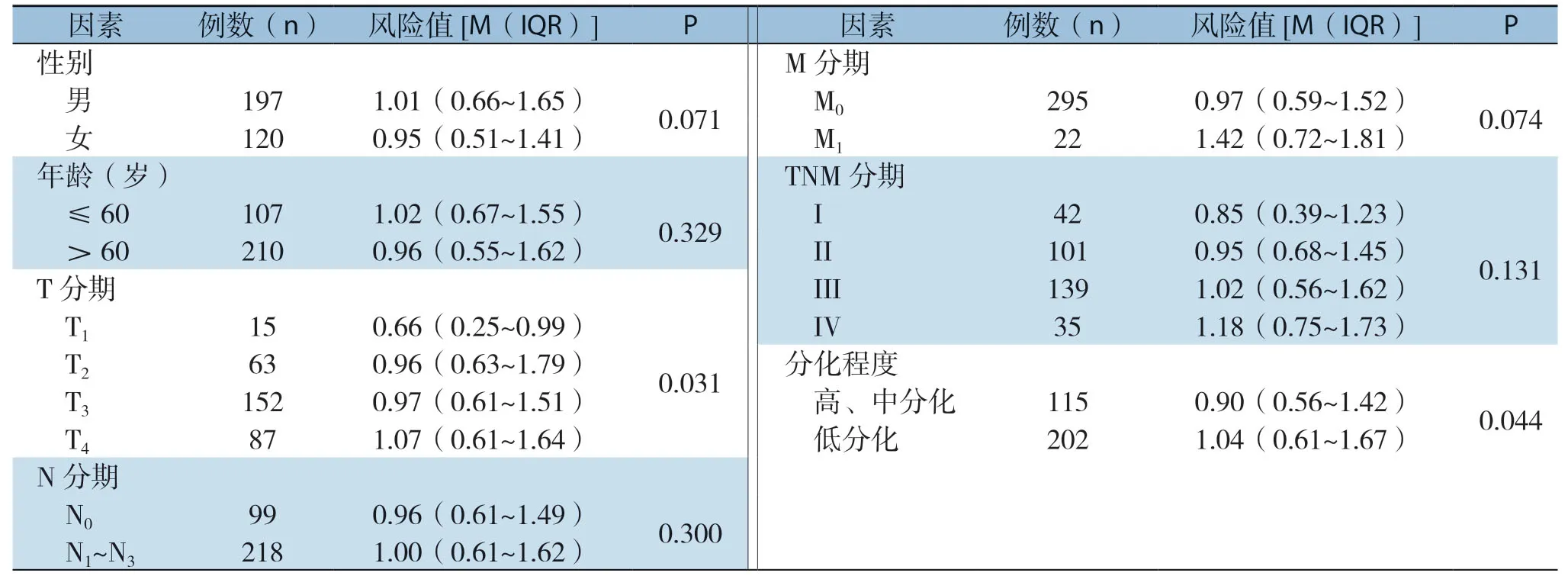
表1 胃癌的临床病理特征与风险值的关系Table1 Relations of clinicopathologic features with risk score in gastric cancer
2.6 预后模型的验证
按照公式计算验证组(GSE62254)中的风险值,并与其临床资料合并,绘制Kaplan-Meier曲线,分析显示,高风险组的总体生存率明显低于低风险组,高及低风险组5年总体生存率存在明显差异[(45.3±4.0)% vs.(59.2±4.0)%,P<0.01](图6A)。另外,高风险组无病生存率显著低于低风险组,高及低风险组5年无病生存率存在明显差异[(42.2±4.4)% vs.(60.1±4.5)%,P<0.01](图6B)。随后进行的单因素(P<0.01)和多因素(P=0.019)Cox回归分析表明,风险值仍为独立预后因素。验证结果提示,该预后模型在不同环境下均具有良好的预测效能。

图6 验证组(GSE62254)中高及低风险组患者Kaplan-Meier 曲线 A:总体生存曲线;B:无病生存曲线Figure6 Kaplan-Meier curves for patients in high-and low-risk groups of the validation cohort (GSE62254) A:Overall survival curves;B:Disease-free survival curves
3 讨 论
过去,人们一直认为lnc RNA是转录过程中的废弃产物。随着研究的深入,lncRNA已经被发现参与许多基本的生物学过程,例如调节细胞周期、细胞凋亡和DNA损伤修复[23-24]。为了探索可用于胃癌的预后风险判断的lncRNA,我们通过挖掘高通量测序数据,构建出能够用于评估患者生存状态的10-lncRNA预后模型。
通过使用10-lncRNA预后模型,可以观察到高及低风险的患者的生存曲线存在着明显的分离。与高风险评分患者相比,低风险评分患者的生存时间显著延长。单因素和多因素Cox回归分析显示,10-lncRNA预后模型与疾病预后独立相关。风险值与T分期及肿瘤分化程度有关,而与N分期及M分期无关,提示10-lncRNA预后模型能够用于判断患者预后的具体机制可能涉及肿瘤的生长及分化,而非迁移及侵袭等生理学过程。
关于这10种lnc RNA的特征,其中5个(DNA J 9-AS1、C15orf54、ERICH3-AS1、LINC00326及LINC01526)迄今为止还没有相关的研究报道,笔者首先报道其表达水平与预后之间关系的研究。另外5个lncRNA,其中母体表达基因3(maternally expressed gene 3,MEG3)在不同的癌细胞(例如乳腺癌、肝癌、结直肠癌及胃癌)中通过调节主要的抑癌基因P53和Rb来发挥抗肿瘤的作用[25]。而ACTA2-AS1的一个转录本的低表达显著促进了肝癌细胞的增殖、细胞周期进程、迁移和侵袭[26]。在卵巢癌中,LINC01210的较高表达与卵巢癌患者较差的总体生存和无病生存相关[27]。在大肠癌和黑色素瘤中,OVAAL与丝氨酸/苏氨酸蛋白激酶3(STK3)的相互作用增强了STK3与RAF-1之间的结合,最终导致RAF/MEK/ERK通路的激活,从而促进了癌细胞的增殖和存活[28]。最后一个lncRNA,POU6F2-AS2参与了电离辐射后食管癌细胞DNA损伤修复并调节细胞的存活[29],且在结肠癌中具有促进肿瘤增殖和耐药的作用[30]。
目前,对于胃癌患者来说,仍然还未有能够有效判断预后的工具。如本研究所示,使用较少量的lncRNA(10个)便可以预测胃癌患者的预后,这为临床医生提供了宝贵而可行的参考。当然本研究还存在局限性,由于高通量测序数据具有一定的误差及背景噪音[31],本研究虽然在分析前已对数据进行标准化及批次校正,且通过独立验证组初步验证了模型的稳健性,但结果仍需临床和基础实验来进一步的研究证实。
总之,本研究构建了与胃癌患者生存相关的10-lncRNA预后模型,并对模型的预测效能进行了验证。未来需要更多细胞、动物功能学实验来探索这些lncRNA的作用。

