区间偏最小二乘结合差分进化算法应用于鱼粉近红外光谱波长筛选
张优优,陈伟豪,唐志敏,辜 洁,莫丽娜,陈华舟,3*
(1.桂林理工大学 理学院,广西 桂林 541004;2.重庆人文科技学院 机电与信息工程学院,重庆 401524;3.桂林理工大学 大数据处理与算法技术研究中心,广西 桂林 541004)
鱼粉是以一种或多种鱼类为原料加工而成的动物饲料,包含许多动物生长所需营养物质,蛋白质含量(约占55%~70%)是评价其质量的主要指标[1]。但传统方法测量鱼粉中蛋白质含量具有过程繁琐、实验操作差异大,容易造成环境污染等问题。近红外(NIR)光谱通过分析待测样品的光谱响应数据来预测样品中的有机物质含量,具有快速、无污染和操作简单等优点[2],已广泛用于农业、医学、食品等领域[3-5]。NIR光谱定量分析时需建立分析模型,由于光谱数据中的波长变量是连续的,且相邻变量的相关性较强,易导致光谱信息冗余[6],因此从测量波长中筛选出具有代表性的特征波长,对NIR定量分析模型进行波长变量筛选具有重要意义。
区间偏最小二乘(iPLS)是近红外光谱特征波段筛选的化学计量学方法[7-8],该法通过对实验波段进行等间距划分,在每个子区间上建立偏最小二乘(PLS)回归模型,再根据模型预测精度选出最优特征波段。iPLS操作简单、便于实现,能够快速地筛选出连续的特征波段,但由于不能选取离散波长点,在连续特征波段中仍存在信息冗余[9-11]。差分进化(DE)算法是模仿自然界中生物的生存行为来构造的优化算法,具有收敛速度快、精准度高等特点[12-13],种群中的个体通过变异、交叉和选择产生新个体,再根据优胜劣汰的原则优选具有更好适应能力的个体,使种群朝最优方向进化[14-15]。DE算法在信号处理、工程优化等方面得到了较好的应用[16-17],在解决连续实值变量问题上展现出优良的性能,然而在解决离散问题时易出现早熟收敛。
基于此,本研究采用iPLS波段优选模式结合二进制变异策略的DE算法[18],提出区间偏最小二乘差分进化(iPLS-DE)算法,以验证集样品的均方根误差最小为目标,在连续特征波段中进一步筛选离散特征波长组合,以寻找更具代表性的特征波长,并将该算法应用于鱼粉光谱数据的波长筛选,通过与iPLS波段优选对比,以期提高鱼粉蛋白质NIR定量分析模型的预测精度,验证iPLS-DE算法在NIR快速定量检测中的应用有效性。
1 实验部分
1.1 实验数据
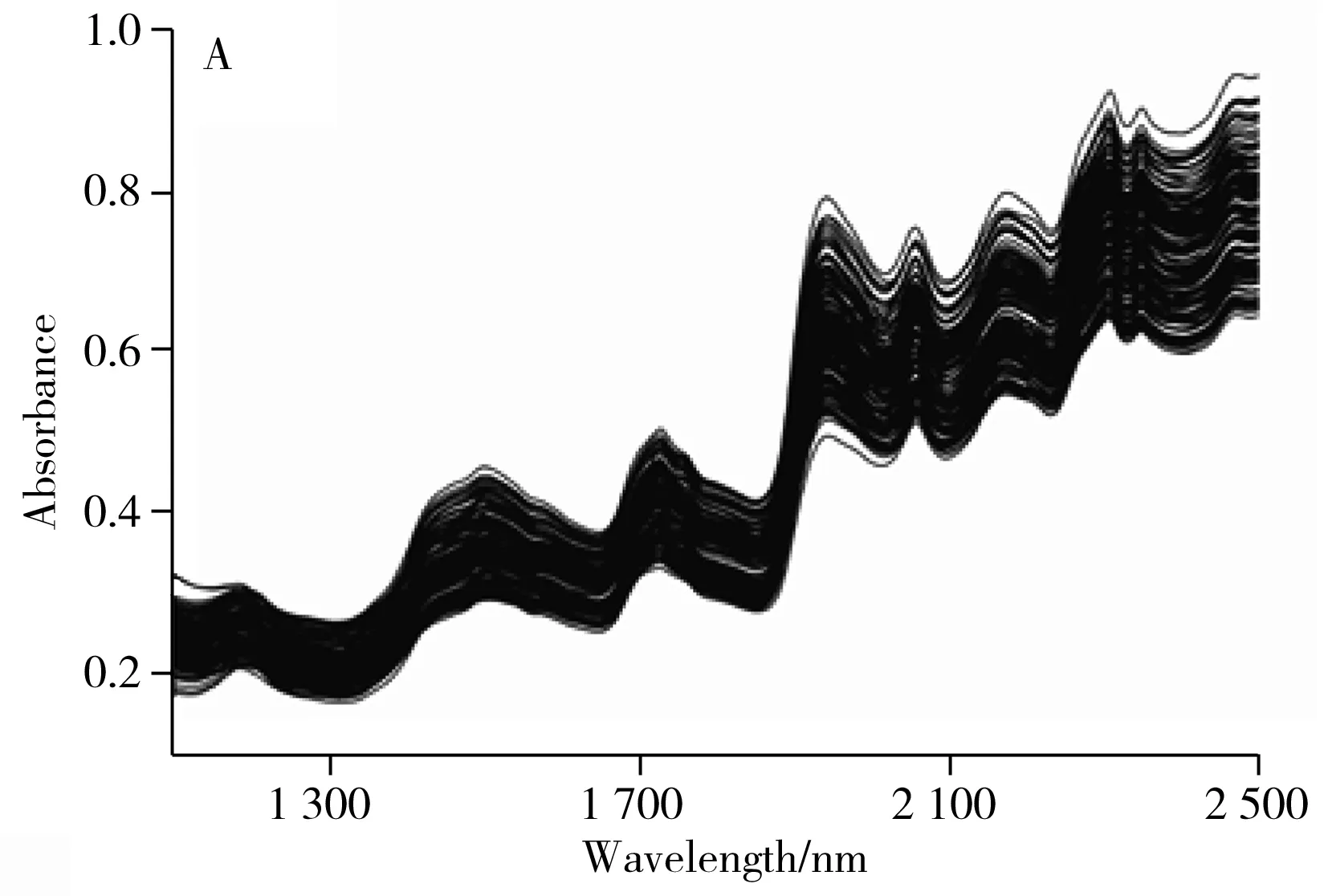
收集来自不同厂家、不同批次的192份鱼粉样品,采用GB/T 6432-1994方法[19]测定各样品中的蛋白质含量作为参考化学值,其范围为53.17%~67.03%,标准偏差和平均值分别为4.38%和60.65%。采用NIR Systems 5000光栅型光谱仪(FOSS公司)以PbS检测器采集鱼粉样品在1 100~2 500 nm的近红外光谱数据,间隔为2 nm,共记录700个波长点,实验在恒温恒湿的条件下进行,实验温度为(25±1) ℃,相对湿度为49%±1%RH,每份样品测量64次后输出平均值。采用标准正态变换(SNV)对鱼粉光谱数据进行建模前降噪预处理,192份鱼粉样品的NIR原始光谱和SNV预处理后的光谱如图1所示。
1.2 样品集划分与模型评价指标
按2∶1∶1的比例划分训练集、验证集和测试集,随机抽取47个样品为测试集,不参与建模训练;采用SPXY方法[20]将剩余样品划分为训练集(97个)和验证集(48个)。各样品集的鱼粉蛋白质含量描述统计数据如表1所示。
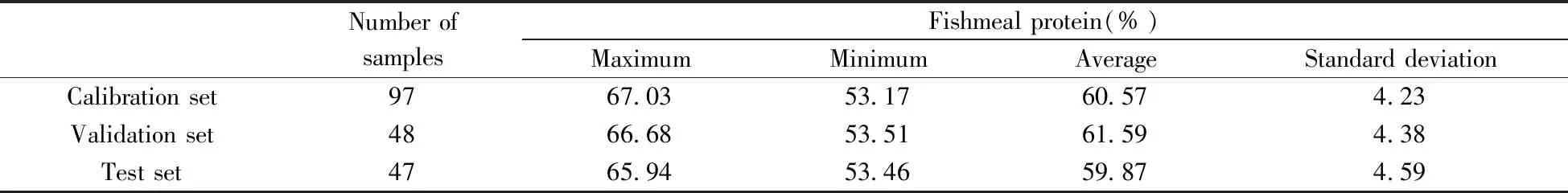
表1 鱼粉蛋白质含量数据的描述性分析Table 1 Descriptive analysis of fishmeal protein content data
近红外分析模型的评价分别包括验证集与测试集样品对模型的评价,本文选用均方根误差(RMSE)和相对分析误差(RPD)进行评价,计算公式如下:

1.3 区间偏最小二乘法
iPLS是将全光谱检测区域分割为n个等长度子区间(n是一个可调试参数)。对不能等分的情况,当剩余波长数超过子区间长度的一半时,则将其作为单独子区间,否则将剩余波长并入最后一个子区间。针对每个固定的n,基于训练集样品分别对每一个子区间构建PLS模型,将模型用于对验证集样品的预测,以验证集的RMSEV最小为原则选择最优光谱子区间内的波段作为iPLS优选的最优特征波段。本文通过调试子区间数量n得到不同的最优特征波段,选择RMSEV最小的最优特征波段所建立的模型为iPLS优选模型。
1.4 iPLS-DE波长优选算法
iPLS-DE是将iPLS和DE相结合的基于iPLS差分进化的特征波长提取方法。首先对全光谱检测区域通过iPLS提取最优特征波段,然后在提取出的最优特征波段内采用二进制变异策略的DE算法,以验证集的RMSEV最小为目标筛选离散的特征波长组合。iPLS-DE算法的主要参数包括种群规模(N)、迭代次数(G)和交叉概率(CR)。具体步骤如下:
Step 1编码。对最优特征波段中的每个波长进行0~1二进制编码,编码0表示该波长未被选中,编码1表示被选中;设置0~1编码的长度等于最优特征波段内的波长数量;
Step 2初始种群。随机生成N个长度为D的0~1编码,用来确定待优化最优特征波段内的波长被选中的状态,形成N个初始种群个体;
Step 3适应度函数。基于训练集样品,针对每个个体(i=1,2,…,N)所选择的特征波长组合建立PLS定标模型,对验证集样品进行预测,再以RMSEV作为个体i的适应度函数值fit(i);
Step 4变异。父代个体xr0,g采用二进制变异策略[18]进行变异操作产生变异个体vi,g+1:
vij,g+1=xr0j,g+(-1)xr0j,g|xr1j,g-xr2j,g|

Step 5 交叉。采用DE传统的单点交叉方式[21],变异个体vi,g+1和父代个体xi,g经交叉算子作用产生新个体ui,g+1,具体如下:
式中,rand为[0,1]之间的随机数,uij,g+1为个体ui,g+1的第j个分量,CR为交叉概率,jrand为[1,D]中随机数。由此定义的新个体ui,g+1同时包含有父代个体xi,g和变异个体vi,g+1的信息;
Step 6 选择。采用贪婪策略对新个体ui,g+1和父代个体xi,g进行选择操作,即适应度值小的个体xi,g+1作为子代个体进入下一代的进化,具体如下:

图2 iPLS不同子区间数量对应的RMSEVFig.2 RMSEV corresponding to different numbers of intervals by iPLS
2 结果与讨论
采用iPLS算法对全谱筛选最优特征波段,其中子区间数量n为可调试参数。为考察不同子区间数量n对iPLS优选最优特征波段和模型性能的影响,设置n从1依次连续增加至30,对每一个n所划分的各个子区间建立PLS模型,将模型用于对验证集样品的蛋白质含量预测,根据RMSEV最小原则可得到iPLS在每个n取值下所对应的最优特征波段,基于每个最优特征波段所建立的定标模型对验证集样品的RMSEV如图2所示。由图2可以看出,当n=5时最优特征波段所建立的定标模型的RMSEV达到最小(0.651%),表示iPLS模型训练效果达到最优,因此将700个波长点等分为5个子区间,其波段范围分别是1 100~1 378、1 380~1 658、1 660~1 938、1 940~2 218、2 220~2 498 nm,在每个子区间上构建PLS定标模型,再将模型用于对验证集样品预测,获得定标模型的RMSEV分别为0.651%、0.913%、1.606%、1.121%和0.922%,以最小RMSEV为原则选择出第一个子区间为最优子区间,其波段1 100~1 378 nm为iPLS在n=5时优选的最优特征波段,包含140个连续波长点。
同时,当n=2、3、4、5、6、7、8、10时,iPLS最优特征波段内所建立的定标模型的RMSEV小于全谱PLS模型(n=1时的iPLS模型)的RMSEV,将其经iPLS优选的最优特征波段分别标记为iPLS(1)、iPLS(2)、iPLS(3)、iPLS(4)、iPLS(5)、iPLS(6)、iPLS(7)、iPLS(8)和iPLS(9),9个最优特征波段的选择如图3所示。由图3可知,9个最优特征波段均出现在等分子区间的第一个子区间,即在第一个子区间中包含与鱼粉蛋白质相关的光谱信息,对9个最优特征波段做进一步的波长筛选,可消除无关信息波长对NIR定量分析模型的影响。
iPLS-DE特征提取是基于iPLS优选的9个最优特征波段分别采用二进制变异策略的DE算法进行特征波长组合筛选,算法参数N、G、CR分别设置为100、500、0.5,迭代的初始适应度函数值为iPLS模型的RMSEV值,经500次优化迭代后所得结果如图4所示。由图4可见,算法迭代的初期收敛速度较快,后期逐渐趋于平稳,针对最优特征波段iPLS(5)内的光谱数据建立的iPLS-DE模型获得最好的迭代优化预测效果,此时验证集样品的RMSEV值更新为0.511%。
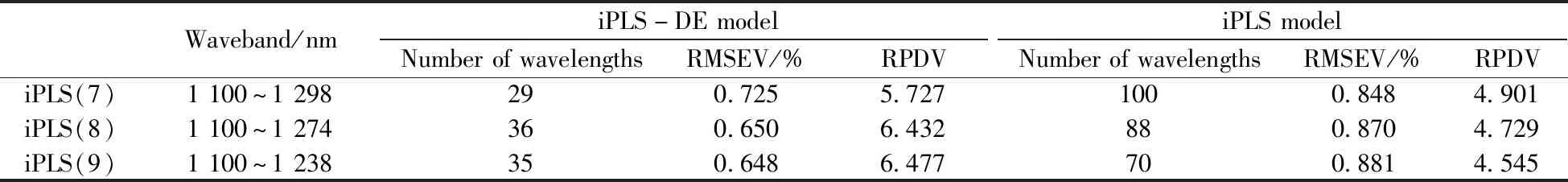
基于9个iPLS优选的最优特征波段,iPLS-DE特征提取方法所建立的鱼粉蛋白质NIR光谱定量分析模型对验证集样品的预测结果如表2所示。结果显示,在9个最优特征波段内,iPLS-DE模型的预测效果均优于iPLS模型,且iPLS-DE的建模波长数量远小于iPLS最优特征波段长度。图5为最优特征波段iPLS(5)上DE迭代筛选出的50个特征波长所建立的iPLS-DE优选模型,其RMSEV和RPDV分别为0.511%和8.302,而iPLS的RMSEV和RPDV分别为0.651%和6.593。






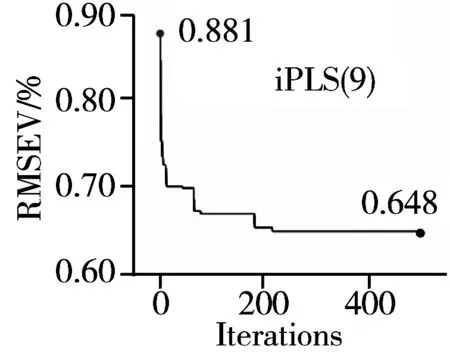
图4 iPLS-DE迭代优化效果Fig.4 Iteration results of iPLS-DE optimization

表2 iPLS-DE特征波长和iPLS特征波段的建模结果对比Table 2 Comparison of the modeling results between the iPLS-DE feature wavelengths and the iPLS feature wavebands
(续表2)

图5 iPLS-DE模型所筛选的50个特征波长Fig.5 The 50 feature wavelengths selected by iPLS-DE
利用iPLS-DE近红外光谱优选模型对测试集样品中鱼粉蛋白质含量定量进行预测。结果显示,测试集样品的RMSET和RPDT分别为1.033%和4.058,而iPLS优选模型的RMSET和RPDT分别为1.131%和3.855。由此可见,iPLS-DE特征提取方法可有效地在全谱范围内筛选出与鱼粉蛋白质含量相关的特征波长信息,降低分析模型的计算复杂度,同时提高了预测精度。
3 结 论
本文研究了iPLS-DE特征波长筛选方法在鱼粉蛋白质NIR光谱定量检测中的应用,发现iPLS-DE可在全谱范围内筛选出50个离散特征波长建立优选模型,对测试集样品的RMSET和RPDT分别为1.033%和4.058。与iPLS优选模型相比,iPLS-DE可更有效地筛选出与鱼粉蛋白含量相对应的波长信息,在简化模型的同时提高了模型的预测精度,克服了iPLS不能筛选离散波长的不足,可尝试推广应用到其他农业的NIR光谱分析。

