5个罗氏沼虾群体微卫星遗传多样性分析
慎佩晶,张宇飞,李喜莲,徐洋,程海华,陈雪峰
(农业农村部淡水渔业健康养殖重点实验室浙江省淡水水产遗传育种重点实验室 浙江省淡水水产研究所,浙江 湖州 313001)
罗氏沼虾(Macrobrachiumrosenbergii),又称大头虾、马来西亚大虾,隶属于节肢动物门,软甲纲,十足目,长臂虾科,沼虾属,原产于东南亚,是目前世界上重要的淡水养殖虾类之一,也是世界上最大的淡水虾。它在水生态系统中分布较广,在淡水区域(如江河、湖泊)和半咸水区域(如河口)均有分布,对水环境中的理化指标变化反应较敏感[1]。它具有生长速度快、食性较广、生长周期短和抗病能力强等特点,自1976年该虾引入我国以来,已在浙江、江苏、上海、广东、广西、湖南、湖北等十多个省市自治区养殖推广。
目前,用于虾类遗传多样性研究的分子标记方法较多。限制性片段长度多态性(RFLP)[2]、随机扩增多态性DNA(RAPD)[3]、简单重复序列标记(SSR)[4]、ISSR[5]、EST-SSR[6]、相关序列多态性(SRAP)[7]、扩增片段长度多态性(AFLP)[8]等多种标记技术已在虾类多样性研究中被采用。其中SSR由2~6个核苷酸为重复单元组成的DNA序列,在真核生物的基因组中分布广泛。这一序列具有共显性、多态性相对丰富、基因组覆盖高等优点。该技术具有易于操作、周期短、等位基因多样性高和稳定性高等特点[9],使得SSR标记成为基因组研究中的一种主要的分子标记,被广泛用于遗传图谱构建[10]、品种鉴定[11]、辅助选择育种[12]、亲缘关系鉴定[13]、遗传多样性分析[14]和系统发育学[15]等研究中。SSR技术也在罗氏沼虾上得到广泛应用。王传聪等[16]对罗氏沼虾肝胰腺组织转录组测序共检测出15 356个SSR位点。蒋飞等[17]应用SSR标记对罗氏沼虾5个选择系子二代共630尾虾的遗传多样性和遗传分化进行了研究。戴习林等[18]利用SSR对罗氏沼虾3个养殖群体进行遗传多样性分析,并探讨了最适宜的样本量及标记量。
本研究选用5个群体127份罗氏沼虾样品,对罗氏沼虾种质进行SSR分子标记遗传多样性分析,可以有效挖掘特异的罗氏沼虾种质材料,促进种质材料的有效利用,这对改良现有品种,提高罗氏沼虾产量和品质,具有极其重要的意义,也为分子标记辅助育种提供理论依据。
1 材料与方法
1.1 样品采集
试验材料为罗氏沼虾南太湖2号,从缅甸与孟加拉引种的缅甸群体和孟加拉群体,2018年新引进的以色列超雌虾和泰国虾群体,取自浙江省淡水水产研究所罗氏沼虾长兴保种基地。其中,南太湖2号为连续第10代选育的核心群,缅甸群体为从缅甸引进的野生种经驯化养殖的第5代,孟加拉群体为从孟加拉引进野生种经驯化养殖的第7代。
分别从以色列群体(I)、缅甸群体(R)、孟加拉群体(B)、核心群体(C)、泰国群体(T)中随机选取8、30、28、30、31个罗氏沼虾个体,采集其腹部肌肉置于无水乙醇中固定,-20 ℃保存备用。
1.2 基因组DNA提取
罗氏沼虾肌肉组织基因组DNA提取采用天根生物公司植物基因组提取试剂盒(DP320)[17],具体步骤按说明书进行。采用NanoDrop2000C分光光度计检测DNA质量和浓度,-20 ℃保存备用。
1.3 微卫星引物设计与合成
9对微卫星引物均为本实验室自行开发的SSR引物(表1),由南京金斯瑞生物科技有限公司合成,并在正向引物5′端添加FAM荧光标记。

表1 微卫星引物的信息
1.4 PCR扩增
以这9对引物分别对127个样品DNA进行PCR扩增,反应体系为20 μL:10×buffer 2 μL,dNTPs(2.5 mmol·L-1)0.5 μL,荧光引物(10 μmol·L-1)各1 μL,DNA模板约50 ng,Taq酶(5 U·μL-1)0.5 μL,加ddH2O至20 μL。PCR反应程序为:94 ℃预变性5 min;94 ℃变性30 s,合适的退火温度下退火30 s,72 ℃延伸30 s(35个循环);72 ℃延伸7 min。各对引物的最适退火温度通过梯度PCR试验确定。PCR产物纯化后,在乙醇已经挥发完全的板中加入内标LIZ500和HiDi,振荡充分混匀,瞬时离心以除去管壁样品。放入PCR仪,95 ℃ 4 min变性。SSR-PCR扩增产物放到3730XL中进行毛细管电泳。
1.5 数据处理与分析
使用Gene Mapper V3.0软件对基因扫描文件进行分析,记录每个位点的相对分子质量大小,得到相应的位点的片段大小和信号值。根据ROX-500标准,自动校准尺寸和峰值。采用Popgen1.32软件计算微卫星座位的有效等位基因数(Ne)、等位基因数(Na)、表观杂合度(Ho)、Shannon’s信息指数(I)、期望杂合度(He)、基因流(Nm)、多态信息含量(polymorphism information content,PIC)、F-统计量(Fis、Fit、Fst)、群体遗传距离等相关参数,并构建UPGMA聚类图。
2 结果与分析
2.1 9个微卫星座位遗传多态性
采用筛选出的9对引物对127份罗氏沼虾种质材料的遗传多样性进行分析,9对引物均具有多态性,扩增出的条带在137~301 bp(如图1为引物11206在罗氏沼虾1、16、28号样品中的扩增结果),共检测到70个等位位点,每个引物检测到5~10个,平均为7.777 8个。其中,引物4179检测到的等位位点最多,有10个;引物168970检测的等位位点次之,有9个。

图1 引物11206在罗氏沼虾1、16、28号样品中的基因分型
各座位的表观杂合度在0.149 6~0.637 8,群体均值为(0.325 6±0.173 6);期望杂合度在0.362 2~0.850 4,群体均值为(0.674 4±0.173 6);9对引物的多态信息量在0.607 4~0.844 9,平均值为0.828 0,其中引物168970的多态信息量最高,为0.844 9,引物10049的多态信息量最低,仅为0.607 4。9对引物的PIC均大于0.5,说明这些标记能提供较高可信度的信息(表2)。
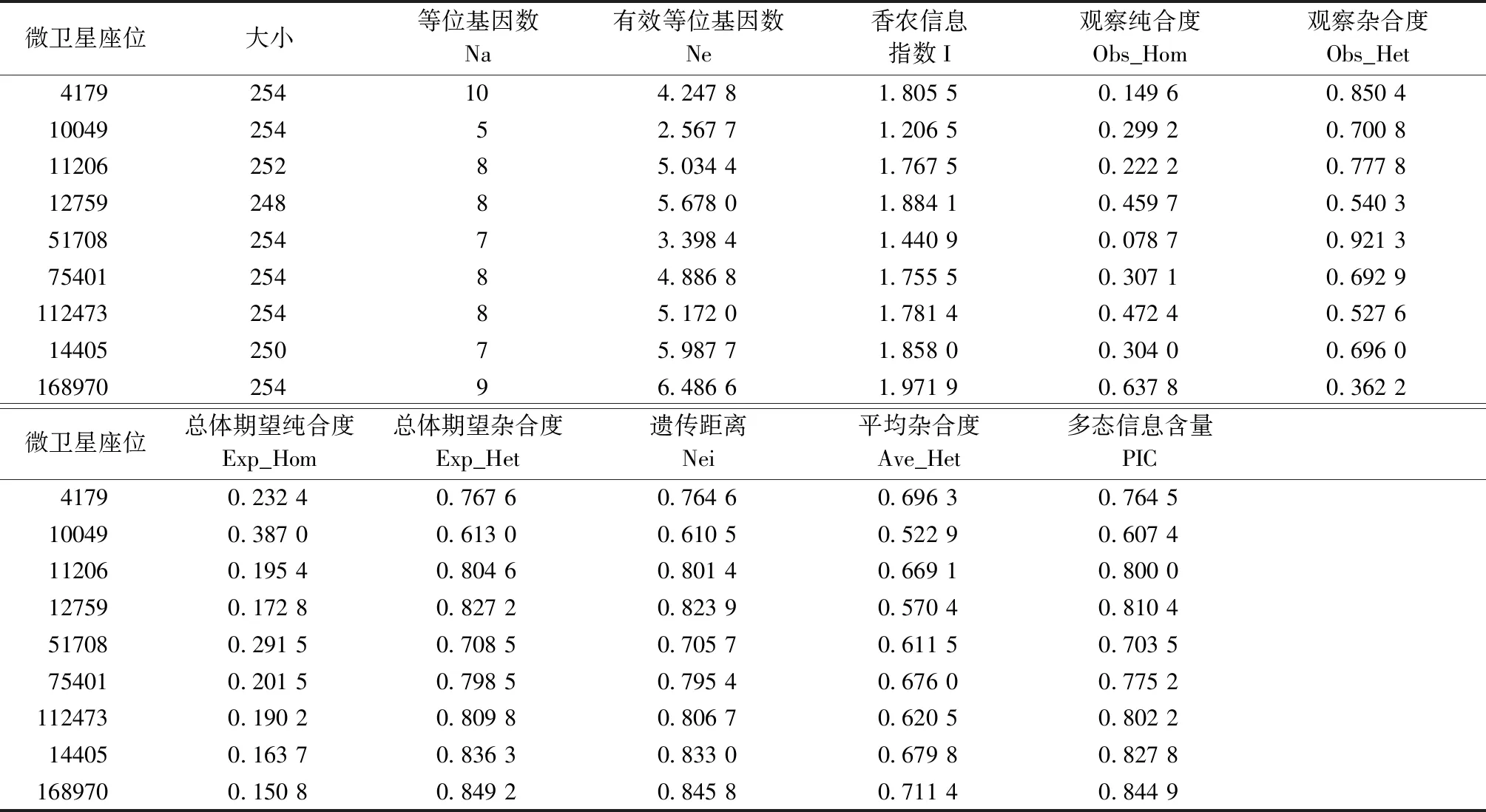
表2 罗氏沼虾5个地理群体9个微卫星位点的遗传多样性
2.2 群体内遗传多态性
9对SSR引物均扩增出清晰的条带,罗氏沼虾种质资源的等位基因(Na)平均为2.777 8~6.666 7。以色列群体最少,泰国群体最多,5个种质资源总体等位基因数平均为5.044 5。有效等位基因(Ne)平均为2.078 8~3.930 5;以色列群体最少,泰国群体最多,5个种质来源总体平均为3.131 4。5个群体PIC值平均在0.453 7~0.548 3,以色列群体最低,泰国群体最高。PIC为衡量基因变异程度高低的多态性信息量指标[19],PIC>0.50时,该引物为高度多态性信息引物;0.25≤PIC≤0.50时,该引物为中度多态性信息引物;当PIC<0.25时,该引物为低度多态性信息引物。核心群体及泰国群体具有较高的遗传多样性;以色列群体、缅甸群体和孟加拉群体具有中度的遗传多样性(表3)。
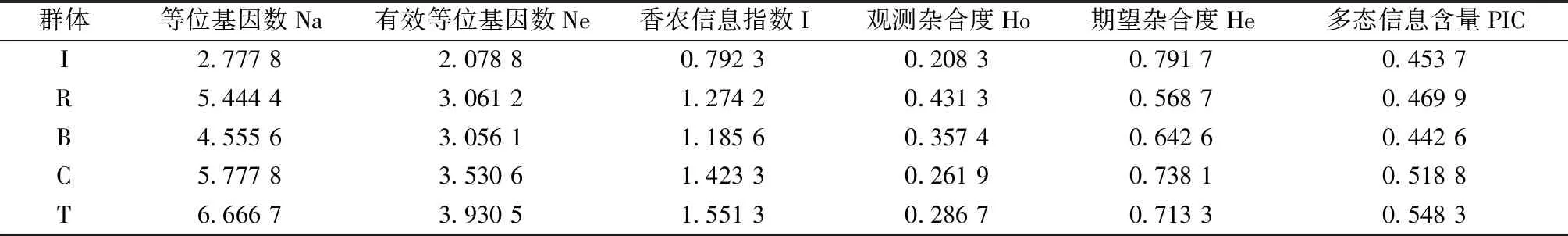
表3 罗氏沼虾5个群体的遗传多样性
2.3 微卫星座位遗传分化
对罗氏沼虾5个群体的Fit、Fis、Nm统计显示,Fis在微卫星位点12759、112473和168970上表现为正值,分别为0.162 9、0.093 7和0.411 9,其余位点均为负值,总体平均为-0.079 3。Fit在位点4179、10049、11206和51708为负值,分别是-0.136 8、-0.221 0、-0.007 0和-0.312 8,平均值为0.105 8。各位点的基因流(Nm)在0.661 5~4.406 0,其中≥1的位点有7个(4179、10049、11206、51708、75401、14405和168970),占所有检测位点的77.78%,均值为1.207 7(表4)。

表4 微卫星座位的F值和基因流
2.4 群体间遗传距离
5个群体间遗传距离范围在0.227 6~1.001 2,其中遗传距离最大的是孟加拉群体与以色列群体之间的距离为1.001 2;遗传距离最小的为孟加拉群体与核心群体之间的距离为0.227 6;平均遗传距离为0.619 4(表5),这与前人的研究结果[20]相比,本研究的遗传多样性更为丰富。这可能与本研究采集的群体样品来源地跨度大、分布广有关。
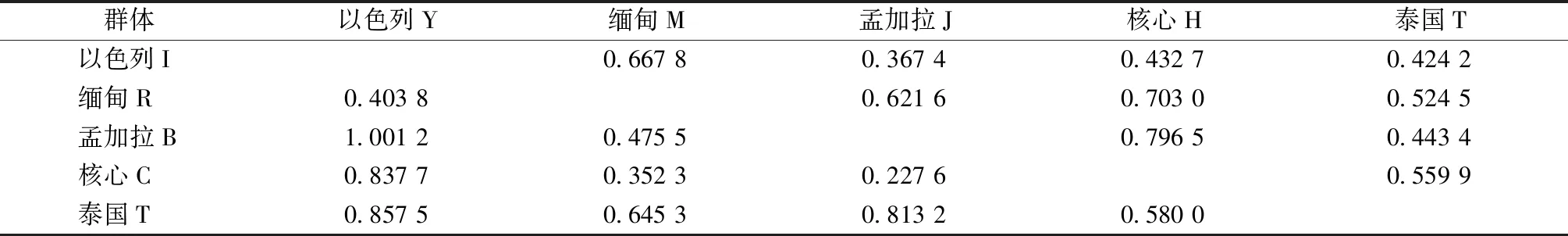
表5 Nei氏遗传距离和遗传相似性
2.5 群体遗传结构
采用Popgen1.32软件,对5个群体的微卫星遗传多样性进行分析,以遗传相似系数为基础进行UPGMA聚类,绘制的遗传聚类树状图能直观表现个体间亲缘。由图2可知,孟加拉群体与核心群体首先聚为一支,再与以色列群体与缅甸群体聚成的一支聚合,聚类最远的为泰国群体。
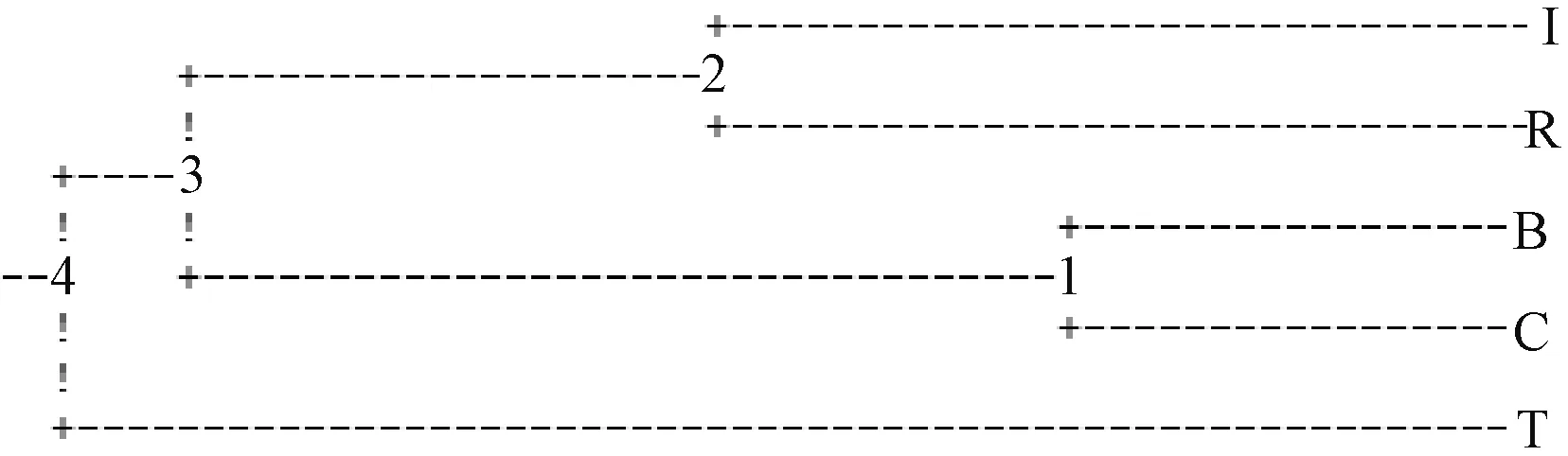
图2 罗氏沼虾5个群体的UPGMA聚类
3 讨论
种质资源是育种的基础,育种材料的遗传多样性能提高育种水平,作为种质评价和利用的基础。遗传多样性是物种长期生存和保持进化的基础[21],也可为基因资源的发掘提供必要的信息[22]。群体遗传结构的研究对物种的保护、利用和改良具有重要的理论和实践意义。
我国自1976年引进罗氏沼虾,由于长期近亲间和在有限的小群体交配繁殖,使这些罗氏沼虾种群基因流失,出现退化现象,抗病性与生长能力都在降低[23]。因此,对罗氏沼虾的群体遗传结构和遗传多样性的研究成为确定优先保护种群、有效的取样策略和近交衰退种群的恢复策略的制定都有极为重要的意义[24-25]。周劲松[26]对罗氏沼虾缅甸引进种和浙江本地种及其杂交种进行SRAP分析,并利用RAPD技术分析了罗氏沼虾单对交配亲本及F1代的遗传关系及分离规律。李明云等[27]利用RAPD技术,对浙江省罗氏沼虾养殖群体和缅甸群体的遗传差异进行了分析,发现缅甸自然群体与浙江养殖群体遗传距离为0.184 5。甘西等[23]采用RAPD技术,对马来西亚引进后代的养殖群体(简称NY)和新加坡F1代与养殖群体交配繁殖的后代(简称NX)的遗传多样性进行了研究,Nx群体内的遗传变异度明显高于Ny群体。
本研究采用9对SSR引物对罗氏沼虾5个群体的127个样品进行遗传多样性及遗传结构分析,共得到70个等位位点,平均多态信息含量(PIC)为0.828 0;群体结构分析结果显示,5个种群遗传变异丰富,可以为新品种的创制提供帮助。

