环状RNA-42398在肝纤维化模型中的表达变化及功能
吕雪幼,周玉平,杨萍,朱林文
(1.宁波大学医学院附属医院 消化内科,浙江 宁波 315020;2.宁波大学 消化疾病研究所,浙江 宁波 315020;3.宁波卫生职业技术学院 护理学院,浙江 宁波 315100;4.宁波大学医学院 生物化学与分子生物学系,浙江 宁波 315211)
环状RNA(circular RNA,circRNA)是非编码RNA(non-coding RNA,ncRNA)研究领域的最新热点之一,在阐明疾病发病机制及相关的诊断标志物及治疗靶点发现方面,取得了重大进展[1]。研究发现circRNA与原发性肝癌、脂肪肝等多种慢性肝病有关,部分学者也初步探讨了其与肝纤维化的关系[2-3]。 本课题组通过基因芯片技术初步分析了肝纤维化模型小鼠肝脏的circRNA表达谱变化,发现mmu_circ_42398的表达变化差异最大,模型组与正常组表达量相差11.74倍[4]。本研究拟在此基础上,进一步验证mmu_circ_42398在肝纤维化模型肝组织和相关的细胞模型的表达变化,并初步探讨其功能。
1 材料和方法
1.1 材料
1.1.1 实验动物:C57BL/6雄性小鼠,SPF级,体质量18~22 g,购于北京维通利华实验动物技术有限公司,生产许可证号:SCXK(京)2016-0006,饲养及实验于宁波大学实验动物中心,实验获得宁波大学动物伦理委员会批准同意。AML12细胞株、RAW264.7细胞株购自中国科学院上海细胞库;JS1细胞株购自深圳豪地华拓生物公司。
1.1.2 主要试剂和仪器:CCl4分析纯及3% H2O2
购自上海国药集团化学试剂有限公司。胎牛血清、细胞培养基购自美国Gibco公司。超氧化物歧化酶(superoxide dismutase,SOD)、谷丙转氨酶 (glutamic pyruvic transaminase,ALT)、谷草转氨酶(glutamic oxaloacetic transaminase,AST)检测试剂盒购自南京建成生物工程研究所。肿瘤坏死因子(tumor necrosis factor-α,TNF-α)检测试剂盒购自武汉USCN公司。重组TGF-β1细胞因子购自南京金斯瑞生物科技公司。脂多糖(lipopolysaccharide, LPS)购自北京索莱宝科技有限公司。RNAhold液购自北京全式金生物公司。TRIzol试剂购自美国 Invitrogen公司。反转录试剂盒GoScriptTMReverse Transcription System、实时荧光定量PCR试剂盒GoTaq®qPCR Master Mix购自美国Promega公司。α-SMA、collagen I(ColI)、GAPDH蛋白一抗购自美国Proteintech公司。mmu_circ_42398过表达质粒及对照质粒由广州吉赛生物科技公司构建。 Lipofectamine 2000脂质体转染试剂购自美国 Invitrogen公司。Agilent-Mx3005P实时荧光定量PCR仪为美国安捷伦公司产品。PCR小鼠引物由上海英骏生物技术有限公司合成,序列见表1。

表1 qPCR所用引物序列
1.2 方法
1.2.1 动物造模和标本取材:小鼠适应性喂养3 d, 随机分为正常组、模型组,每组8只。模型组按体质量以15% CCl4-橄榄油溶液2 mL/kg,腹腔注射,每周3次,正常组不处理。5周末采集小鼠肝脏,戊巴比妥钠腹腔注射麻醉,下腔静脉采血,切取肝脏最大叶放入4%多聚甲醛固定用于石蜡切片;其余肝组织液氮中速冻,后转入-80 ℃长期保存备用。
1.2.2 组织病理学观察:制作肝组织石蜡切片,行HE染色和Masson染色,观察组织炎症和胶原增生情况。组织切片由2位病理专家盲法进行阅片。
1.2.3 复制肝星状细胞活化模型:培养JS1细胞至对数生长期细胞,铺6孔板,每孔1.5×105个细胞,完全培养基(含10% FBS)培养24 h后,换成无血清DMEM饥饿培养细胞12 h,再给予5 ng/mL(DMEM稀释)TGF-β1刺激细胞24 h,收集细胞进行qPCR实验。
1.2.4 复制肝细胞氧化应激损伤模型:培养AML12细胞至对数生长期,铺6孔板,每孔3×105个细胞,完全培养基DMEM+F12(1:1)培养24 h后,换成无血清培养基饥饿培养细胞12 h,添加H2O2孵育(终浓度为250 μmol/L)、12 h,收集细胞上清液采用试 剂盒检测ALT、AST、SOD,收集细胞进行qPCR实验。
1.2.5 复制巨噬细胞炎症激活模型:培养RAW.264.7 细胞至对数生长期,铺6孔板,每孔1.5×105个细胞,完全培养基(含10% FBS1640培养基)培养24 h后,换成无血清DMEM饥饿培养12 h,再给予100 ng/mL(DMEM稀释)LPS刺激12 h,收取培养液用ELISA试剂盒检测TNF-α含量,收集细胞行qPCR实验。
1.2.6 生物信息学分析:在目标序列上使用最新miRBase中的microRNA信息进行靶点预测,根据与目标序列上MRE的microRNA覆盖度以及整合其他信息(mRNA/microRNA表达谱数据),选择适当的过滤阈值(一般为site coverage≥0.3),得到符合要求的CeRNA关系对。采用Cytoscape软件绘制CeRNA关系网络图,以说明circRNA-miRNA-mRNA的关系。
1.2.7 qPCR:TRIzol法提取总RNA,将2 μg RNA反转录成cDNA(20 μL体积),5倍稀释后-20 ℃保存备用。PCR扩增反应体系为20 μL,条件设置为:95 ℃ 预变性10 min;40个PCR循环(95 ℃变性10 s, 60 ℃退火60 s)。采用2-ΔΔCT法计算目的基因的相对表达量。每组设3个复孔,重复3次实验。
1.2.8 Western blot:提取JS1细胞蛋白,BCA法定量,SDS-PAGE电泳,转PVDF膜,洗膜,5%BSA/PBS封闭1 h,加一抗(α-SMA、ColI及GAPDH),4 ℃轻摇孵育过夜,洗膜,加二抗,室温孵育1 h。进行ECL化学发光显影,获取条带后Image J软件分析灰度值,以GAPDH为内参计算蛋白相对表达量。每次设3个复孔,重复3次独立实验。
1.2.9 过表达质粒构建和细胞转染:构建pLC5- mmu_circ_42398过表达质粒载体,JS1细胞分为mmu_circ_42398组、阴性对照组(vector组)和control组,利用Lipofectamine 2000转染试剂,将过表达载体或对照空载体转染到细胞,48 h后收集细胞,提取细胞样品总RNA,反转录后进行qPCR检测 mmu_circ_42398表达情况。同时设计测序引物(F:5’-CCTGAGTTCTTCAAGTCTGCTC-3’;R:5’-CCAGCTCCA TTAGCACCTGGAAG-3’),PCR后回收条带测序验证环化位点接头。另提取细胞总蛋白,Western blot检测细胞α-SMA、ColI蛋白表达情况。
1.3 统计学处理方法 采用SPSS20.0软件进行分析。计量资料以表示,2组比较采用两独立样本t检验。P<0.05为差异有统计学意义。
2 结果
2.1 肝纤维化模型小鼠肝组织病理学及mmu_circ_42398的表达变化 HE染色和Masson染色显示,模型组小鼠肝细胞广泛坏死,正常肝小叶结构消失,可见大量的炎性细胞浸润,肝组织胶原广泛增生,由汇管区向四周放射延伸,部分形成大小不一的完整假小叶结构,肝纤维化模型制备成功(见图1A-B)。qPCR结果显示,模型组肝组织mmu_circ_42398表达显著低于正常组(P<0.05)(见图1C)。
2.2 mmu_circ_42398在肝纤维化相关的细胞模型中的表达变化 qPCR结果显示,TGF-β1刺激JS1细胞后α-SMA、ColI mRNA表达显著升高(P<0.01),提示JS1细胞活化模型复制成功;qPCR结果显示,相比对照组,模型组mmu_circ_42398表达显著降低(P<0.05)(见图2A)。生化试剂盒检测结果显示, 250 μmol/L的H2O2刺激AML12细胞12 h后ALT、AST酶活性显著升高(P<0.05),提示肝细胞氧化应激模型复制成功;qPCR结果显示,相比对照组,模型组circRNA_42398表达显著降低(P<0.01)(见图2B)。ELISA检测结果显示,100 ng LPS刺激RAW264.7细胞12 h后细胞上清中TNF-α水平显著升高(P<0.05),提示细胞炎症损伤模型复制成功;qPCR结果显示,相比对照组,模型组mmu_circ_42398表达显著降低(P<0.05)(见图2C)。
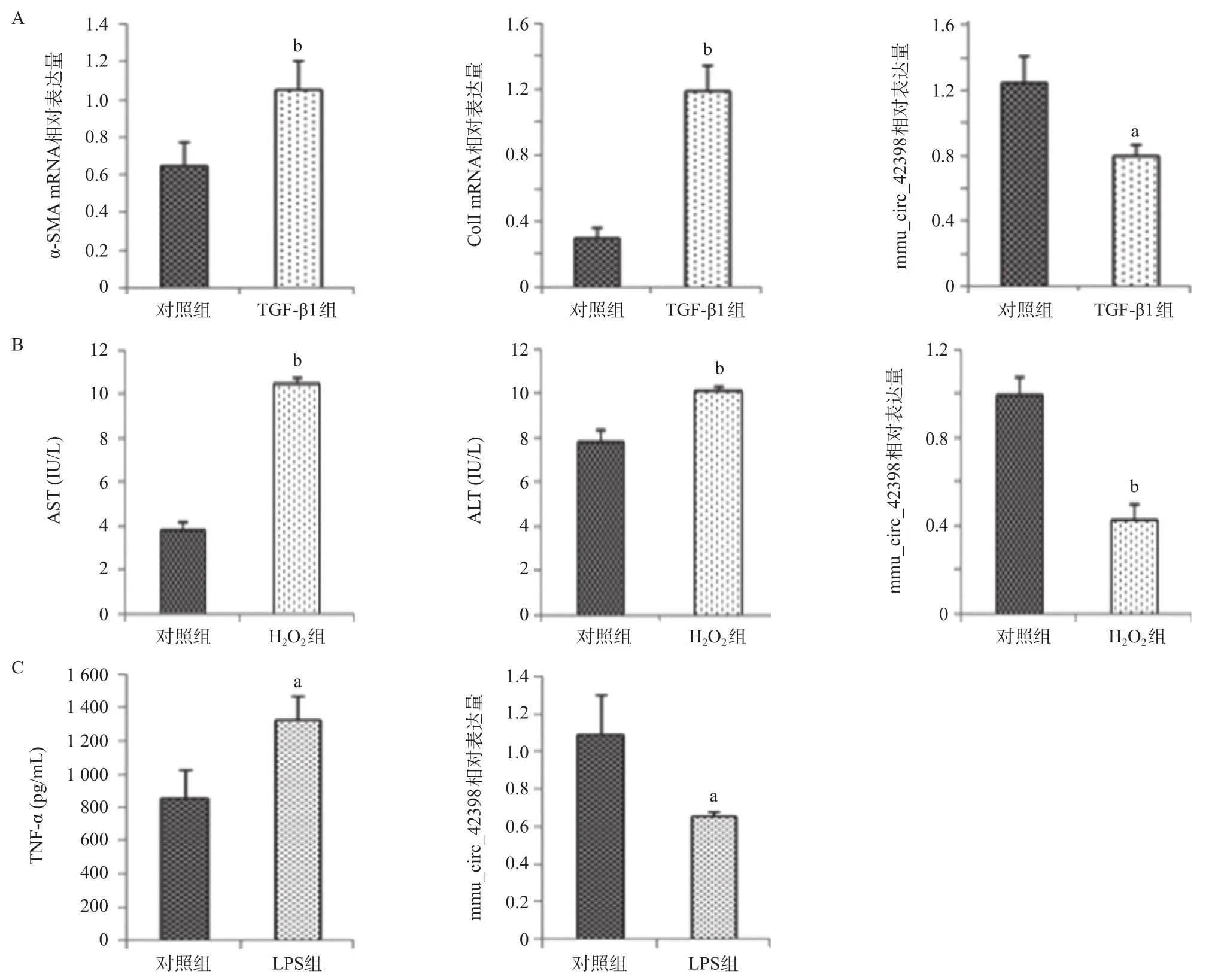
图2 mmu_circ_42398及炎性指标在肝纤维化相关的细胞模型中的表达变化
2.3 mmu_circ_42398靶基因的生物信息学分析 通过生物信息学分析预测,得到mmu_circ_42398作用的靶基因,同时利用cytoscape软件建立 circRNA靶基因参与的信号通路网络关系图,粉红色代表其可能结合的miRNA,黄色代表靶miRNA可能结合的mRNA,见图3。经过文献检索及分析发现,mmu_circ_42398的靶基因miR-338-3p可能和肝星状细胞(hepatic stellate cell,HSC)活化相关信号通路有关,从而影响肝纤维化进展。
2.4 mmu_circ_42398过表达对JS1细胞活化的影响 qPCR检测结果显示,过表达组mmu_circ_42398的基因表达较vector增加几百万倍(P<0.01)。将PCR产物进行一代测序,确定其环化位点,并将其序列信息与数据库circbase中的序列信息对比,结果是一致的,证明该cicrRNA确实为环状RNA,而非线性RNA结构。qPCR结果显示,mmu_circ_42398过表达的JS1细胞α-SMA及ColI的蛋白表达显著降低(P<0.01)。见图4。
3 讨论
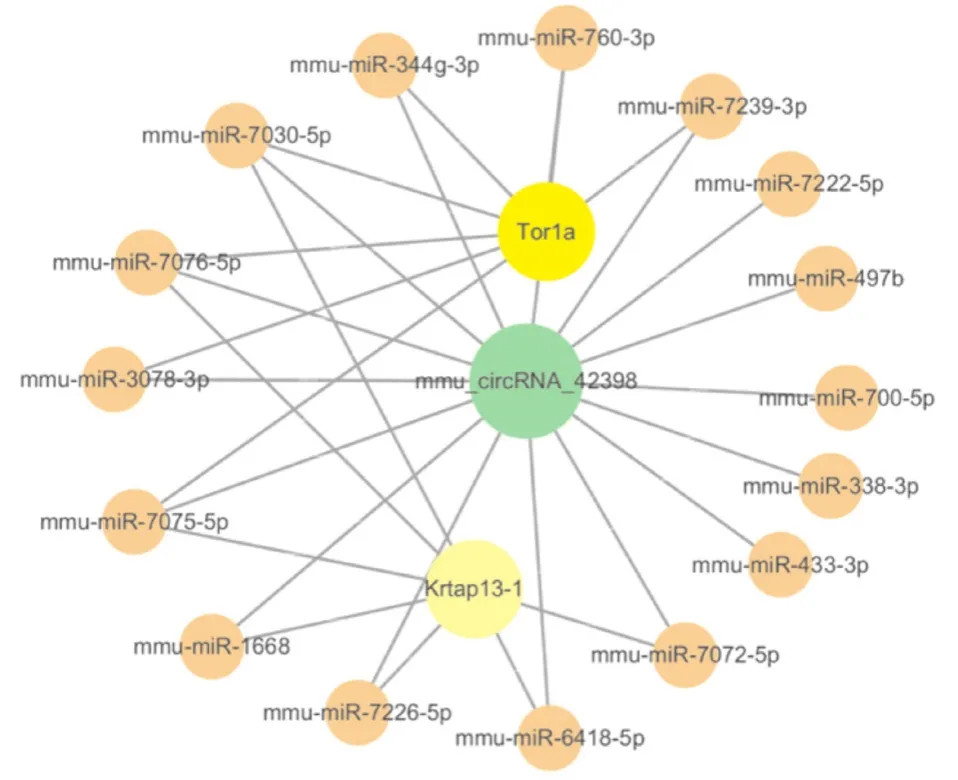
图3 mmu_circ_42398靶基因参与的信号通路网络图
近几年circRNA研究取得了较大进展,其与肝脏疾病的关系也逐渐被揭示,但目前的研究主要聚焦在肝癌、脂肪肝等领域,与肝纤维化关系的研究报道不多[2-3]。2017年CHEN等[5]首次报道了circRNA 与HSC活化有关,采用芯片技术分析了放射线诱导的LX-2细胞活化模型的circRNA差异表达谱,并进一步证实了hsa_circ_0071410可通过“miRNA海绵作用”结合miR-9-5p,从而影响HSCs的活化;WANG等[6]发现患者血清CircMTO1表达水平与肝纤维化疾病进展显著负相关,通过体外HSC细胞活化模型进一步证实了CircMTO1可通过结合miR-17-5p,提高Smad7的表达水平,从而抑制肝纤维化进展;另外,ZHU等[7]的研究发现circRNA-0067835可通过调节miR-155/FoxO3信号通路影响HSC的活化。上述几项研究表明,部分circRNA可影响HSC活化这一肝纤维化进展的关键细胞生物学事件,但circRNA与肝纤维化的关系仍有待进一步探究。
本课题组围绕circRNA与肝纤维化的关系也开展了探索性研究,通过基因芯片技术筛选出了肝纤维化模型肝组织中差异表达的circRNA谱,其中变化差异最大的为mmu_circ_42398[4]。本研究在既往工作基础上,采用qPCR验证了mmu_circ_42398在动物模型组织水平表达显著低于对照组,与前期芯片结果是一致的。为了探讨mmu_circ_42398在肝纤维化进展细胞生物学机制中的作用,复制了肝细胞氧化应激损伤、巨噬细胞炎症、HSC活化肝纤维化相关的3种细胞模型,然后检测了mmu_circ_42398在其中的表达变化,结果显示其在3种细胞模型中均表达降低。接着,我们通过生物信息学分析预测其生物学功能,发现可能与HSC活化相关的信号通路有关。mmu_circ_42398具有miR-344-3p的miRNA的反应元件(microRNA response element,MRE),能靶向与之结合,从而进一步调节其下游mRNA的表达,发挥生物学功能。既往有研究发现,miR-344具有激活Wnt/β-catenin信号通路作用[8],而经典Wnt信号通路在HSCs活化中非常重要,激活β-catenin将促进HSCs的活化[9],这提示miR-344可能通过激活Wnt/β-catenin信号通路促进HSCs的活化。推测mmu_circ_42398可能调控miR-344/Wnt/β-catenin信号通路抑制HSCs的活化。
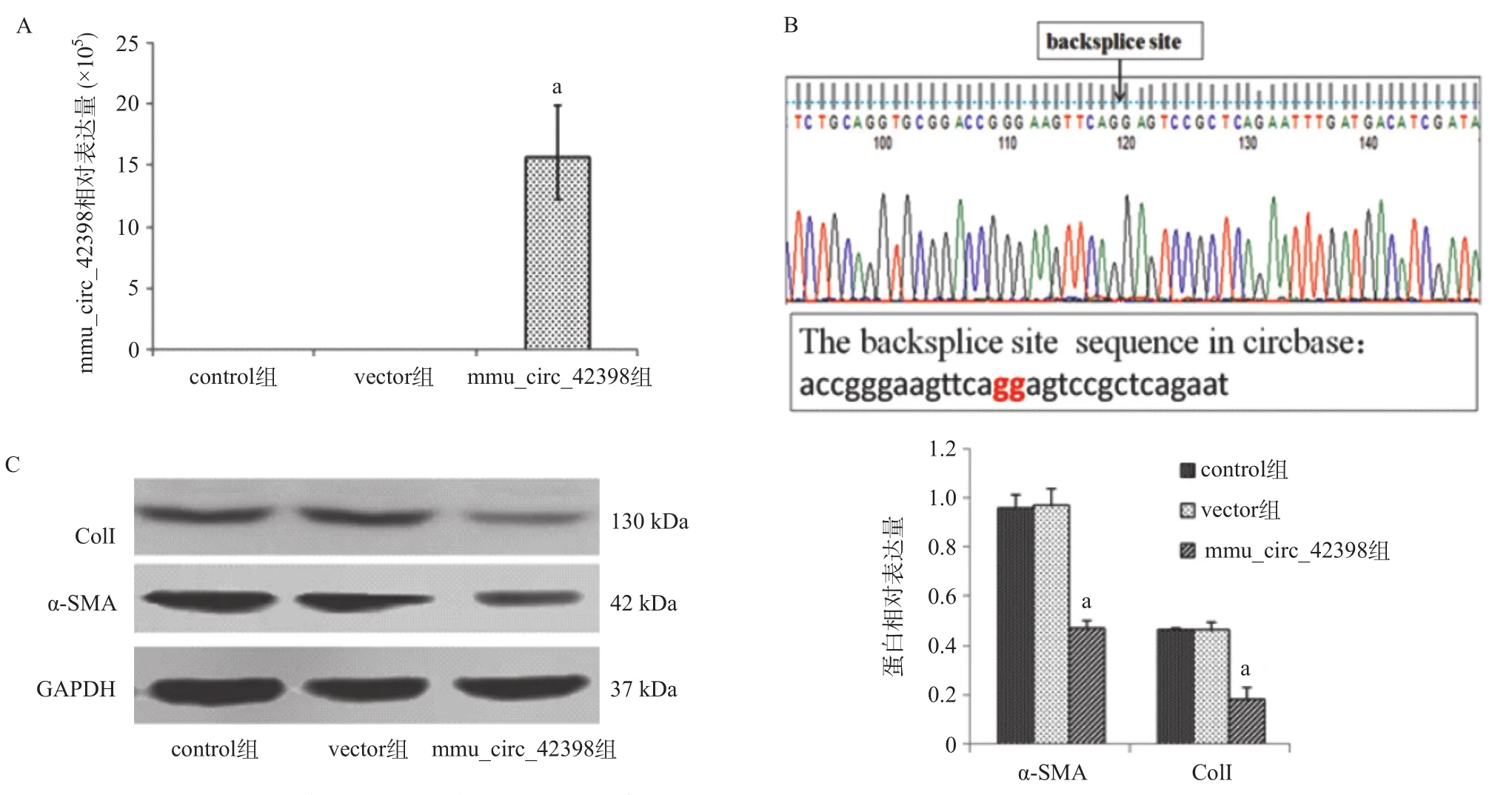
图4 mmu_circ_42398过表达对JS1细胞活化的影响
为了证实这一推测,我们构建了mmu_circ_ 42398过表达质粒载体,并将其转染到JS1细胞中,观察了HSC活检标志物α-SMA蛋白表达及ColI胶原产生的情况,结果证实mmu_circ_42398过表达能显著抑制HSC的活化。本研究通过深入研究circRNA与肝纤维化发生发展机制的关系,有助于推进肝纤维发病机制的研究及其有关治疗新靶点的发现。本研究的不足之处为尚未通过荧光素酶报告基因等技术明确mmu_circ_42398与其靶基因miR-338-3p的直接作用及其下游的信号转导通路的关系,其具体的分子机制尚不清楚,这些是未来课题组要开展的工作。

