地黄C3H基因的克隆及生物信息学分析
周延清 邵露营 郭萌萌 朱佳琳

摘 要:香豆酸-3-羟化酶属于植物中最大的蛋白酶细胞色素P450家族之一,在植物生命活动中发挥着重要作用。为了解地黄香豆酸-3-羟化酶基因RgC3H合成毛蕊花糖苷的功能,该研究基于地黄代谢组学分析获得KEGG途径中的C3H,采用多重比对在NCBI中获得同源基因的一个保守序列,并基于该保守序列和地黄SRA数据库,采用电子克隆和RT-PCR克隆技术获得地黄C3H基因全长CDS(RgC3H),对其进行生物信息学分析。结果表明:RgC3H基因全长为1 530 bp,且编码一个含509个氨基酸、分子量为57.91 kD、无信号肽的蛋白质;基于氨基酸序列的结构分析显示,RgC3H有一个保守区域-P450结构域;系统进化分析结果显示,RgC3H与芝麻和猴面花的C3H基因具有很高的同源性。上述结果为进一步研究RgC3H基因在地黄毛蕊花糖苷生物合成途径中的作用奠定了基础。
关键词:地黄,C3H基因,基因克隆,生物信息学分析
中图分类号:Q943
文献标识码:A
文章编号:1000-3142(2020)09-1281-07
Abstract:Coumarin-3-hydroxylase belongs to the cytochrome P450 family,one of the largest protease families,and plays an important role in plants. However,its role for verbascoside biosynthesis remains deficient. In order to understand the role of P-coumarate 3-hydroxylase for verbascoside biosynthesis in Rehmannia glutinosa (RgC3H),C3H was mined from a candidate KEGG pathway based on the metabolomics analysis of R. glutinosa,and a conserved sequence of homologous gene was obtained in NCBI by multiple alignment. According to its fragment and SRA database of R. glutinosa,we cloned its full length CDS (RgC3H) by electro cloning and RT-PCR,and performed its bioinformatics analyses. The results were as follows:RgC3H gene was 1 530 bp in length and encoded a 509-amino acid protein with a molecular weight of 57.91 kD and without signal peptide; According to its amino acid sequence and structural analysis,RgC3H contained one conserved domain-cytochrome P450 domain,suggesting that it belonged to cytochrome P450 family; Phylogenetic tree analysis showed that RgC3H had high homology with the C3H genes of Sesamum indicum and Erythranthe guttata. This study laid a foundation for further research on the role of RgC3H gene in the pathway of verbascoside biosynthesis in Rehmannia glutinosa.
Key words:Rehmannia glutinosa,C3H gene,gene cloning,bioinformatics
地黃是“四大怀药”之一,属于玄参科地黄属多年生草本植物,是一种重要的传统中药药材,种类繁多,具有益阴生津、清凉热血等功效。目前,有关地黄的基因不断被挖掘和分析,如地黄纤维素合酶基因(周延清等,2015)、RgTPI基因(邵露营等,2018)、RghBNG基因(张喻,2014)等,但明确相应功能的基因并不多。
香豆酸-3-羟化酶(p-coumarate 3-hydroxylase,C3H)属于细胞色素P450中的CYP98亚家族,细胞色素P450参与木质素中间物的生物合成过程中,在植物生长发育过程中发挥重要作用(贺丽虹等,2008)。目前,C3H基因已在拟南芥(Schoch et al.,2001)、慈竹(周美娟等,2012)、苎麻(袁有美等,2017)等植物中逐步被鉴定。C3H是毛蕊花糖苷等多种物质生物合成途径中的一条共有途径-苯丙素途径中的一个限速酶,地黄转录组测序挖掘出其Unigenes(Wang et al.,2017),地黄代谢组分析挖掘出C3H及其催化途径(Zhou et al.,2018)。其与毛蕊花糖苷合成相关的作用被初步证实(Wang et al.,2017)。但是,地黄全长C3H基因的克隆及生物信息学分析尚未见报道。本研究基于地黄代谢组分析挖掘的C3H,采用同源克隆、电子克隆技术结合RT-PCR的方法首次获得了RgC3H全长CDS,并对其碱基序列及其编码的蛋白质序列进行了生物信息学分析,为进一步研究其在地黄毛蕊花糖苷途径中的作用及其他功能的开发应用奠定理论基础。
1 材料与方法
1.1 材料
地黄品种温85-5块根,种植于河南师范大学实验田。获得的RgC3H基因部分保守序列:GAGTTCAGACCCGAGAGGTTTCAAGAGGAGG
ACATCGACATGAAGGGGACCGATTATCGGCTACTTCCGTTCGGGTCAGGACGGCGTATTTGCCCCGGTGCACAACTTGCTATCAACTTGGTG。RgC3H基因引物序列为(F:5′-ATGGCTATCCCTCTCCTCAT-3′,R:5′- GCAAAAGAAAAGAACCATA-3′),引物序列合成于英潍捷基(上海)贸易有限公司。琼脂糖、溴化乙锭(四川维克奇生物科技有限公司),DNA Marker(DL2 000)(宝生物生物技术公司)
1.2 方法
1.2.1 目的基因RgC3H全长CDS的获取 依据地黄代谢组学分析侯选KEGG通路挖掘出RgC3H酶(Zhou et al.,2018),并在NCBI数据库中下载与地黄亲缘关系较近物种的已知C3H的核苷酸序列进行同源比对,获得C3H部分保守序列。找到地黄相关的SRA数据库在Nucleotide Blast中输入已知的部分序列,选择高度相似的序列检索地黄SRA数据库进行电子克隆。用ORF Finder软件预测RgC3H的完整ORFs,利用软件primer primer 5.0设计用于RT-PCR的引物,并以温85-5块根的cDNA为模板,RT-PCR反应体系参照王向楠(2017)。RT-PCR 程序为95 ℃预变性5 min;95 ℃变性30 s,引物退火温度为53 ℃,退火时间为30 s,72 ℃延伸时间2 min,35 cycle;72 ℃终延伸10 min。RT-PCR结束后,进行1%琼脂糖凝胶电泳检测。产物胶回收后送至上海金唯智公司进行测序得到RgC3H基因的编码序列。
1.2.2 RgC3H的生物信息学分析 将测序得到的RgC3H完整CDS区,用NCBI在线分析工具ORF Finder查找该基因开放阅读框ORF;用ProtParam对RgC3H蛋白进行理化性质分析;用TMHMM 2.0和NetPhos 3.1 Server对RgC3H蛋白质的跨膜区和磷酸位点进行预测分析;应用Signal P-5.0对地黄RgC3H氨基酸序列进行信号肽的分析。用SOPMA及SWISS-MODEL分别对RgC3H蛋白的二级和三级结构进行分析。用NCBI的CD search和InterProScan对RgC3H蛋白的结构域进行分析。利用NCBI在线工具Blast查找与RgC3H同源性较高的序列,运用软件DNAMAN6.0进行同源性分析及采用MEGA 6.0软件构建系统进化树。
2 结果与分析
2.1 目的基因RgC3H全长CDS的获取
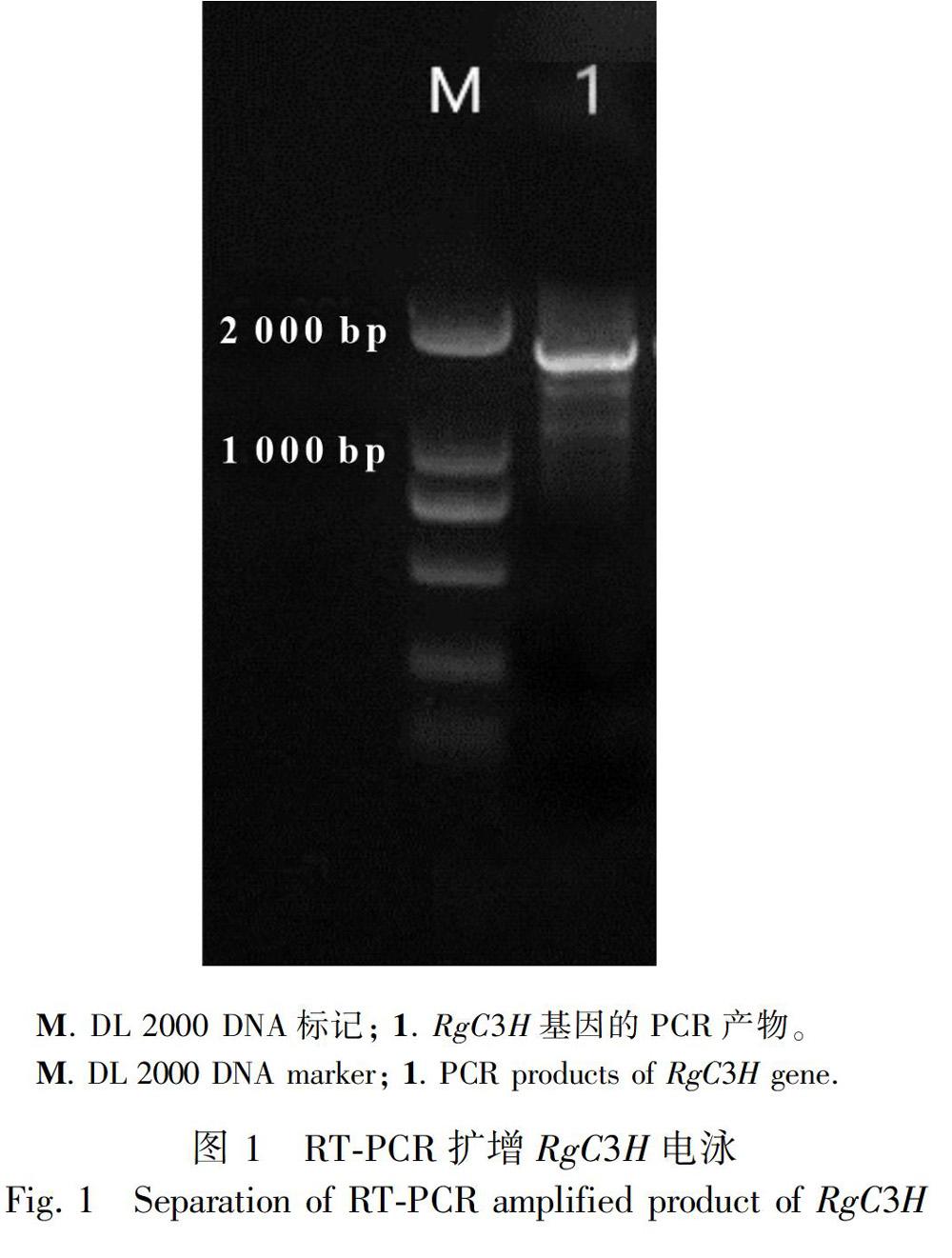
根据拼接得到的CDS序列设计引物,利用RT-PCR进行扩增,结果如图1所示。PCR产物切胶回收送至上海金唯智公司进行测序所得基因序列结果如图2所示。CDS全长为1 530 bp,编码的蛋白共有509个氨基酸残基。
2.2 RgC3H同源性分析及系统进化分析
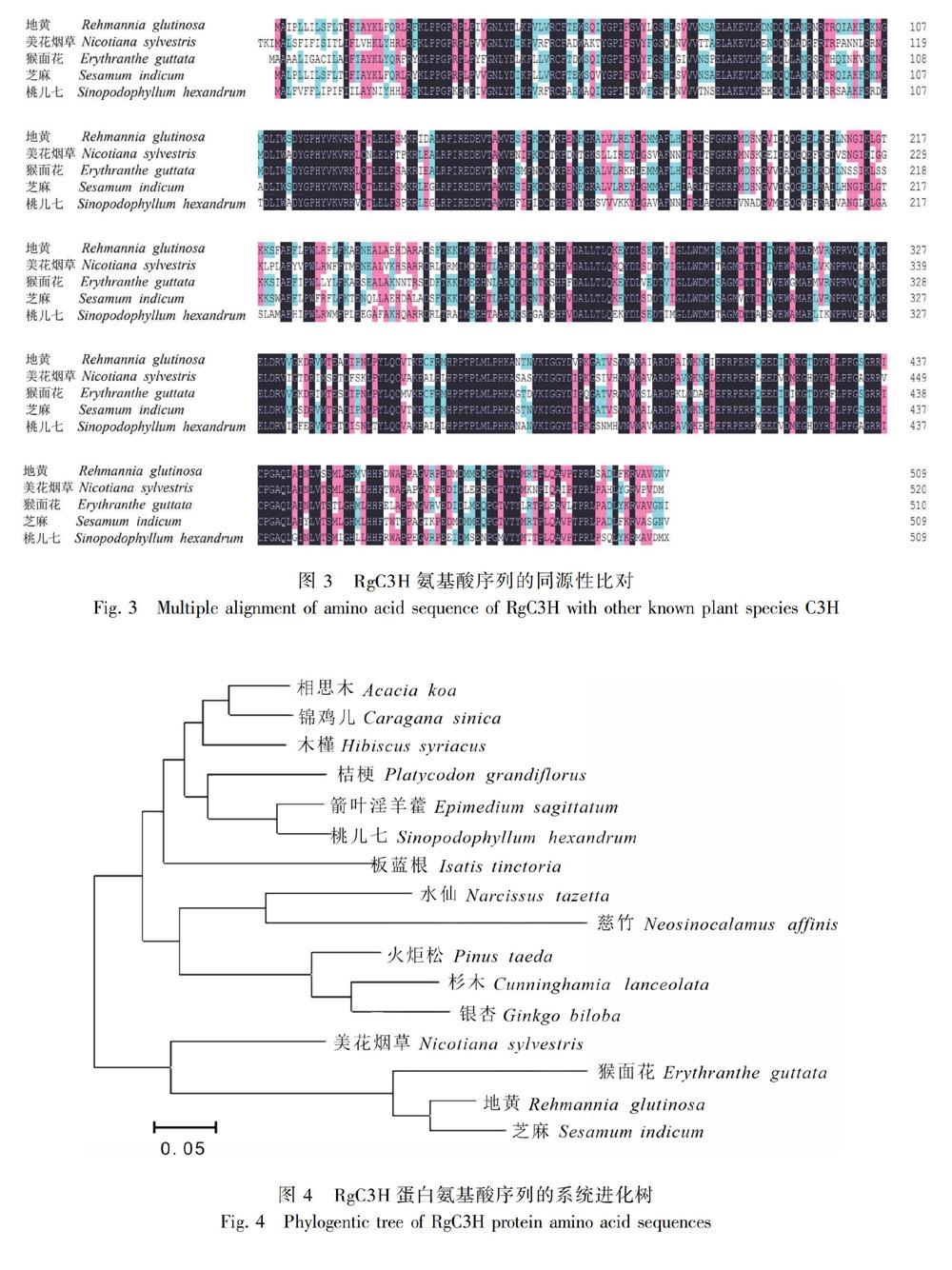
通过NCBI的Blastn搜索与RgC3H基因核苷酸序列相似性较高的物种,用DNAMAN6.0多重比对结果表明RgC3H与芝麻[Sesamum indicum(NM_001304410.1)]、猴面花[Erythranthe guttata(XM_012972625.1)]、桃儿七[Sinopodophyllum hexandrum(KC110989.1)]、美花烟草[Nicotiana sylvestris(XM_009766650.1)]等物种的相似性分别依次为91%、82%、72%和70%(图3)。
为进一步研究RgC3H在其他植物之间的进化关系,利用MEGA 6.0进行C3H蛋白系统进化树的构建结果显示,芝麻、猴面花与地黄亲缘关系最近,率先聚为一支,后与美花烟草聚为一支,与相思木(Acacia koa,KX784942.1)、锦鸡儿(Caragana sinica,HQ829858.1)、木槿(Hibiscus syriacus,JX003249.1)的亲缘关系最远(图4)。
2.3 RgC3H理化性质、功能及结构域预测
2.3.1 RgC3H理化性质 应用ProtParam对RgC3H进行蛋白理化性质分析,发现其分子式为C2608H4136N706O727S28,分子量为57 911.50,氨基酸总数为509,等电点(pI)为8.89,带负电荷的残基(Asp+Glu)总数为60,带正电荷的残基(Arg+Lys)总数为67,估计的半衰期为30 h,不稳定指数为31.11,蛋白质类型为稳定蛋白质。脂肪指数为90.22,亲水性的平均值(GRAVY)為-0.202。
2.3.2 RgC3H保守结构域预测 利用NCBI的Conserved Domain Database数据库分析结果表明RgC3H的保守结构域含有P450超家族,即属于细胞色素P450家族成员(图5)。
2.3.3 RgC3H的跨膜区和磷酸化位点分析 用TMHMM2.0对RgC3H进行分析,结果表明RgC3H没有跨膜结构,且是膜外蛋白质。用NetPhos 3.1 Server对RgC3H进行磷酸化结构预测得出,苏氨酸磷酸化位点有26个;丝氨酸磷酸化位点有20个;酪氨酸磷酸化位点有12个。
2.3.4 RgC3H信号肽分析 利用Signal P-5.0 对地黄RgC3H 氨基酸序列进行信号肽的可能性分析,结果表明,RgC3H无信号肽,可推测该蛋白没有跨膜运输功能,不是分泌型蛋白。
2.4 RgC3H二级结构、三级结构预测分析
本文应用SOPMA及SWISS-MODEL分别对RgC3H的二级和三级结构进行分析,发现其中α螺旋占最多,为50.69%,其次是无规卷曲占33.99%,延伸链占9.43%和β转角占5.89%(图6、图7)。
3 讨论与结论
地黄是一种大宗中草药,具有很高的观赏、食用及药用价值,其主要生物活性成分毛蕊花糖苷对其药效意义重大。毛蕊花糖苷具有抗炎、抗菌、抗氧化、抗肿瘤、光保护、护肝、保护神经和免疫调节等多种生物活性(Li et al.,2019)。但是,地黄毛蕊花糖苷含量很低,我国药典规定地黄毛蕊花糖苷含量不小于0.02%。因此,需要解析地黄毛蕊花糖苷生物合成途径,探索提高地黄毛蕊花糖苷含量。毛蕊花糖苷生物合成途径有一条重要的苯丙烷合成途径,与木质素合成途径具有重叠步骤,而C3H正是两者重叠途径中的限速酶(Schoch et al.,2001),且前期研究已挖掘出了地黄C3H(Wang et al.,2017;Zhou et al.,2018 )及其编码基因RgC3H的Unigene(Wang et al.,2017)。但是,没有克隆RgC3H全长基因和深入研究其合成毛蕊花糖苷的作用。
本研究基于地黄代谢组分析挖掘出C3H,采用同源克隆、电子克隆技术结合RT-PCR的方法首次获得了RgC3H全长CDS,并对其碱基序列及其编码的蛋白质的氨基酸序列进行了生物信息学分析,这些结果体现了我们建立的基因克隆技术具有综合性、先进性和有效性;RgC3H测序结果为1 530 bp,生物信息学分析表明其编码509个氨基酸,具有C3H酶类的保守P450结构域,这与前人从慈竹、苎麻、石榴等植物中克隆的C3H基因在基因大小、氨基酸残基个数等方面的结果一致(周美娟等,2012;袁有美等,2017;熊枫等,2018);RgC3H在氨基酸水平上与多种植物的C3H具有较高的同源性,且系统进化树结果显示,地黄RgC3H基因与芝麻(NM_001304410.1)和猴面花(XM_012972625.1)的C3H基因相似性分别高达91%和82%,这与他们同属于管状花目植物的分类地位相一致;采用SOPMA预测RgC3H蛋白二级结构表明该蛋白中主要由α螺旋和无规卷曲组成,延伸链和β转角相对较少;经信号肽预测分析得出该蛋白是一种膜外蛋白质,无信号肽,没有跨膜运输功能。
综上所述,本研究成功克隆并分析了地黄RgC3H基因,证明其属于植物C3H基因家族。本研究工作的开展,扩大了已知地黄基因数据,为下一步揭示RgC3H在地黄毛蕊花糖苷生物合成途径中的作用提供了有价值的基础数据。但是,RgC3H合成地黄毛蕊花糖苷的真正作用有待进一步深入研究。
参考文献:
ALIPIEVA K,KORKINA L,ORHAN IE,et al.,2014. Verbascoside—A review of its occurrence,(bio)synthesis and pharmacological significance [J]. Biotechnol Adv,32:1065-1076.
HE LH,ZHAO SJ,HU ZB,2008. Gene and function research progress of plant cytochrome p450s [J]. Pharm Biotechnol,15(2):142-147. [賀丽虹,赵淑娟,胡之壁,2008. 植物细胞色素p450基因与功能研究进展 [J]. 药物生物技术,15(2): 142-147.]
KIM YS,RYUK JA,KO BS,2012. Discrimination of Korean Rehmannia glutinosa from Chinese Rehmannia glutinosa using sequence-characterized amplified region marker [J]. J Korean Soc Appl boil,55(1):1-6.
LI G,YANG Q,ZHANG Y,et al.,2013. Cloning and sequence analysis of the ckc3h gene from Caragana korshinskii kom and preliminary studies of its function [J]. China Biotechnol,33(4):61-67.
LI WT,DENG RX,JING XS,et al.,2019. Acteoside ameliorates experimental autoimmune encephalomyelitis through inhibiting peroxynitrite-mediated mitophagy activation [J]. Free Radical Biol Med. DOI:10.1016/j.freeradbiomed.2019.10.408.
SCHOCH G,GOEPFERT S,MORANT M,et al.,2001.CYP98A3 from Arabidopsis thaliana is a 3′-hydroxylase of phenolic esters,a missing link in the phenylpropanoid pathway [J]. J Biol Chem,276(39):36566-36574.
SHAO LY,ZHOU YQ,YANG K,et al.,2018. Cloning and spatial expression of Triosephosphateisomerase gene in Rehmannia glutinosa [J]. J Henan Agric Sci,47 (11):50-55. [邵露营,周延清,杨珂,等,2018. 地黄磷酸丙糖异构酶基因的克隆与空间表达 [J]. 河南农业科学,47(11):50-55.]
WANG FQ,ZHI JY,ZHANG ZY,et al.,2017. Transcriptome analysis of salicylic acid treatment in Rehmannia glutinosa hairy roots using RNA-seq technique for identification of genes involved in acteoside biosynthesism [J]. Front Plant Sci,8(17):787-801.
WANG XN,2017. Mining and identification of verbascoside biosynthesis associated genes based on transcriptome sequencing of Rehmannia glutinosa [D]. Xinxiang:Henan Normal University:27-45. [王向楠,2017. 基于转录组测序的地黄毛蕊花糖苷生物合成相关酶基因的发掘与鉴定 [D]. 新乡:河南师范大学:27-45.]
XIONG F,CHEN L,LIU TR,et al.,2018. Cloning and expression analysis of coumarate-3-hydroxylase C3H gene in Punica granatum [J]. Mol Plant Breed,16(23):7628-7633. [熊枫,陈磊,刘同瑞,等,2018. 石榴对香豆酸-3-羟基化酶C3H基因的克隆和表达分析 [J]. 分子植物育种,16(23):7628-7633.]
XIU ZM,PENG XP,CHEN JH,2017. Effects of down-regulated expression of Poplar lignin synthesis genes C3H and HCT on cell well spatial morphology [J]. J Shandong Sci Technol,47(1):37-40. [修则明,彭霄鹏,陈建辉,2017. 杨树木质素合成基因C3H与HTC表达下调对细胞壁空间形态的影响 [J]. 山东林业科技,47(1):37-40.]
YUAN YM,CHEN JR,LIU F,et al.,2017. Gene cloning,bioinformatics and expression analysis on C3H gene from Boehmeria nivea L. [J]. Chin Agric Sci Bull,33(32):21-27. [袁有美,陈建荣,刘芳,等,2017. 苎麻C3H基因的克隆、生物信息学分析及表达分析 [J]. 中国农学通报,33(32): 21-27.]
ZHANG DD,2018. Discovery and espressions of key verbascoside biosynthesis and glycolysis-associated enayme genes in Rehmannia glutinosa [D]. Xinxiang:Henan Normal University:25-28. [张丹丹,2018. 地黄毛蕊花糖苷生物合成和糖酵解途径相关关键酶基因的挖掘和表达分析 [D]. 新乡:河南师范大学:25-28.]
ZHANG Y,2014. Cloning and bioinformatics analysis of RghBNG-like gene in Rehmannia glutinosa [D]. Xinxiang:Henan Normal University:29-35. [張喻,2014. 地黄RghBNG基因的结构分析和功能验证 [D]. 新乡:河南师范大学:29-35.]
ZHOU MJ,HU SL,CAO Y,et al.,2012. Cloning and bioinformation analysis of C3H gene in Neosinocalamus affinis [J]. Bull Bot Res,32(1):38-46. [周美娟,胡尚连,曹颖,等,2012. 慈竹C3H基因克隆及其生物信息学分析 [J]. 植物研究,32(1):38-46.]
ZHOU YQ,YANG K,ZHANG DD,et al.,2018. Metabolite accumulation and metabolic network in developing roots of Rehmannia glutinosa reveals its root developmental mechanism and quality [J]. Sci Rep,8(1):14127-14137.
ZHOU YQ,WANG XN,WANG WS,et al.,2016. De novo transcriptome sequencing-based discovery and expression analyses of verbascoside biosynthesis-associated genes in Rehmannia glutinosa tuberous roots [J]. Mol Breed,36(10):139-149.
ZHOU YQ,ZHANG Y,LI JY,et al.,2015. Cloning and bioinformatics analysis of cellulose synthase gene in Rehmannia glutinosa [J]. Jiangsu Agric Sci,1(43):21-24. [周延清,张喻,李静云,等,2015. 地黄纤维素合酶基因的克隆及生物信息学分析 [J]. 江苏农业科学,1(43):21-24.]
(责任编辑 何永艳)

