基于SSR的光敏型饲草高粱分子辅助育种体系
牛皓,平俊爱,王玉斌,张福耀,吕鑫,李慧明,楚建强
基于SSR的光敏型饲草高粱分子辅助育种体系
牛皓1,2,平俊爱1,2,王玉斌1,2,张福耀1,2,吕鑫1,2,李慧明1,2,楚建强1,2
(1山西农业大学(山西省农业科学院)高粱研究所/高粱遗传与种质创新山西省重点实验室,山西晋中 030600;2农业部黄土高原作物基因资源与种质创制重点实验室,太原 030031)
【】利用分子标记技术对光敏型饲草高粱新品种传统选育方法进行改良,构建分子辅助育种体系,从而提高品种选育效率,降低育种成本,为形成新的育种体系奠定理论基础。以光敏型(不抽穗)饲草高粱品种晋光1R去雄后为母本、非光敏型(抽穗)饲草高粱BMRC-3-2为父本进行杂交,构建F2群体。按照光敏与非光敏性状将群体分为两类,各选30株,取叶片提取DNA,利用微卫星分子标记技术(SSR)和集团分离分析法(BSA)对晋光1R/BMRC-3-2的F2群体进行光敏感基因定位分析,以及特异性SSR引物的设计、筛选;将筛出的特异性引物对F1杂交种(以晋光1R为亲本选育的高代稳定恢复系作为父本与其他不育系测配得到的F1代杂交种)进行光敏性鉴定,通过与田间表型对照,确定最终目标引物,从而构建新的光敏型饲草高粱新品种选育体系。经过SNP index分析,选取99%阈值线最高点前后约250 kb的区域作为性状相关的候选区域,区域总长度为500 kb,共400个SNP位点,对这些SNP位点注释,其中非同义突变或者stop-gain或者stop-loss的SNP位点有6个,最终确定2个候选基因与高粱光敏感性相关,这两个基因位于高粱第7染色体。利用微卫星标记技术开发引物51对,对光敏和非光敏的亲本及F1杂交种进行毛细管电泳检测,选定1对特异性引物70.2-3,结果表明,以251 bp处出现“单峰”为判断依据,引物70.2-3对50个非光敏型F1杂交种的鉴选准确率达到100%,所有材料均在251 bp附近出现峰值。以214和251 bp两处附近出现“双峰”为判断依据,该引物对另外50个光敏型F1杂交种鉴选的准确率达到90%,其中F1-69、F1-70、F1-71这3个材料在214和262 bp附近出现“双峰”;F1-81仅在251 bp处有“单峰”,而F1-86则在251和232 bp两处附近出现“双峰”。发现了控制高粱光敏性的候选基因和,位于高粱第7染色体810 000—1 310 000 bp,获得特异性引物70.2-3,可在一定程度上改良传统育种手段,用实验室验证替代田间测交观察,节省了育种成本,提高了育种效率。
饲草高粱;光敏型;分子标记
0 引言
【研究意义】饲草高粱是中国主要青贮饲料,由于其再生能力强、产量高、蛋白质丰富、易消化等特点,被广泛应用于农牧饲喂及渔业养殖领域[1-2]。光敏感特性是影响饲草高粱产量和品质的主要因素[3-4]。在北方夏季长日照地区,当光照时长超过12.3 h时,具有光敏感特性的饲草高粱不抽穗结实,只进行营养生长,其高度可超过4 m,部分材料在新疆地区可长至6 m,因而产量、蛋白质含量比普通饲草高粱高,若部分替代玉米作为哺乳期奶牛饲料,在增加牛奶乳脂肪含量上有显著效果[5-9]。然而,在光敏型饲草高粱品种选育的过程中,稳定后代是否具有光敏特性,必须将其与普通的不育系测交,第二年种植于大田中,秋天观察抽穗性状才能判断,选育过程中物力、人力、地力投入较多,育种成本非常高,因此,利用分子标记辅助育种技术对传统育种方法进行改良,提高育种效率显得尤为重要。【前人研究进展】生物质产量是将木质素转化为生物燃料或生物能源的生物质或生物能源作物最重要的属性之一,生长期是生物质产量的首要决定因素。因此,假设环境条件允许产量潜力得以发挥,光敏型高粱积累最多的生物质,可达到普通高粱的2倍以上[10]。目前,已知与高粱光敏性相关的基因共有6个,为Ma—Ma[10-11]。早在1945年,美国学者Quinby等[12]认为有3个基因影响高粱的花期和成熟期,分别为Ma、Ma和Ma。Ma、Ma在没有Ma的情况下不会表达,Ma在显性Ma存在的情况下也不会表达。同时Ma的作用受光周期的影响,开花时间控制着叶片的数量、生长时间和植株的最终大小。1965年,Quinby[13-14]又发现了第4个与高粱成熟期相关的基因,并将它命名为Ma,认为这4个基因的表达受环境的影响,特别是受光周期和温度的影响,且花期开始的早晚取决于品种的成熟基因型。20世纪90年代,Morgan等[11]、Major等[15]从环境、温度等因素对这一概念又进行了阐述,之后有关高粱光敏基因的研究丰富起来。Childs等[16]认为Ma是第一个被克隆的与高粱成熟期相关的基因,并且有3个等位基因分别为Ma、ma、ma。Rooney等[17-18]和Morgan等[19]将高粱恢复系EBA-3去雄后与其他非光敏(photo-insensitive)恢复系杂交,发现8个F2群体中PS/PI分离比率为9﹕7,表明2个独立的基因位点在互补显性上位中相互作用,而这两个与光周期敏感度相关的基因与先前的Ma、Ma、Ma和Ma不同,从而定名为Ma和Ma,并发明了基于Ma/Ma系统的生物质能源高粱的选育方法。Murphy等[20]认为蛋白PRR37控制高粱的开花时间。Yang等[21-22]认为高粱CONSTANS是一种开花的激活剂,在开花抑制剂PRR37的长时间内在转录后受到抑制,有助于光敏感型短日照高粱的开花。Wang等[23]对Ma的进化做了分析。另外,针对饲草高粱生物产量与光敏感特性的研究,Wolabu等[24-25]认为不同类型的高粱在温带地区的应用主要取决于其开花时间的控制,光周期不敏感的高粱用于粮食生产,而光周期敏感基因型则用于饲料和生物量原料生产。关于光敏感基因定位的研究,Murphy等[20]将Ma定位在高粱第6染色体上的标记Xtxi62和Xtxi58之间约86 kb的区段内。Mullet等[26]将Ma定位在高粱第2染色体67 923 811—68 393 290 bp,认为该基因可能通过抑制由光敏色素B和C介导的调节高粱光照依赖性产物来调控高粱的开花时间。Ma是第一个被成功克隆的调控高粱感光性的基因,被定位于高粱第1染色体长臂的末端,在60 910 479—60 917 763 bp。Ma至少有3种等位基因,分别为显性Ma、隐性Ma和一种特殊的隐性基因Ma[27-29]。Ma被定位在高粱第10染色体,与RFLP标记txs1163相邻[30-31]。Ma被定位到高粱第1染色体6 762 743—6 767 650 bp,在遗传图谱上的位置为23—26 cM,对应的基因编码光敏色素C[26]。Ma被定位在高粱第6染色体39 379 760—42 610 705 bp,它和Ma均受Ma、Ma的遗传上位性影响,通过抑制一系列成花素基因的表达,使高粱在长日照条件下开花时间延迟。Ma是调控高粱籽粒数量、株高和抽穗期的基因——[26]。牛皓等[32]对144对SSR引物(包括PRR37(Ma)、GHD7(Ma)、PHYB(Ma)、PHYC(Ma)等基因对应的引物)进行筛选,筛出4对疑似光敏感引物,但经过与F1杂交种扩大群体验证,发现它们并不是特异性引物,不能将光敏和非光敏的材料区分开,因而不具备生产使用价值。【本研究切入点】高粱光敏感基因的理论研究丰富,但利用已有成果形成育种体系应用于田间育种的研究鲜有报道。【拟解决的关键问题】本研究以具有光敏感特性(photosensitive)的饲草高粱恢复系晋光1R和普通非光敏(photo-insensitive)饲草高粱恢复系BMRC-3-2为研究对象,通过杂交构建F2群体,利用微卫星分子标记技术,定位光敏基因,筛选特异性引物,为今后改良高粱品种传统育种方式,提高育种效率,降低育种成本,进而形成新的育种体系奠定理论基础。
1 材料与方法
1.1 试验材料
亲本材料:具有光敏基因的饲草高粱恢复系晋光1R,普通饲草高粱恢复系BMRC-3-2。
F2群体:2017年,在山西榆次以光敏恢复系晋光1R为母本,非光敏恢复系BMRC-3-2为父本,经人工去雄有性杂交得到F0种子;2017年冬,在海南三亚种植F0种子,并自交,得到F1种子,2018年,获得晋光1R/BMRC-3-2的F2群体。
F1杂交种:以晋光1R为亲本选育的高代稳定恢复系作为父本,与其他不育系测配得到的F1杂交种。
部分SSR标记序列来源于NCBI网站,部分由上海派森诺生物科技有限公司依据候选基因序列设计合成。
材料种子由山西省农业科学院高粱研究所提供。所有试验材料均种植于山西省农业科学院高粱研究所东白试验基地。田间管理与其他普通高粱一致,2018年8月底调查抽穗情况。
1.2 表型鉴定及遗传分析
取样前,对亲本及群体单株进行光敏和非光敏性状调查统计。根据表型鉴定结果计算分离比例,并进行卡方检测,确定光敏性状在F2群体中的分离情况,用于遗传分析。
1.3 DNA提取及近等基因池的构建
为确保取样的准确性,在8月底高粱抽穗尾期,按照光敏(不抽穗)与非光敏(抽穗)将F2分两类,每类随机各选30株,分别挂牌标记,取顶部新鲜幼叶放入2 ml离心管中,液氮速冻,由上海派森诺生物科技有限公司提DNA及进行后续相关试验。同时,将1.1内容中F1杂交种,也按照光敏与非光敏分两类,每类随机各取50株顶端新鲜幼叶放入2 ml离心管中,液氮速冻,由上海派森诺生物科技有限公司进行微卫星标记验证试验。
1.4 BSA试验
构建基于Illumina HiSeq测序平台,利用第二代测序技术(next-generation sequencing,NGS),对插入片段为400的文库进行双末端(paired-end,PE)测序。通过对目标性状区域分析,确定基因位置。
1.5 微卫星标记验证试验
将F1杂交种DNA进行毛细管电泳检测,验证特异性引物。
2 结果
2.1 表型鉴定和遗传分析
晋光1R与BMRC-3-2杂交,对F2进行性状调查,其中,251株不抽穗,169株抽穗,经c2检验光敏与非光敏(PS/PI)的分离比例符合9﹕7(表1)。
2.2 基因初步定位及功能注释
经过测序,选取99%阈值线最高点前后约250 kb的区域作为性状相关的候选区域,区域总长度为500 kb,这个范围内满足条件的SNP位点共400个,对这些SNP位点注释,其中,非同义突变或者stop-gain或者stop-loss的SNP位点有6个,分布在4个基因。关联区域位于第7染色体810 000—1 310 000 bp(图1)。

表1 F2光敏性状的遗传模式
c20.05,1=3.84

图1 子代混池 SNP index和△(SNP index)在染色体上的分布
4个候选基因分别为、、和。通过与GO、KEFG、Swiss Prot、NR等数据库比对注释,其中和都是剪切因子基因,因而,认为它们可能就是与饲草高粱光敏感相关的基因。
2.3 特异性SSR引物的获得及分子辅助育种体系的建立
在F1材料的DNA中选取光敏与非光敏单株各2株,根据候选基因序列设计合成的引物共51对,进行荧光定量PCR,每组引物前2条带为非光敏F1,后2条带为光敏F1(图2)。
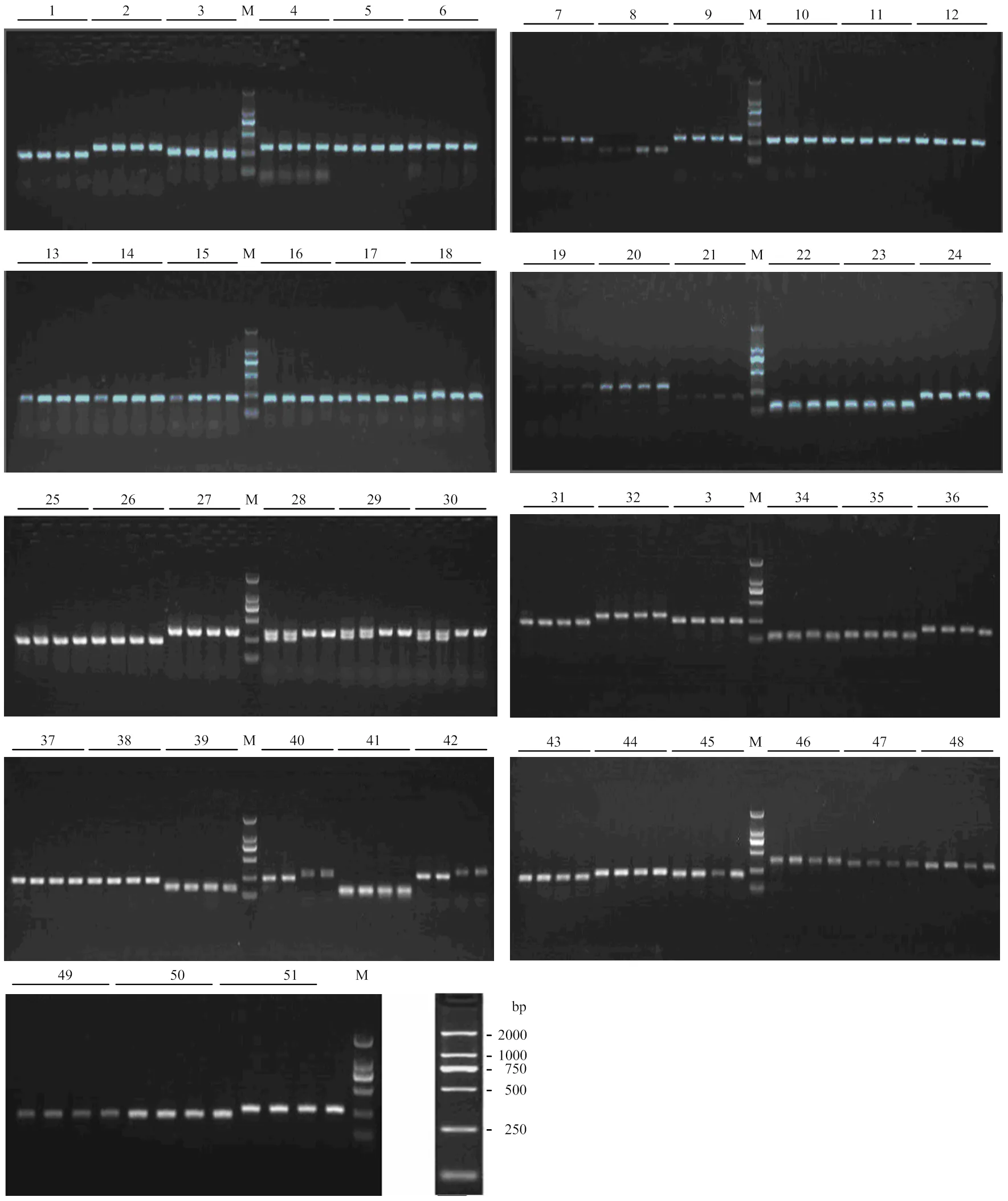
M:DL2000 DNA Maker;1—51:70.2-1、70.2-2、70.2-3、70.2-152-1、70.2-152-2、70.2-152-3、71.2-229-1、71.2-229-2、71.2-229-3、72.2-1、72.2-2、72.2-3、73.2-1、73.2-2、73.2-3、75.2-833-1、75.2-833-2、75.2-833-3、75.2-392-1、75.2-392-2、75.2-392-3、75.2-384-1、75.2-384-2、75.2-384-3、75.2-036-1、75.2-036-2、75.2-036-3、75.2-258-1、75.2-258-2、75.2-258-3、77.2-751-1、77.2-751-2、77.2-751-3、77.2-050-1、77.2-050-2、77.2-050-3、78.2-409-1、78.2-409-2、78.2-409-3、78.2-005-1、78.2-005-2、78.2-005-3、79.2-488-1、79.2-488-2、79.2-488-3、79.2-924-1、79.2-924-2、79.2-924-3、79.2-797-1、79.2-797-2、79.2-797-3
根据图2结果,将个别差异极不明显的引物排除,其余引物扩大样本至100个F1材料进行毛细管电泳验证。结果表明,仅有引物70.2-3可以区分光敏与非光敏F1材料(图3)。非光敏型F1杂交种在251 bp 附近出现“单峰”;而光敏型则在214和251 bp两处附近出现“双峰”;光敏亲本和非光敏亲本则分别在214和251 bp附近出现“单峰”。以251 bp处出现“单峰”为判断依据,引物70.2-3对50个非光敏型F1杂交种的鉴选准确率达到100%,所有材料均在251 bp附近出现峰值。以214和251 bp两处附近出现“双峰”为判断依据,该引物对另外50个光敏型F1杂交种鉴选的准确率达到90%,其中F1-69、F1-70、F1-71这三个材料在214和262 bp附近出现“双峰”;F1-81仅在251 bp处有“单峰”而F1-86则在251和232 bp两处附近出现“双峰”。据此,认为所选引物70.2-3为特异性引物,可以区分光敏和非光敏杂交种,可以作为田间生产指导。其序列为FORWARD PRIMER:5′-CCTCCTCTTCCTCGGATAGC-3′和REVERSEPRIMER:5′-ATGATCGGTGGT TG GAAGAG-3′。

Sample1和Sample2为非光敏型F1杂交种,Sample3和Sample4为光敏型F1杂交种,Sample5为光敏亲本晋光1R,Sample6为非光敏亲本BMRC-3-2
综上所述,对传统的光敏感饲草高粱选育体系进行改良,利用实验室筛选代替田间测交,形成新的分子辅助育种体系(图4)。

图4 新旧育种体系对比示意图
3 讨论
3.1 传统选育方式
光敏型饲草高粱恢复系的选育,以传统高粱恢复系选育方法为基础,选择具有光敏特性的饲草高粱恢复系与普通非光敏但具有其他优良性状的饲草高粱恢复系人工去雄有性杂交,获得F0种子,通过南繁加代也可下一年种植大田得到F1,F1套袋自交后第二年得到F2群体。从这一步开始传统的育种方法需另选不育系A与选出的F2群体单株一一测交,得到杂交种T,翌年田间种植后再观察抽穗情况,从而判定上年测交F2群体中哪株为光敏型。
3.2 分子辅助育种选育方式
分子辅助育种选育方式与传统育种方式在获得F2群体的方法上保持一致,但将工作量最繁琐的田间测交验证这一步用实验室荧光定量PCR方法取代,利用筛选的特异性SSR引物70.2-3将所有F2群体中的个体进行毛细管电泳检测,观察214和251 bp处的“峰值”情况,两处均出现“峰”则判定为光敏型,非光敏型则淘汰。结果表明,该引物筛选光敏材料准确度非常高,但仍有极少部分材料出现峰值位移,这与部分“中间型”材料取样分类误差有关。同时,候选基因和定位在第7染色体,与目前所有已知的与高粱光敏感性相关的基因位置都不同,是否是新的光敏基因,其调控机理如何还需进一步研究确定,但F2群体中光敏与非光敏(PS/PI)分离比例为9﹕7,这说明饲草高粱光敏感特性是由2个独立的互补显性基因控制,这与Rooney[17]研究结果相同。
3.3 分子辅助育种体系优点
本研究创造的光敏型饲草高粱分子辅助育种体系是对传统育种体系的改良,优点主要体现在:1)节约育种时间;2)节省育种成本。通过对比可发现,传统的育种体系比分子辅助育种体系的育种年限要多1年,而多出来的这一年必然会引起育种成本的增加。假设F2群体共200株,每株均与不育系A测交,收获测交种子200份,第二年以每15 m2小区种6行,每2行种1个材料来计算,大约需要67个小区0.1 hm2,以2019年每公顷地育种成本来计算,需约6 000元,而实验室荧光定量PCR耗材费用不超过2 000元。如果F2群体数量庞大,后者将节省更多的育种成本。
综上所述,调控高粱抽穗期性状的基因很多,而且等位基因也很丰富,并且影响不同杂交组合产生后代的抽穗期性状的QTL位点各不相同,调控高粱抽穗期性状的基因之间互作方式也是多种多样。因此,尽可能多的发掘调控高粱抽穗期性状的基因位点,明确基因功能及互作模式,对于揭示高粱光敏感特性的机理、提高育种效率、节约育种成本有着重要的意义。
4 结论
发现控制高粱抽穗期的候选基因和,位于高粱第7染色体810 000— 1 310 000 bp。同时特异性SSR引物70.2-3的获得,在饲草高粱光敏性鉴定上有极高的准确率,基于该引物构建的光敏型饲草高粱选育体系,利用实验室验证替代田间测交观察,节省育种成本,提高育种效率。
[1] 平俊爱, 谢国强, 张福耀, 何余湧, 吴志勇, 王荣民, 吕鑫, 杜志宏, 李慧明, 牛皓, 王玉斌. 晋牧1号在南方红壤区的产草量: 营养价值及饲养效果研究. 安徽农业科学, 2018, 46(35): 94-96.
PING J A, XIE G Q, ZHANG F Y, HE Y Y, WU Z Y, WANG R M, Lü X, DU Z H, LI H M, NIU H, WANG Y B. Breeding and characteristic ofבJinmu1’., 2018, 46(35): 94-96. (in Chinese)
[2] 平俊爱, 张福耀, 牛皓,杨慧勇, 吕鑫, 杜志宏, 李慧明, 王玉斌. 基于SSR标记的饲草高粱种质资源遗传多样性分析. 分子植物育种, 2018, 16(14): 4663-4670.
PING J A, ZHANG F Y, NIU H, YANG H Y, Lü X, DU Z H, LI H M, WANG Y B. Genetic diversity analysis of germplasm resources of forage sorghum based on SSR marker., 2018, 16(14): 4663-4670. (in Chinese)
[3] Bhosale S U, Benjamin S, Rattunde H F W, Weltzien E, Haussmann B I, Haussmann C T, Ramu P, Cuevas H E, Paterson A H, Melchinger A E, Parzies H K. Association analysis of photoperiodic flowering time genes in west and central[(L.) Moench]., 2012, 12: 32-41.
[4] Casto A L, Mattison A J, Olson S N, Thakran M, Rooney W L, Mullet J E. Maturity2, a novel regulatorof flowering time in, increases expression of SbPRR37 and SbCO in long days delaying flowering., 2019, 14(4): e0212154.
[5] Sukumaran S, Li X, Li X R, Zhu C S, Bai G H, Perumal R, Tuinstra M R, Prasad P V V, Mitchell S E, Tesso T T, Yu J. QTL mapping for grain yield, flowering time, and stay-green traits in sorghum with genotyping-by-sequencing markers., 2016, 56(4): 1429-1442.
[6] Tuinstra M R, Grote E M, Goldsbrough P B, Ejeta G. Genetic analysis of post-flowering drought tolerance and components of grain development in(L.) Moench., 1997, 3(6): 439-448.
[7] Spindel J E, Dahlberg J, Colgan M, Hollingsworth J, Sievert J, Staggenborg S H, Hutmacher R, Jansson C, Vogel J P. Association mapping by aerial drone reveals 213 genetic associations for Sorghum bicolor biomass traits under drought., 2018, 19: 679-696.
[8] Wang Y H, Upadhyaya H D, Burrell A M, Sahraeian S M, Klein R R, Klein P E. Genetic structure and linkage disequilibrium in a diverse, representative collection of the C4 model plant., 2013, 3(5): 783-793.
[9] Harper M T, Oh J, Giallongo F, Lopes J C, Roth G W, Hristov A N. Using brown midrib 6 dwarf forage sorghum silage and fall-grown oat silage in lactating dairy cow rations., 2017, 100(7): 5250-5265.
[10] Mullet J E, Rooney W L, Klein P E, Bryan D M, Bryan R M, Brady J A. Discovery and utilization of sorghum genes (Ma5/Ma6). 13/744405[P].2013-09-12.
[11] Morgan P W, Finlayson S A, Lee I J, Childs K L, He C J, Creelman R A, Drew M C, Mullet J E. Regulation of circadianly rhythmic ethylene production by phytochrome b in sorghum., 1997(1): 105-111.
[12] Quinby J R, Karper R E. The inheritance of three genes that influence time of floral initiation and maturity date in Milo1., 1945(11): 916-936.
[13] Quinby J R. Fourth maturity gene locus in sorghum., 1965(6): 516-518.
[14] Quinby J R. The maturity genes of sorghum., 1967(19): 267-305.
[15] Major D J, Rood S B, Miller F R. Temperature and photoperiod effects mediated by the sorghum maturity genes., 1990, 30(2): 305-310.
[16] Childs K L, Lu J L, Mullet J E, Morgan P W. Genetic regulation of development in: X. Greatly attenuated photoperiod sensitivity in a phytochrome-deficient sorghum possessing a biological clock but lacking a red light-high irradiance response., 1995, 108(1): 345-351.
[17] Rooney W L, Jürg B, Bean B, Mullet J E. Designing sorghum as a dedicated bioenergy feedstock., 2007, 1(2): 147-157.
[18] Rooney W L, Aydin S. Genetic control of a photoperiod-sensitive response in(L.) Moench., 1999, 39(2): 397-400.
[19] Morgan P W, Finlayson S A, Childs K L, Mullet J E, Rooney W L. Opportunities to improve adaptability and yield in grasses., 2002, 42(6): 1791-1799.
[20] Murphy R L, Klein R R, Morishige D T, Brady J A, Rooney W L, Miller F R, Dugas D V, Klein P E, Mullet J E. Coincident light and clock regulation of pseudo response regulator protein 37 (PRR37) controls photoperiodic flowering in sorghum., 2011, 108(39): 16469-16474.
[21] Yang S S, Weers B D, Morishige D T, Mullet J E. CONSTANS is a photoperiod regulated activator of flowering in sorghum., 2014, 14(1): 148-162.
[22] Yang S S, Murphy R L, Morishige D T, Klein P E, Rooney W L, Mullet J E. Sorghum phytochrome B inhibits flowering in long days by activating expression of SbPRR37 and SbGHD7, repressors of SbEHD1, SbCN8 and SbCN12., 2014, 9(8): e105352.
[23] Wang Y, Tan L B, Fu Y C, Zhu Z F, Liu F X, Sun C, Cai H W. Molecular evolution of the sorghum maturity gene Ma3., 2015, 10(5): e0124435.
[24] Wolabu T W, Tadege M. Photoperiod response and floral transition in sorghum., 2016, 11(12): e1261232.
[25] Wolabu T W, Zhang F, Niu L F, Kalve S, Mathur P B, Muszynski M G, Tadege M. Three FLOWERING LOCUS T-like genes function as potential florigens and mediate photoperiod response in sorghum., 2016, 210(3): 946-959.
[26] Mullet J E, Rooney W L. Method for production of sorghum hybrids with selected flowering times. 13/886130[P].2016-08-30.
[27] Childs K L, Miller F R, Cordonnier Pratt M M, Pratt L H, Morgan P W, Mullet J E. The sorghum photoperiod sensitivity gene, Ma3encodes a phytochrome B., 1997, 113(2): 611-619.
[28] Childs K L, Cordonnier Pratt M M, Pratt L H, Morgan P W. Genetic regulation of development in Sorghum bicolor: VII. Ma3Rflowering mutant lacks a phytochrome that predominates in green tissue., 1992, 99(2): 765-770.
[29] Childs K L, Pratt L H, Morgan P W. Genetic regulation of development in: VI. The Ma3Rallele results in abnormal phytochrome physiology., 1991, 97(2): 714-719.
[30] Hart G E, Schertz K F, Peng Y, Syed N H. Genetic mapping of(L.) moench QTLs that control variation in tillering and other morphological characters., 2001, 103(8): 1232-1242.
[31] Chantereau J, Trouche G, Rami J F, Deu M, Barro C, Grivet L. RFLP mapping of QTLs for photoperiod response in tropical sorghum., 2001, 120(2):183-194.
[32] 牛皓, 平俊爱, 张福耀, 吕鑫, 杜志宏, 李慧明, 王玉斌. 基于SSR的高粱光敏特性分析. 安徽农业科学, 2018, 46(35): 103-105.
NIU H, PING J A, ZHANG F Y, Lü X, DU Z H, LI H M, WANG Y B. Analysis on the photosensitivity of sorghum based on SSR., 2018, 46(35): 103-105. (in Chinese)
Molecular Aided Breeding System of Photosensitive Forage Sorghum Based on SSR
NIU Hao1,2, PING JunAi1,2, WANG YuBin1,2, ZHANG FuYao1,2, Lü Xin1,2, LI HuiMing1,2, CHU JianQiang1,2
(1Sorghum Institute of Shanxi Agricultural University (Shanxi Academy of Agricultural Sciences)/Shanxi Key Laboratory of Sorghum Genetic and Germplasm Innovation, Jinzhong 030600, Shanxi;2Key Laboratory of Crop Gene Resources and Germplasm Enhancement on Loess Plateau, Ministry of Agriculture, Taiyuan 030031)
【】The aim of this study is to use molecular marker to construct a breeding system to improve the traditional breeding method,thereby reducing breeding costs and improving breeding efficiency.At the same time, it lays a theoretical foundation for the formation of a new breeding system.【】The F2population was constructed by crossing the photosensitive(non-heading) forage sorghum variety Jinguang 1R as female and the photoinsensitive(heading) sorghum variety BMRC-3-2 as male.According to heading and non-heading phenotypes, F2population was divided into two groups. Thirty plants were selected from each group and DNA was extracted from leaves,SSR and BSA were used to map photosensitive genes in F2population of Jinguang 1R/BMRC-3-2 and screen for specific SSR primers.The specific primers were used to identify the photosensitivity trait in F1generation(F1Hybrids breeded from high generation stable restorer line with Jinguang 1R as parent and matched with other male sterile lines), and the final target primers were determined by comparing with the phenotype in the field, so as to construct a new photosensitive breeding system for forage sorghum.【】After SNP index analysis, the region of about 250 Kb before and after the highest point of 99% threshold line was selected as the candidate region for trait correlation. The total length of the region was 500 kb, with 400 SNP loci, and six of them were non-synonymous mutations or stop gain or stop loss SNP loci.Finally, two candidate genes were identified to be related to photosensitivity, which were located on chromosome 7.A pair of specific primers 70.2-3 was developed by SSR. The primerscan distinguish between heading and non-heading sorghum plants to a great extent.The results showed that according to the "single peak" at 251bp, the accuracy of primers 70.2-3 in the identification and selection of 50 non-photosensitive F1hybrids reached 100%, and all the materials showed a peak value near 251bp.Based on the "double peaks" in the vicinity of 214bp and 251bp, the accuracy rate of the primer for the identification and selection of the other 50 photosensitive F1hybrid species reached 90%. Three materials, No. F1-69, F1-70 and F1-71, showed "double peaks" near 214 bp and 262 bp. Material F1-81 has a "single peak" at 251bp, while material No. 86 has a "double peak" near 251 bp and 232 bp.【】In this study, we revealed that the candidate genes controlling heading date of sorghum were LOC8068537 and LOC8068548, which were between 810000 and 1310000 bp on chromosome 7 of sorghum. The acquisition of specific primers 70.2-3 improved the traditional breeding methods. The primer validation in laboratory could replace the field crossing observation, saving the breeding cost and improving the breeding efficiency.
foragesorghum;photosensitive type; molecular markers

10.3864/j.issn.0578-1752.2020.14.004
2019-06-06;
2019-08-08
山西省农业科学院优势课题组自选项目(YYS1706)、现代农业产业技术体系建设专项资金(CARS-06)、山西省农业科学院生物育种工程(17yzgc031)、山西省重点研发计划重点项目(201703D211002-9-1)、酿造专用高粱育种及利用山西省科技创新重点团队运行补助(201805D131012-6)
牛皓,E-mail:nkyglsnh@126.com。通信作者平俊爱,E-mail:pingja1029@163.com
(责任编辑 李莉)

