肽核酸荧光原位杂交技术检测生肉中沙门氏菌方法的建立及应用
杨 彤,吴 姗,李 可,帅江冰,叶子弘,张晓峰*
(1. 中国计量大学 生命科学学院,浙江省生物计量与检验检疫技术重点实验室,浙江 杭州 310018;2. 浙江省检验检疫科学技术研究院,浙江 杭州 310016)
沙门氏菌(Salmonella)是最常见的食源性致病菌之一,由其在世界各地引起的食物中毒中常居于首位,据2009 年~2014 年美国国家疾病控制和预防中心(CDC)的数据显示,沙门氏菌病的暴发通常与食物来源有关,在484 次疫情中,385 起事故因食源性致病菌传播导致[1]。因此食品中沙门氏菌高效检测方法的建立,有助于在源头控制由沙门氏菌引起的食源性疾病的暴发。目前食品中沙门氏菌的检测方法主要包括分离培养、免疫学方法和分子生物学方法。分离培养法是实验室最常用和最经典的方法,但其自动化程度低、耗时长、步骤繁琐。ELISA、荧光抗体染色法(免疫荧光法)和同位素标记抗体法(放射免疫)等,尽管具有操作简便、快速的特点,但特异性较差,所需耗材价格昂贵,因此目前在国内应用较少。以PCR 技术为代表的分子生物学方法操作简便、灵敏度高,但对检测人员、仪器设备及环境的依赖性高,实验室往往不能杜绝假阴性与假阳性的结果。
肽核酸(Peptide nucleic acids,PNA)是一种人工合成的核酸模拟物,结合荧光原位杂交技术(Fluorescence in situ hybridization,FISH)可用于食品及环境中的致病菌检测。Almeida 等人首先将该方法应用到沙门氏菌的检测,并在粪便和水中检测到了沙门氏菌[2]。在此基础上,为进一步提高探针的特异性,本研究基于沙门氏菌invA 毒力基因,设计特异性PNA 探针Sal-invA,经条件优化,建立快速、准确、灵敏的PNA-FISH 检测方法,为快速检测致病性沙门氏菌提供了一种可行的技术手段。
1 材料与方法
1.1 主要实验材料本研究所使用的全部细菌见表2,其中标准菌株均购自中国医学细菌保藏管理中心,分离株由本实验室保存;本研究中弧菌用APW(3%氯化钠碱性蛋白胨水,北京陆桥生物技术公司)培养基培养;其余菌株均用BHI(脑心浸出液,北京陆桥生物技术公司)培养基培养。上述细菌培养至对数生长期后,取增菌液离心沉淀,再用PBS 悬浮洗涤,制成初始菌悬液。BPW(缓冲蛋白胨水,北京陆桥技术股份有限公司)用于非选择性增菌;TTB(四硫磺酸钠煌绿,北京陆桥技术股份有限公司)用于选择性增菌;沙门氏菌显色培养基(CHROMagarMT法国科玛嘉)用于培养法中可疑菌落鉴定。
83 份生鲜肉制品用于沙门氏菌的检测,其中生鸡肉18 份,生猪肉51 份,生牛肉14 份,分别购自杭州市某屠宰场、农贸市场、超市和网络。
1.2 PNA 探针设计本研究涉及的探针见表1,其中Bacuni 是细菌通用型探针[3];SalPNA1873 是Almeida 等设计的沙门氏菌特异性探针[2];Sal-invA 是本研究根据沙门氏菌毒力基因设计的沙门氏菌特异性探针。探针设计方法如下:从NCBI 数据库中选择了常见的16 种血清型致病性沙门氏菌中的27 个invA毒 力 基 因 序 列(https://www.ncbi.nlm.nih.gov),利 用MegAlign 软件(version 5.0;DNASTAR、Madison、WI)的ClustalV 算法进行排序,在其保守区设计沙门氏菌属特异性探针Sal-invA。并通过BLAST(http://blast.ncbi.nlm.nih.gov/)对探针的特异性进行预测。本研究所用探针5'均用Cy3标记,由韩国Daejeon公司合成。
1.3 PNA-FISH 方法特异性试验本研究利用19个血清型的25 株沙门氏菌和12 株有代表性的非沙门氏菌用于PNA-FISH 方法的特异性检测,利用本研究室前期优化好的杂交体系,对表1 中的3 种探针均采用如下相同的杂交步骤,进行探针的特异性试验。杂交方法:用PBS 调整菌液浓度至OD600nm=1.0,取10 μL菌液于100%酒精清洗过的玻片上涂匀,火焰固定,将玻片于4%多聚甲醛中浸泡10 min,再置于50%的酒精加50%的PBS中浸泡10 min,风干;25 μL杂交缓冲液(除3 种探针外,其余成分均相同)[4](500 pmol/mL PNA 探针;pH 7.5,10%(w/v)硫酸葡聚糖;10 mmol/L NaCl;30%(v/v)甲酰胺;0.1%(w/v)焦磷酸钠;0.2%(w/v)聚乙烯吡咯烷酮;0.2%(w/v)聚 蔗 糖;5 mmol/L Na2EDTA;0.2%(v/v)Triton X-100;50 mmol/L Tris/HCl)滴加于玻片上(1 cm×1 cm),55 ℃~57 ℃作用1.5 h;杂交后玻片于55 ℃~57 ℃预热的洗涤 缓 冲 液(PH 10,5 mmol/L Tris;15 mmol/L NaCl;0.1%(v/v)Triton X-100)中洗涤2 次,每次10 min。将风干后的涂片,滴上无自发荧光的香柏油,利用荧光显微镜(Leica DM 6000B)100 倍物镜及N21 通道(515 nm~560 nm)观察荧光亮度及菌的形态。不加任何探针作为阴性对照。显微镜下具有明显发光的菌体且符合目标菌形态学特点即判定为阳性,观察15个视野,若无目标菌体,则判定为阴性。

表1 PNA 探针序列Table 1 PNA Probes used in this study
1.4 灵敏度试验以肠炎沙门氏菌(Salmonella enteritidis)ATCC 15611 株 为 目 标 菌,10 倍 倍 比 稀 释(101~109),取其中107~1010稀释的沙门氏菌进行平板计数:每个浓度取100 μL 涂布于计数营养琼脂平板上,培养48 h。记录菌落数,每个浓度做3个平行,取平均值,根据标准GB4789.2-2016[5]计算不同菌悬液的浓度,以此得到纯培养原始菌落数,同时测定其OD600nm值。
根据上述平板计数结果,利用PNA-FISH 法、荧光定量PCR 法和传统生化培养法同时对100cfu/mL~109cfu/mL 的沙门氏菌进行灵敏度的检测。荧光定量PCR 检测方法中:菌体DNA 的提取(热裂解法)、荧光PCR 的引物和探针序列、检测方法及判定标准均参照行业标准SN/T 1059.7-2010[6]。以Ct 表示检测结果,每个浓度的菌液重复检测3 次,3 次结果中若有2 次或2 次以上的结果为阳性,则表示可检测到该浓度的菌液。引物和探针由北京擎科生物科技有限公司合成。沙门氏菌的培养鉴定方法参照国标法《GB 4789.4—2016 食品安全国家标准食品微生物学检验沙门氏菌检验》[7],但本研究不作血清型分型鉴定,疑似目标菌落利用全自动微生物鉴定仪(VITEK 2 COMPACT 30)鉴定,具体操作步骤参照VITEK 2 Compact 仪器用户手册。
1.5 人工污染肉制品的检测在6 份经GB 4789.4-2016[7]检测为沙门氏菌阴性的25 g新鲜猪肉糜中分别添加100cfu、101cfu、102cfu、103cfu、104cfu、105cfu的沙门氏标准菌,静置10 min,按照国标法增菌:装入有225 mL BPW 增菌液的均质过滤袋中,在均质机中拍打1 min,36 ℃培养12 h,再取1 mL菌液转接于10 mL TTB内,于42 ℃培养20 h。所有增菌步骤均按照国标法实施,增菌后的样品,经上述3 种方法检测,每个浓度做3个平行,比较3种方法的灵敏度。
1.6 肉制品的检测将购买的83 份生鲜肉制品按照1.5 的方法增菌后,利用上述PNA-FISH 法、荧光定量PCR 法和培养法,同时检测生鲜肉制品中的沙门氏菌,比较3 者的检测结果,并计算本研究建立的方法分别与其余两种方法的符合率。
2 结 果
2.1 PNA 探针设计及3 种探针对比结果由于细胞内含有丰富的rRNA,针对微生物检测中,肽核酸探针通常是靶向于细菌保守区域中的16S rRNA 或23S rRNA序列设计的,如Almeida等[2]设计的用于检测沙门氏菌的探针SalPNA1873。invA为沙门氏菌的主要毒力因子,与其致病性相关,沙门氏菌粘附和侵入宿主细胞都是由其介导的。因此本研究靶向于沙门氏菌特异的invA基因设计探针,以提高探针的特异性。
利用BLAST 程序对探针的特异性进行预测。结果显示,所设计的探针Sal-invA 与10 余种沙门氏菌同源性均较高。但同时也和一些非目标菌具有相同序列,如:喜温硫杆菌(Acidithiobacillus caldus)、硫酸盐还原菌(Desulfobulbus propionicus)、解木聚糖拟杆菌(Bacteroides xylanisolvens)、不透明红球菌(Rhodococcus opacus)等,但上述细菌均与沙门氏菌的亲缘关系较远,也并非食品中常见的致病菌。因此,Sal-invA 探针特异性较强。
2.2 特异性试验结果通过探针的杂交试验进一步检测了Sal-invA 探针的特异性,并和SalPNA1873 探针进行了比较。结果显示,全部沙门氏菌与两种特异性探针Sal-invA 与SalPNA1873 均呈阳性杂交结果,但SalPNA1873 探针与肺炎克雷伯氏菌(Klebsiella pneumoniae)、痢疾志贺氏菌(Shigella castellani)、大肠杆菌O157(Escherichia coli O157∶H7)等非沙门氏菌产生了非特异性杂交,Sal-invA 探针与非目标菌株均未产生杂交反应,而Bacuni探针与全部细菌杂交结果均为阳性(表2)。表明,本研究建立的PNA-FISH检测方法中Sal-invA探针特异性强于SalPNA1873探针。
2.3 灵敏度试验结果根据平板计数结果计算得到标准菌沙门氏菌ATCC 15611 株的初始菌液浓度为9.8×109cfu/mL。将其10 倍倍比稀释后(109cfu/mL~100cfu/mL)利用本研究建立的PNA-FISH 方法检测。结果显示,109cfu/mL~105cfu/mL 菌液涂片均可以稳定观察到发光菌体,104cfu/mL 的3 个涂片中仅一处观察到目标菌且在多数视野中未见目标菌体,故判定为阴性(图1),低于该浓度镜检均不能检出,因此本研究建立的方法对沙门氏菌的最低检测限为105cfu/mL。同时以105cfu/mL~100cfu/mL 的沙门氏菌标准菌株ATCC 15611 裂解液提取的DNA 为模板,进行荧光定量PCR 扩增。结果显示,102cfu/mL 菌液及以上浓度的菌液DNA 均有特异曲线扩增,低于该浓度则无特异性曲线扩增。因此,102cfu/mL 为荧光定量PCR 方法的最低检出限(图2)。因为PNA-FISH检测方法无细菌的扩增步骤,其灵敏度低于荧光定量PCR 方法,因此在实际检测中要有增菌步骤或者富集增菌液是不可或缺的。
取不同浓度的菌液于沙门氏菌显色培养基划线接种,培养后观察各个平板上菌落生长情况。结果显示, 接种105cfu/mL 及以上浓度的平板均可出现沙门氏菌的特征性菌落,经后续生化试验,确定特征性菌落为沙门氏菌,因此,培养法的灵敏度为105cfu/mL,与PNA-FISH 方法检测限相同。
2.4 人工污染肉制品的检测结果本研究将不同浓度的沙门氏菌分别人工污染生鲜肉样品,经增菌后取增菌液进行PNA-FISH 检测。经显微镜观察结果显示,在初始接种量为100cfu/mL 的菌液经增菌后在涂片上看到大量发光菌体,因此,本研究建立的PNA-FISH 方法对沙门氏菌增菌后的最低检测限可达100cfu/mL,3个重复试验中均无杂交失败的涂片。
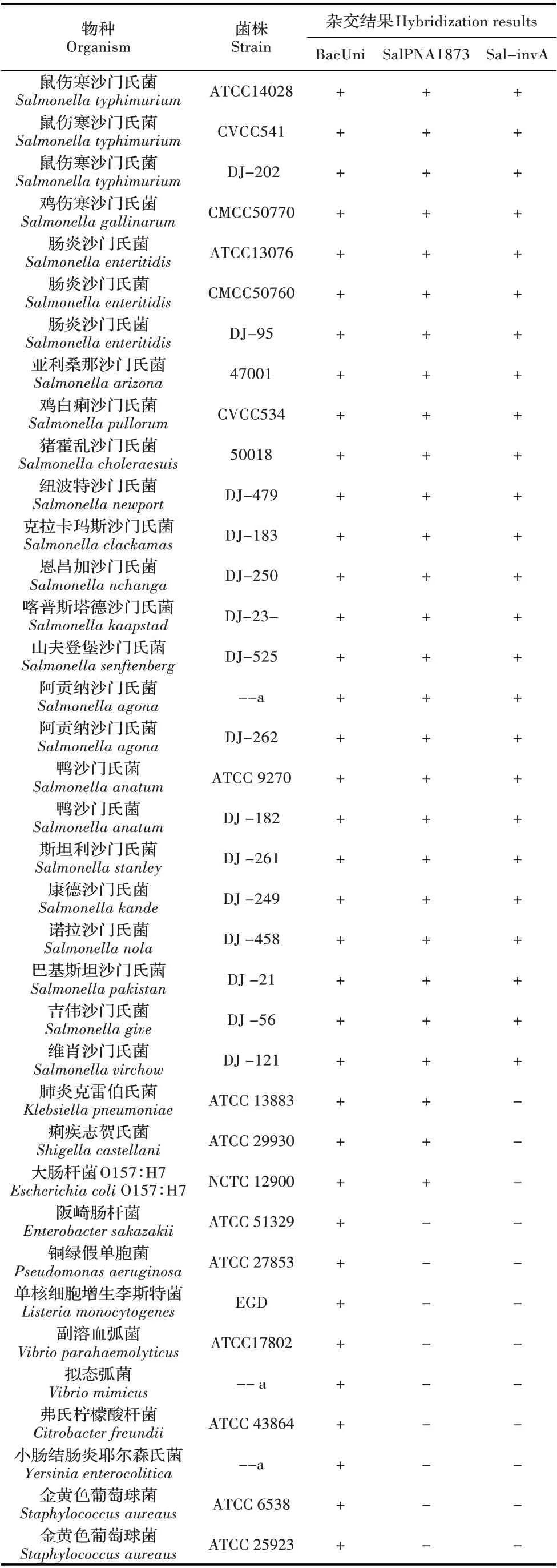
表2 PNA-FISH 方法的探针特异性试验结果Table 2 Results of specificity test by PNA-FISH method

图1 PNA-FISH 方法灵敏度试验结果Fig.1 Results of sensitivity test by PNA-FISH method
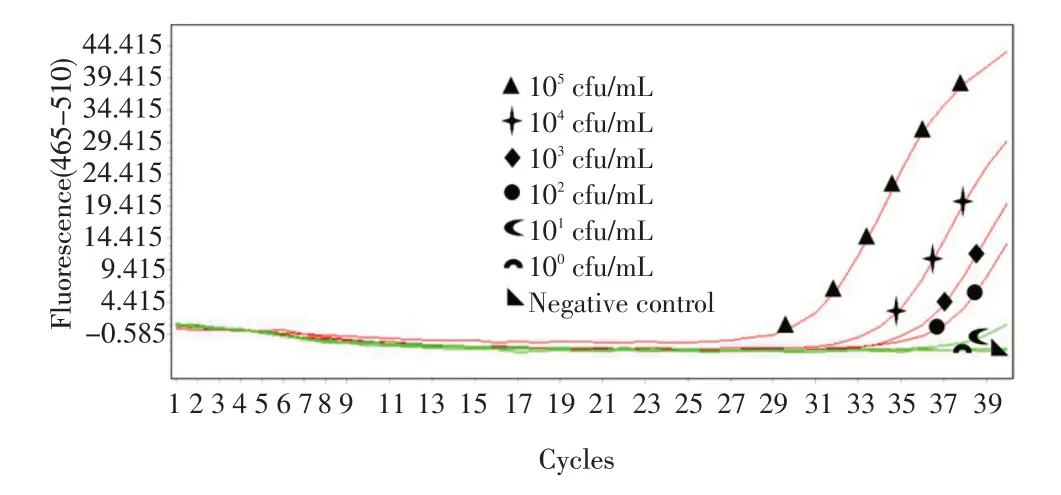
图2 荧光定量PCR 方法灵敏度试验结果Fig.2 Results of sensitivity test by qPCR method
以不同浓度的沙门氏菌人工污染生鲜肉的增菌裂解液为模板,进行荧光定量PCR 扩增。结果显示,109cfu/mL~100cfu/mL 菌液样品经增菌后均出现特异性扩增曲线,而阴性样品则无扩增曲线(图3)。因此,荧光定量PCR方法能稳定检测100cfu/mL的样品。
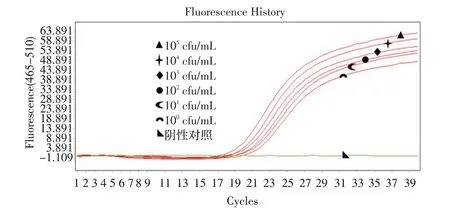
图3 荧光定量PCR方法对人工污染不同浓度沙门氏菌的肉制品的检测结果Fig.3 Detection of different concentrations Salmonella in artificially contaminated samples by qPCR methodd
取增菌后的菌液于沙门氏菌显色培养基划线接种,培养后观察各个平板上菌落生长情况。结果显示,109cfu/mL~100cfu/mL 的沙门氏菌经增菌后均能在显色培养基上出现沙门氏菌的特征性菌落,并经生化试验鉴定均为沙门氏菌。因此,3 种检测方法对沙门氏菌检测的灵敏度均达到了100cfu/mL。结果表明,经过国标法中的增菌步骤,PNA-FISH 检测方法可以达到与培养法和荧光定量PCR 法相同的检测限。
2.5 生鲜肉制品的检测结果对市场上购买的83份生鲜肉样品同时采用上3 种方法进行沙门氏菌检测。结果显示,3 种检测方法结果基本相同,荧光定量PCR 方法灵敏度较高, PNA-FISH 方法则具有较强的特异性,而沙门氏菌的分离培养法是3 种方法中唯一可以只对活菌检测的方法且作为目前微生物检测的金标准,是判定检测结果的根本依据。经计算,PNA-FISH 方法与分离培养法、荧光定量PCR 方法的符合率均为98.8%(表3)。上述结果表明,经国标法增菌后,本研究建立的PNA-FISH方法与另两种方法符合率较高,可以用于生鲜肉制品的检测。

表3 培养法、荧光定量PCR法和PNA-FISH法对肉制品中沙门氏菌的检测结果Table 3 Detection of Salmonella in food-related samples byculture method、qPCR method and PNA-FISH method
3 讨 论
本研究所用的PNA 探针Sal-invA 结合FISH 检测方法,针对常见的主要致病性沙门氏菌的检测表现出了较高的灵敏度和较强的特异性。虽然Almeida等人也用类似的方法检测出了更为广谱的沙门氏菌, 但其采用23S rRNA 基因片段作为靶标设计的SalPNA1873 探针[2]经本研究检测,特异性较差。而本研究选择了沙门氏菌侵袭基因invA 片段作为靶标设计探针,对致病性沙门氏菌的检测具有更强的特异性,本研究结果也证实了这一点。
本研究比较了PNA-FISH 法、荧光定量PCR 法与培养法检对沙门氏菌检测的灵敏性。PNA-FISH方法在纯培养情况下仅可观察到105cfu/mL 浓度的菌体,这与Almeida 等的研究中同样采用PNA-FISH 方法检测李斯特菌的灵敏性相当[8]。荧光定量PCR 法灵敏度达到102cfu/mL,高于PNA-FISH 方法和培养法,其主要原因是荧光定量PCR 方法有扩增步骤,能够使目标菌靶序列呈指数增长且依赖高精度仪器判定结果,灵敏度更高。在随后对人工污染沙门氏菌的肉制品的检测中,经国标法增菌后再利用该3种方法检测,其灵敏度均达100cfu/mL,表明增菌步骤是目前所有检测方法中不可或缺的步骤,且对于依赖人工判定结果的检测方法该步骤更为重要。
分离培养法是目前微生物检测的金标准,该方法是对活菌数目进行统计,而在生鲜肉制品采集运输过程中,不适宜的储存条件可能会造成细菌损伤,冷冻肉还可能造成细菌活力降低甚至死亡,在细菌培养时其活力不易恢复到初始水平,因而会影响检出率,造成假阴性。荧光定量PCR 方法是对目标菌的DNA 进行扩增,消除了细菌活力对检测结果的影响,但其高灵敏度易对检测造成假阳性结果。PNA-FISH 方法的可视化可以基于细菌形态学特点经人工做出判定,降低假阳性率,提高了检测的特异性。 同一检测方法中,高灵敏度与高特异性不可兼得,新型检测方法的开发旨在获得最接近实际情况的可靠结果,本研究中PNA-FISH 检测方法的建立对现有检测方法是一个有力的补充。
由于PNA-FISH 方法无法区分目标菌是死菌还是活菌,但通过检测前的增菌过程增大了活菌与死菌的比例,降低了死菌对检测结果的影响。Machado 等人在PNA-FISH 方法的建立中,利用4',6-二脒基-2-苯基吲哚(DAPI)染料对活细胞染色,从而区分了死菌与活菌[9],但是如何利用分子生物学方法鉴别,仍有待研究。通过细菌预富集手段(免疫学磁珠富集等)对大量杂菌中目标菌进行特异性吸附收集,代替国标法中的增菌步骤从样品中迅速、有选择性地分离出目的细菌,可以显著缩短检测时间[10-12],将会成为PNA-FISH 方法的另一创新点。目前PNA 探针通常是13~18 个碱基的短肽核酸,其长度比DNA 探针更短,设计出特异性强的PNA 探针是目前阻碍该方法建立的一大障碍, 这也是PNA-FISH 方法不能像荧光定量PCR 方法一样普及的重要原因。本研究设计了一种具有强特异性的探针,用于沙门氏菌的检测,为开发快速、特异的沙门氏菌检测方法提供了一个新思路。

