珠江流域9个大鳞副泥鳅群体遗传多样性的微卫星分析*
刘 洁 高风英 陈 刚 曹建萌 刘志刚 王 淼 卢迈新
珠江流域9个大鳞副泥鳅群体遗传多样性的微卫星分析*
刘 洁1,2高风英1①陈 刚2曹建萌1刘志刚1王 淼1卢迈新1①
(1. 中国水产科学研究院珠江水产研究所 农业农村部热带亚热带水产资源利用与养殖重点实验室 广州 510380;2. 广东海洋大学水产学院 湛江 524025)
为探索我国珠江流域大鳞副泥鳅()群体的遗传分化及亲缘关系,本研究采用8个微卫星分子标记,对我国珠江流域9个大鳞副泥鳅群体(佛山、高要、封开、肇庆、乳源、乐昌、韶关、河源和惠州)进行了遗传多样性分析。结果显示,8个微卫星位点共检测到 69个等位基因,平均等位基因(a)和有效等位基因(e)分别为8.6和4.0个,平均观测杂合度(o)和期望杂合度(e)分别为0.4426和0.7030。9个大鳞副泥鳅群体间的遗传分化系数(st)和基因流(m)分别为0.2452和0.7697,表明群体间遗传分化水平较高,遗传交流水平低。采用UPGMA法,对 9个群体基于遗传距离进行聚类,可分为两大支,韶关、佛山和乳源群体聚为一支;另一支包含了其余的6个大鳞副泥鳅群体,分为3个小分支,分别为乐昌与肇庆群体、河源与惠州群体及高要与封开群体。研究表明,珠江流域的9个大鳞副泥鳅群体具有较高的遗传多样性,且群体间存在着一定的遗传分化,具有进一步选育的价值。
大鳞副泥鳅;微卫星;遗传多样性
大鳞副泥鳅()主要分布于我国东部地区的河流、水田及湖泊水域,其外部形态与泥鳅()极为相似(杜启艳等, 2007)。大鳞副泥鳅肉质细嫩、味道鲜美、营养丰富,素有“水中人参”之称(赵睿等, 2014)。近年来,我国向日本、韩国出口大量大鳞副泥鳅,随着市场需求的增加,大鳞副泥鳅的养殖规模也不断扩大,而良种选育工作的滞后使苗种的人工繁育量已不能满足养殖规模不断扩大的需要(李彩娟等, 2014)。此外,环境污染和近亲繁殖导致野生泥鳅的资源量锐减,种质资源不断退化(邱楚雯等, 2017)。亲鱼捕捞后的异地养殖和人工养殖个体的逃逸干扰了泥鳅的自然分布和群体结构。因此,研究大鳞副泥鳅的遗传多样性,对合理开发和保护我国现有大鳞副泥鳅资源具有重要意义。
遗传多样性为生物多样性的重要组成部分。一个群体或物种的遗传多样性越高或遗传变异越丰富,越能适应多变的环境,其进化潜力也越大(沈浩等, 2001)。对物种遗传多样性的数据分析,可为物种资源的开发和生物多样性的保护提供科学依据(罗静等, 2000)。遗传多样性的研究方法随着遗传学和分子生物学的发展,已到DNA分子水平。其中,微卫星又称简单重复序列(Simple sequence repeats, SSR),是指以少数几个核苷酸(2~6)经多次重复组成的简单串联重复序列(刘萍等, 2008)。SSR具有高多态性、共显性、稳定性强等优点,被广泛应用于水产动物遗传多样性评估、遗传结构和亲缘关系分析等研究中(王军等,2018; 李可等, 2018; 叶竹青等, 2015; 苏天凤等, 2010)。目前,有研究用到微卫星技术对我国各地区大鳞副泥鳅进行遗传多样性分析。李彩娟等(2014)采用17个多态性微卫星标记,发现四大湖泊大鳞副泥鳅自然群体具有丰富的遗传多样性。游翠红等(2012)筛选出6对微卫星引物,对我国长江水系及广州地区的7个野生大鳞副泥鳅群体遗传结构分析显示,大鳞副泥鳅种群遗传多样性较低。珠江流域大鳞副泥鳅群体遗传多样性分析还未见报道。
本研究采用8个微卫星,对我国珠江流域共9个群体大鳞副泥鳅的遗传多样性与遗传分化进行研究,充分探索大鳞副泥鳅的遗传背景,以期为其种质资源保护策略的制定以及良种选育基础群体的选择提供理论依据。
1 材料与方法
1.1 实验材料
2017年8~10月于珠江水系采集泥鳅活体,采集后于中国水产科学研究院珠江水产研究所实验室暂养,取尾鳍并保存于无水乙醇中。9个大鳞副泥鳅群体野生样品分别采自珠江流域的西江(佛山,24尾;高要,30尾;封开,35尾;肇庆,27尾)、北江(乳源,25尾;乐昌,30尾;韶关,34尾)和东江(河源,29尾;惠州,20尾) (图1),共计254尾个体。
鉴别泥鳅和大鳞副泥鳅的特异性引物:上游引物(5′CCGCCCAGGAGTATTTTATG3′);下游引物(5′TGGCATTTCATCAGGGGTG3′) (侯吉伦等, 2015)。8个微卫星(Mac37、Mac45、Mac49、Mac60、Mac425、Mac456、Mac576和Mac627)摘自泥鳅微卫星连锁图谱中等位基因较多的位点(Kagayaki, 2008),扩增片段长度为100~300 bp,便于电泳分型。上述引物均由生工生物工程(上海)股份有限公司合成。
1.2 实验方法
1.2.1 基因组DNA的提取 使用美基DNA提取试剂盒提取泥鳅尾鳍基因组DNA。用1%琼脂糖凝胶120 V电泳20 min检测所提DNA质量;用微量分光光度计检测DNA浓度和质量;加ddH2O稀释DNA至50 ng/μl,–20℃保存备用。

图1 大鳞副泥鳅采集地点()
FS:佛山;GY:高要;FK:封开;ZQ:肇庆;RY:乳源;LC:乐昌;SG:韶关;HY:河源;HZ:惠州
FS: Foshan; GY: Gaoyao; FK: Fengkai; ZQ: Zhaoqing; RY: Ruyuan; LC: Lechang; SG: Shaoguan; HY: Heyuan; HZ: Huizhou
1.2.2 泥鳅和大鳞副泥鳅鉴别 用鉴别引物进行PCR扩增,PCR反应体系(25 μl):50 ng/μl DNA模板2.5 μl,10 μmol/L上下游引物各2.5 μl,10×LA PCR Buffer(含Mg2+)2.5 μl,2.5 mmol/L dNTP Mixture 2.0 μl,5 U/μl LADNA聚合酶0.25 μl,ddH2O 12.75 μl。PCR反应程序:94℃预变性5 min;94℃变性30 s,54℃退火90 s,72℃延伸1 min,35个循环;72℃延伸10 min。用1%琼脂糖凝胶120 V电泳20 min,检测PCR扩增产物,电泳图谱显示,被测样品条带大小鉴别泥鳅(285 bp)和大鳞副泥鳅(214 bp)。
1.2.3 PCR扩增 采用10对微卫星引物进行PCR扩增,同时,筛选最佳的退火温度。PCR反应体系(20 μl):50 ng/μl DNA模板1.0 μl,10 μmol/L上下游引物各0.2 μl 2×EasyTaq®PCR SuperMix for PAGE 10.0 μl,ddH2O 8.6 μl。PCR反应程序:94℃预变性 3 min;94℃变性30 s,退火30 s,72℃延伸30 s, 23个循环;72℃延伸5 min。各对引物的特异退火温度见表1。
表1 本研究使用的微卫星位点及其相关信息

Tab.1 Microsatellites used in this study and their associated information
*:(X)代表核心序列重复次数
*: (X)means repeat number of core sequence
1.2.4 聚丙烯凝胶电泳及银染 扩增产物经12%非变性聚丙烯酰胺凝胶电泳分离,上样量为5 μl,内外电泳槽中各加适量的1×TBE电泳缓冲液,50 V电泳30 min后再150 V电泳150 min。银染参考徐绍斌等(2002)的方法并适当调整,同一位点等位基因由大到小记为A、B、C…V。
1.2.5 数据分析 使用Popgene32 1.32软件分析各微卫星基因座的等位基因数(Number of allele,a)、有效等位基因数(Effective mumber of allele,e)、观测杂合度(Observed heterozygosity,o)、期望杂合度(Expected heterozygosity,e)、群体内近交系数(Fixation index,is)、遗传分化系数(Genetic differentiation coefficient,st)、基因流(Number of migrants per generation,m)、遗传距离(Genetic distance,A)、遗传相似系数(Genetic similarity index,) (Kagayaki, 2008),使用PIC-CALC 0.6软件计算各位点的多态信息含量(Polymorphism information content, PIC)。
基于群体间Nei氏标准遗传距离,使用MEGA6.0构建7个群体UPGMA(Unweighted pair group method with arithmetic means)系统进化树。
2 结果
2.1 泥鳅和大鳞副泥鳅鉴别
鉴别引物在所有样品中均能扩增出清晰的条带,且能根据PCR扩增的产物片段大小来区分泥鳅(285 bp)和大鳞副泥鳅(214 bp)。部分大鳞副泥鳅鉴定的电泳结果见图2。

图2 大鳞副泥鳅鉴定的部分电泳结果
2.2 微卫星引物的PCR扩增
选取的10对泥鳅微卫星引物在9个大鳞副泥鳅群体中,有8对能扩增出清晰的条带,且重复性好,在12%非变性聚丙烯酰胺凝胶上能分离等位基因。带型统计分析后发现,8个微卫星位点共获得了69个等位基因,微卫星基因座的等位基因范围为5~15个,平均每个位点检测出8.6个等位基因,其中,Mac49存在等位基因最少(5个),Mac456最多(15个)。部分位点的电泳结果见图3。
2.3 9个大鳞副泥鳅群体遗传多样性
从表2可以看出,8个微卫星位点的平均观测等位基因为8.6个,平均有效等位基因数为4.0个,平均观测杂合度为0.4426,平均期望杂合度为0.7030。8个位点在9个大鳞副泥鳅群体中表现出不同水平的多态。参照Botstein等(1980)提出的衡量基因变异程度高低的PIC指标,除位点Mac45和Mac49为中度多态(0.25

图3 位点Mac425和Mac576的部分电泳结果
表2 8个微卫星位点总遗传参数
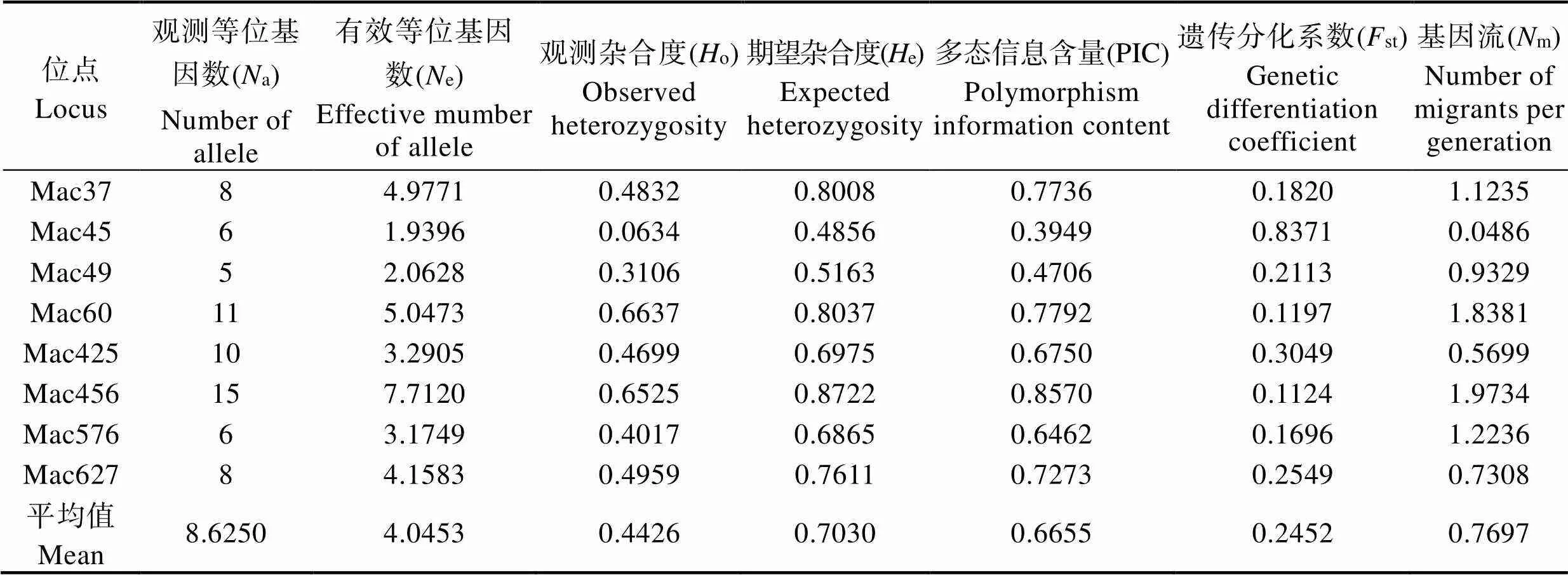
Tab.2 Total genetic parameters of 8 microsatellite loci
从表3可见,9个大鳞副泥鳅群体的等位基因数差别较小,其中,高要群体的等位基因数(a)最少(2.25),韶关群体最多(5.38);有效等位基因数(e)高要群体最少(1.98),韶关群体最多(3.15);各群体平均观测杂合度(o)为0.3307~0.5626;期望杂合度(e)为0.3499~0.6144;PIC为0.4629~0.6777,其中,乐昌和河源群体表现为中度多态,其余7个群体表现为高度多态。
2.4 9个大鳞副泥鳅群体间的遗传相似度和遗传距离
遗传距离和遗传相似性指数是衡量群体亲缘关系远近的重要指标(朱滔等, 2013)。根据Nei指数法计算9个泥鳅群体间遗传相似性指数和遗传距离(表4)。9个大鳞副泥鳅群体间遗传相似度为0.1965~0.9467,遗传距离为0.0548~1.6271。肇庆与乐昌群体的遗传相似度最高(0.9467),遗传距离最近(0.0548);高要与乳源群体的遗传相似度最低(0.9467),遗传距离最远(1.6271)。
根据遗传距离运用UPGMA法对7个泥鳅群体进行了聚类分析。系统树结果表明(图4),9个大鳞副泥鳅群体分为两大支。其中,韶关和佛山群体聚在一起,再与乳源群体聚为一支;另一支包含了其余的6个大鳞副泥鳅群体,其中,乐昌与肇庆群体聚为一支,河源与惠州群体聚为一支,这2个分支聚为一个分支后,再与高要和封开群体的这个分支聚为一大分支。
表3 8个微卫星位点在9个大鳞副泥鳅群体中的遗传多样性参数

Tab.3 Genetic diversity indices of 8 microsatellite loci in 9 P. dabryanus populations
表4 9个大鳞副泥鳅群体的遗传相似性(对角线之上)与遗传距离(对角线之下)

Tab.4 Genetic identity (above diagonal) and genetic distance (below diagonal) among 9 P. dabryanus populations

图4 基于Nei´s无偏遗传距离构建的9个大鳞副泥鳅群体UPGMA树
3 讨论
3.1 微卫星引物多样性
本研究8个微卫星位点多态性丰富,共获得 69个等位基因,平均等位基因为8.6个,与游翠红等(2012)利用6对泥鳅微卫星引物跨种扩增大鳞副泥鳅,每个位点检测出的平均等位基因数相近。PIC可反映基因座位变异程度的高低,同时,也关系到该位点的可用性和使用效率,PIC越大,提供的遗传信息越高,越具有可用性(邢晶晶, 2012)。本研究的8个微卫星位点中,除Mac45和Mac49两个位点为中度多态(0.25
3.2 种内遗传多样性
9个大鳞副泥鳅群体平均PIC为0.5602,其中,乐昌和河源群体表现为中度多态,其余7个群体PIC均高于0.5,表现为高度多态,具有进一步选育的潜力。李雅娟等(2010)采用5个微卫星分子标记对我国3种野生泥鳅进行分析显示,平均观测杂合度和期望杂合度分别为0.8696和0.7283,平均多态信息含量为0.6193,均高于本研究结果的平均观测杂合度(0.4477)、期望杂合度(0.5409)和PIC(0.5602),在一定程度反映出泥鳅的遗传多样性高于大鳞副泥鳅。Dewoody等(2000)采用微卫星标记对13种淡水鱼进行分析,得出平均期望杂合度为0.46,与本研究结果中9个大鳞副泥鳅的平均期望杂合度相近。李彩娟等(2014)采用 17个微卫星分子标记,分析四大湖泊大鳞副泥鳅的平均期望杂合度为0.577,高于本研究的结果,可能研究对象中有一部分个体为逃逸到野生环境中的养殖个体的原因。
3.3 遗传分化与亲缘关系
遗传分化指数(st)是评价群体间遗传分化程度的重要指标,本研究中9个大鳞副泥鳅群体的遗传分化指数均值为0.2452,表明群体间遗传分化水平较高(刘志刚等, 2018)。丁海燕等(2015)利用6个微卫星标记分析我国泥鳅,遗传分化指数均值为0.4990,高于本研究的遗传分化指数值,多种原因可造成遗传分化的严重,人类活动造成的遗传漂变可能是最主要的原因。Wright(1931)认为,基因流指数m>1,基因流则能发挥均质化作用,即能有效抑制种群间遗传分化的产生;m<1时,则表明各亚群间基因流受阻,群体可能走向遗传分化。本研究中8个座位的m均值为0.7697(m<1),表明9个群体间基因交流水平低,进而导致遗传分化程度较高。这可能也是由于大鳞副泥鳅自身迁移能力有限、地理距离、自然屏障的隔离和自然选择等导致了较大程度的遗传分化(王坤, 2009)。
聚类分析结果显示,9个大鳞副泥鳅群体大多数按地理位置聚类。乳源和韶关群体位于珠江水系的北江及其支流,与其他地区群体因地理距离远而缺乏基因交流,使得与其他地区间遗传距离较大,在聚类图上这2个大鳞副泥鳅群体聚为一大支与其他6个群体分开。另外的一大支中,位于珠江东江流域的河源和惠州群体聚在一起,位于珠江西江流域的高要和封开群体聚在一起。在游翠红等(2012)对我国7个大鳞副泥鳅群体的研究中发现,多个地区群体也表现出相似的遗传分化现象。可见地理隔离是影响大鳞副泥鳅群体间遗传分化的主要原因。但在9个群体中,乐昌群体属于珠江水系的北江,却与位于珠江西江流域的肇庆群体遗传相似度最高(0.9467),遗传距离最近(0.0548)。此外,位于珠江水系西江的佛山群体与位于珠江北江的韶关群体的遗传距离(0.2201)也较近。单磊等(2009)对长江中下游泥鳅的遗传多样性的研究也发现,武汉和太湖群体之间地理位置较远而遗传关系较近,表现出遗传距离与地理距离不一致的现象。丁海燕等(2015)对我国7个泥鳅群体的遗传多样性研究也表明,四大流域东部盆地各泥鳅群体遗传距离却与地理距离没有明确的关联性。这说明人类活动或养殖行为可能在一定程度上扰动了泥鳅资源的分布,也可能与实验样品量不足有关,后续应扩大采样范围和增加实验群体。本研究中,9个珠江水系的大鳞副泥鳅群体具有较高的遗传多样性,且群体间存在一定的遗传分化,可对其进行种质保护,防止与其他苗种混杂,造成种质退化。
Balloux F, Lugon MN. The estimation of population differentiation with microsatellite markers. Molecular Ecology, 2002, 11(2): 155–165
Botstein D, White RL, Skolnick M,. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980, 32(3): 314–331
DeWoody JA, Avise JC. Microsatellite variation in marine, freshwater and anadromous fishes compared with other animals. Journal of Fish Biology, 2000, 56(3): 461–473
Ding HY, Zhu M, Peng C,. Preliminary analysis of the microsatellite diversity of Chinese loach). Journal of Fisheries of China, 2015, 39(10): 1433–1442 [丁海燕, 朱明, 彭冲, 等. 我国泥鳅微卫星遗传多样性初步分析. 水产学报, 2015, 39(10): 1433–1442]
Du QY, Nan P, Yan SG,. Observation on the embryonic development of. Journal of Henan Normal University (Natural Science), 2007(2): 146–149 [杜启艳, 南平, 燕帅国, 等. 豫北地区大鳞副泥鳅胚胎发育的观察. 河南师范大学学报(自然科学版), 2007(2): 146–149]
Hou JL, Liu YF, Liu HJ,. Identification primer and identification method forandPatent: CN104498604A, 2015- 04-08 [侯吉伦, 刘永富, 刘海金, 等. 一种泥鳅和大鳞副泥鳅种质鉴别引物和鉴别方法. 专利: CN104498604A, 2015-04-08]
Kagayaki M, Ichiro N, Katsutoshi A. Genetic linkage map of the loach(Teleostei: Cobitidae). Genetica, 2008, 132(3): 227–241
Li CJ, Ling QF, Ge C,. Genetic diversity offrom four big lakes in China. Jiangsu Journal of Agricultural Sciences, 2014, 30(5): 1087–1094 [李彩娟, 凌去非, 葛辰, 等. 中国4大湖泊大鳞副泥鳅群体遗传多样性分析. 江苏农业学报, 2014, 30(5): 1087–1094]
Li K, Ma XF, Xie P,. Genetic diversity and population genetic structure ofbased on microsatellite genetic markers. Journal of Huazhong Agricultural University, 2018, 37(4): 112–120 [李可, 马徐发, 谢鹏, 等. 基于微卫星标记的拟鲤遗传多样性及群体遗传结构分析. 华中农业大学学报, 2018, 37(4): 112–120]
Li YJ, Yu Z, Zhang MZ,. Genetic diversity in Chinese loach species analyzed by microsatellite DNA markers. Journal of Northeast Agricultural University, 2010, 41(12): 75–79 [李雅娟, 于卓, 张明昭, 等. 我国三种野生泥鳅遗传多样性的微卫星分析. 东北农业大学学报, 2010, 41(12): 75–79]
Liu P, Song LP, Li J,. Development of microsatellite DNA markers and their applications in genetic studies in crabs. Periodical of Ocean University of China, 2008, 38(5): 712–718 [刘萍, 宋来鹏, 李健, 等. 蟹类微卫星DNA标记的筛选及其在遗传学研究中的应用. 中国海洋大学学报, 2008, 38(5): 712–718]
Liu ZG, Lu MX, Cao JM,. Genetic diversity and genetic relationship analysis of tilapia “Yuemin No.1” and its breeding populations. Progress in Fishery Sciences, 2018, 39(6): 31–41 [刘志刚, 卢迈新, 曹建萌, 等. 罗非鱼“粤闽1号”及其繁育群体的遗传多样性和遗传关系分析. 渔业科学进展, 2018, 39(6): 31–41]
Luo J, Yang JX, Zhang YP,. Genetic basis of fish diversity. Zoological Research, 2000, 21(2): 158–164 [罗静, 杨君兴, 张亚平. 鱼类多样性的遗传基础. 动物学研究, 2000, 21(2): 158–164]
Qiao LY, Yuan YN. The measurement indices and influencing factors of microsatellite polymorphisms. Chinese Animal Husbandry and Veterinary Medicine, 2010, 37(1): 107–111 [乔利英, 袁亚男. 微卫星标记遗传多样性的度量指标及影响因素. 中国畜牧兽医, 2010, 37(1): 107–111]
Qiu CW, Wang HX. Advances in research on genetic diversity of. Fisheries Science and Technology Information, 2017, 44(2): 78–86 [邱楚雯, 王韩信. 泥鳅属鱼类遗传多样性的研究进展. 水产科技情报, 2017, 44(2): 78–86]
Shan L, Wei KJ, Zhang GR,. Genetic diversity of four diploid loach) populations in the middle and lower Yangtze River Basin. Journal of Huazhong Agricultural University, 2009, 28(4): 453–458 [单磊, 魏开建, 张桂蓉, 等. 长江中下游二倍体泥鳅4个种群的遗传多样性. 华中农业大学学报, 2009, 28(4): 453–458]
Shen H, Liu DY. Summary of genetic diversity. Journal of Biology, 2001(3): 5–7 [沈浩, 刘登义. 遗传多样性概述. 生物学杂志, 2001(3): 5–7]
Su TF, Xiong XF, Jiang SG,. Microsatellite analysis of genetic relationship among 7 full-sib families of. South China Fisheries Science, 2010, 6(6): 1–7 [苏天凤, 熊小飞, 江世贵, 等. 斑节对虾7个全同胞家系间亲缘关系的微卫星分析. 南方水产, 2010, 6(6): 1–7]
Wang J, Wang QY, Kong J,. SSR Analysis on genetic diversity in breeding and wild populations of. Progress in Fishery Sciences, 2018, 39(2): 104–111[王军, 王清印, 孔杰, 等. 中国明对虾人工选育群体与野生群体遗传多样性的SSR分析. 渔业科学进展, 2018, 39(2): 104–111]
Wang K. A primary study on moleculargenetic differences and growth of different loach populations in the Yangtze River Basin. Master´s Thesis of Soochow University, 2009 [王坤. 长江流域不同泥鳅群体的分子遗传差异及生长的初步研究. 苏州大学硕士研究生学位论文, 2009]
Wright. Evolution in mendelian population. Genetics, 1931, 16: 97–159
Xing JJ. The types of molecular markers and the research and applications of molecular technology on the aquatic creature. Chinese Journal of Fisheries, 2002(1): 61–70 [邢晶晶. 分子遗传标记及其技术在水产生物中的研究与应用. 水产学杂志, 2002(1): 61–70]
Xu SB, Tao YF, Yang ZQ. A simple and rapid methods used for silver staining and gel preservation. Hereditas, 2002, 24(3): 335–336 [许绍斌, 陶玉芬, 杨昭庆. 简单快速的DNA银染和胶保存方法. 遗传, 2002, 24(3): 335–336]
Ye ZQ, Ling QF, Li CJ,. Genetic structure analysis of five families of. Journal of Hydroecology, 2015, 36(1): 66–73 [叶竹青, 凌去非, 李彩娟, 等. 5个大鳞副泥鳅家系的遗传结构分析. 水生态学杂志, 2015, 36(1): 66–73]
You CH, Tong JG, Yu XM. Microsatellite DNA analysis on genetic diversity of seven populations of. Journal of Hydroecology, 2012, 33(1): 84–91 [游翠红, 童金苟, 俞小牧. 大鳞副泥鳅7个群体遗传变异的微卫星分析. 水生态学杂志, 2012, 33(1): 84–91]
Zhao R, Shen F, Na LH. Artificial breeding technology of. China Fisheries, 2014(9): 59–60 [赵睿, 沈芾, 那立海. 大鳞副泥鳅人工繁育技术. 中国水产, 2014(9): 59–60]
Zhu T, Liang XF, Peng MH,. Isolation and characterization of polymorphic EST-SSRs markers in threepopulation. Journal of Jinan University, 2013, 34(3): 347–352 [朱滔, 梁旭方, 彭敏燕, 等. 翘嘴鳜EST-SSR标记的开发及3个群体遗传多态性分析. 暨南大学学报, 2013, 34(3): 347–352]
Genetic Diversity Analysis of Nine Loach () Populations in the Pearl River Basin Based on Microsatellite Markers
LIU Jie1,2, GAO Fengying1①, CHEN Gang2, CAO Jianmeng1, LIU Zhigang1, WANG Miao1, LU Maixin1①
(1. Pearl River Fisheries Research Institute, Chinese Academy of Fishery Science, Key Laboratory of Tropical and Subtropical Fishery Resource Application and Cultivation, Ministry of Agriculture and Rural Affairs, Guangzhou 510380;2. College of Fisheries, Guangdong Ocean University, Zhanjiang 524025)
Human activities and environmental pollution have caused varying degrees of damage toresources in China. To explore the genetic differentiation and phylogenetic relationship of.populations in China’s Pearl River Basin, eight microsatellite markers were used to analyze the genetic diversity of nine geographicalpopulations (Foshan, Gaoyao, Fengkai, Zhaoqing, Ruyuan, Lechang, Shaoguan, Heyuan, and Huizhou) of.in the Pearl River Basin. The analysis detected 69 alleles in eight microsatellite loci. The average number of alleles (a) and effective alleles (e) per locus were 8.6 and 4.0, respectively, and the average observed heterozygosity (o) and expected heterozygosity (e) were 0.4426 and 0.7030, respectively. The average genetic differentiation coefficient (st) and number of migrants per generation (m) of the nine.populations were 0.2452 and 0.7697, respectively, which indicates that genetic differentiation among populations was high and genetic communication was low. Cluster analysis was performed by the unweighted pair group method with arithmetic mean (UPGMA) method, based on the genetic distance among nine populations. The resulting UPGMA tree was divided into two main branches. The Shaoguan, Foshan, and Ruyuan populations were monophyletic and formed oneclade. The remaining six.populations formed the other branch, which contained three smaller branches, which were composed of the Lechang and Zhaoqing populations, the Heyuan and Huizhou populations, the Gaoyao and Fengkai populations. These results suggest that the genetic diversity of nine.populations in the Pearl River Basin was high, and there was a certain genetic differentiation among the populations, which could be used for further breeding.
; Microsatellite; Genetic diversity
GAO Fengying, E-mail: fengyinggao2011@163.com;LU Maixin, E-mail: mx-lu@163.com
S931.5
A
2095-9869(2020)04-0050-08
10.19663/j.issn2095-9869.20190731002
http://www.yykxjz.cn/
刘洁, 高风英, 陈刚, 曹建萌, 刘志刚, 王淼, 卢迈新. 珠江流域9个大鳞副泥鳅群体遗传多样性的微卫星分析. 渔业科学进展, 2020, 41(4): 50–57
Liu J, Gao FY, Chen G, Cao JM, Liu ZG, Wang M, Lu MX. Genetic diversity analysis of nine loach () populations in the Pearl River Basin based on microsatellite markers. Progress in Fishery Sciences, 2020, 41(4): 50–57
* 现代农业产业技术体系专项资金(CARS-46)资助[This work was supported by China Agriculture Research System (CARS-46)]. 刘 洁,E-mail: ljie0624@163.com
高风英,副研究员,E-mail: fengyinggao2011@163.com;卢迈新,研究员,E-mail: mx-lu@163.com
2019-07-31,
2019-08-15
(编辑 冯小花)

