大豆生物钟基因GmLNK1/2、GmRVE4/8和GmTOC1 CRISPR/Cas9组织表达分析及敲除靶点的鉴定
甘卓然 石文茜 黎永力 侯智红 李海洋 程 群 董利东 刘宝辉 芦思佳
研究简报
大豆生物钟基因、和CRISPR/Cas9组织表达分析及敲除靶点的鉴定
甘卓然 石文茜 黎永力 侯智红 李海洋 程 群 董利东 刘宝辉 芦思佳*
广州大学生命科学学院, 广东广州 510006
生物钟基因能够参与调控植物的整个生命进程, 对提高作物产量具有重要的作用。(-)、()、()和()是植物中重要的生物钟基因。本研究利用BLAST同源比对和进化树分析的方法分别鉴定、、、和在大豆中的同源基因, 通过qRT-PCR实验证明这些生物钟基因在大豆根、茎、叶等组织中均有表达。成功构建这些基因的CRISPR/Cas9敲除载体, 并利用大豆根毛转化体系成功鉴定出13个基因的CRISPR/CAS9有效靶点。为进一步获得稳定的大豆突变体材料及研究其生物钟基因的功能提供了理论基础。
大豆; 基因敲除; 生物钟基因; CRISPR/Cas9
单产低是我国大豆面临的最大问题, 因此提高大豆单产是首要任务[1]。影响大豆产量的因素可归为两大类, 一是外部因素, 如耕作技术、气候、土壤和自然灾害等; 二是内部因素, 即基因, 两方面的因素相互影响、协调, 共同调控大豆的产量[2-3]。大豆作为固着生物, 必须准确地感知环境信号, 如光和温度, 以适应其生长发育的周围环境[4]。实际上, 大多数生物并不是简单地对外部环境做出反应, 而是对外部环境的变化做出预测, 并根据自己的生物习性进行调整[5]。这种内源性生物节律就是生物钟。生物钟使生物体能够预测和准备应对周围环境中的变化[4]。生物种基因在控制植物开花、产量、生物胁迫和非生物胁迫中都扮演重要的角色。我们前期的研究发现, 大豆长童期性状关键位点是拟南芥生物钟关键基因()的一个同源基因, 并证明在短日照下的等位变异能够延迟大豆开花, 使大豆产量提高30%以上[6]。植物生物钟由许多基因共同调控, 如()、()、()、()、(),()、()、()、()等[7]。在拟南芥中,/与是植物生物钟的核心元件,并且形成反馈调节回路[8-9]。、和是与同源的生物钟基因, 作为生物钟激活因子调控下游基因的表达[9-10]。与作为转录共激活因子, 能够与及相互作用, 共同调控基因的表达[11]。番茄基因的突变可导致番茄生物钟周期延迟[12]。这些生物钟基因在不同植物中引起其他性状的变化也开始被解析。功能研究中发现, 拟南芥//三突变体中, 光敏色素互作因子()和()表达量提高, 从而导致其叶片面积显著大于野生型拟南芥植株[10]。功能研究中发现, 番茄基因突变可导致番茄生物钟周期延后, 叶绿素含量明显增加, 有效防止番茄晒伤, 该研究指出昼夜节律中的这种条件变化可能对各物种(包括植物和动物)的纬度适应性至关重要[12-13]。尽管这些重要的生物钟基因在拟南芥、番茄等植物中的功能相继被解析, 但在大豆中仍不清楚。
CRISPR/Cas9技术能够精准快速地进行基因编辑, 因此被广泛应用在动物和植物中[14-17]。近些年在大豆中利用CRISPR/Cas9技术已经成功对多个大豆基因进行定点编辑。例如、、、、等, 为更加深入研究这些基因的功能, 提供了良好的遗传材料[18-20]。大豆的基因组复杂, 生长周期长, 转化效率低, 所以我们在利用CRISPR/Cas9技术进行大豆稳定转化前, 确定目的基因靶点(gRNA)的有效性是十分重要的。本研究首先利用蛋白同源比对和进化树分析的方法成功鉴定出与拟南芥同源的大豆生物钟基因和。并利用qRT-PCR技术, 检测这些基因在大豆植株的各个组织中的表达情况。随后, 利用CRISPR/Cas9技术, 构建了大豆、、基因的敲除载体, 并利用大豆发根体系成功检测出大部分基因的有效编辑靶点。为利用CRISPR/Cas9技术创造这些生物钟基因的稳定大豆突变体和进一步研究这些生物钟基因的功能提供了理论基础。
1 材料与方法
1.1 材料
大豆发根转化材料为大豆品种Williams 82 (W82), 大肠杆菌菌株DH5α、发根农杆菌菌株K599均由本实验室保存, 载体构建质粒AtU3d、AtU3b、AtU6-1、AtU6-29、Cas9载体由华南农业大学刘耀光院士提供。
1.2 植物材料处理及RNA提取
将大豆品种W82种子播于钵中(草炭土∶蛭石=2∶1), 每钵6粒, 在温度25℃、光照16 h/8 h (光照/黑暗)的温室内培养。大约14 d后, 大豆幼苗长到V1期, 取大豆的根、茎、子叶、下胚轴、初生叶、三出复叶和生长点于液氮中速冻。大豆生长到R1期(约48 d), 取大豆的花于液氮中速冻。参照RNA提取试剂盒说明书(RNA pure Plant Kit, China)提取RNA。qRT-PCR实验分别进行3次生物重复。
1.3 大豆生物钟基因的鉴定和进化树分析
分别利用拟南芥生物钟基因、、、、的蛋白序列, 在Phytozome 12数据库进行BLAST分析, 分别鉴定与拟南芥同源的大豆生物种基因。根据同源性由高到低, 分别将每个拟南芥生物钟基因在大豆中4个同源基因命名为a、b、c和d。利用DANMAN软件进行同源比对分析。
1.4 靶点设计
利用CRISPR direct在线网站(http://crispr.dbcls.jp/)进行靶点设计, 载体构建所用基因靶点引物信息, 如表1所示。

表1 基因靶点引物列表
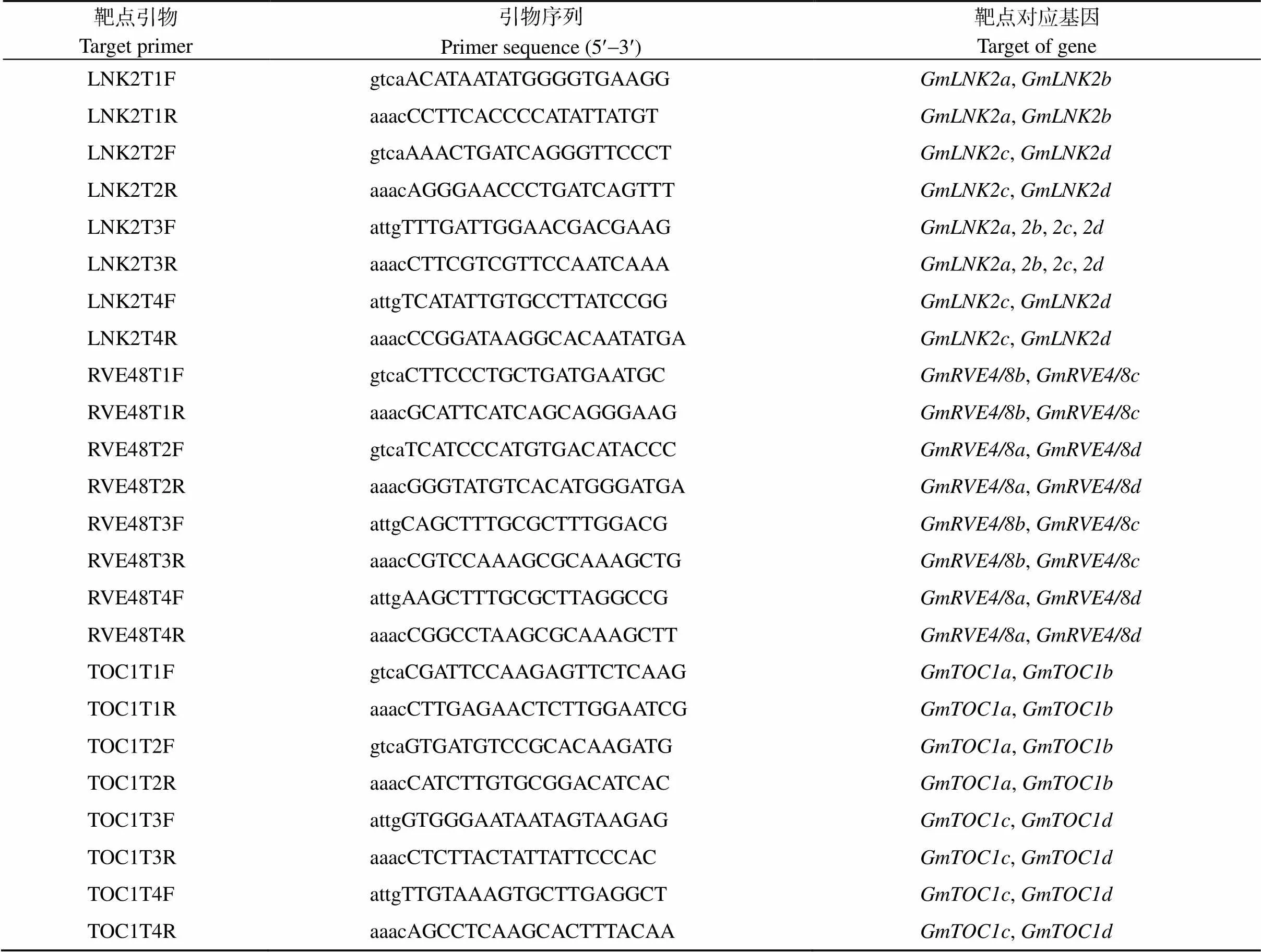
(续表1)
1.5 CRISPR/Cas9载体构建
参照曾栋昌等[21]、侯智红等[22]研究方法进行载体构建。实验步骤如下: (1)靶点引物退火。(2)已退火的靶点与小载体AtU3d、AtU3b、AtU6-1和AtU6-29一一对应连接, 并进行第一次边切边连。(3)使用第一次边切边连产物进行第一次PCR扩增。(4)以第一次PCR产物为模板, 进行第二次PCR扩增。(5)回收第二次PCR的产物, 进行第二次边切边连, 连接Cas9载体。(6)第二次边切边连产物转化大肠杆菌DH。(7)挑取阳性单菌落, 摇菌, 提取质粒, 转化农杆菌K599。
1.6 大豆根毛转化
参照表2配制萌发及根诱导培养基, 并参照Cheng等[23]方法进行根毛转化。步骤如下: (1)将大豆种子灭菌, 取饱满没有病斑的籽粒置干燥器中, 在干燥器中放一个200 mL烧杯, 盛96 mL次氯酸钠和4 mL浓盐酸, 灭菌12~16 h。(2)根据表2配制大豆萌发培养基, 在无菌操作台中将已灭菌的大豆种子均匀摆放到萌发培养基。(3)大豆种子萌发5 d左右, 根据表2配制发根培养基, 在无菌操作台中利用手术刀在大豆的子叶上面切一个小洞。然后将处理的大豆子叶均匀放置到发根培养基上, 取20 µL含有目的载体的农杆菌K599放到大豆子叶的小洞中。(4)将培养基放在大豆无菌培养室中, 培养15 d左右, 从大豆子叶的侵染部位会有根毛长出, 分别收集大豆的根毛于液氮中速冻。
1.7 基因靶点编辑有效性检测
大豆生物钟基因靶点编辑有效性检测步骤如下: (1)用1.6中收集的大豆根毛提取DNA。(2)利用CRISPR/Cas9载体通用引物SP1和SP3 (表3)检测大豆根毛是否为转基因根毛(目的片段的长度约为2000 bp左右), 检测每个载体20个转基因事件以检测靶点编辑效率。(3)根据每个基因的DNA序列, 分别设计扩增目的基因的靶点检测特异引物。利用每个基因的靶点特异引物(表3)进行PCR扩增目的片段, 将PCR产物送广州天一辉远测序服务公司测序。(4)分析测序结果的峰图, 如果在靶点附近发生套峰, 证明目的基因的靶点是有效的。
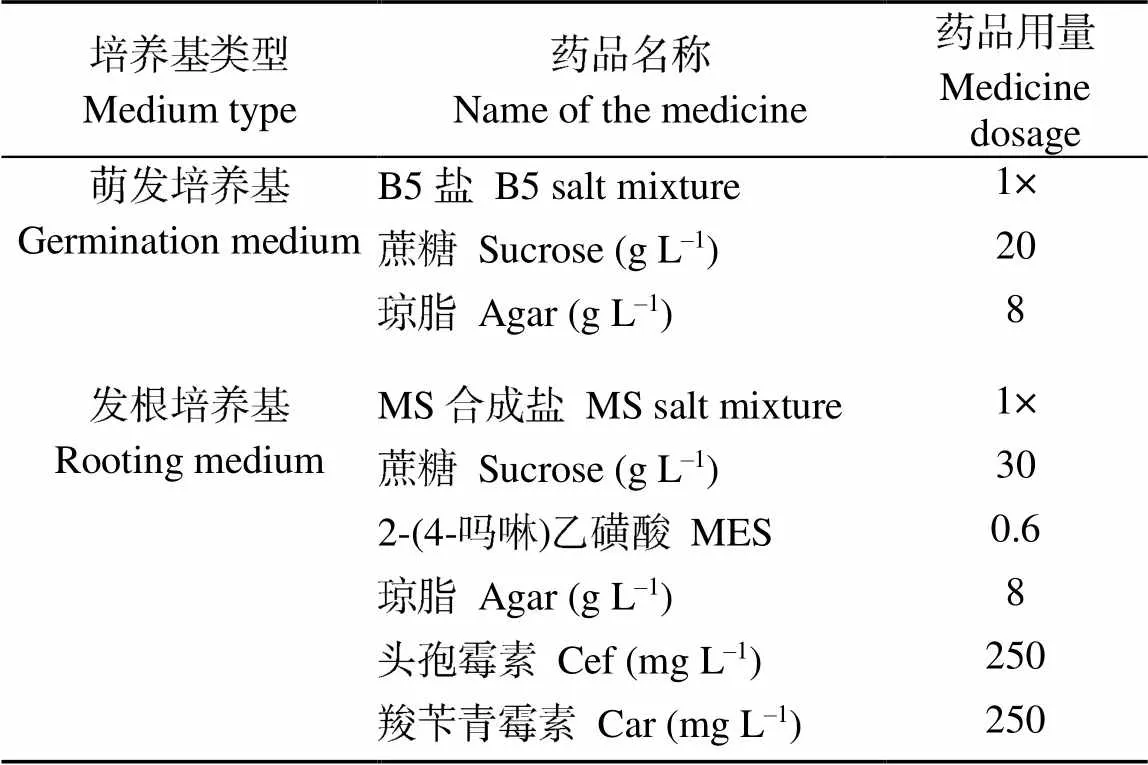
表2 萌发培养基与根诱导培养基
MES: 2-Morpholinoethanesulfonic acid.
2 结果与分析
2.1 进化树与同源性比对分析
在Phytozome 12数据库中, 搜索并下载与拟南芥生物钟基因()、()、()、()、()同源的大豆基因序列, 发现大豆中分别含有4个同源拷贝, 命名为()、()、()、(); 大豆中分别含有4个同源拷贝, 命名为()、()、()、(); 大豆中分别含有4个同源拷贝, 命名为()、()、()、(); 大豆中分别含有4个与同源拷贝, 命名为()、()、()、()。
利用 DNAMAN软件, 分别与AtLNK1、AtLNK2、AtTOC1、AtRVE4、AtRVE8进行同源性比对。表明, GmLNK1a/b/c/d在第503~618位氨基酸比较保守, 保守率约为63.38% (图1-A)。GmLNK2a/b/c/d在第176~224、438~471和511~620位氨基酸比较保守, 保守率约为68.81% (图1-B)。GmTOC1a/b/c/d在第27~205、553~622位氨基酸比较保守, 保守率约为72.07% (图1-C)。GmRVE4/8a/b/c/d在第35~124、226~282位氨基酸比较保守, 与AtRVE4比对, 保守率约为73.37%, 与AtRVE8比对, 则保守率约为76.66% (图1-D)。
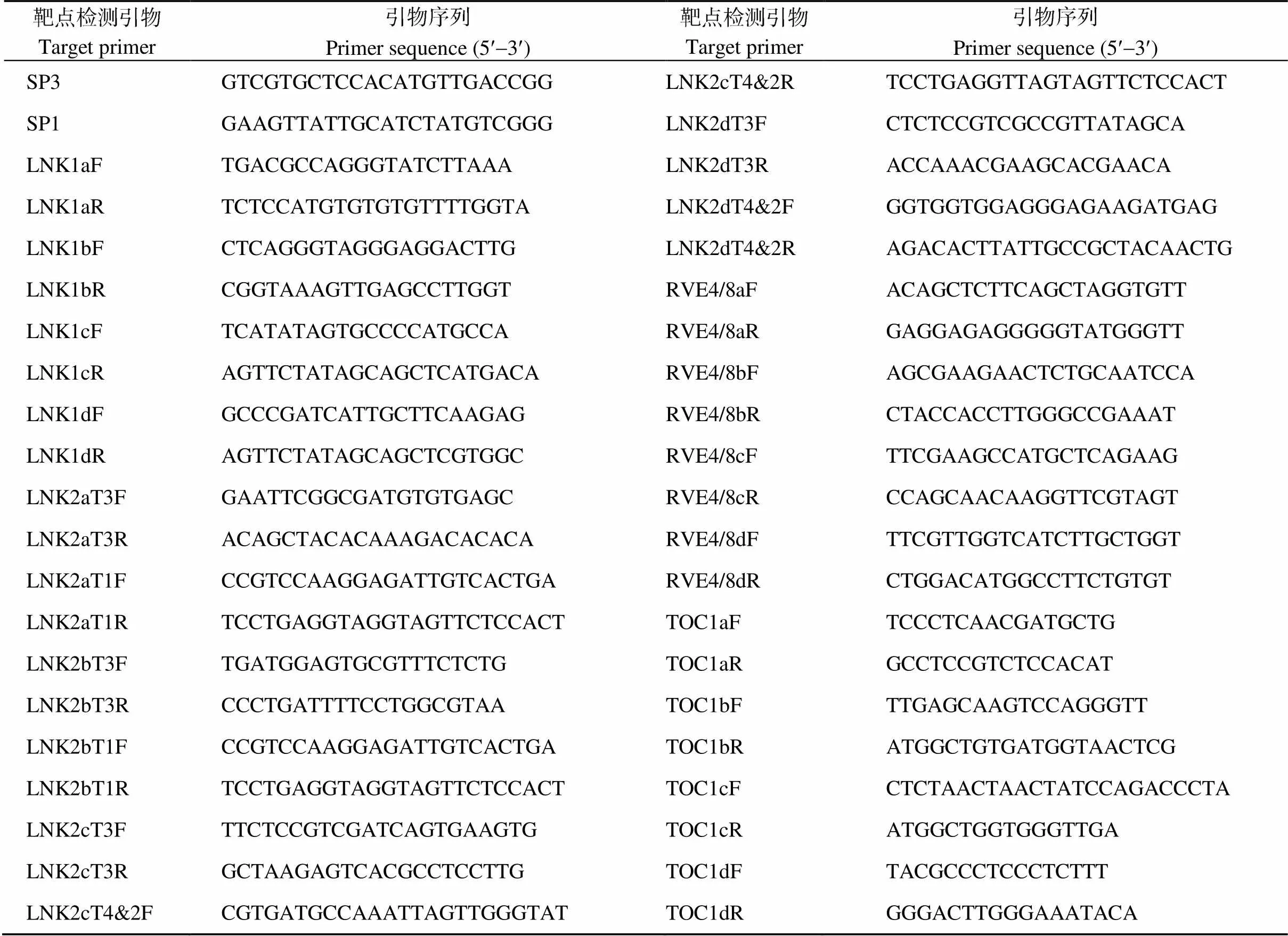
表3 靶点检测引物

(图1)
A: AtLNK1与GmLNK1a、GmLNK1b、GmLNK1c、GmLNK1d氨基酸序列对比; B: AtLNK2与GmLNK2a、GmLNK2b、GmLNK2c、GmLNK2d氨基酸序列对比; C: AtTOC1与GmTOC1a、GmTOC1b、GmTOC1c、GmTOC1d氨基酸序列对比; D: AtRVE4、AtRVE8与GmRVE4/8a、GmRVE4/8b、GmRVE4/8c、GmRVE4/8d氨基酸序列对比。
A: amino acid sequences comparison of AtLNK1, GmLNK1a, GmLNK1b, GmLNK1c, and GmLNK1d; B: amino acid sequences comparison of AtLNK2, GmLNK2a, GmLNK2b, GmLNK2c, and GmLNK2d; C: amino acid sequences comparison of AtTOC1, GmTOC1a, GmTOC1b, GmTOC1c, and GmTOC1d; D: amino acid sequences comparison of AtRVE4, AtRVE8, GmRVE4/8a, GmRVE4/8b, GmRVE4/8c, and GmRVE4/8d.
2.2 组织特异性表达分析
生物钟基因在整个植物生命进程及应答环境胁迫过程中扮演至关重要的角色, 因此检测生物钟基因在植物中的表达部位非常重要。通过qRT-PCR技术检测显示,、和、在大豆各个组织中均有表达, 但表达量较低, 而在下胚轴、子叶、茎、生长点中表达较高(图2-A);在大豆各个组织中均有表达, 且在初生叶
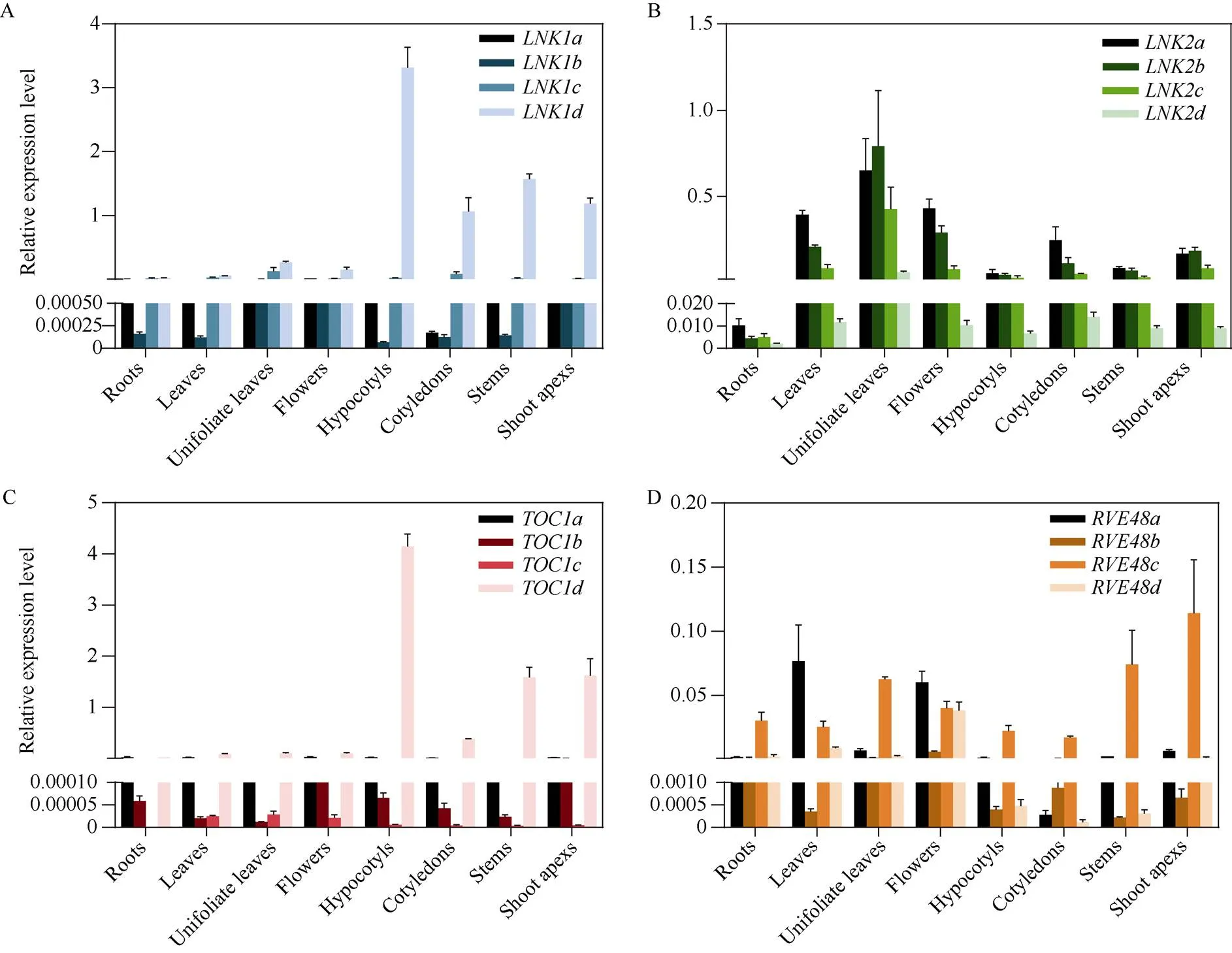
图2 4个生物钟基因组织特异表达
A:///在W82中的组织特异表达; B:///在W82中的组织特异表达; C:///在W82中的组织特异表达; D:///在W82中的组织特异表达。
A: the expression of///in soybean W82 tissues; B: the expression of///in soybean W82 tissues; C: the expression of///in soybean W82 tissues; D: the expression of///in soybean W82 tissues.
中表达最高(图2-B);家族中4个的表达情况与家族相似,、和的表达量都很低,在下胚轴的表达量最高(图2-C);家族则没有明显的规律,在三出复叶处的表达量最高,在花处的表达量最高,在生长点处的表达量最高,在花处的表达量最高(图2-D)。
2.3 CRISPR/Cas9载体构建
为了鉴定这些基因有效的基因编辑靶点, 分别将、、、16个基因的16个靶点构建到4个CRISPR/Cas9载体上, 每个基因包含2~3个靶点, 各基因所对应靶点如表1所示。载体结构如图3所示,、、和基因的T1靶点连接拟南芥的启动子, T2靶点连接拟南芥的启动子, T3靶点连接拟南芥的启动子, T4靶点连接拟南芥的启动子。
2.4 GmLNK1、GmLNK2、GmTOC1和GmRVE4/8基因的靶点有效性检测
从图4-A可以看出,基因的T1靶点测序峰图在靶点附近发生套峰, 说明这个靶点是有效的。根据测序发现的16个基因中, 有13个被成功编辑,的LNK1T1靶点、的LNK1T2靶点、和的LNK1T4靶点可被有效编辑(图4-A);和的靶点LNK2T1靶点,和的LNK2T4可被有效编辑(图4-B),的RVE48T1靶点和的RVE48T4靶点可被有效编辑(图4-C),的TOC1T2靶点, 以及和的TOC1T4可被有效编辑(图4-D)。
3 讨论
基因组定点编辑是将外源的DNA导入受体细胞染色体的特定位点上, 特异地改造基因组, 是目前研究基因功能的重要方法之一[24]。2013年, 科学家发现了一个突破性的基因编辑技术CRISPR/Cas系统, 这个系统作为防御系统存在于部分古生菌和细菌的基因组中[24-27]。在植物中, CRISPR/Cas9 系统已成功应用于包括拟南芥、烟草、水稻、烟草、高粱、小麦、玉米、柑橘和苔类植物在内的多种植物中[28-35]。CRISPR/Cas9系统较传统基因编辑技术更加便捷高效, 应用广泛[36]。
大豆是古四倍体植物, 约75%的基因重复[37]。在大豆中一个基因往往存在2~4个拷贝, 而且这些基因大多是功能冗余的, 随机突变一个基因往往不会产生表型[38]。随着对CRISPR/Cas9系统的不断完善, 现已成功应用到大豆中, 实现对目标基因的定点编辑[18-19,39]。但由于大豆的转化效率低, 基因组复杂, 生长周期长等特点, CRISPR/ Cas9技术在大豆中的应用仍然会出现靶点无效的问题。生物钟基因在植物的生长发育、生物胁迫和非生物胁迫中都扮演着重要的角色。但大部分生物钟基因在大豆中的功能都没有被解析。本试验通过蛋白序列比对和进化树分析鉴定了大豆中的、、、同源基因, 初步分析了它们在大豆植株不同组织中的表达情况, 为进一步预测这些基因在大豆的不同组织中是否具有功能提供了理论基础。例如,在三出复叶中的表达量最高, 推测在大豆的叶片中具有重要功能;和在大豆的花中表达量最高, 这2个基因可能对大豆花的发育具有重要作用; 而在生长点处的表达量最高,可能与大豆的开花和生长习性有关。
CRISPR/Cas9系统在应用于大豆的过程中靶点的编辑效率存在相对低的问题。本研究利用CRISPR/Cas9系统成功构建多个用于诱导大豆、、、突变的CRISPR/Cas9载体。共设计了16个靶点, 其中9个能有效编辑目的基因, 同时也发现9个有效的靶点能够有效敲除13个基因。本研究设计大豆多个拷贝的同源基因CRISPR/Cas9基因敲除靶点的策略尽可能设计一个能够同时敲除2个或2个以上基因的靶点, 在构建一个多基因敲除载体时保证每个基因含有2个或2个以上的靶点。本研究中, 构建的CRISPR/Cas9载体能够同时敲除的4个同源基因和的4个同源基因证明CRISPR/Cas9靶点设计策略是有效的。本研究提供了一个简单高效的大豆多拷贝同源基因CRISPR/Cas9靶点设计策略, 保证每个基因设计2个或2个以上靶点, 同时利用大豆发根转化体系快速检测靶点的编辑效率, 为更加快速创造大豆关键基因的稳定突变体提供了理论基础。
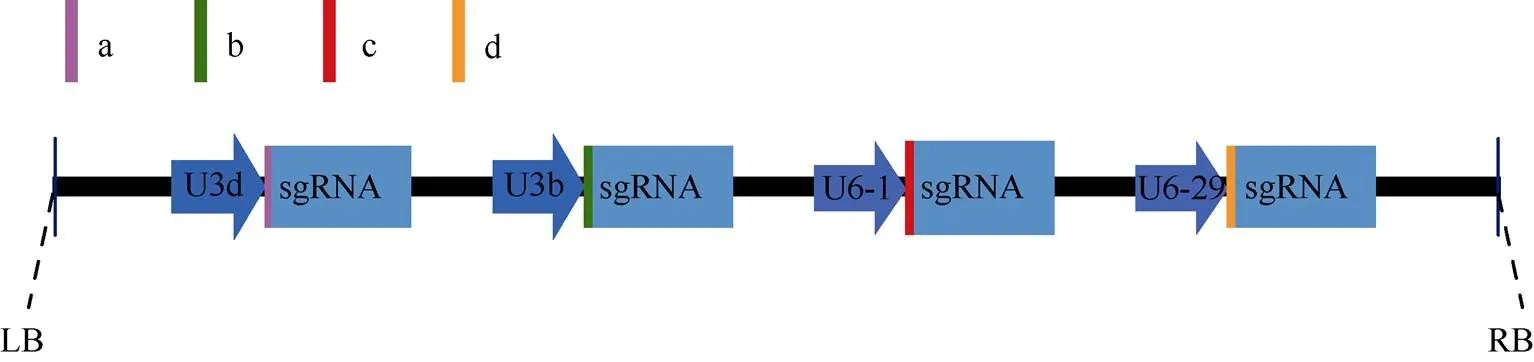
图3 Cas9载体结构图
a: 靶点LNK1T1、LNK2T1、TOC1T1和RVE4/8T1; b: 靶点LNK1T2、LNK2T2、TOC1T2和RVE4/8T2; c: 靶点LNK1T3、LNK2T3、TOC1T3和RVE4/8T3; d: 靶点LNK1T4、LNKT4、TOC1T4和RVE4/8T4。
a: target of LNK1T1, LNK2T1, TOC1T1, and RVE4/8T1; b: target of LNK1T2, LNK2T2,T2, and RVE4/8T2; c: target of LNK1T3, LNK2T3, TOC1T3, andRVE4/8T3; d: target LNK1T4, LNK2T4,TOC1T4, and RVE4/8T4.
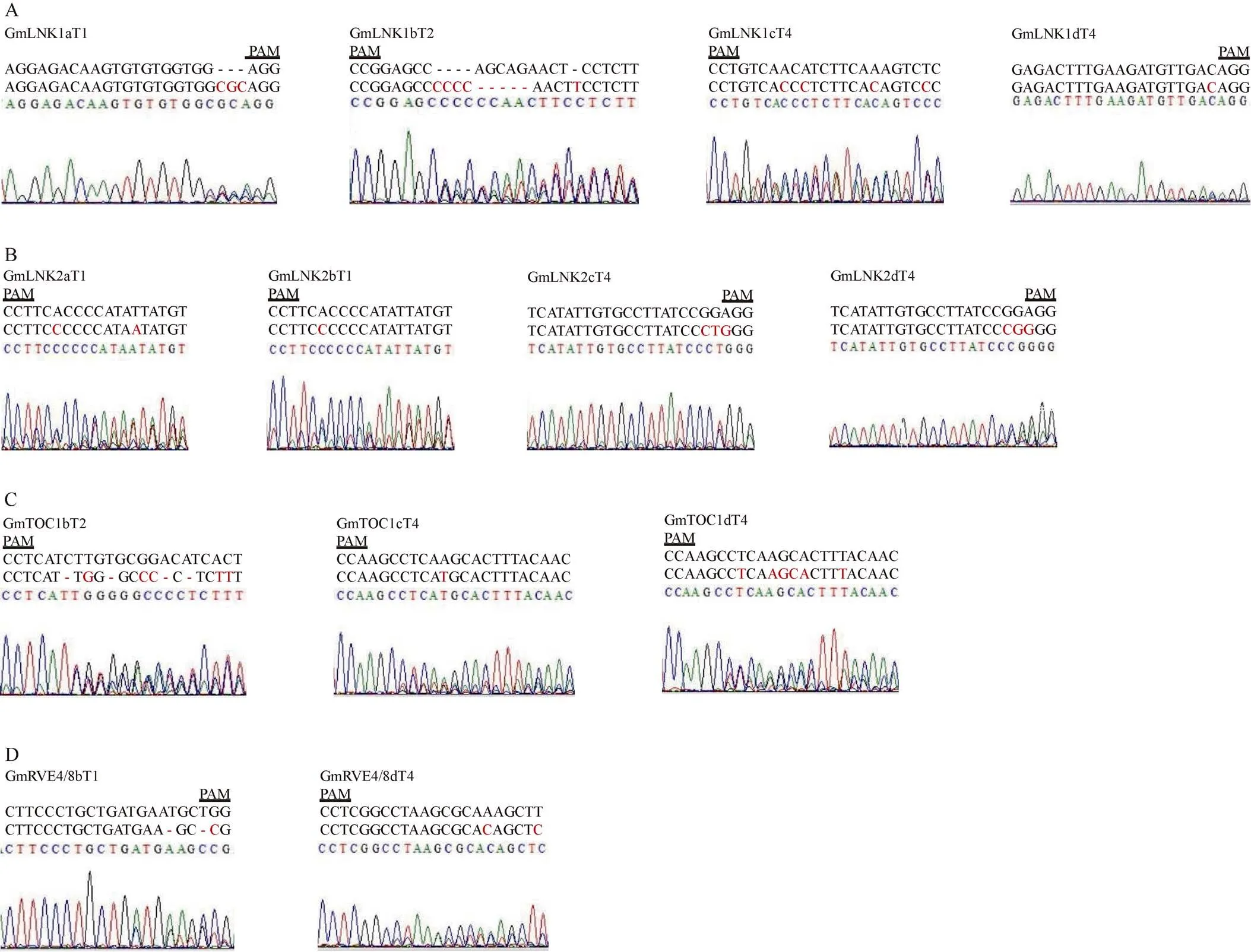
图4 发生编辑靶点的测序峰图
A:的靶点LNK1T1、的靶点LNK1T2、的靶点LNK1T4和的靶点LNK1T4突变情况; B:的靶点LNK2T1、的靶点LNK2T1、的靶点LNK2T4和的靶点LNK2T4突变情况; C:的靶点TOC1T2、的靶点TOC1T4和的靶点TOC1T4突变情况; D:的靶点RVE4/8T1和的靶点RVE4/8T4突变情况。
A: mutations were found in target LNK1T1 of, target LNK1T2 of, target LNK1T4 ofand target LNK1T4 of; B: mutations were found in target LNK2T1 of, target LNK2T1 of, target LNK2T4 ofand target LNK2T4 of; C: mutations were found in target TOC1T2 of, target TOC1T4 ofand target TOC1T4 of; D: mutations were found in target RVE4/8T1 ofand target RVE4/8T4 of.
[1] 田志喜, 刘宝辉, 杨艳萍, 李明, 姚远, 任小波, 薛勇彪. 大豆分子设计育种成果与展望. 中国科学院院刊, 2018, 33: 915–922.Tian Z X, Liu B H, Yang Y P, Li M, Yao Y, Ren X B, Xue Y B. Update and prospect of soybean molecular module-based designer breeding in China., 2018, 33: 915–922 (in Chinese with English abstract).
[2] Li M W, Xin D, Gao Y, Li K P, Fan K, Muñoz N B, Yung W S, Lam H M. Using genomic information to improve soybean adaptability to climate change., 2017, 68: 1823–1834.
[3] Raju S K K, Shao M R, Sanchez R, Xu Y Z, Sandhu A, Graef G, Mackenzie S. An epigenetic breeding system in soybean for increased yield and stability., 2018, 16: 1836–1847.
[4] Inoue K, Araki T, Endo M. Circadian clock during plant development., 2018, 131: 59–66.
[5] McClung C R. Plant circadian rhythms., 2006, 18: 792–803.
[6] Lu S, Zhao X, Hu Y, Liu S, Nan H, Li X, Fang C, Cao D, Shi X, Kong L, Su T, Zhang F, Li S, Wang Z, Yuan X, Cober E R, Weller J L, Liu B, Hou X, Tian Z, Kong F. Natural variation at the soybeanlocus improves adaptation to the tropics and enhances yield., 2017, 49: 773–779.
[7] Inoue K, Araki T, Endo M. Circadian clock during plant development., 2018, 131: 59–66.
[8] Green R M, Tobin E M. Loss of the circadian clock-associated protein 1 inresults in altered clock-regulated gene expression., 1999, 96: 4176–4179.
[9] Bendix C, Marshall C M, Harmon F G. Circadian clock genes universally control key agricultural traits., 2015, 8: 1135–1152.
[10] Gray J A, Shalit-Kaneh A, Chu D N, Hsu P Y, Harmer S L. The REVEILLE clock genes inhibit growth of juvenile and adult plants by control of cell size., 2017, 173: 2308–2322.
[11] Xie Q, Wang P, Liu X, Yuan L, Wang L, Zhang C, Li Y, Xing H, Zhi L, Yue Z, Zhao C, McClung C R, Xu X. LNK1 and LNK2 are transcriptional coactivators in thecircadian oscillator., 2014, 26: 2843–2857.
[12] Müller N A, Zhang L, Koornneef M, Jiménez-Gómez J M. Mutations inandcaused light-conditional clock deceleration during tomato domestication., 2015, 115: 7135–7140.
[13] Müller N A, Wijnen C L, Srinivasan A, Ryngajllo M, Ofner I, Lin T, Ranjan A, West D, Maloof J N, Sinha N R, Huang S, Zamir D, Jiménez-Gómez J M. Domestication selected for deceleration of the circadian clock in cultivated tomato., 2016, 48: 89–93.
[14] 高耀辉, 马斌, 肖凤洁, 魏光普. CRISPR/Cas9系统在园林植物中的研究展望. 北方园艺, 2019, (15): 133–140. Gao Y H, Ma B, Xiao F J, Wei G P. Prospect of research on CRISPR/Cas9 system in garden plants., 2019, (15): 133–140 (in Chinese with English abstract).
[15] 暴会会, 尹竹君, 王少坤, 马瑞红, 谢俊俊, 张杰, 杨正安. CRISPR-Cas9系统在蔬菜育种上应用研究进展. 江西农业学报, 2019, 31(7): 38–44.Bao H H, Yin Z J, Wang S K, Ma R H, Xie J J, Zhang J, Yang Z A. Research advances in application of CRISPR/Cas9 system in vegetable breeding., 2019, 31(7): 38–44 (in Chinese with English abstract).
[16] Zhang J, Zhu Z, Yue W, Li J, Chen Q, Yan Y, Lei A, Hua J. Establishment of CRISPR/Cas9 mediated knock-in system for porcine cells with high efficiency., 2019, 189: 26–36.
[17] Lin C Y, Su Y H. Genome editing in sea urchin embryos by using a CRISPR/Cas9 system., 2016, 409: 420–428.
[18] Cai Y, Wang L, Chen L, Wu T, Liu L, Sun S, Wu C, Yao W, Jiang B, Yuan S, Han T, Hou W. Mutagenesis ofandmediated by CRISPR/Cas9 contributes for expanding the regional adaptability of soybean., 2020, 18: 298–309.
[19] Bao A, Chen H, Chen L, Chen S, Hao Q, Guo W, Qiu D, Shan Z, Yang Z, Yuan S, Zhang C, Zhang X, Liu B, Kong F, Li X, Zhou X, Tran L P, Cao D. CRISPR/Cas9-mediated targeted mutagenesis ofgenes alters plant architecture in soybean., 2019, 19: 131.
[20] Do P T, Nguyen C X, Bui H T, Tran L T N, Stacey G, Gillman J D, Zhang Z J, Stacey M G. Demonstration of highly efficient dual gRNA CRISPR/Cas9 editing of the homeologousandgenes to yield a high oleic, low linoleic and α-linolenic acid phenotype in soybean., 2019, 19: 311.
[21] 曾栋昌, 马兴亮, 谢先荣, 祝钦泷, 刘耀光. 植物CRISPR/Cas9多基因编辑载体构建和突变分析的操作方法. 中国科学: 生命科学, 2018, 48: 783–794.Zeng D C, Ma X L, Xie X R , Zhu Q L , Liu Y G. A protocol for CRISPR/Cas9-based multi-gene editing and sequence decoding of mutant sites in plants., 2018, 48: 783–794 (in Chinese with English abstract).
[22] 侯智红, 吴艳, 程群, 董利东, 芦思佳, 南海洋, 甘卓然, 刘宝辉. 利用CRISPR/Cas9技术创制大豆高油酸突变系. 作物学报, 2019, 45: 839–847. Hou Z H, Wu Y, Cheng Q, Dong L D, Lu S J, Nan H Y, Gan Z R, Liu B H. Creation of high oleic acid soybean mutation plants by CRISPR/Cas9., 2019, 45: 839–847 (in Chinese with English abstract).
[23] Cheng Q, Dong L D, Gao T J, Liu T F, Li N H, Wang L, Chang X, Wu J J, Xu P F, Zhang S Z. The bHLH transcription factorfacilitates resistance toin., 2018, 69: 2527–2541.
[24] 解莉楠, 宋凤艳, 张旸. CRISPR/Cas9系统在植物基因组定点编辑中的研究进展. 中国农业科学, 2015, 48: 1669–1677. Xie L N, Song F Y, Zhang Y. Progress in research of CRISPR/Cas9 system in genome targeted editing in plants., 2015, 48: 1669–1677 (in Chinese with English abstract).
[25] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna J A, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity., 2012, 337: 816–821.
[26] Bhaya D, Davison M, Barrangou R. CRISPR-Cas systems in bacteria and archaea: versatile small RNAs for adaptive defense and regulation., 2011, 45: 273–297.
[27] Wiedenheft B, Sternberg S H, Doudna J A. RNA-guided genetic silencing systems in bacteria and archaea., 2012, 482: 331–338.
[28] Nekrasov V, Staskawicz B, Weigel D, Jones J D, Kamoun S. Targeted mutagenesis in the model plantusing Cas9 RNA-guided endonuclease., 2013, 31: 691–693.
[29] Shan Q, Wang Y, Li J, Zhang Y, Chen K, Liang Z, Zhang K, Liu J, Xi J J, Qiu J L, Gao C. Targeted genome modification of crop plants using a CRISPR-Cas system., 2013, 31: 686–688.
[30] Li J F, Norville J E, Aach J, McCormack M, Zhang D, Bush J, Church G M, Sheen J. Multiplex and homologous recombination-mediated genome editing in Arabidopsis andusing guide RNA and Cas9., 2013, 31: 688–691.
[31] Feng Z Y, Zhang B T, Ding W, Liu X D, Yang D L, Wei P L, Cao F Q, Zhu S H, Zhang F, Mao Y F, Zhu J K. Efficient genome editing in plants using a CRISPR/Cas system., 2013, 23: 1229–1232.
[32] Miao J, Guo D, Zhang J, Huang Q, Qin G, Zhang X, Wan J, Gu H, Qu L J. Targeted mutagenesis in rice using CRISPR-Cas system., 2013, 23: 1233–1236.
[33] Jiang W, Zhou H, Bi H, Fromm M, Yang B, Weeks D P. Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis, tobacco, sorghum and rice., 2013, 41: e188.
[34] Jia H, Wang N. Targeted genome editing of sweet orange using Cas9/sgRNA., 2014, 9: e93806.
[35] Fauser F, Schiml S, Puchta H. Both CRISPR/Cas-based nucleases and nickases can be used efficiently for genome engineering in., 2014, 79: 348–359.
[36] 梁丹, 吴宇轩, 李劲松. CRISPR-Cas9技术在干细胞中的应用. 生命科学, 2015, 27(1): 93–98. Liang D, Wu Y X, Li J S. Progress of CRISPR-Cas9 in stem cell research., 2015, 27(1): 93–98 (in Chinese with English abstract).
[37] Chen K, Gao C. Targeted genome modification technologies and their applications in crop improvements., 2014, 33: 575–583.
[38] Curtin S J, Zhang F, Sander J D, Haun W J, Starker C, Baltes N J, Reyon D, Dahlborg E J, Goodwin M J, Coffman A P, Dobbs D, Joung J K, Voytas D F, Stupar R M. Targeted mutagenesis of duplicated genes in soybean with zinc-finger nucleases., 2011, 156: 466–473.
[39] Cai Y, Chen L, Liu X, Sun S, Wu C, Jiang B, Han T, Hou W. CRISPR/Cas9-Mediated genome editing in soybean hairy roots., 2015, 10: e0136064.
Identification of CRISPR/Cas9 knockout targets and tissue expression analysis of circadian clock genes,, andin soybean
GAN Zhuo-Ran, SHI Wen-Qian, LI Yong-Li, HOU Zhi-Hong, LI Hai-Yang, CHENG Qun, DONG Li-Dong, LIU Bao-Hui, and LU Si-Jia*
School of Life Sciences, Guangzhou University, Guangzhou 510006, Guangdong, China
Circadian clock genes play an important role in improving crop yield.,,,, andare important circadian clock genes in plants. Homologous genes of,,,, andin soybean were found by evolutionary tree analysis. These genes were expressed in soybean roots, stems and leaves. The knockout vectors of these genes were successfully constructed by using CRISPR/Cas9 gene editing technology. Transformation system of soybean root hair and RT-PCR were used to identify 13 genes targets effectively. The results of this study provide important target informations for further obtaining soybean mutant materials, and a foundation for further studying the function of circadian clock genes.
soybean; gene knockout; clock gene; CRISPR/Cas9
10.3724/SP.J.1006.2020.94169
本研究由国家自然科学基金项目(31771815, 31701445)和作物遗传与种质创新国家重点实验室开放课题(ZW201901)资助。
This study was supported by the National Natural Science Foundation of China (31771815, 31701445) and the State Key Laboratory of Crop Genetics & Germplasm Enhancement (ZW201901).
芦思佳, E-mail: lusijia@gzhu.edu.cn
E-mail: gggzzr@126.com
2019-11-07;
2020-03-24;
2020-04-07.
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20200404.0851.002.html

