基于MAGIC群体的水稻抽穗期和产量相关性状全基因组关联分析
魏秀彩 刘金栋 刘利成 黎用朝 潘孝武 董铮 刘文强 熊海波 闵军 李小湘 , *
(1湖南大学研究生院隆平分院, 长沙 410125; 2湖南省农业科学院 水稻研究所, 长沙 410125; 3 中国农业科学院 农业基因组研究所, 深圳 518124;4农业部长江中下游籼稻遗传育种重点实验室, 长沙 410125; #共同第一作者;*通信联系人, E-mail: xiaoxiang66196@126.com)
水稻是世界上最重要的粮食作物之一,养育着全世界半数以上的人口。我国是世界上最大的稻米生产和消费国之一。近 30年来,由于全球人口快速增长,气候急剧变化、城市化建设和盐碱地面积扩张导致人均可耕地面积迅速减少,严重威胁全球粮食安全。预计到 2030年,我国粮食缺口将达到1.5亿t。高产是当前和今后水稻育种的主导目标[1]。虽然化肥的大量施用可有效提升稻米产量,但同时也会对自然生态环境带来严重威胁。发掘水稻产量相关性状位点及其关联标记,筛选携带大量优异等位基因的重要种质,通过分子标记辅助选择的方法选育高产品种,是提升水稻产量,维护粮食安全最为经济、有效且环保的方法。
水稻产量性状是由多基因控制的复杂数量性状,受环境影响显著。水稻的产量性状主要包括单株有效穗数、单穗粒数和千粒重。抽穗期是水稻重要农艺性状之一,抽穗期(播种至抽穗期的时间)是水稻生育期长短的直接反应。适宜的抽穗期可使植株在特定的生态条件下最大程度地利用当地的光热资源,决定了水稻适宜种植的区域和时间[2]。迄今为止,已有多项研究针对水稻抽穗期和产量相关性状遗传机制解析展开。在水稻抽穗期方面,Ehd1、Hd3a、RFT1、RID1、SDG724、Ehd4 等已被克隆QTL/基因能够诱导植株提早抽穗,DTH7、Ghd7、EL1和DHD1等负调控因子能延迟水稻开花。关于产量构成因子,正向调控水稻产量的有GIF1、GS5、GS2、GLW7、DEP1、DST、GNP1 和 NOG1等;负向调控水稻产量的有PROG1、D53、GS3、GW2、qGL3、qTGW3和An-1等。除此之外,还有数十个抽穗期和产量相关的基因/QTL被发掘[3]。然而,水稻产量性状遗传机制十分复杂,尚未完全解析。亟需发掘新的产量相关基因为育种提供参考。
连锁分析和全基因组关联分析(genome-wide association analysis,GWAS)是挖掘微效基因的两种主要方法。长期以来,水稻遗传学研究主要采用双亲群体开展连锁分析。连锁分析以双亲群体为材料,遗传背景简单且定位结果可靠,但也存在一定局限性。连锁分析具有构建双亲群体耗时长,成本偏高,双亲群体遗传背景单一,无法反映自然界广泛存在的遗传变异的弊端。相较于连锁分析,基于连锁不平衡的关联分析则具有成本低、效率高且来源广泛,可反映自然界广泛存在的遗传变异的优点。但是 GWAS也有采用材料存在显著的亲缘关系和群体结构,关联结果假阳性高且稀有等位变异检出率低、准确性低的缺点[4]。近年来,多亲本重组自交系群体(multi-parent advanced generation inter-crosses population, MAGIC)的出现则在一定程度上弥补了二者的缺陷。MAGIC群体是由多亲本的杂交和多代的互交构建而来的新一代作图群体。Bandillo等[5]利用来自于中国、哥伦比亚、国际水稻研究所等国家或组织的8个籼稻和8个粳稻优良品种(系)经过多年多代杂交构建了 4个 MAGIC群体,即籼稻MAGIC群体(8个籼稻亲本)、MAGIC Plus群体(8个籼稻亲本和2个额外的8籼稻亲本互交的F1)、粳稻MAGIC群体(8个粳稻亲本)、Global MAGIC群体(16个亲本:8个籼稻和8个粳稻亲本)。随后,利用这些MAGIC群体,针对抽穗期、株高、粒长、粒宽、直链淀粉含量、耐盐性、耐淹性、稻瘟病及白叶枯病抗性等方面进行了连锁分析[5-7]。这些群体在长沙抽穗期和产量相关性状的表现型评价及全基因组关联分析未见报道。
本研究以 Bandillo等[5]所构建的 8亲本MAGIC-Hei群体为材料,连续两年在湖南省水稻研究所长沙马坡岭试验田开展抽穗期和产量相关性状鉴定,基于基因分型(GBS)测序技术获得基因型,通过 GWAS发掘水稻抽穗期和产量相关性状关联位点,筛选携带有利等位基因的优良株系,以期为分子标记辅助育种提供新基因和优异种质。
1 材料与方法
1.1 材料种植及农艺性状调查
将MAGIC-Hei群体的8个亲本[Fedearroz 50,Shan-Huang Zhan-2 (SHZ-2),IR64633-87-2-2-3-3(PSBRc82),IR4630-22-2-5-1-3,IR45427-2B-2-2B-1-1,IR84196-12-32(SAMBHA MAHSURI+SUB1),IR77298-14-1-2-10,IR77186-122-2-2-3(PSBRc 158)]及其395个株系MAGIC群体分别于2017年和2018年5月底种植在湖南省水稻研究所马坡岭试验基地。播种25 d后单本移栽,每份材料种植3行,10株/行,株行距为20.0 cm×26.4 cm,两次重复。记载抽穗期(单株系全小区50%稻穗抽穗时),田间肥水管理按照当地一季晚稻正常种植管理技术进行,及时防治病虫害。
水稻齐穗后30 d,按抽穗期不同,分批收获每个材料中间行的 3株稻穗,分别装袋,两次重复。收获后将穗子置于45℃烘箱中烘72 h,对单株有效穗数(number of tillers per plant,NTP)、每穗粒数(grain number per panicle,GNPP)、结实率(seed setting rate,SR)、千粒重(1000-grain weight,GW)、单株产量(grain yield per plant,YD)等表型性状进行鉴定。由于MAGIC群体有些株系没稳定有分离,有些株系在长沙不能正常抽穗,未能收集到所有株系表型数据,只收集到234份(MAGIC群体226份和8个亲本)材料表型数据。
1.2 SNP基因型分析
Bandillo等[5-7]通过GBS测序技术对8个亲本和MAGIC-Hei群体395个株系进行基因型分型。待种子萌发后,取新鲜叶片,按CTAB法提取DNA,通过琼脂糖凝胶电泳和紫外分光光度计对DNA的纯度和核酸浓度进行检测。用限制性内切酶ApeKⅠ对基因组进行酶切,采用Illumina Hiseq测序平台进行双末端(Paired-end,PE)测序,测序完成后参照日本晴基因组筛选SNP标记。
1.3 数据分析
用Microsoft Excel 2019整理表型数据,以2个重复的平均值作为相应性状的后续分析表型值,并计算 MAGIC-Hei群体的平均值、标准差、变幅、变异系数。利用DPS 14.10对抽穗期和产量性状的相关性进行分析,两年间表型的差异显著性分析采用t检验。
1.4 关联分析
利用基于R语言的GAPIT包中的MLM(Mixed linear model)模型,以主成分分析(PCA)和亲缘关系(K)作为协变量,结合SNP基因型数据,对MAGIC-Hei群体的抽穗期和产量相关性状进行关联分析。当P≤0.00001(-lgP=5)时,认为该标记与性状显著关联。关联分析结果的可视化通过 R 3.2.2的 CMplot软件包绘制曼哈顿图(Manhattan Plot)和Q-Q图(Quantile-quantile plot)来实现。用R2评价关联位点对表型性状的总体贡献率[8],QTL的命名参照McCouch等[9]提出的方法。
2 结果与分析
2.1 亲本和 MAGIC群体抽穗期及产量性状的表现及相关性分析
分别对2017年和2018年两个环境下的亲本及群体的抽穗期及产量性状进行统计。2017年8个亲本抽穗期变幅为9~125 d,MAGIC-Hei群体的抽穗期为83~106 d,平均抽穗期为93.0 d;2018年8个亲本抽穗期变幅为0~122 d,MAGIC-Hei群体的抽穗期为76~114 d,平均抽穗期100.0 d。两年结果表明抽穗期在不同株系间差异明显(表 1)。在2017年 8个亲本单株有效穗数变幅为 12~21,MAGIC-Hei群体的单株有效穗数变幅为 10~27,平均单株有效穗数为17.25;2018年8个亲本单株有效穗数变幅 11~18,MAGIC-Hei群体的单株有效穗数变幅为7~22,平均单株有效穗数为13.68。2017年 8个亲本单株产量变幅 24.07~46.49 g,MAGIC-Hei群体的单株产量变幅为 11.56~62.96 g,平均单株产量为34.92 g;2018年8个亲本单株产量变幅为23.52~37.77 g,MAGIC-Hei群体的单株产量变幅为1.14~67.73 g,平均单株产量为27.81 g。产量性状结果表明,MAGIC-Hei群体的所有性状均分离明显,呈连续分布,说明水稻产量性状属于典型的数量性状遗传。
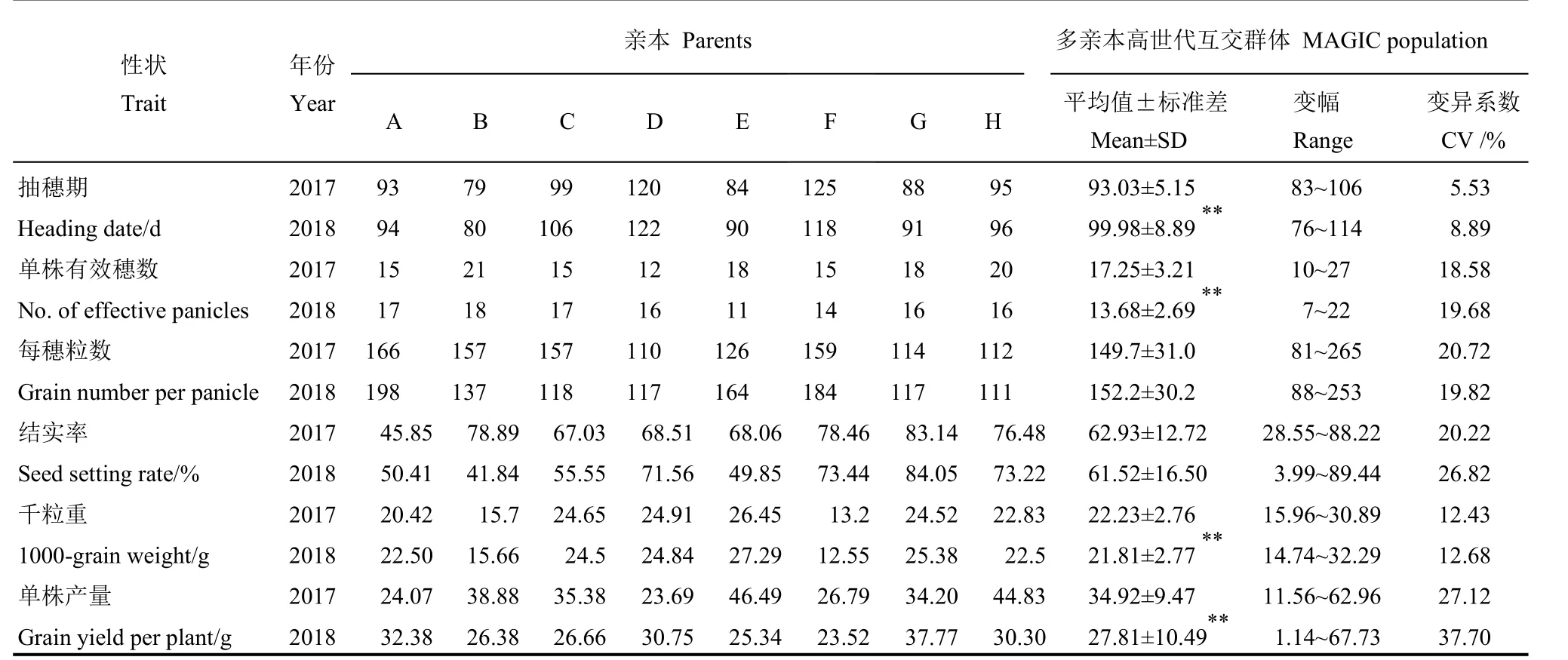
表1 亲本和MAGIC群体在不同环境下抽穗期和产量相关性状的表现及变异系数Table 1. Performance of parents and MAGIC populations for heading date and yield related traits.
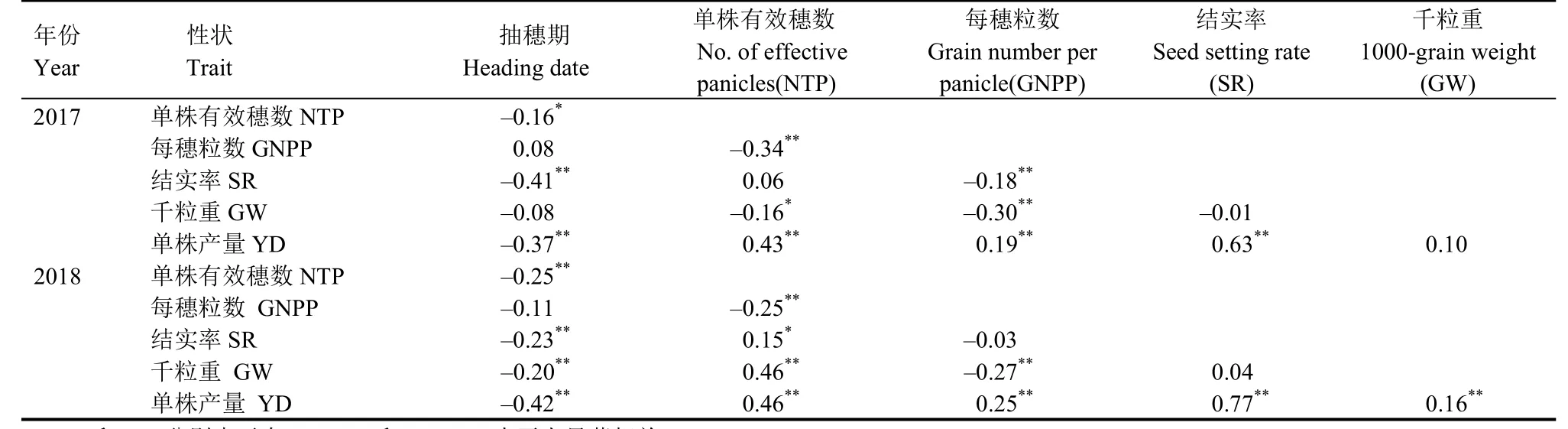
表2 MAGIC群体抽穗期和产量性状间的相关性分析Table 2. Correlation analysis on heading date and yield traits in MAGIC population.
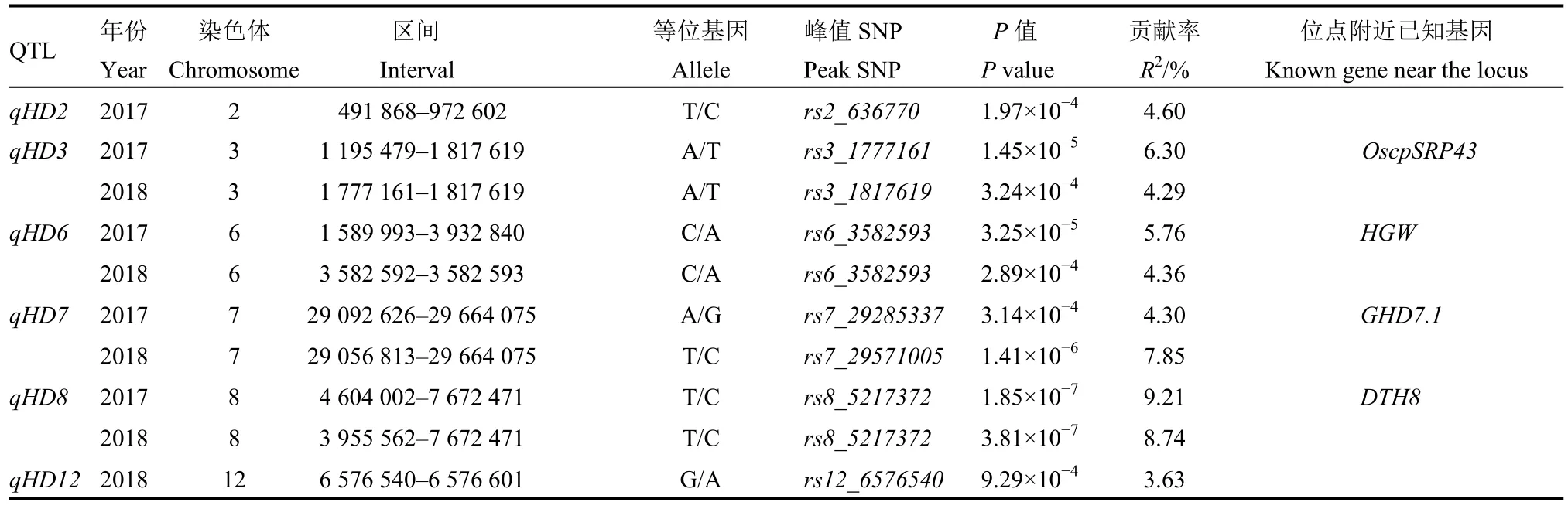
表3 全基因组关联分析检测到与抽穗期显著关联的位点Table 3. Loci significantly associated with heading date detected by genome-wide association analysis.
方差分析表明,每穗粒数、结实率年度间无显著差异,但2017年抽穗期极显著短于2018年,单株有效穗数、千粒重和单株产量则极显著高于2018年。
性状间的相关分析结果表明(表 2),每穗粒数与单株有效穗数、千粒重呈极显著负相关,每穗粒数与单株有效穗数两年相关系数分别为-0.34和-0.25,每穗粒数与千粒重两年相关系数分别为-0.30和-0.27。
2.2 抽穗期及产量性状关联分析结果
在2017和2018两个环境下共计检测到控制抽穗期的 QTL 6个,分别位于第 2、3、6、7、8和12染色体上,对表型的贡献率变幅为 3.63%~9.21%(表 3)。其中,4 个 QTL(qHD3、qHD6、qHD7、qHD8)在两年中均被检测到,对表型贡献率最高的是qHD8,在2017年环境下解释9.21%的表型贡献率。对表型贡献率最小的是qHD3,2018年的贡献率为4.29%。
在2017和2018环境下共计检测到控制产量性状的QTL 20个,包括控制单株有效穗数、每穗粒数、结实率、千粒重和单株产量的位点分别为3、4、3、5和5个,单个QTL对表型的贡献率范围5.13%~10.68%。在两年间共定位的QTL有4个(qNTP9、qGNPP3、qSR8和qGW3),分别控制单株有效穗数、每穗粒数、结实率和千粒重,对表型的平均贡献率分别为5.80%、8.59%、10.29%和7.07%(表4)。
2.3 优良株系的筛选
根据抽穗期和产量性状表型数据,结合SNP基因型筛选到5个携带较多有利等位基因的优良株系,分别是 GID4170482、GID4172993、GID4173982、GID4173992和GID4171060(表5)。其中,双基因聚合优异株系 GID4170482,两年单株有效穗数分别为27和22,两年的抽穗期分别为87和92 d,携带有控制单株有效穗数的qNTP9,该位点前后SNP基因型为 TGCGC,同时携带控制抽穗期的qHD6,该位点前后 SNP基因型为 TGCCC。材料GID4172993两年每穗粒数的平均值为237,两年抽穗期平均值为91 d。材料GID4173982两年的结实率均达到81.27%以上,材料GID4173992两年的千粒重均达到26.88 g以上,材料GID4171060两年单株产量的平均值为57.32 g。这些优良株系可用于水稻高产育种。
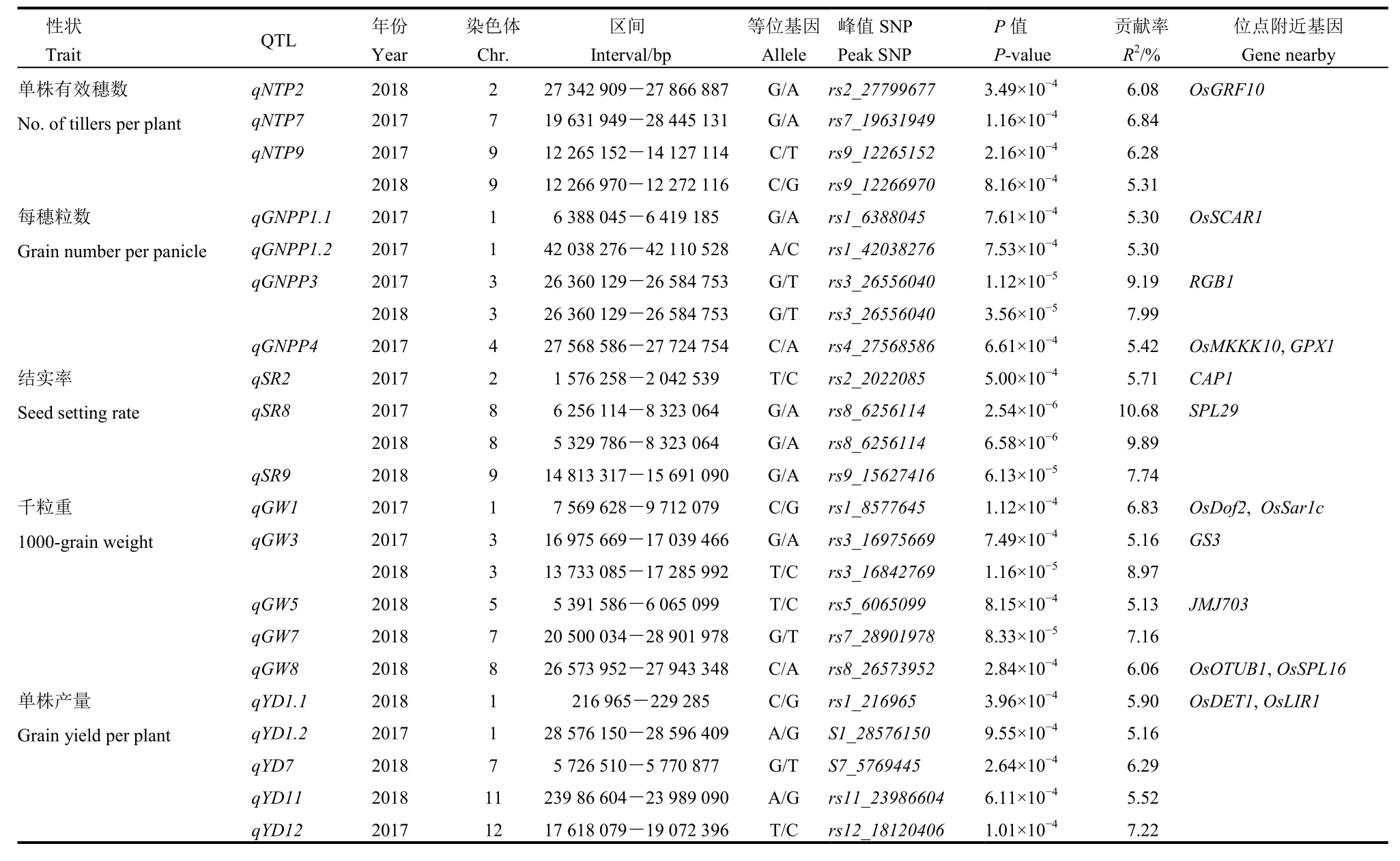
表4 全基因组关联分析检测到与产量性状显著关联的位点Table 4. Loci significantly associated with the yield traits detected by genome-wide association analysis.
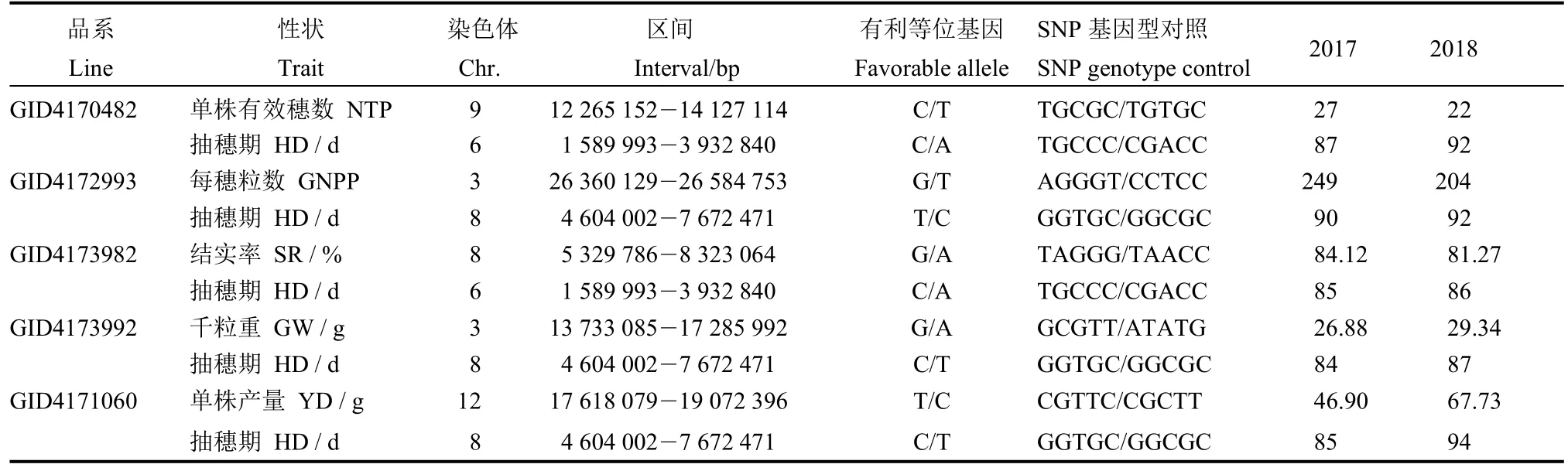
表5 双基因(抽穗期和产量因子)聚合的优异株系Table 5. Excellent lines with two genes (heading date and yield factors) polymerization.
3 讨论
水稻是我国的第一大粮食作物,高产始终是水稻育种进程的最重要目标之一。水稻抽穗开花期遇极端高温天气不仅影响水稻产量,同时会降低稻米品质[10],适宜的抽穗期可使植株在特定的生态条件下最大程度地利用当地的光热资源,也可使水稻开花灌浆期避开极端高温天气,降低高温天气对水稻产量和品质的影响。因此,亟需发掘新的水稻抽穗期和产量相关性状关联位点,为水稻高产育种利用。
相关性分析结果表明,抽穗期与结实率、单株产量呈极显著负相关,推测这可能是因为部分材料在抽穗开花期遇高温天气。材料 GID4173666、GID4174244、GID4171059等在始穗期至齐穗期,遭遇高温天气(2018年8月7日-8月14日,日最高温 36.5℃~38.1℃),导致结实率和单株产量明显下降;GID4173365、GID4171060、GID4171107等材料两年在抽穗扬花期均遇上了高温,但结实率都高。由此推测本研究两年重复定位到的某些水稻抽穗期和产量性状相关基因很可能也是温度钝感相关基因。
本研究在两年环境下分别检测到6个和20个与抽穗期和水稻产量相关的QTL,两年均被检测到的QTL有8个。对于抽穗期,两年均定位到4个QTL(qHD3、qHD6、qHD7、qHD8)分别覆盖已克隆基因 OscpSRP43、HGW、GHD7.1、DTH8[11-14]。Raghavan等[5]对AGIC-Hei群体关于抽穗期做了关联分析,检测到8个与水稻抽穗期显著关联的QTL,分布于第 1、3、4、5、6、7和 8染色体上,其中定位于第3和8染色体的QTL与本研究结果一致,在本研究中连续两年均被检测到,而其他 QTL与本研究结果没有重叠,可能是由于受不同生态区域光周期、温度等环境因素影响。
与前人结果比较,关于水稻产量性状,我们发现 11个定位区间内或附近已有克隆的基因。qGNPP1.1、qGNPP4、qGNPP4、qGW1 在 2017 年被检测到的,区间内包含已克隆基因 OsSCAR1、OsMKKK10、OsGPX1、CAP1、OsDof2 和OsSar1c[15-20]。qNTP2、qYD1.1、qGW5 和 qGW8 在2018年被检测到,覆盖已克隆基因 OsGRF10、OsDET1、OsLIR1、JMJ703、OsOTUB1、OsSPL16[21-26]。其中,qGNPP3、qSR8、qGW3在两年中能同时被检测到,分别覆盖已克隆QTL/基因RGB1、SPL29、GS3[27-29]。RGB1是ABA响应和干旱适应性的一个正调控因子,位于第3染色体,RGB1敲除植株表现出不育籽粒数目增多、植株矮化、籽粒变小、结实率和单株产量降低[27]。SPL29编码尿苷二磷酸-N-乙酰葡糖胺焦磷酸化酶(UAP1),spl29突变体的株高、穗长、总粒数、灌浆率、结实率以及千粒重均显著降低[28]。GS3位于水稻第3染色体近着丝粒区,在调节籽粒和器官大小中发挥负调节子的功能,是控制水稻粒重和粒长的主效QTL,同时也是控制水稻粒宽和籽粒充实度的微效QTL[29]。本研究两年均检测到的 QTL相对较少,究其原因,可能是控制抽穗期和产量相关性状的 QTL易受环境的影响,
导致两年检测到的QTL数量不同。
除上述已有克隆基因的区间外,本研究定位结果中还有多个与水稻抽穗期和产量性状相关的新位点,分别是与水稻抽穗期显著关联的 2个QTL(qHD2、qHD12),与水稻产量性状显著关联的QTL(qNTP7、qNTP9、qGNPP1.2、qSR9、qGW7、qYD1.2、qYD7、qYD11、qYD12)。其中,与单穗粒数显著关联的位点qNTP9,被定位于第9染色体的12-14 Mb区间内,在两年均被检测到,说明该位点受环境影响较小,可用于进一步的精细定位和基因克隆。
本研究采用一个8亲本的MAGIC群体,基于GBS测序的方法,对抽穗期和产量相关性状进行关联分析,发掘到一批关联显著标记和农艺性状优良且含有优异等位基因的优异种质,为MAS育种方法加速水稻高产育种进程提供了基因资源。
谢辞: 感谢国际水稻研究所叶国友博士提供MAGIC群体及其基因型,感谢本实验室金建楚和赵文锦协助抽穗期的调查。

