胃癌中异常表达剪接因子的生物信息学分析及功能
张爱东,史 娟
中国医学科学院 北京协和医学院 基础医学研究所医学分子生物学国家重点实验室,北京 100005
胃癌作为一种常见的消化系统恶性肿瘤之一,死亡率一直居高不下。据调查,胃癌发病率在中国所有癌症中居第2位,在全世界的发病率居第5位,致死率在所有肿瘤中居第3位[1]。胃癌患者的年龄主要集中在40~70岁,其他年龄段患者人数较少,但近年来青年人的胃癌发病率呈上升趋势[2]。早期胃癌患者行根治性切除术后的5年生存率可达90%,但是由于早期诊断中缺少有效的分子标志物等原因,我国胃癌患者就诊时约80%已到晚期,导致疾病进一步发展、肿瘤转移,甚至步入终末期[3]。
选择性剪接的发生与细胞的生理病理、发育调控甚至疾病的发生有关[4]。有研究表明,在癌症发生发展过程中,选择性剪切的发生以及在这个过程中产生的异构体蛋白的功能,对癌症的诊断和治疗都具有非常重要的意义[5]。选择性剪接对癌基因以及抑癌基因的活性和异常表达均有调控作用,从而对癌症的发生及恶化产生一定影响。研究还表明,选择性剪接可以影响蛋白质结构域家族,这些蛋白质家族在肿瘤中经常发生突变并可能破坏癌症相关途径中的蛋白质-蛋白质相互作用,该发现为癌症的治疗提供了新思路[6]。富含丝氨酸苏氨酸蛋白家族(SR)是选择性剪接调节的重要因子之一,有多个拷贝的RNA识别基序(RNA recognition motif,RRM)。富含丝氨酸苏氨酸蛋白家族中的剪接因子10(SR Splicing Factor,SRSF10)基因作为SR蛋白家族中重要的成员之一,具有经典的SR蛋白结构域,是一个重要的剪切因子。
癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库是由美国国家癌症研究院(National Cancer Institute,NCI)和美国国家人类基因组研究院(National Human Genome Research Institute,NHGRI)在2006年共同合作承担开展的TCGA数据库计划[7-9],旨在编目和发现主要的致癌基因组改变,以创建癌症基因组谱的全面“图谱”。 到目前为止,TCGA研究人员通过大规模基因组测序和综合多维分析,分析了30多种人类肿瘤,对癌症类型的研究以及全面的泛癌分析已经扩展了肿瘤发生的当前知识。该项目的一个主要目标是提供公开的数据集,以帮助改进诊断方法、治疗标准,并最终预防癌症[10]。本研究采用生物信息学方法分析了胃癌中有表达差异的剪接因子,研究了异常表达的选择性剪接因子SRSF10对胃癌细胞表型的影响,以期为胃癌的早期诊断、预后判断以及靶向治疗提供新的研究方向。
材料和方法
实验细胞和材料SGC-7901胃癌细胞由本实验室保存,在含有双抗及10%胎牛血清的RPMI-1640培养基中培养,培养条件为37 ℃、5% CO2。Lipofectamine 2000、TRIzol(美国赛默飞世尔科技有限公司),RPMI-1640培养基、胎牛血清(中国HyClone Laboratories公司),Anti-SRSF10抗体(美国Abcam公司),Anti GAPDH抗体(中国Proteintech公司),HRP标记的山羊抗兔抗体、HRP标记的山羊抗小鼠抗体(中杉金桥生物技术有限公司),MTS、siRNA(锐博生物有限公司)。
胃癌及癌旁组织RNA-seq数据差异表达分析在TCGA数据库中下载胃癌及癌旁RNA-seq数据,使用Perl语言处理RNA-seq数据格式的脚本[11],在Linux服务器上运行Perl脚本提取并整合RNA-seq数据[12],以获得胃癌和癌旁组织的RNA-seq矩阵。同时在ENSEMBLE数据库中下载Homo_sapiens.GRCh38.88.chr.gtf人类基因组注释文件[13],并使用人类基因组注释文件将矩阵中的整体id转换为基因id,最后获得含基因id的RNA-seq矩阵。利用R语言以及R软件包对胃癌中差异表达基因进行分析[14],绘制差异基因表达的火山图。利用Perl脚本 gene.namelistto info.pl、构建的选择性剪接因子库文件ASFs.txt以及得到的表达差异所有基因文本,依据|logFC|≥0.5,P<0.05条件筛选出胃癌中表达差异的选择性剪接因子[15](FC表示倍数变化;logFC表示对FC取以2为底的对数)。在线网站gepia(http://gepia.cancer-pku.cn/index.html)输入SRSF10按照教程进行在线分析。
细胞转染技术转染前1d将细胞铺到6孔板中,密度40%~60%,置于5%CO2、37 ℃培养箱中过夜培养。当细胞密度达到80%左右时,更换无血清培养基,饥饿细胞2 h。转染试剂的准备:用高温高压过的2个1.5 ml EP管配制A液和B液。A液:100 μl无血清培养基加入6~8 μl转染试剂Lipofectamine 2000,混匀。B液:100 μl无血清培养基加入20 pmol siRNA或者4 μg DNA,混匀。A和B液室温放置5 min后混合,静置20 min,使得DNA或者siRNA与脂质体形成复合物,将该复合物加入到细胞培养基中,缓慢温和地混匀。放入培养箱中继续培养。待4 h后将6孔板的液体替换成完全培养基,继续培养进行后续实验。
TRIZOL法提取RNA经过处理后的细胞培养到一定数量时,用冷PBS清洗细胞2次,加入1 ml Trizol吹打细胞收集到1.5 ml EP管中,再向管中加入200 μl三氯甲烷,剧烈混匀,冰上放置10 min。4℃离心机中,15 402×g离心20 min。取上层水层到新的无RNA酶的EP管,加入等量的异丙醇,混匀,冰上放置20 min,4 ℃离心机中15 402×g离心30 min。弃上清,加入75%的酒精(用DEPC水配制)震荡混匀,于4℃离心机中,12 579×g离心8 min。弃去上清,室温放置20 min,加入30~50 μl DEPC水在EP管中溶解RNA。取2 μl RNA溶液在NANODROP 2000C仪器上测量提取的RNA浓度,并标注在EP管壁。
提取细胞总蛋白待细胞经过处理后,数量达到一定数目时,用配置好的PBS清洗细胞2~3次,加入适当胰酶消化细胞(不同类型细胞消化时间不同),然后加入完全培养基终止消化,用滴管吹打细胞将细胞悬液加入离心管中,然后在离心机里770 ×g离心5 min,弃上清,加入细胞裂解液和PMSF(100∶1)混合液(该混合液现用现配,因为PMSF在水溶液中易降解)重悬细胞,裂解1 h左右。待细胞裂解充分后,在4℃离心机中,15 402×g离心20 min,取上清到新的EP管中。
细胞划痕实验利用直尺,以Marker笔在6孔板背面均匀划竖线,每隔0.5~1.0 cm划1道,每孔至少穿过4条线。在每个孔中加入5×105个细胞,具体细胞数量因细胞种类不同而不同,过夜能铺满。次日用枪头比着直尺,尽量垂直6孔板背面竖线,枪头要垂直,不能倾斜。用PBS清洗细胞3次,去除漂浮的细胞,加入不含血清的培养基培养细胞。放入37℃、5%CO2培养箱中,分别在0、6、12、24、48、72 h取样拍照,每个时间点的数据重复测量3次。
MTS法测细胞增殖曲线将细胞均匀铺于96孔板中,每孔种2000个细胞。6 h待细胞贴壁后,避光配制MTS试剂,1 ml电子偶联化合物染料中加入20 μl四氮唑,混匀后,按每孔15 μl加入96孔板中,于37℃、5%CO2培养箱中避光孵育2 h后,在492 nm处测OD值。检测时间点分别为0、24、48、72、96 h,每个时间点的数据重复测量3次。
Transwell实验在Transwell小室中加入(1~2)×105个细胞,上室加入500 μl无血清培养基,下室内加入600 μl含10% FBS培养基。待细胞迁移14 h后,弃去滤膜上下室的培养基,用预热的PBS清洗小室,重复1次。在24孔板的1个孔中加入4%多聚甲醛,将小室移动到该孔中,使滤膜下表面浸没在4%多聚甲醛中,固定15~20 min。弃去固定液,倒置Transwell小室20 min,自然风干。将Transwell小室浸没在结晶紫溶液中,染色20~60 min。蒸馏水清洗Transwell小室下表面,风干后用棉球擦去小室内未迁移的细胞,在倒置显微镜下计数。
统计学处理采用GraphPad Prism7软件[16],数据均由多次(大于等于3次)重复实验得出,以均数±标准差表示,两样本比较采用独立样本t检验,P<0.05为差异有统计学意义。
结 果
获得TCGA数据库中胃癌及癌旁RNA-seq数据下载TCGA数据库中胃癌及癌旁组织RNA-seq数据,得到407个胃癌及癌旁样本数据,其中胃癌样本375个,癌旁样本32个。
形成剪接因子库通过统计已经报道的剪接因子以及对收录剪接因子的在线网站ASTD(ftp://ftp.ebi.ac.uk/pub/databases/astd/altsplice/human/latest)的查阅,组建成了含有975个剪接因子的剪接因子库,部分剪接因子见表1。
胃癌中差异表达的选择性剪接因子对胃癌中表达差异的选择性剪接因子进行筛选,共得到胃癌差异表达的剪接因子48个,其中,表达上调35个,表达下调13个(表2、图1)。
SRSF10在胃癌组织中表达上调在TCGA可视化数据库网站gepia在线分析了SRSF10基因在胃癌和正常组织中的表达情况,结果显示,与正常组织相比,胃癌组织中SRSF10基因表达与上述结果一致,SRSF10在胃癌组织中的mRNA表达量上调(图2)。
siRNA使用的确定以及干扰目的基因的效率利用qRT-PCR实验和Western blot实验分别从RNA水平和蛋白质水平检测siRNA的干扰效率,结果显示,与对照组siNC相比,SRSF10的siRNA-1有较为明显的干扰效率(图3、4)。
敲低SRSF10基因可减慢胃癌细胞SGC-7901的增殖将SRSF10的siRNA和非特异性siRNA转染至胃癌SGC-7901细胞中,MTS方法检测结果显示,敲低SRSF10基因的胃癌细胞SGC-7901增殖减慢(图5)。

表2 胃癌中表达差异的剪接因子Table 2 Differentially-expressed splicing factors in gastric cancer
FC:倍数变化;FDR:错误发现率
FC:fold change;FDR:false discovery rate

FC:倍数变化;FDR:错误发现率;图中每个点代表1个基因,红色表示基因表达上调,绿色表示基因表达下调
FC:fold change;FDR:false discovery rate;each point in the figure represents a gene,among which red indicates up-regulated gene expression and green indicates down-regulated gene expression
图1胃癌中表达差异剪接因子的火山图
Fig1Volcanic map of differentially-expressed splicing factors in gastric cancer

SRSF10:富含丝氨酸苏氨酸蛋白家族中的剪接因子10;左边是胃癌组织样本,右边是正常组织样本,SRSF10基因在胃癌组织中mRNA表达量(测序量的reads数)上调
SRSF10:serine/arginine-rich splicing factor 10;on the left is a sample of gastric cancer tissue,and on the right is a sample of normal tissue,the mRNA of the SRSF10 gene is up-regulated in gastric cancer tissue
图2gepia网站中分析SRSF10在胃癌中表达上调
Fig2SRSF10 is up-regulated in gastric cancer(Gepia website)

aP<0.05,bP<0.01
图3qRT-PCR检测siSRSF10的干扰效果
Fig3The interference effect of siSRSF10(by qRT-PCR)
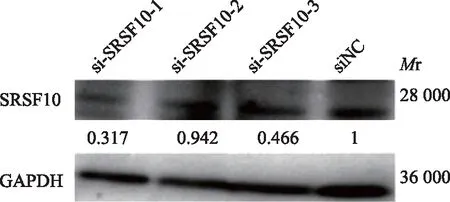
Mr:相对分子质量;GAPDH表达相同时,siSRSF10-1具有最浅的条带,siSRSF10-1干扰靶基因表达的效率最高
Mr:relative molecular mass;when the expression of GAPDH is the same,siSRSF10-1 has the shallowest band,and siSRSF10-1 has the highest efficiency in interfering with target gene expression
图4Western blot检测siSRSF10的干扰效率
Fig4Interference efficiency of siSRSF10(by Western blotting)

图5SRSF10敲低后的胃癌细胞增殖曲线
Fig5Growth curve of gastric cancer cells after SRSF10 knockdown
敲低SRSF10基因可降低胃癌细胞SGC-7901的划痕愈合能力将SRSF10的siRNA和非特异性siRNA转染至胃癌SGC-7901细胞中,显微镜下观察细胞在0、12、24、48、72 h时划痕愈合情况,结果显示,敲低SRSF10基因的胃癌细胞SGC-7901划痕愈合能力降低(图6)。
敲低SRSF10基因可降低胃癌细胞SGC-7901的迁移能力将SRSF10的siRNA和非特异性siRNA转染至胃癌SGC-7901细胞中,倒置显微镜下拍摄不同区域的照片,结果显示,敲低SRSF10基因的胃癌细胞SGC-7901迁移能力明显下降(t=13.03,P=0.002)(图7)。
讨 论
胃癌作为一种常见的消化系统恶性肿瘤之一,其死亡率一直居高不下[16]。为了提高胃癌患者的诊断率和术后治愈率,研究人员从胃癌的早期诊断、治疗方法和术后愈合等不同方面进行了研究。虽然治疗胃癌方法以及术后愈合的研究比较普遍,但在胃癌早期诊断中缺少有效的分子标志物,80%的胃癌患者就诊时已到晚期,导致疾病进一步发展、肿瘤转移,甚至步入终末期[17],错过了最佳治疗时间,因此急需加强对早期胃癌诊断标志物的寻找。
越来越多的证据表明,选择性剪接与细胞的生理病理、发育调控甚至疾病的发生有关。尽管选择性剪接位点对于组装功能性剪接是必须的,但是调节选择性剪接事件的辅助元件也必不可少。SR蛋白家族成员是选择性剪接调节的重要因子,其成员是多细胞生物中高度保守的剪接因子家族,具有多个拷贝RRM,参与选择性剪接过程中通常作为反式作用因子而发挥作用,因此,SR蛋白家族成员发挥着调控选择性剪接的作用[18]。SRSF10基因是SR蛋白家族中重要的成员之一,具有经典的SR蛋白结构域,是一个重要的剪接因子。本研究在TCGA数据库中下载了胃癌组织与正常组织RNA-seq数据,样本量共计407个,其中胃癌组织样本包含375个,癌旁样本32个。分析结果发现,胃癌患者中存在较多表达差异的剪接因子,它们可能调控转录后mRNA以及lncRNA的表达[19]。剪接因子SRSF10在临床胃癌样本中mRNA表达量上调,这可能是其在胃癌组织中拷贝数扩增的结果。生物学实验检测到敲低SRSF10的胃癌细胞增殖曲线减慢,Transwell细胞能力下降,划痕愈合能力降低,证实了SRSF10可促进胃癌增殖和转移。

A.对照组中SGC-7901细胞0和72h划痕宽度比较(×50);B.敲除SRSF10基因的SGC-7901细胞0和72h划痕宽度的比较(×50);C.siNC组和siSRSF10组中SGC-7901细胞划痕愈合能力的比较
A.comparison of 0 and 72 h scratch widths of SGC-7901 cells in the control group(×50);B.comparison of 0 and 72 h scratch widths of SGC-7901 cells after SRSF10 knockdown(×50);C.comparison of scratch healing ability of SGC-7901 cells in siNC group and siSRSF10 group
图6SRSF10敲低后的胃癌细胞划痕愈合能力
Fig6Scratch wound-healing of gastric cancer cell after SRSF10 knockdown

A.倒置显微镜观察结果(×200);B.siNC和si-SRSF10 组细胞数的比较
A.under inverted microscope(×200);B.comparison of cell numbers between siNC and si-SRSF10 groups
图7SRSF10敲低前后胃癌细胞迁移侵袭能力
Fig7Migration and invasion of gastric cancer cells before and after SRSF10 knockdown
与其他胃癌研究相比,本研究从TCGA数据库中的RNA-seq着手研究分析,充分利用了TCGA公共数据的资源,分析得到大量胃癌中具有显著表达差异的剪切因子,筛选出具有巨大潜力的剪接因子进行研究,为胃癌早期诊断提供了新的研究方向。另一方面本实验在研究思路方面需要再提高创新性,对于选择性剪接因子SRSF10对胃癌发生发展中的调控机制研究需要进一步加深研究,后续实验会着重研究SRSF10的调控机制。本研究得到的数据具有统计学意义,表型实验结果预示着剪接因子SRSF10很可能在胃癌中作为原癌基因对胃癌的发生发展发挥一定作用,具有成为胃癌早期诊断、预后判断乃至治疗的标志物的潜力,为胃癌临床靶向治疗提供了新的研究方向,这也是本研究的意义所在。
近年来对胃癌早期诊断标志物的研究不胜枚举,为胃癌早期筛选提供了多种可能。本实验中还存在一些未解决的问题,显著表达差异的剪接因子SRSF10对胃癌发生发展调控机制,下游作用靶标等问题都需要进一步研究确定。之后的研究方向是利用RNA-seq以及RIP-seq高通量测序分析剪接因子的结合位点以及下游的作用靶标,详细阐述剪接因子SRSF10的调节机制。从国际形势以及医学诊断的需求来看,我国作为胃癌的高发区,到目前为止一直未能实现全国性胃癌普查,因此,寻找胃癌早期诊断和筛查标志物的任务还很艰巨。同时寻找胃癌早期诊断的生物标志物,提高胃癌早期诊断率也是医学诊断的必然要求和趋势。
综上,本研究结果发现,胃癌患者中存在较多表达差异的剪接因子,它们可能调控转录后靶mRNA以及lncRNA的表达来影响胃癌发生。剪接因子SRSF10在临床胃癌样本中mRNA表达量被上调,这可能是其在胃癌组织中拷贝数扩增的结果。生物学实验证实了SRSF10可促进胃癌增殖和转移,从而影响胃癌的发生。本研究的不足之处在于,需要后续实验深入研究剪切因子SRSF10影响胃癌发生发展的调控机制。

