龙眼HD-Zip 家族固有无序蛋白的预测与分析
张春渝,许小琼,张梓浩,陈裕坤,林玉玲,赖钟雄
(福建农林大学园艺植物生物工程研究所,福建福州350002)
长期以来,在人类的固有思维里都认为蛋白质需要折叠成一定的三维结构,才能发挥生物学功能。直到20世纪90 年代,这种固有思维被逐渐打破,人们发现有一类蛋白在天然孤立状态下,没有一个明确唯一的三维结构,局部或完全不折叠,但是也能发挥一定的生物学功能[1-2]。研究者们将这类蛋白称之为固有无序蛋白质(intrinsically disordered protein,IDPs)或天然无折叠蛋白质(nativelyun folded protein,NUP)[3]。在发现的初期,人们曾将这类蛋白称为垃圾蛋白质组(junk proteome)[4],认为它不具有什么功能。但伴随着人们对蛋白质研究的深入,这类蛋白的生物学功能逐渐被挖掘,如多肽类激素(polypeptide hormones),是植物体内一类重要的信号物质,在植物的防御、受精以及生长和发育方面起着重要作用,据研究发现其在溶液状态下是无序的[5-6]。值得注意的是,无序蛋白中存在着特殊的作用机制,当无序蛋白和其他蛋白质相结合时,能够折叠成有序结构,因而存在一系列快速互变的构象[2]。目前,预测蛋白是否具有无序区已然成为当下的一个热点。据悉,在著名的蛋白质结构预测的比赛Critical Assessment of Structure Prediction 中[7],预测无序蛋白已经成为一项比赛的内容,可见固有无序蛋白在蛋白质研究中的重要性。
无患子科龙眼属龙眼(Dimocarpus longan Lour.)是热带和亚热带的常绿果树之一,具有丰富的营养与药用价值。HD-Zip 作为植物中特有的转录因子,在植物的应激胁迫中发挥着重要作用[8-9]。同样,有研究结果表明,固有无序蛋白因为自身结构的特点,可在植物受到逆境胁迫时快速反应,从而减少逆境对植物的伤害[1]。如脱水蛋白(DHNs)可稳定细胞膜从而减少拟南芥受到冷冻胁迫时的伤害[10];拟南芥的ANAC019 介导的ABA 超敏反应与其无序C-端的TRD(transcription regulatory domain)有关[11]。故固有无序蛋白在植物应答以及适应逆境方面发挥着重要作用[12]。目前对于龙眼HD-Zip 无序蛋白的研究尚属空白,因此有必要对龙眼HD-Zip 固有无序蛋白进行预测,进一步探究龙眼HD-Zip 参与植物应激胁迫的分子机制。
该文通过对实验室基因组数据库中提取出来的19 条龙眼HD-Zip 的蛋白序列进行无序蛋白区域的预测、无序区域的特征以及无序区域与有序区域氨基酸序列偏好性的分析,以期为进一步研究龙眼HD-Zip 的结构与功能、蛋白互作奠定基础。
1 材料与方法
1.1 材料
福建农林大学园艺植物生物工程研究所实验室构建的龙眼基因组数据库(NCBI 登录号:BioProject PRJNA305337)中提取的19 条HD-Zip 成员的蛋白序列。
1.2 方法
于 DISOPRED3[13](http://bioinf. cs. ucl. ac.uk/psipred/disopred=1)在线网站对19 条HD-Zip 成员的蛋白进行无序区域预测,并用WPS 以及Excel 对其蛋白质的无序区以及有序区的氨基酸数目进行相关统计,以便后续分析(其中由于Dlo-026005.1、Dlo-027079.2 的蛋白质中含有部分未知氨基酸序列,故不进行后续分析)。
2 结果与分析
2.1 无序区域的特征
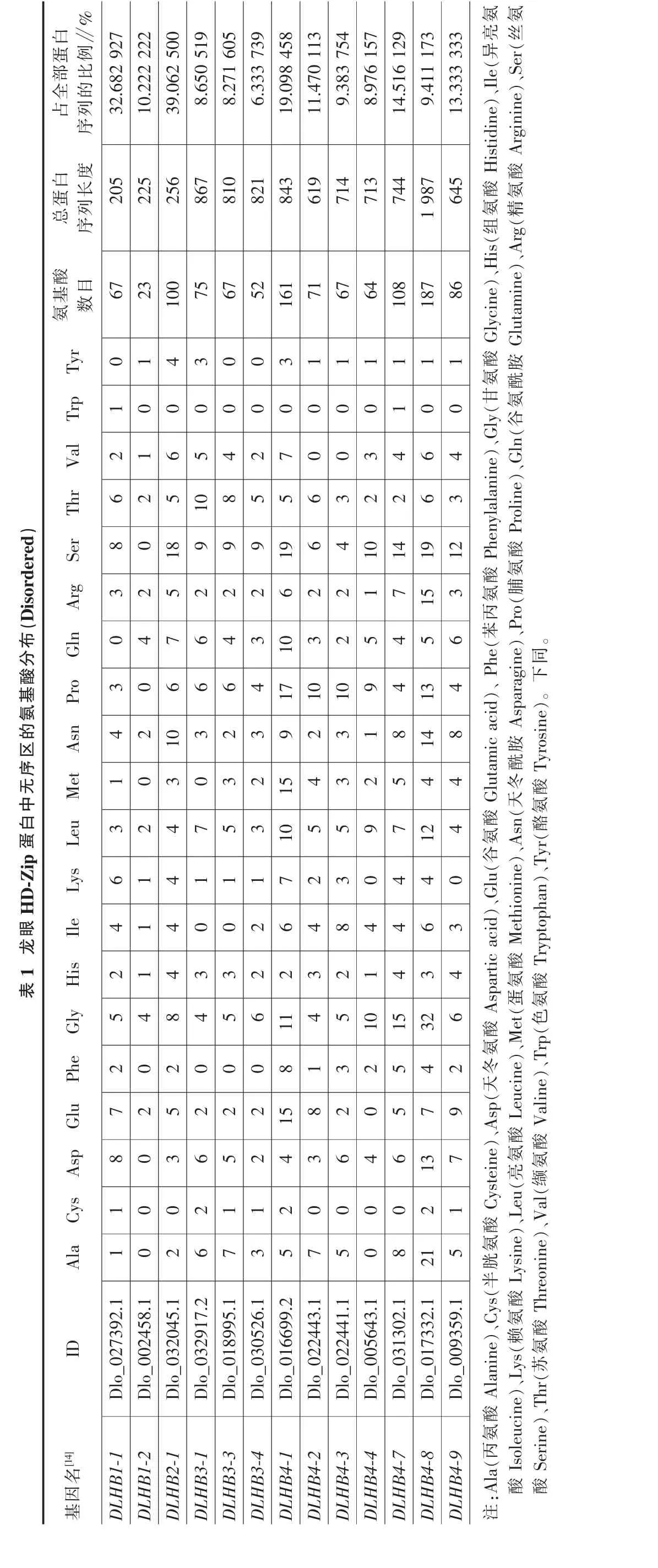
注:(Ala丙氨酸)Alanine、(半Cys胱氨酸)Cysteine、(Asp天冬氨酸)Aspartic acid、(Glu谷氨酸)Glutamic acid、 (Phe苯丙氨酸)Phenylalanine、(Gly甘氨酸)Glycine、(组His氨酸)、Histidine(Ile异亮氨酸)Isoleucine、(赖Lys氨酸)、Lysine(Leu亮氨酸)Leucine、(蛋Met氨酸)Methionine、(Asn天冬酰胺)Asparagine、(Pro脯氨酸)Proline、(Gln谷氨酰胺)、Glutamine(Arg精氨酸)Arginine、(丝Ser氨酸)Serine、(苏Thr氨酸)Threonine、(缬Val氨酸)Valine、(Trp色氨酸)Tryptophan、(酪Tyr氨酸)Tyrosine。 下同。表1 龙眼HD-Zip 蛋白中无序区的氨基酸分布(Disordered)基因名[14]ID Ala Cys Asp Glu Phe Gly His Ile Lys Leu Met Asn Pro Gln Arg Ser Thr Val Trp Tyr氨基酸数目总蛋白序列长度占全部蛋白序列的比例∥%DLHB2-1 DLHB3-1 Dlo_032045.1 Dlo_032917.22 60 23 65 22 08 44 34 04 14 73 010 36 67 65 218 95 106 50 04 3100 7525686739.0625008.650519 DLHB3-3 DLHB3-4 Dlo_018995.1 Dlo_030526.17 31 15 22 20 05 63 20 21 15 33 22 36 44 32 29 98 54 20 00 067 528108218.2716056.333739 DLHB4-1 DLHB4-2 Dlo_016699.2 Dlo_022443.15 72 04 315 88 111 42 36 47 210 515 49 217 1010 36 219 65 67 00 03 1161 7184361919.09845811.470113 DLHB4-3 DLHB4-4 Dlo_022441.1 Dlo_005643.15 00 06 42 03 25 102 18 43 05 93 23 110 92 52 14 103 20 30 01 167 647147139.3837548.976157 DLHB4-7 DLHB4-8 Dlo_031302.1 Dlo_017332.18 210 26 135 75 415 324 34 64 47 125 48 144 134 57 1514 192 64 61 01 1108187744198714.5161299.411173 DLHB4-9 Dlo_009359.15179264304484631234018664513.333333 DLHB1-1 DLHB1-2 Dlo_027392.1 Dlo_002458.11 01 08 07 22 05 42 14 16 13 21 04 23 00 43 28 06 22 11 00 167 2320522532.68292710.222222
通过DISOPRED3 对龙眼HD-Zip 成员的蛋白进行无序区域的预测,发现19 个龙眼HD-Zip 成员中共有15 个成员的蛋白具有无序区域,并且这些蛋白的无序区域都是由连续30 个以上的氨基酸组成。按照固有无序蛋白的分类,此类蛋白质应属于部分无序的固有无序蛋白[15]。其中,预测结果中龙眼HD-Zip 蛋白的无序区又根据是否具有蛋白质的结合位点分为Disordered 与Disordered,portein binding。对其中13 个成员(除Dlo-026005.1、Dlo-027079.2)无序区的这2 种情况的氨基酸分布进行统计(表1~2)。结果表明:首先,龙眼HD-Zip 蛋白的Disordered 的氨基酸总数大于Disordered,portein binding,其中Disordered 的氨基酸总数为1128,Disordered,portein binding 的氨基酸总数为593,可见无序区域中存在参与蛋白质结合位点的区域约为全部无序区域的1/3;其次,13 个龙眼HD-Zip 在Disordered区域各成员具有的氨基酸种类为12-19,在Disordered,portein binding 各成员具有的氨基酸种类也为12-19,但从整体氨基酸分布的情况上来看,Disordered 区域相较于Disordered,portein binding 氨基酸的种类在分布上更加丰富;通过计算每个成员蛋白中Disordered 与Disordered,portein binding 区域序列所占的比例发现,同一亚家族的含量相近(除DLHB1-1 与DLHB1-2 存有一定的差异),猜测无序区域的形成以及无序区域的大小可能与蛋白的结构域有着一定的联系。

%白∥蛋例部比全的占列9.75609832.00000021.8750004.1522494.6913587.3081614.6263357.9159947.8431377.1528756.3172041.6104685.736434序白度蛋长205225256867810821843619714713744645总列序1987酸基目 207256 36 38603949 56 51473237氨数Tyr 031 0 1100 1 1110 Trp 010 0 0000 0 0000),portein binding Val 061 1 1141 0 1123 Thr 367 2 1632 1 4210 Ser 2107 5 61059 9 11321(Disordered Arg 122 1 3115 3 5211 Gln 120 4 0310 1 1201布Pro 141 3 0320 0 0310分酸65 2 3565 5 2534 Asn 13基氨的Met 33 3 2201 2 1131区序43 2 3610 2 0212无Leu 0中白Lys 040 1 2312 4 2002蛋HD-Zip Ile 221 2 0023 2 01161 His 001 1 132 3 4102眼龙Gly 110 5 54454426 8 1210表Phe 033 0 1100 1 1111 Glu 155 1 1153 5 1763 Asp 223 1 3414 3 4334 Cys 001 0 1111 1 0100 Ala 242 2 4515 5 1122 ID Dlo_027392.1 Dlo_002458.1 Dlo_032045.1 Dlo_032917.2 Dlo_018995.1 Dlo_030526.1 Dlo_016699.2 Dlo_022443.1 Dlo_022441.1 Dlo_005643.1 Dlo_031302.1 Dlo_017332.1 Dlo_009359.1名因基DLHB1-1 DLHB1-2 DLHB2-1 DLHB3-1 DLHB3-3 DLHB3-4 DLHB4-1 DLHB4-2 DLHB4-3 DLHB4-4 DLHB4-7 DLHB4-8 DLHB4-9
为分析在龙眼HD-Zip 蛋白无序区域中哪些氨基酸种类有促进无序区域中蛋白质结合区域的组成,对20 种氨基酸在无序区域的含量进行统计。从氨基酸的倾向性来分析(表3)可以发现:Cys、Asp、Glu、Gly、His、Lys、Met、Asn、Arg、Ser、Thr、Tyr 这12 种氨基酸在Disordered,portein binding 区域的含量较高,其中Gly 表现出最强的倾向性。说明这12 种氨基酸较倾向于无序区中蛋白结合位点的序列组成。
2.2 氨基酸偏好性特征

表3 龙眼HD-Zip 蛋白无序区域氨基酸含量
对上述13 个龙眼HD-Zip 成员蛋白质的无序区以及有序区的氨基酸统计表明,无序区氨基酸的总数为1721,约占蛋白质全部序列的18.2%,因此有序区的氨基酸总数约为无序区的4.5 倍。可见,龙眼HD-Zip 蛋白的有序区氨基酸总数要大大高于无序区域,龙眼HD-Zip 蛋白质主要还是以有序区为主。
为分析氨基酸在龙眼HD-Zip 蛋白中的偏好性,对无序区以及有序区的不同氨基酸含量进行统计,并且计算有序区与无序区之间各类氨基酸的含量(表4),可以看出,在龙眼HD-Zip 蛋白中Asp、Gly、His、Met、Asn、Pro、Ser、Thr这几种氨基酸在无序区的含量高于有序区,表明这8 种氨基酸有利于无序区的组成;而Ala、Cys、Glu、Phe、Ile、Lys、Leu、Gln、Arg、Val、Trp、Tyr 则在有序区的含量高于无序区,表明这12 种氨基酸在一定程度上比较有利于有序区域的组成。在20 种氨基酸中,Gly 最有利于龙眼HD-Zip 蛋白中无序区域的组成,其在无序区域的含量与有序区域大约相差5.637288 个百分点;而Leu 则最有利于龙眼HD-Zip蛋白中有序区域的组成,其在有序区的含量比无序区高出了4.114621 个百分点。可见在龙眼HD-Zip 中,20 种氨基酸对无序区域以及有序区域的形成各有所偏向。

表4 龙眼HD-Zip 蛋白有序区及无序区的氨基酸含量对比
3 讨论
3.1 龙眼HD-Zip 固有无序蛋白可能通过分子调控网络参与应激反应
通过预测发现,19 个龙眼HD-Zip 成员中15 个成员为固有无序蛋白。据悉,固有无序蛋白通过分子调控网络参与植物对逆境的胁迫和适应。同时,HD-Zip 转录因子在植物的应激反应中发挥着重要作用[9,16]。因此猜测龙眼HD-Zip 固有无序蛋白可能参与应激反应并且存在分子调控网络。根据以往的研究报道[12],固有无序蛋白的分子调控具有多种模式。其中,与完全无序的蛋白质相比,部分无序蛋白中的有序区域可能起到了结合特定的DNA 序列,启动光信号等作用。同时部分无序蛋白又根据无序区与有序区是否存在分子内的相互作用分为两类。分子间不存在明显相互作用的蛋白,其无序区可能与其他的蛋白质相互作用,有序区在此过程中起协作作用。分子间存在明显相互作用时,又存在3 种情况:其一,无序域可在有序域释放活性位点;其二,无序域可从有序区域中释放出来从而与其他蛋白相互作用;其三,无序区域在有序区域与无序区域同时释放活性位点,进而与其他蛋白相互作用。这些分子调控网络在植物的非生物胁迫、转录调控等方面发挥着重要作用。据报道,转录因子NAC[17]、HY5[18]存在第一类分子调控模式;GRAS[19]、CRY[20]存在第二类分子调控模式。至于龙眼HD-Zip 中固有无序蛋白通过哪种分子调控网络参与应激反应,还需后续进一步探究。
3.2 龙眼HD-Zip 固有无序蛋白具有氨基酸偏好性
对具有完整氨基酸序列的13 个成员进行无序区域特征的分析发现,龙眼HD-Zip 存在固有无序区域,且在这些无序区域中存在蛋白质结合位点。此前有研究者对IDPs与其他蛋白的结合位点残基做了分析,其认为疏水性、侧链净电荷量较少、极性较小、溶剂可及性表面积较大、侧链体积较大、极化率较大的氨基酸残基比较有利于IDPs 与蛋白质作用位点的形成[3]。而该研究未对此做深入分析,龙眼HD-Zip 固有无序蛋白的作用位点残基是否也具有这些特点,值得后续更加深入地分析。
对龙眼HD-Zip 蛋白的有序区域以及无序区域的氨基酸含量进行统计,结果表明在龙眼HD-Zip 蛋白的氨基酸序列中,Asp、Gly、His、Met、Asn、Pro、Ser、Thr 促进无序区域的 形 成,Ala、Cys、Glu、Phe、Ile、Lys、Leu、Gln、Arg、Val、Trp、Tyr 促进有序区域的形成。早前,Radivojac 对固有蛋白的氨基酸偏好性做了分析,他认为Phe、Val、Leu、His、Thr、Cys、Trp、Tyr、Ile、Asn 这几种氨基酸相对促进有序结构的形成;Asp、Met、Lys、Arg、Ser、Gln、Pro、Glu 则促进无序区域的形成;Ala 和Gly 则表现中性[21]。通过对比发现,该研究和Radivojac 都认为Cys、Trp、Tyr、Ile、Phe、Val、Leu 促进有序区域的形成;Asp、Met、Ser、Pro 促进无序区域的形成。但仍存在一些不同,笔者认为His、Thr、Asn 促进无序区域的形成,而Radivojac 认为其促进有序区域的形成;笔者认为Lys、Arg、Glu、Gln 促进有序区域的形成,而Radivojac 认为是促进无序区域的形成。猜测形成这种差异的原因可能是:其一,该研究选取的范围较小,仅为龙眼HD-Zip 成员;其二,对固有无序蛋白的预测方法有所不同。
4 展望
本次预测固有无序蛋白的工具为DISOPRED3,其相较于上一代DISOPRED2 更加准确[13]。除此之外,还有PONDR VL3[22]、POODLE-L[23]等一些计算机预测软件。通过试验的方法预测无序蛋白的结构存在诸多困难,比如费用高昂、需要花费大量时间,因此预测软件的发展在固有无序蛋白的研究上显得尤为重要。但目前固有无序蛋白的预测还存在许多局限之处,如预测的精确和准确程度。据日前报道[24],人们已经利用AI 技术,进行了蛋白3D 结构的预测:谷歌“深度思维”(DeepMind)公司将人工智能“阿法狗”转型,研究出一种“阿法折叠”(AlphaFold)的程序,成功根据基因序列预测出蛋白质的3D 结构。以此趋势,未来人工智能必定在蛋白的研究与发展中起到更加重要的作用,无序蛋白的精准预测也将指日可待。

