活性蓝4与牛血清白蛋白的相互作用研究
任钰婷,邱智军,张 彬,赵 渊,马琼琼,原江锋
(河南科技大学 a.食品与生物工程学院; b.农学院/牡丹学院,河南 洛阳 471023)
0 引言
随着生物技术,尤其是分子生物学技术的快速发展,下游分离技术逐渐成为制约整个生物产业发展的瓶颈[1]。因此,开发高效的分离纯化方法对于生物产业发展至关重要。亲和色谱技术由于具有高选择性、易于放大和易于工艺开发等优势而备受关注[2]。亲和色谱成功的关键在于亲和配基的选择。在目前已开发的亲和配基中,染料分子配基,特别是三嗪基染料分子配基由于具有价格低廉、稳定性强且不易降解等特点而得到广泛应用[3]。
作为三嗪基染料分子的代表,以汽巴蓝(cibacron blue)为配基的亲和色谱介质已广泛用于人血清白蛋白[4]、激酶[5]、限制性内切酶[6]、脂酶[7]和木聚糖酶[8]等蛋白的纯化,在生物、医药和食品等领域具有重要的应用价值。近年来,与汽巴蓝具有类似分子结构的其他三嗪基染料分子也作为亲和配基,逐步应用于多种蛋白质的分离和纯化。例如,以活性棕10(reactive brown 10)作为亲和配基,通过亲和色谱法可以从转基因棉籽中一步纯化获得磷酸肌醇乙酰转移酶[9];以活性绿5(reactive green 5)为亲和配基,使用自由基共聚法制备的亲和超大孔聚(2-羟乙基甲基丙烯酸酯)整体柱一步纯化获得了木瓜蛋白酶[10]。
尽管越来越多的三嗪基染料分子作为亲和配基在蛋白色谱应用中取得成功,但目前的研究主要集中在新型色谱介质的制备和色谱方法的建立等方面[11-12],而对于三嗪基染料分子与目标蛋白质相互作用的研究较少,这极大地限制了三嗪基染料配基的进一步应用以及新型配基分子的设计。因此,为了明确三嗪基染料配基与蛋白质相互作用的分子机制,本研究基于前期对一系列结构类似的三嗪基染料分子的筛选结果,选择具有良好吸附效果的活性蓝4(reactive blue 4,RB-4)作为亲和配基,通过紫外吸收光谱、荧光光谱和同步荧光光谱等光谱学手段和分子对接方法,研究了RB-4与模式蛋白牛血清白蛋白(bovine serum albumin,BSA)之间的结合作用机制,可为三嗪基染料分子作为配体的亲和色谱方法的建立,以及新型配基的设计提供理论依据。
1 材料与方法
1.1 试剂
RB-4和BSA,购自Sigma-Aldrich公司;三羟甲基氨基甲烷(Tris),购自生工生物工程(上海)股份有限公司;磷酸氢二钠、磷酸二氢钠、氢氧化钠和乙醇等试剂均为分析纯,购自天津市德恩化学试剂有限公司;试验用水均为超纯水。
1.2 仪器与设备
Agilent Cary Eclips型荧光分光光度计,安捷伦科技(中国)有限公司;L5S型紫外分光光度计,上海精密科学仪器有限公司;STARTER 3100型pH计,上海奥豪斯仪器有限公司;AR1502CN型电子天平,上海奥豪斯仪器有限公司;HB120-S型数显恒温金属浴加热器,大龙兴创实验仪器(北京)有限公司。
1.3 紫外吸收光谱分析
用浓度为20 mmol/L的醋酸钠缓冲液(pH为4.5)将BSA溶解,配制成浓度为0.025 mmol/L的溶液。将所配制的BSA溶液与不同浓度的RB-4溶液等体积混合,在25 ℃静置反应2 h。反应结束后,在270~300 nm波长扫描各混合物的紫外吸收光谱。
1.4 荧光光谱分析
用浓度为20 mmol/L的醋酸钠缓冲液(pH为4.5)将BSA溶解,配制成浓度为0.025 mmol/L的溶液。将所配制的BSA溶液与不同浓度的RB-4溶液等体积混合,分别在25 ℃、35 ℃和45 ℃下静置反应2 h。反应结束后,测定各混合物的荧光光谱。荧光光谱测定仪器参数:激发波长λex为280 nm,激发与发射狭缝宽度均为5 nm,电压600 V,荧光扫描发射波长λem为270~450 nm,同步荧光扫描激发波长为270~310 nm。三维荧光光谱主要参数:扫描模式为3-D Scan,数据模式为Fluorescance,发射波长λem为200~450 nm,激发波长λex为200~350 nm,激发狭缝为5.0 nm,发射狭缝为5.0 nm,光电倍增管电压为600 V,扫描速度为120 nm·min-1。
1.5 分子对接
BSA的晶体结构取自蛋白质数据库RCSB Protein Data Bank(PDB)(http://www.rcsb.org/pdb/home/home.do),PDB ID:3V03。RB-4结构由Chemical draw软件绘制。分析之前用UltraEdit软件去除BSA结构中自带的配体、水和游离氢原子。用Discovery Studio软件优化RB-4的结构模型(加力场),分析BSA的活性区域。通过软件识别分析得出BSA有36个活性位点可供配体进行对接,选择评分第一的位点作为对接位点,用LibDock 模块进行分子对接。对接完成后产生14个对接构象,选择评分最高(107.365)的对接构象作为对接结果进行分析。
2 结果与分析
2.1 紫外吸收光谱分析
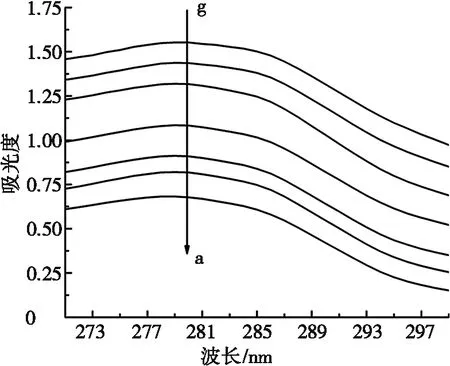
图1 RB-4与BSA相互作用的紫外吸收光谱图
图1为RB-4与BSA相互作用的紫外吸收光谱图。图1中,g→a线表示曲线所对应的RB-4浓度的变化方向,即从上至下曲线所对应的RB-4浓度分别为0.125 0 mmol/L、0.100 0 mmol/L、0.075 0 mmol/L、0.050 0 mmol/L、0.025 0 mmol/L、0.012 5 mmol/L和0 mmol/L。由图1可以看出:在不添加RB-4的情况下,BSA在波长279 nm处具有特征吸收峰。随着RB-4溶液浓度的增加,BSA的特征吸收峰位置未发生改变,但吸光度随之增大。BSA在波长279 nm处的特征吸收峰主要是由其肽链上色氨酸和酪氨酸的苯杂环π-π*跃迁所引起的[13],由此可见,RB-4的加入会诱导BSA分子发生蛋白质肽链伸展,使内部疏水性的色氨酸和酪氨酸残基的芳杂环基团暴露,吸光度增强。基于此,紫外吸收光谱分析显示RB-4与BSA之间存在特定的相互作用,但具体的作用模式需要进一步确定。
2.2 内源荧光光谱和荧光淬灭机制
BSA分子中含有色氨酸、酪氨酸和苯丙氨酸等一系列能够发射内源荧光的氨基酸残基,因此,可以通过内源荧光的变化来反映RB-4与BSA之间的相互作用以及RB-4所引起BSA的构象变化。图2为RB-4与BSA相互作用的内源荧光光谱与Stern-Volmer方程拟合图。图2中,a→g线表示曲线所对应的RB-4浓度的变化方向,即从上至下曲线所对应的RB-4浓度分别为0 mmol/L、0.012 5 mmol/L、0.025 0 mmol/L、0.050 0 mmol/L、0.075 0 mmol/L、0.100 0 mmol/L和0.125 0 mmol/L,下同;F0为未加入RB-4时BSA的荧光强度;F为加入不同浓度RB-4时BSA的荧光强度。如图2a所示,在280 nm激发波长下,BSA具有明显的荧光发射,且在342 nm具有最大发射峰。随着RB-4浓度的增加,荧光发射强度逐渐降低并伴随最大发射峰轻微蓝移,表明随着RB-4的加入,BSA发生荧光淬灭。
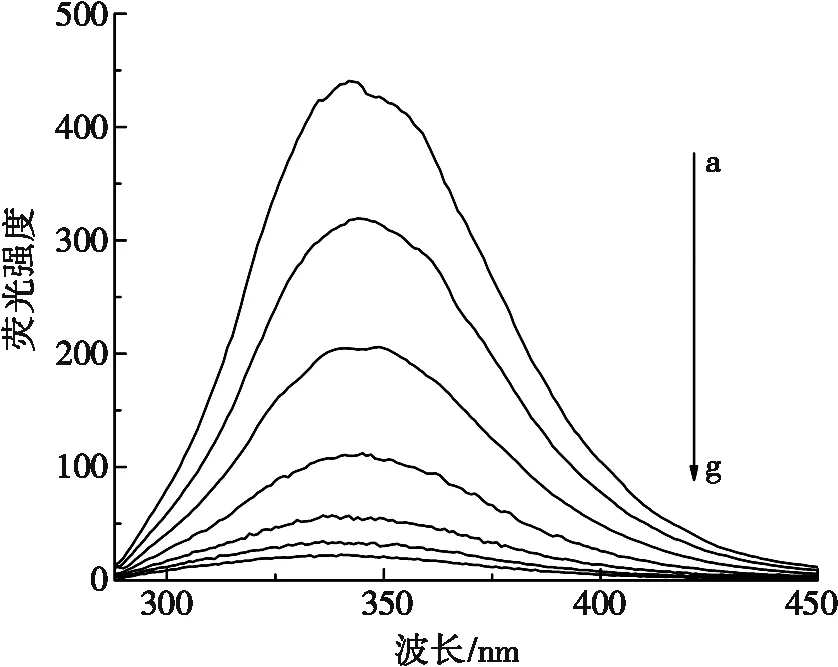
(a) 25 ℃时的内源荧光光谱
引起蛋白质发生荧光淬灭的原因包括动态淬灭、静态淬灭,以及基于不同淬灭机制的动态淬灭和静态淬灭的组合[14]。在动态淬灭过程中,蛋白质与淬灭剂通过碰撞引起动态淬灭。因此,随着温度的升高,扩散系数增大,电子转移增强,动态淬灭常数也相应增大。在静态淬灭条件下,蛋白质与淬灭剂形成基态复合物,所形成复合物的稳定性随温度的升高而降低,因此,静态淬灭常数随温度的升高而降低。动态淬灭和静态淬灭的组合,则是由蛋白质与淬灭剂通过碰撞以及基态复合物的形成共同引起的[15]。对于动态淬灭和静态淬灭,Stern-Volmer方程中的F0/F与Q之间成线性关系[13]。基于此,使用Stern-Volmer方程[13]分析了BSA的荧光淬灭过程。
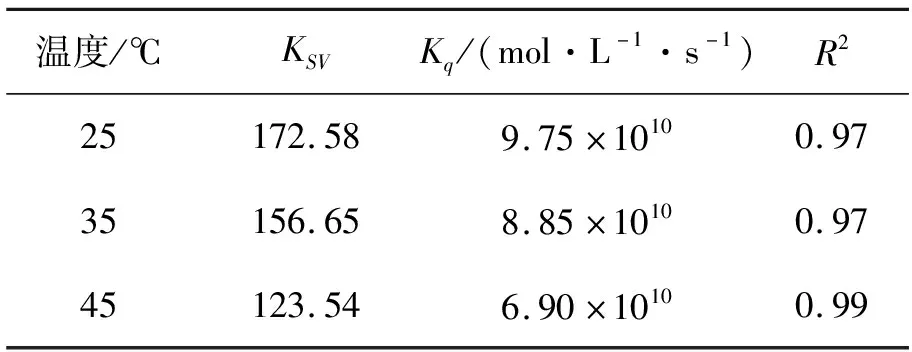
表1 RB-4与BSA相互作用的Stern-Volmer常数
使用Stern-Volmer方程对不同温度条件下BSA的荧光强度进行拟合(见图2b),拟合结果即RB-4与BSA相互作用的Stern-Volmer常数(见表1)。表1中,KSV为Stern-Volmer淬灭常数;Kq为由扩散过程控制的双分子动态荧光淬灭速率常数与单分子衰变速率常数的比值。从表1可以看出:在RB-4与BSA的相互作用过程中,拟合后的R2均不低于0.97,表明F0/F与Q之间成线性关系且斜率为负值,RB-4对BSA的淬灭机制为静态淬灭;另外,在RB-4对BSA的淬灭过程中,Kq>2.0×1010mol·L-1·s-1,且随着温度升高,Kq减小,表明RB-4与BSA所形成的复合物在高温下不稳定,进一步证明该过程为静态淬灭[16]。
2.3 RB-4对BSA的结合常数和结合位点数
对于静态淬灭过程,可根据Lineweaver-Burk双对数方程进行计算,获得RB-4与BSA相互作用的结合常数KA和结合位点数n[17]。
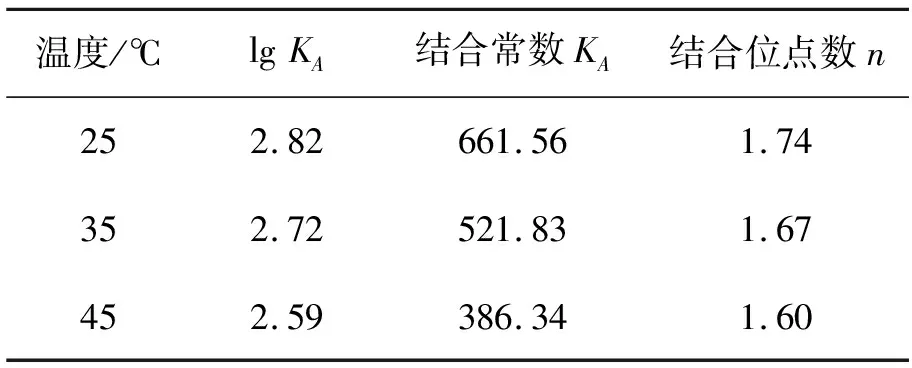
表2 Lineweaver-Burk双对数方程的拟合结果
表2为Lineweaver-Burk双对数方程的拟合结果。由表2可知:RB-4与BSA之间的结合位点数n接近2,表明RB-4与BSA大致具有2个结合位点;随温度升高,n值变化不大,表明RB-4与BSA具有较强的相互作用。随着温度的升高,KA减小,符合静态淬灭的特征[18]。
蛋白质与小分子化合物之间的结合强度可以通过结合常数KA来表示,KA值越大,则结合强度越大。在药物分子(例如罗利环素、卡比多巴)与BSA的结合过程中,其KA值通常为103~104,表明这些药物分子与BSA之间形成了强结合[19-20]。然而,在本研究中,RB-4与BSA之间的结合常数KA为102~103,明显小于这些药物分子与BSA之间的结合强度。与药物分子不同,色谱配基除了要与目标蛋白质形成稳定结合外,还需要考虑在分离结束时,能够将目标蛋白质从色谱介质上洗脱下来,因此结合强度不宜过大。
由于目前对于使用光谱法研究色谱配基与蛋白质相互作用的报道较少,因此无法根据文献判断色谱配基适合的KA范围。然而,在后续研究中,用RB-4官能化的色谱介质对BSA表现出良好的吸附和解吸性能。因此,可以推断102~103的KA对于色谱配基是较为适宜的。
2.4 RB-4与BSA相互作用的热力学参数
热力学参数,如吉布斯自由能变化(△G)、焓变(△H)和熵变(△S)可以指示小分子化合物与蛋白质的相互作用模式和作用力类型。使用van′t-Hoff方程计算了RB-4与BSA相互作用过程中的热力学参数△G、△H和△S,计算结果见表3。

表3 RB-4与BSA相互作用的热力学参数
由表3可知:在RB-4与BSA的反应过程中△G<0,表明RB-4与BSA之间的结合是自发进行的。小分子化合物与蛋白质的作用力类型主要包括疏水作用、范德华力、静电作用及氢键。根据△S和△H值的大小,可以判断相互作用过程中所存在的主要作用力,即当△H<0、△S<0时,表明该过程的主要作用力可能是氢键和范德华力;当△H>0、△S>0时,表明该过程的主要作用力是典型的疏水作用;当△H<0、△S>0时,表明该过程的主要作用力是静电作用。在本研究中,△H和△S均大于0,因此可以认为RB-4与BSA之间的主要作用力为疏水作用。
2.5 同步荧光光谱
同步荧光光谱可以用于指示荧光团附近的分子微环境。当将激发波长和发射波长之间的波长差△λ固定为15 nm和60 nm时,所获得的同步荧光光谱分别代表了蛋白质中酪氨酸残基和色氨酸残基的光谱特征[21]。因此,可以基于荧光发射波长的改变来判断蛋白质构象的变化情况。
图3为RB-4与BSA相互作用的同步荧光光谱。如图3所示,当△λ=15 nm(见图3a)和△λ=60 nm(见图3b)时,同步荧光光谱显示RB-4的加入使BSA的特征吸收峰降低,且使最大荧光发射波长分别蓝移约7 nm和5 nm。表明RB-4与BSA的结合导致酪氨酸残基和色氨酸残基周围的疏水性增强,且对酪氨酸残基的影响更大,结合过程导致BSA的构象发生变化。
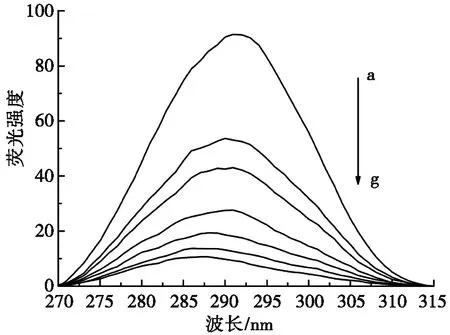
(a) △λ=15 nm
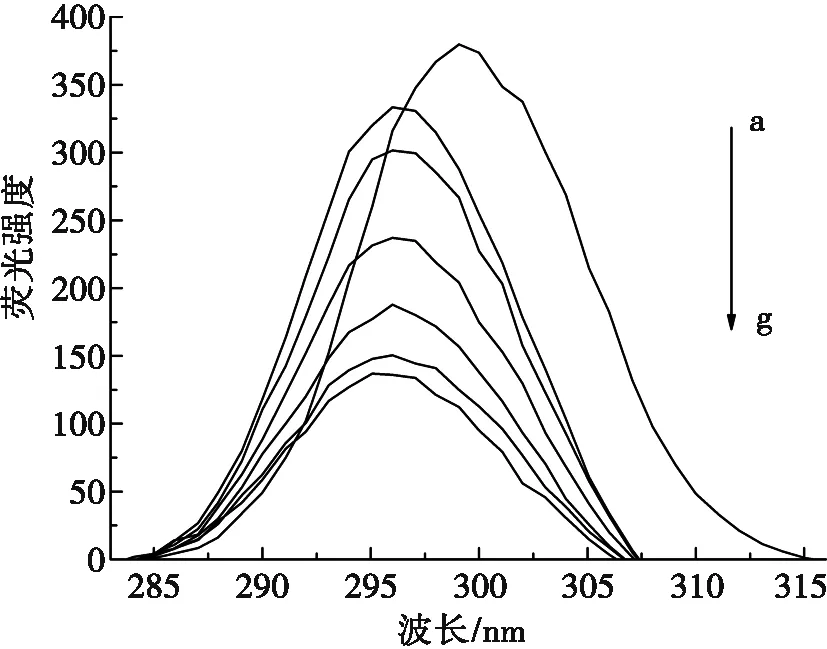
(b) △λ=60 nm
2.6 三维荧光光谱
三维荧光光谱能准确、可靠地检验蛋白质的结构和微环境变化。图4为RB-4与BSA相互作用的三维荧光光谱。由图4可知:在两个体系中存在特征为△λem=△λex的瑞利散射,即图4中所示的“山脊”峰(峰A)[22]。另外,对于BSA(见图4a),在△λem=345 nm处存在峰B,这是由BSA中氨基酸的π-π*跃迁所造成的。而对于RB-4与BSA结合的反应体系(见图4b),峰B的荧光强度显著降低,且λem发生轻微蓝移(λem=343 nm),表明在酪氨酸残基和色氨酸残基周围微环境中的疏水性增强,这与同步荧光光谱的结果一致。
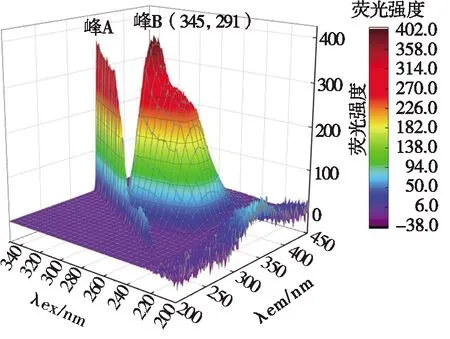
(a) BSA

(b) RB-4与BSA结合
2.7 分子对接
为了进一步阐明RB-4与BSA之间的结合特性,进行分子对接试验以确定配基和蛋白质分子之间的最佳结合位点。
BSA分子的三维结构呈心形,具有3个相似的结构域:结构域Ⅰ(1~183氨基酸残基)、结构域Ⅱ(184~376氨基酸残基)和结构域Ⅲ(377~583氨基酸残基),其中大部分的小分子与BSA的作用位置位于由亚结构域Ⅱ和亚结构域Ⅲ所形成的疏水性腔内[23]。
图5为RB-4与BSA的分子对接模型。图5a显示了RB-4与BSA分子对接的整体结果,在此构象下,结合自由能最低。RB-4与BSA对接时,周边存在11个氨基酸残基,包括离子型氨基酸残基:Glu-564和His-509;疏水型残基:Phe-567、Ala-568、Ala-510、Val-575、Pro-572、Phe-508和Phe-506;亲水型残基:Gly-571和Thr-507。其中,Phe-508、Ala-510、Glu-564与RB-4形成5个氢键(见图5b)。分子对接结果表明:BSA与RB-4之间的主要作用力为疏水作用,同时还存在一定的静电作用与氢键作用,该结果与光谱试验结果一致。
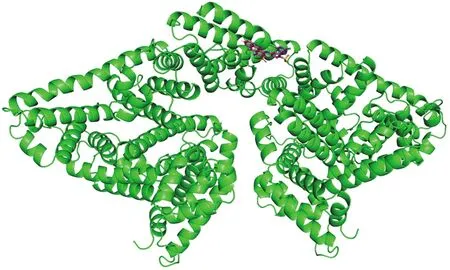
(a) 整体图
3 结论
(1)RB-4与BSA发生相互作用,使BSA内部疏水性的色氨酸和酪氨酸残基的芳杂环基团暴露。
(2)RB-4与BSA通过静态淬灭机制形成稳定的复合物,从而使BSA荧光淬灭。RB-4与BSA的结合过程是自发放热过程,低温有利于两者的结合,且疏水相互作用力是维持RB-4与BSA结合的主要作用力。
(3)RB-4与BSA的结合导致BSA的三级结构被破坏,空间结构变得松散,从而使酪氨酸残基和色氨酸残基周围微环境的疏水性增强。
(4)BSA与RB-4之间的主要氨基酸作用位点为Phe-567、Ala-568、Ala-510、Val-575、Pro-572、Phe-508和Phe-506,同时还存在一定的静电作用与氢键作用。

