寨卡病毒非结构蛋白研究进展
赵汝佳, 杨海涛, 王泽方
(天津大学 生命科学学院, 天津 300072)
寨卡病毒(Zika virus,ZIKV)是黄病毒科(Flaviviridae)、黄病毒属(Flavivirus)的成员之一,主要通过蚊虫叮咬进行传播[1]。自2007年起,该病毒在雅浦、法属波利尼西亚相继发生大规模爆发,2015年在美洲、加勒比海和太平洋地区报道了超过了几十万的感染病例[2]。经研究发现,该病毒还会导致严重的神经系统疾病(格林-巴氏综合征)和新生儿小头畸形[3-4],这给全球人类的健康造成了巨大的威胁,抗病毒药物的研发也因此变得非常迫切。ZIKV的基因组为一条长约11 kb的单股正链RNA,在复制过程中该基因组会被翻译成一条多聚蛋白,在宿主和病毒蛋白酶的切割下,该多聚蛋白最终形成3种结构蛋白:衣壳蛋白(C)、膜蛋白(prM/M)和包膜蛋白(E),以及7种非结构蛋白(NS1~NS5)(图1),其中结构蛋白参与病毒组装,非结构蛋白参与病毒增殖过程,如NS1不仅参与病毒复制同时帮助病毒逃避宿主免疫系统,NS3同时具有蛋白酶和解旋酶功能,参与多聚蛋白的水解和RNA解旋,NS5作为最大的非结构蛋白同时具有甲基转移酶和RNA聚合酶功能[5-6]。非结构蛋白在病毒复制过程中承担的重要角色使其成为了非常具有吸引力的药物靶点。近几年来,国内外多个研究组相继报道了这些蛋白的三维空间结构,为抗病毒药物的开发提供了重要的理论基础。根据这些科研成果,本文对ZIKV非结构蛋白的三维空间结构及相关药物靶点的最新进展进行了综述,以期为抗寨卡病毒的药物开发提供参考。
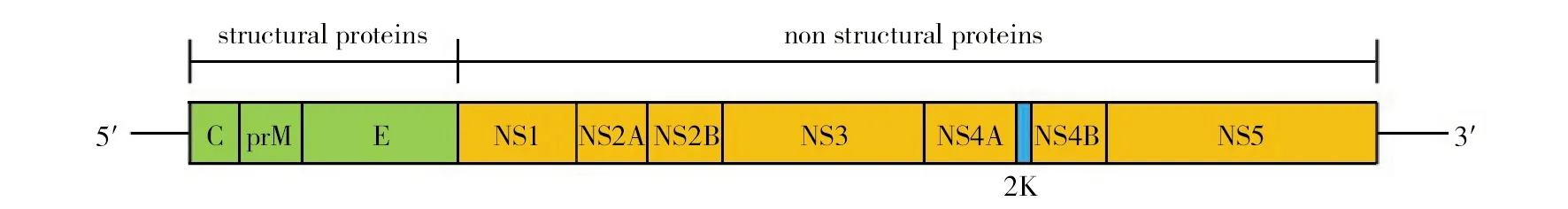
图1 寨卡病毒基因组组成[7]Fig.1 Genome Organization of Zika Virus[7]
1 ZIKV NS1研究进展
在病毒增殖过程中,NS1不仅能够与NS4A和NS4B构成复制复合物,参与病毒基因组的复制,还能以六聚体脂蛋白颗粒(sNS1)的形式分泌到细胞外,通过与宿主免疫系统组分(如补体成分C4)相互作用,帮助病毒免疫逃避[8-9],同时,NS1也是黄病毒早期感染的标志物[10]。
2016年,Song H研究团队和Brown WC研究团队先后报道了ZIKV NS1 的C端结构(PDB ID:5IY3)[11]和全长结构(PDB ID:5GS6,5K6K)[12-13]。分析表明,NS1为同源二聚体,每个单体由N端β-发夹结构域(β-hairpin domain),侧翼结构域(wing domain)和C端β-梯形结构域(β-ladder domain)组成,两个单体的β-hairpin domain相互缠绕形成二聚化结构域“β-roll”[12-13](图2)。值得注意的是,wing domain中的一段疏水loop(intertwined loop)可能参与了同膜的相互作用及NS1六聚体的形成,结构比对表明,ZIKV NS1与WNV、DENV相比显示出不同性质的表面电荷[12-13],这些都为开发特异性针对ZIKV的药物提供了新思路。以上研究成果同样在George F Gao老师的综述中得到了详细的介绍[14]。
在以上二聚体结构的基础上,本实验室在2017年也解析了NS1的C端结构(PDB ID:5X8Y)[15],通过对ZIKV中的病理突变T233A(图2)进行研究,证明了该突变影响了二聚体形成,可能与ZIKV的致病机制相关。除此之外,通过与WNV保护性抗体22NS1所对应的表位进行对比,分析了与抗体结合部位氨基酸的保守性与可变性,为未来保护性抗体的设计和开发提供了一定的结构基础[15]。
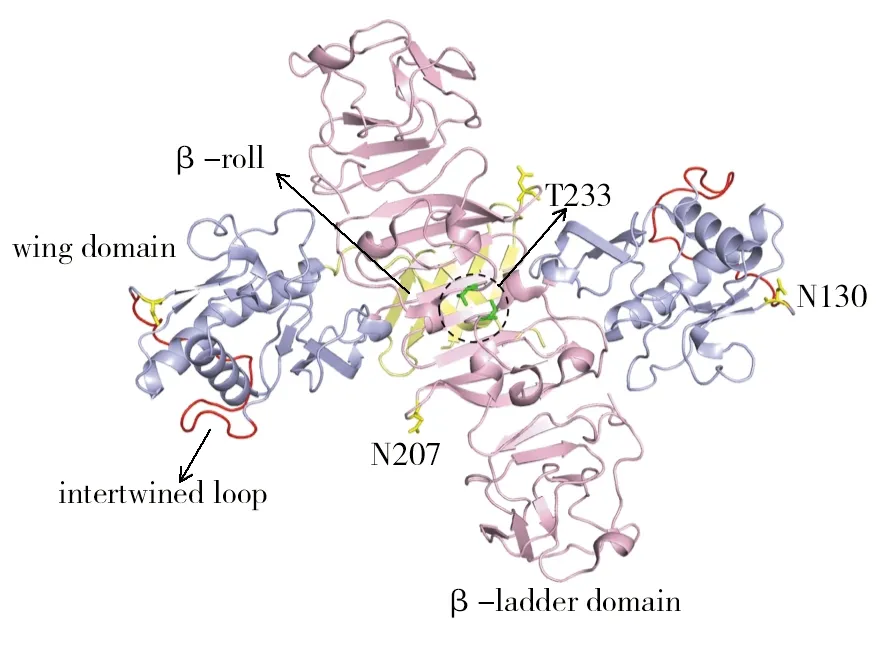
图2 ZIKV NS1全长的二聚体结构[12]Fig.2 Overall structure of the full-length dimeric ZIKV NS1[12]ZIKV NS1的二聚体结构(PDB ID:5GS6),糖基化位点N130和N207(黄色)、T233(绿色)为球棍模型,intertwined loop被标记为红色Overall structure of the dimeric ZIKV NS1(PDB ID:5GS6),glycans N130 and N207(yellow) and T233 (green) are shown in stikcs, the intertwined loop is marked in red
除了以上提到的疏水loop可作为潜在的药物靶点,对于NS1的药物设计还可以从与NS1结合的脂肪入手。之前有对DENV分泌型NS1六聚体的分析,发现确实存在多种脂质成分,如甘油三酯、胆固醇酯和磷脂等,这些脂质影响着sNS1的组装和分泌,因此也可以作为药物开发的新靶点[16]。此外,NS1中保守的糖基化位点N130和N207对病毒基因组的复制及NS1的分泌非常重要[17],也是抑制剂开发的重要方向。
NS1在ZIKV的感染诊断和疫苗开发方面也具有很大的应用价值。2017年,Brault研究团队[18]将ZIKV NS1序列插入到改良型痘苗病毒安卡拉中,设计出了第一个以NS1为基础的ZIKV疫苗。该疫苗不仅对输入了致死剂量ZIKV的免疫小鼠模型具有100%的保护作用,而且能够有效避免抗体依赖性增强作用,具有很大的开发前景。近期,Li研究组[19]首次采用共表达的方式同时以prM、E及NS1作为免疫抗原,设计了一种以水泡性口炎病毒为载体的减毒疫苗,该疫苗可诱导产生大量的特异性保护抗体和强烈的T细胞免疫应答,不仅揭示了NS1在细胞免疫中的重要作用,也为ZIKV疫苗的设计开发提供了新的方向。除了以上功能,NS1作为病毒早期感染的生物标记还可用于诊断试剂的开发。Zhang研究组[20]最近报道了一种用于检测ZIKV NS1的双抗体夹心酶联免疫吸附测定法,该方法快速、灵敏,可作为未来诊断ZIKV的新工具。Rong研究团队[21]最新开发了一种基于智能手机的荧光横流免疫分析(LFIA)方法,可对ZIKV NS1进行即时检测,为ZIKV诊断方法开发和应用提供了新的见解。
2 ZIKV NS3研究进展
NS3的N端为蛋白酶(NS3-pro)结构域,在NS2B存在时才能够被激活,主要对病毒的多聚蛋白进行加工处理,切割位点包括NS2A/NS2B、NS2B/NS3和NS3/NS4A[7];NS3的C端为解旋酶(NS3-hel)结构域,同时具有NTPs水解活性、双链RNA解旋活性和RNA 5′端三磷酸酶活性[7]。NS3的多重功能使其成为非常具有吸引力的药物靶点。
2.1 NS2B-NS3pro的结构及其抑制剂设计
2016年,Lei研究团队和Chen研究团队先后报道了以linker连接的NS2B-NS3pro与硼酸盐抑制剂cn-716的复合物结构(PDB ID: 5LC0)和母体结构(PDB ID: 5GXJ)[22-23]{Chen, 2016 #45;Chen, 2016 #45;Lei, 2016 #44}。复合物结构中,NS2B呈现“闭合构象”,而母体结构的NS2B呈“开放构象”(图3A),通过对比两个结构在结合口袋处的差异,揭示了一种蛋白酶从抑制状态到激活状态可能的分子机制[23]。Phoo研究组[24]使用NS2B C端的几个氨基酸直接与NS3pro相连的方式解析了ZIKV NS2B-NS3pro 1.84Å的结构(PDB ID: 5GJ4),在该结构中NS2B最后四个氨基酸(TGKR)反向结合在了NS3pro的活性位点上,揭示了生理状态下多聚蛋白被蛋白酶酶切后的状态(图3B)。此外,该研究组还得到了一种“非连接”的蛋白酶结构(PDB ID: 5H4I)[25],在该结构中同时存在结合与非结合两种状态的蛋白酶。结合态的NS3pro与相邻NS3pro的 N端氨基酸(KKGE)结合(图3B)。以上内容也在George F Gao老师的综述中得到了阐述[14]。
以上述“非连接”的NS2B-NS3pro为基础,Dahai Luo课题组在近两年相继报道了多个NS2B-NS3pro与底物模拟肽的复合物结构。这几种复合物的结构与母体结构相比变化不大,但是不同多肽与蛋白酶的结合方式却存在差异,进一步为底物肽抑制剂的开发提供了新的研究思路。例如,在NS2B-NS3pro与二肽抑制剂Acyl-KR-aldehyde的复合物结构(PDB ID: 5H6V)中[26],精氨酸和赖氨酸分别占据了蛋白酶的S1和S2结合口袋(图3C),其醛基与催化位点Ser135形成共价键,对蛋白酶的抑制作用很明显。在NS2B-NS3pro与三种底物肽模拟物的复合物结构(PDB ID:5ZMQ、5ZMS、5ZOB)中[27],compound3在P1′位置引入了4-amidino benzyl amide,其不能被蛋白酶水解,也具有很强的抑制效果(图3D)。
根据上述蛋白酶及其复合物不同的结构特点,抑制剂的设计方法可以从以下几个方面考虑:①使用不同构建方式得到的蛋白酶进行抑制剂筛选。研究发现,共表达方式得到的蛋白酶因没有linker的存在而不会产生空间位阻,活性更高,更加适合进行药物筛选[25,28]。但是随后也有研究表明,蛋白酶与TGKR在溶液中的结合也是动态的,小分子抑制剂同样可以和TGKR竞争结合活性位点,这种构建方式得到的蛋白酶可能更加接近天然生理状态,因此也可以用于抑制剂的筛选[29]。②对底物肽类似物进行加工修饰。例如前面提到的二肽抑制剂,因其分子量小、侧链可被修饰而具有很大的成药潜力,通过添加醛、硼酸弹头等,都可以大大提高模拟肽抑制剂对蛋白酶的亲和力[22,26]。此外,结构研究表明多肽抑制剂中P1位置为精氨酸时抑制剂与蛋白酶的结合更加紧密,同时P4取向较为灵活(图3D),且并未完全占据蛋白酶的S4口袋,可以进一步对P4进行修饰以改善抑制剂的性质[27]。③通过高通量药物筛选对现有药物进行测试。如有的课题组筛选出了替莫卟吩,其不仅能抑制ZIKV在胎盘和神经祖细胞中的复制,还能够有效降低小鼠模型的死亡率,是一种非常具有开发潜力的抑制剂[30]。

图3 ZIKV NS2B-NS3pro的结构[20-27]Fig.3 Structural comparison of ZIKV NS2B-NS3pro[22-27]A:NS2B-NS3pro与cn-716的复合物结构(PDB ID: 5LC0)同母体结构(PDB ID: 5GXJ)的叠加对比,cn-716为黄色球棍模型,黑色箭头处指出了复合物中NS2B的闭合构象; B:NS2B-NS3pro与TGKR的复合物结构(PDB ID: 5GJ4)同结合状态的“非连接”的蛋白酶结构(PDB ID: 5H4I)叠加对比,TGKR(绿色)和KKGE(红色)为球棍模型;C:NS2B-NS3pro与Acyl-KR-aldehyde的复合物结构(PDB ID: 5H6V),二肽抑制剂(紫色)和Ser135(黄色)为球棍模型;D:NS2B-NS3pro与3种底物肽抑制剂的复合物结构(PDB ID:5ZMQ,5ZMS,5ZOB)对比,compound 1-3分别为绿色、粉色和黄色球棍模型A: Overlay of the complex structure of NS2B-NS3pro with cn-716 (PDB ID: 5LC0) and the apo structure (PDB ID: 5GXJ), the black arrow indicates the closed conformation of NS2B in the complex; B: Overlay of the NS2B-NS3pro-TGKR complex structure (PDB ID: 5GJ4) and the "unlinked" protease structure (PDB ID: 5H4I),TGKR(green)、KKGE(red) are shown as sticks;C: Complex structure of NS2B-NS3 and Acyl-KR-aldehyde (PDB ID: 5H6V),Acyl-KR-aldehyde and Ser135(yellow) are shown as sticks; D: Overlay of the complex structure of NS2B-NS3pro with three different peptidomimetic inhibitors(PDB ID: 5ZMQ, 5ZMS, 5ZOB), compound 1-3(green,pink,yellow) are shown as sticks
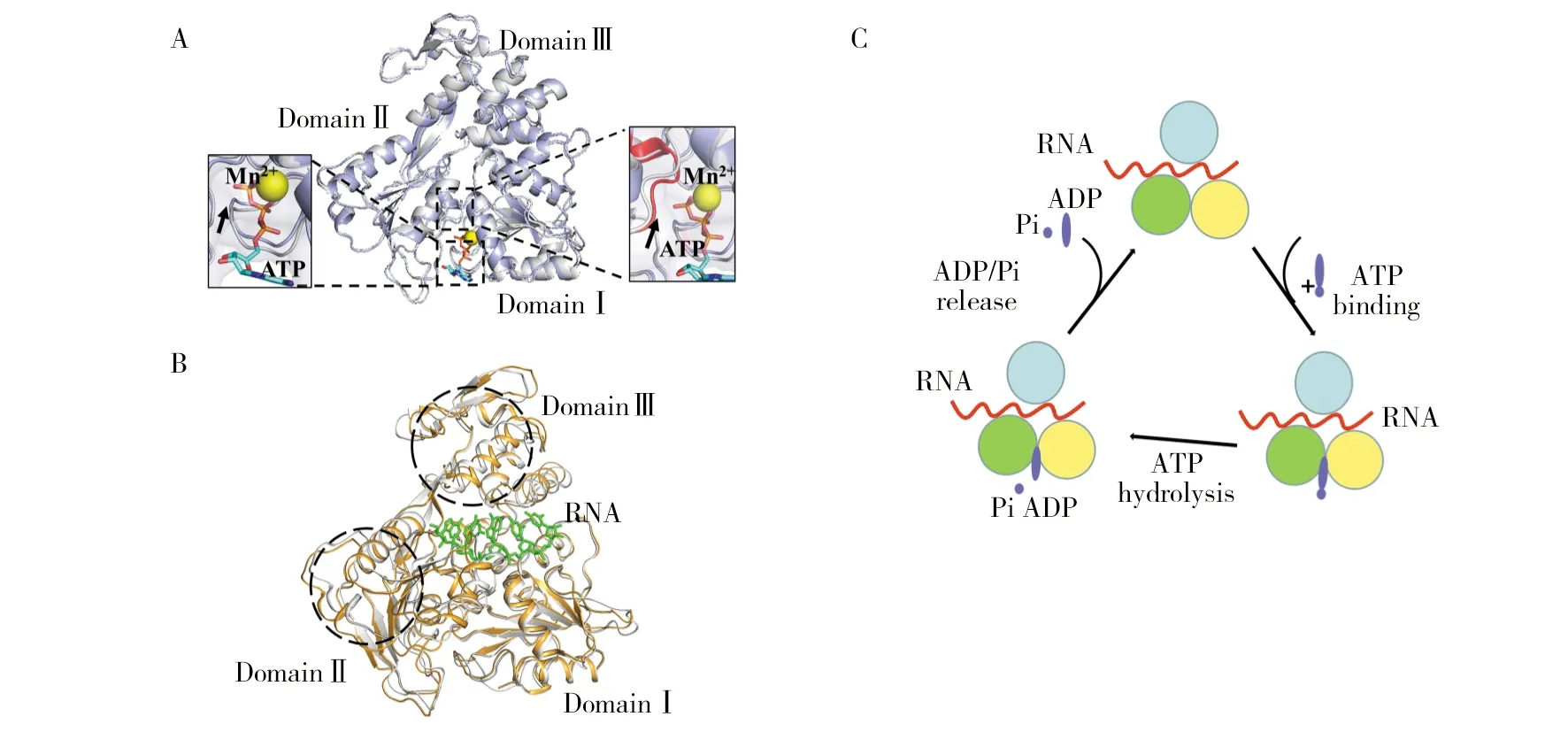
图4 ZIKV helicase的结构[31-32]
A:NS3-hel母体结构(PDB ID:5JMT, 灰色)与NS3-hel-ATP/Mn2+的复合物结构(PDB ID:5GJC,蓝色)叠加图及ATP结合处的构象变化;B:NS3-hel母体结构(PDB ID:5JMT,灰色)与NS3-hel-RNA的复合物结构(PDB ID:5GJB,黄色)叠加图,RNA 显示为黄色球棍模型;C:NS3-hel水解循环示意图,Domain I-III分别为黄色、绿色和蓝色,RNA为红色,ATP/ADP/Pi为紫色
A: Overlay of the NS3-hel apo form(PDB ID: 5JMT, gray) and the NS3-hel-ATP/Mn2+complex structure (PDB ID: 5GJC, blue) and the conformational change at the ATP binding sites; B:Overlay of the NS3-hel apo form(PDB ID: 5JMT, gray) and the NS3-hel-RNA complex structure (PDB ID: 5GJB, yellow), the RNA is shown as sticks(yellow);C:Schematic diagram of NS3-hel ATP hydrolysis cycle, Domain I-III are colored yellow、green and blue, RNA are red and ATP/ADP/Pi are purple
2.2 NS3-hel的结构及药物靶点的筛选
2016年,本实验室率先解析了ZIKV NS3-hel的母体结构(PDB ID:5JMT)、与ATP/Mn2+的复合物结构(PDB ID:5GJC)以及与RNA的复合物结构(PDB ID:5GJB)[31-32],以此揭示了解旋酶的底物识别方式。结构分析表明,NS3-hel共包含三个结构域:Domain I、Domain II和Domain III,在与ATP/Mn2+的复合物中,其催化位点的P-loop向内部中心移动且411~416位氨基酸主侧链发生了偏移(图4A),在同RNA结合时Domain II和Domain III发生转动,一条贯穿整个解旋酶分子的“隧道”容纳了RNA(图4B)。
除了上面提到的ATP和RNA识别机制,NS3-hel的ATP水解机制也是当前研究的热点。水解过程包括ATP的结合、水解和释放三个步骤(图4C),期间还需要二价金属离子的参与。2016年,Cao等[33]通过解析NS3-hel的母体结构(PDB ID:5JWH)、与ATP/Mn2+及ADP/Mn2+(PDB ID:5K8I,5K8U)以及与GTPγS、GTPγS/Mg2+(PDB ID:5K8L,5K8T)的复合物结构,首次揭示了二价离子在解旋酶结合和水解NTP过程中的变构调节作用。2018年,本实验室发表了ZIKV NS3-hel水解ATP过程的中间状态(NS3-hel+ADP+AlF3+Mn2+)和完成态(NS3-hel+ADP+Mn2+)的复合物结构(PDB ID:5Y6M,5Y6N),结合之前的NS3-hel-ATP/Mn2+复合物,提出了NS3-hel水解ATP完整的循环机制[34]。最近,Fang研究组[35]报道了NS3-hel与ADP/Mn2+的复合物结构(PDB ID:6ADX,6ADY),在该结构中存在两种不同状态的反应水,提出反应水可以在Pi和ADP释放过程之间进行补充,为解旋酶的水解循环过程提供了更加精确的见解。
NS3-hel的抑制剂研究目前相对较少,主要通过虚拟筛选和分子动力学模拟的方式来寻找潜在的药物靶点。2016年Jain团队[36]通过FTmap预测出了两个最具有潜力的抑制剂结合位点,分别位于DomainI和DomainII之间及RNA结合通道处,这两处位点都极性疏水,非常适合进行高通量药物筛选和模拟筛选。随后,Mottin等[37]通过对RNA结合位点进行分子动力学模拟,观察到了其与RNA结合时的动态构象变化,为下一步抑制剂的设计提供了新的思路,如可以设计抑制剂阻止RNA与蛋白的结合。最近还有研究组通过基于片段的筛选技术和X-射线衍射技术最终得到了四种可以有效结合于解旋酶的小分子,它们分别结合于解旋酶的两个部位:NS3和NS5的接面处及三个结构域的交汇处[38]。这两个结合位点相对保守且对于复制复合物的形成及解旋酶结构域的转动非常重要,因此是很具有潜力的药物靶点。同时通过晶体学技术分析了小分子与解旋酶的结合方式,对于抑制剂的进一步改造优化提供了重要信息。
3 NS5的结构及其抑制剂设计
NS5包含两个结构域,其N端为甲基转移酶(methyl transferase,MTase),主要功能是甲基化基因组 RNA 5′端的帽子结构,对RNA的稳定、有效翻译及病毒逃避宿主免疫应答起重要作用[39]。近几年多个研究组相继报道了ZIKV NS5 MTase的结构。例如2016年Coloma研究组[40]解析的MTase与S-腺苷基甲硫氨酸(S-adenosyl methionine,SAM)及7-MeGpp的复合物结构(PDB ID:5KQR,5KQS)。2017年,Duan研究组[41]解析了MTase与SAM及RNA加帽类似物(m7GpppA)的复合物结构(PDB ID:5WZ1、5WZ2)。同年MTase与SAM类似物(MS2042)的结构也得到了报道(PDB ID:5ULP)[42]。除此之外,还有课题组解析了MTase与SAH/GTP、SAH/7meGpppA的复合物结构(PDB ID:5GOZ,5GP1)[43],同时筛选出了三个可能的抑制剂结合位点。以上复合物的结构在整体上相似,但因结合底物不同,SAM结合口袋及其附近的构象也有所变化,这都为MTase的分子机制研究和药物靶点的筛选提供了一定的理论基础(图5A)。
2017年,本实验室解析了MTase与SAH的复合物结构(PDB ID:5WXB)[44]。与之前的结构相似,该结构中的螺旋和折叠组成了典型的罗斯曼折叠(Rossman fold),SAH通过氢键网络结合于Rossman fold的N端。通过与MTase和SAM、7-MeGpp的复合物结构(PDB ID:5KQS)对比,揭示了MTase在将SAM转化为SAH过程中结合口袋的“缩小”变化(图5B),为MTase的催化过程提供了一定的结构基础[41,44]。最近,Chatrin研究团队[45]报道了MTase与SAM、SAH的复合物结构(PDB ID:5NJV、5NJU),在MTase与SAH复合物中发现一种loop L3/Helix A3新构像,与之前解析的结构不同,该位点出现了摆动,阻止了与新RNA链的结合,揭示了一种加帽完成后的MTase新状态(图5C),为MTase的分子机制提供了新的结构模型。
对于MTase,药物设计的难点在于宿主的MTase与ZIKV MTase相似度较高,所以开发的抑制剂需要更具特异性{Qadir, 2018 #18;Duan, 2017 #59;Coutard, 2016 #63}。之前的结构研究表明,MTase的SAM结合口袋及与其相邻的疏水腔在黄病毒中是一个非常保守的位点,但是与人类的氨基酸序列和构象却不同,因此适用于抑制剂的开发[40]{Lim, 2011 #122;Coloma, 2016 #58}。MS2042作为SAM的类似物,以4-氟苯基代替了甲基,在与MTase结合时同时占据了RNA的结合位点,阻止了MTase与RNA的结合,如果对这个基团进行进一步的修饰,也将是抑制剂开发的新方向[42]。基于模拟筛选技术,Zhang等[43]还筛选出了GTP/RNA结合位点,结合SAM结合位点及两者之间的极性疏水区域,有望联合这三个位点设计出亲和力更高的小分子抑制剂。近期更有研究人员通过基于片段的筛选技术筛选出了多个对MTase具有抑制作用的小分子片段,通过计算机建模和片段连接技术将其连接起来,逐步优化获得了具有更强抑制作用的小分子[46]。
NS5的C端为RNA依赖的RNA聚合酶(RNA-dependent RNA polymerase,RdRp),主要负责病毒的RNA的合成[39]。对于RdRp的结构,已经有几个研究组进行了解析(PDB ID:5WZ3、5U04、5U0C)[41,47-48]。RdRp呈典型“右手”球形构象,包括手指(fingers)、手掌(palm)和拇指(thumb)三个子结构域,同时存在两个锌离子结合口袋(图5D),在fingers和thumb之间包含了模板RNA结合通道、新合成的双链RNA结合通道以及NTPs的进入通道[41,47]。在三个通道的交汇部位,存在一段priming loop,负责RNA模板3′末端的变构定位,该部分的构象和表面电荷在不同黄病毒之间存在一定差异,成为特异性药物设计的新靶点[48](图5D)。
RdRp的药物开发包括核苷类或非核苷类抑制剂。有研究表明,将核苷2′位置上进行修饰得到的2′-C-甲基化核苷对ZIKV的复制具有很好的抑制效果,且细胞毒性较低[49-50],具有很大的开发潜力。非核苷类抑制剂主要通过变构作用来抑制RdRp活性,Pattnaik研究组[51]通过计算机筛选确定了一种非核苷类抑制剂TPB,分子对接实验表明其结合于RdRp的活性位点,能够有效抑制小鼠模型的病毒血症。由于RdRp目前还没有复合物结构得到解析,基于结构的药物设计还存在一些困难,2018年,ebera等[52]建立了RdRp与RNA的分子模型,为后续的分子建模等提供了结构基础。最近,Sáez-lvarez研究组[53]最新发明了一种基于荧光的检测技术,不仅能够实时检测RdRp的活性,同时也为RdRp进行高通量药物筛选提供了新的技术基础。除了以上技术的应用,一些其他黄病毒NS5抑制剂也可以用于ZIKV药物的开发,例如sofobuvir等[54-56]。
近两年,Zhao研究组[47]和Upadhyay[55]研究组先后报道了NS5的全长结构(PDB ID:5UOB、5TFR),分析表明MTase和RdRp两个结构域通过氢键产生相互作用,MTase对于RNA的合成有着不可或缺的作用,因此开发针对两者接触界面的抑制剂也具有一定的可行性。最新研究表明,黄病毒NS5的N端存在能够与小泛素样蛋白结合的保守基序,NS5的泛素化修饰对于基因组的复制及免疫逃避非常重要,因此针对小泛素样蛋白抑制剂的研发或许成为将来ZIKV药物开发的新方向[56]。
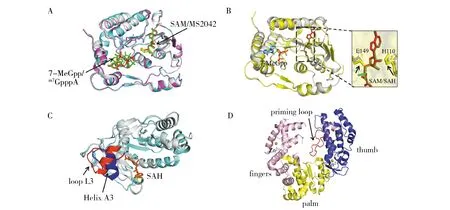
图5 ZIKV NS5 MTase与RdRp的结构[40-42,44-45]Fig.5 structure of ZIKV NS5 MTase and RdRp[40-42,44-45]A:MTase的复合物结构5KQS(MTase为灰色,SAM和7-MeGpp为红色球棍模型)、5WZ2(MTase为蓝色,SAM和m7GpppA为绿色球棍模型)与5ULP(MTase为紫色,MS2042为黄色球棍模型)的叠加对比;B:MTase与SAH的复合物结构(PDB ID:5WXB)与复合物5KQS(灰色)的对比及SAH/SAM结合口袋的比较;C:MTase与SAH的复合物结构(PDB ID:5NJU)与复合物5KQR的对比,它们的loop L3/Helix A3分别标为蓝色和红色;D:ZIKV NS5-RdRp的结构(PDB ID:5WZ3),priming loop标为红色,Zn2+为黄色球型A:Overlay of the MTase complex structure 5KQS (MTase is gray, SAM and 7-MeGpp are red ball stick models), 5WZ2 (MTase is blue, SAM and m7GpppA are green stick models) and 5ULP (MTase is purple, MS2042 is yellow ball),the three structures are overall similar;B:The comparison of the complex structure of MTase with SAH (PDB ID: 5WXB) and the complex 5KQS (gray), the comparison of SAH/SAM binding pockets is shown in details;C: The complex structure of MTase and SAH (PDB ID: 5NJU,cyan) is compared with the complex 5KQR(gray). The loop L3/Helix A3 of the two structures are marked as blue and red, respectively;D:ZIKV NS5-RdRp structure(PDB ID:5WZ3),the priming loop is marked in red,the yellow ball is Zn2+
4 展 望
ZIKV在全世界范围内的流行,引起了人们的广泛关注。但是,特异性疫苗和药物的缺乏使ZIKV成为了全球人类身体健康的潜在威胁。非结构蛋白NS1、NS3、NS5作为病毒增殖过程中的关键蛋白,成为小分子抑制剂筛选和设计的理想靶点。其三维空间结构的解析不仅从分子水平上揭示了蛋白的作用机制,为病毒复制等过程的解读奠定了结构基础,同时也为基于结构的药物设计提供了重要的理论依据。
目前针对ZIKV 非结构蛋白的药物开发方法主要可以归纳为以下几个方面:①对已知的DENV、HCV抑制剂的重新运用;②对现有药物进行高通量筛选;③通过计算机虚拟筛选预测可能的药物结合位点;④设计底物类似物抑制剂。应注意的是,ZIKV的非结构蛋白虽然在整体上与其他黄病毒相似,但是也存在一些特殊性,例如前面提到的NS1特殊的表面电荷、RdRp priming loop的不同构象等。这提示开发特异性针对ZIKV药物的必要性,同时也说明了针对蛋白的结构特点进行药物开发将是未来的主要方向。当然,抑制剂的开发还面临着许多困难和挑战,比如MTase与人体RNA MTase相似而导致的药物细胞毒性、现有小分子抑制剂的药代动力学差等,如何选择具有特异性的药物靶点和改善小分子的性质还需要我们进一步的研究探索。

