刺萼龙葵种子中适宜内参基因的筛选
赵丹丹 陈景超 黄兆峰 姜翠兰 黄红娟 张朝贤 李香菊 魏守辉
摘要 入侵杂草刺萼龙葵Solanum rostratum Dunal传播扩散的主要载体是种子,研究其种子休眠萌发基因的激素调控对于其防除具有重要意义,而选择合适的内参基因可以提高相关基因表达分析的准确性。本研究以赤霉素、脱落酸和水处理的刺萼龙葵种子为材料,利用geNorm、NormFinder、BestKeeper和RefFinder 4种软件对15个候选内参基因进行表达稳定性评价,并通过检测ABI5(abscisic acid-insensitive 5)的表达验证所筛选的内参基因的适用性。结果表明,对于赤霉素、脱落酸和水处理过的种子,最稳定的内参基因分别为eIF(eukaryotic initiation factor)、SAND(SAND protein family)和ACT(β-actin);对所有种子样本而言,PP2Acs(a catalytic subunit of protein phosphatase 2A)是最稳定的内参基因。研究结果将为刺萼龙葵种子休眠萌发的遗传调控研究提供重要参考。
关键词 刺萼龙葵; 内参基因; 种子; 赤霉素; 脱落酸
中图分类号: S 451.1
文献标识码: A
DOI: 10.16688/j.zwbh.2019105
Selection of suitable reference genes in Solanum rostratum seeds
ZHAO Dandan, CHEN Jingchao, HUANG Zhaofeng, JIANG Cuilan, HUANG Hongjuan,
ZHANG Chaoxian, LI Xiangju, WEI Shouhui
(Key Laboratory of Weed and Rodent Biology and Management, Institute of Plant Protection,
Chinese Academy of Agricultural Sciences, Beijing 100193, China)
Abstract
The main transmission way of the invasive weed buffalobur (Solanum rostratum) is seed dispersal. It is important to understand the hormonal regulation of seed dormancy or germination related genes in buffalobur. Selection of appropriate reference genes can improve the accuracy of expression analysis of these genes. Here, 15 candidate reference genes were selected to evaluate their expression stability in hormone-treated and water-treated seeds of buffalobur using geNorm, NormFinder, BestKeeper and RefFinder statistical algorithms. Validation of recommended reference genes were achieved by assessing the relative expression levels of abscisic acid-insensitive 5 (ABI5). The results showed that the most stable reference genes for gibberellin, abscisic acid and water-treated seeds were eukaryotic initiation factor (eIF), SAND protein family (SAND) and β-actin (ACT), respectively. For total seeds, PP2Acs, the gene encoding a catalytic subunit of protein phosphatase 2A was the most stable reference gene. This work would provide references for further research on the genetic regulation of seed dormancy and germination in this invasive plant.
Key words
Solanum rostratum; reference gene; seed; gibberellin; abscisic acid
外來入侵杂草刺萼龙葵Solanum rostratum原产于北美洲,是茄科茄属一年生草本植物[1]。1981年刺萼龙葵首次在我国辽宁朝阳被发现[2],随后逐渐扩散蔓延至吉林、河北、北京、内蒙古、山西等地[3]。刺萼龙葵可以降低入侵地的生物多样性、破坏其生态系统。入侵农田的刺萼龙葵除了直接与农作物竞争之外,还是多种病虫害的中间寄主,其植株含有毒素茄碱,可以使误食的牲畜中毒死亡[1]。刺萼龙葵的入侵对生态环境和农业生产均造成了严重影响,其防控问题受到人们的广泛关注[4]。由于化学除草剂的大量使用会对生态环境造成污染,而基于竞争替代的生态调控技术也存在应用范围较窄的局限性。因此,从刺萼龙葵种子休眠与萌发的遗传调控路径入手,有望发掘更为有效的防控方法[56]。
刺萼龙葵主要通过种子进行传播,其单株结实量可以达1万~2万粒。刺萼龙葵的种子具有复合休眠特性(物理休眠与生理休眠),在土壤中可以保持较长时间的种子活力[7]。前人研究发现赤霉素可以显著提高刺萼龙葵种子的萌发率,其种子的休眠和萌发主要受激素相关的内在遗传基因调控[5]。明确刺萼龙葵种子休眠和萌发调控的分子机理对合理调节其萌发出苗,进而制定有效的杂草防治措施具有重要价值。基因表达分析是揭示植物生命周期中基因功能和调控机制的重要手段。因此,确保相关基因表达分析的准确性十分重要。荧光定量PCR(real-time quantitative PCR, qPCR)因其灵敏度高、特异性强等优点,被广泛用于基因表达检测[8]。选用表达水平不随组织类型、发育阶段或浸种处理而显著变化的内参基因进行qPCR可以确保结果分析的准确性[9]。因此,在使用qPCR技术对基因表达进行检测分析之前,应筛选合适的内参基因并验证其稳定性。目前,有关刺萼龙葵种子适宜内参基因筛选的研究尚未见报道。
本研究以外源激素(赤霉素、脱落酸)处理的刺萼龙葵种子为材料,检测了三磷酸甘油醛脱氢酶基因(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)、肌动蛋白基因(β-actin,ACT)、谷氧还蛋白基因(glutaredoxin protein,GR)、泛素蛋白基因(ubiquitin,UBQ)、液泡膜内在蛋白基因(tonoplast intrinsic protein/aquaporin,TIP41)、18S核糖体RNA基因(18S rRNA,18S)、核糖体蛋白L8基因(ribosomal protein L8,RPL8)、肌动蛋白真核起始因子基因(actin eukaryotic initiation factor,eIF)、熱休克蛋白40基因[DnaJ-like protein (hsp40),DNAJ]、β-微管蛋白基因(β-tubulin,TUB)、亲环蛋白基因(cyclophilin,CYP)、转录延伸因子(elongation factor 1-alpha,EF1α)、蛋白磷酸酶2A的催化亚单位基因(a catalytic subunit of protein phosphatase 2A,PP2Acs)、1,5-二核酸核酮糖基因(ribulose 1,5 bisphosphate,RUBP)和SAND家族蛋白基因(SAND protein family,SAND)共15个内参基因的表达情况,用geNorm[10]、NormFinder[11]、BestKeeper[12]和RefFinder[13]4种软件对其表达稳定性进行分析,并对筛选出的内参基因进行了稳定性验证。其中geNorm和NormFinder均是通过计算内参基因表达稳定值大小来评价其稳定性,前者可选择出两个或两个以上基因作为标准化指标,而后者只能选择一个合适的内参基因;BestKeeper依据各基因间配对的相关系数、变异系数和标准偏差等值对内参基因稳定性进行评价;RefFinder内置的算法包括以上3种,其对候选内参基因的综合排序结果更可靠,避免了单个软件分析的片面性。本研究结果可以为准确检测刺萼龙葵种子休眠萌发相关基因的表达提供参考。
1 材料与方法
1.1 植物材料
刺萼龙葵种子于2018年采于北京市密云区(40°24′4″N, 116°50′21″E)。选取成熟、饱满、大小均匀的刺萼龙葵种子摆放在垫有两层滤纸的培养皿(直径15 cm)中,每皿摆放300粒。向培养皿中分别加入12 mL 0.35 mmol/L赤霉素(gibberellin,GA)、12 mL 0.35 mmol/L脱落酸(abscisic acid,ABA)或12 mL无菌水,以未处理的干种子作为空白对照。然后将培养皿转入光照培养箱中,在30℃±2℃、黑暗条件下培养24、48 h和72 h后分别收集种子样品。将所有的样品分为4组,即赤霉素、脱落酸、水及以上3组的混合样品(种子总体),每个样品设3个生物学重复,取样后立即放入液氮中冷冻并保存在-80℃冰箱中。
1.2 RNA提取及cDNA合成
使用RNAprep Pure植物总RNA提取试剂盒(天根生化科技(北京)有限公司)进行样品RNA的提取,RNA样品经1%琼脂糖凝胶电泳检测在28S和18S处均显示清晰单一的条带,无明显降解。利用NanoDrop TM One/OneC超痕量UV分光光度计测量RNA的浓度和纯度,其A260/A280在1.90~2.10之间,RNA的总体质量较好。采用TransScriptAll-in-One First-Strand cDNA Synthesis SuperMix for qPCR(北京全式金生物技术有限公司)反转录试剂盒对质量合格的RNA进行反转录,体系为20 μL,RNA质量均为800 ng。合成的cDNA保存在-80℃冰箱中。
1.3 候选基因的选择与引物设计
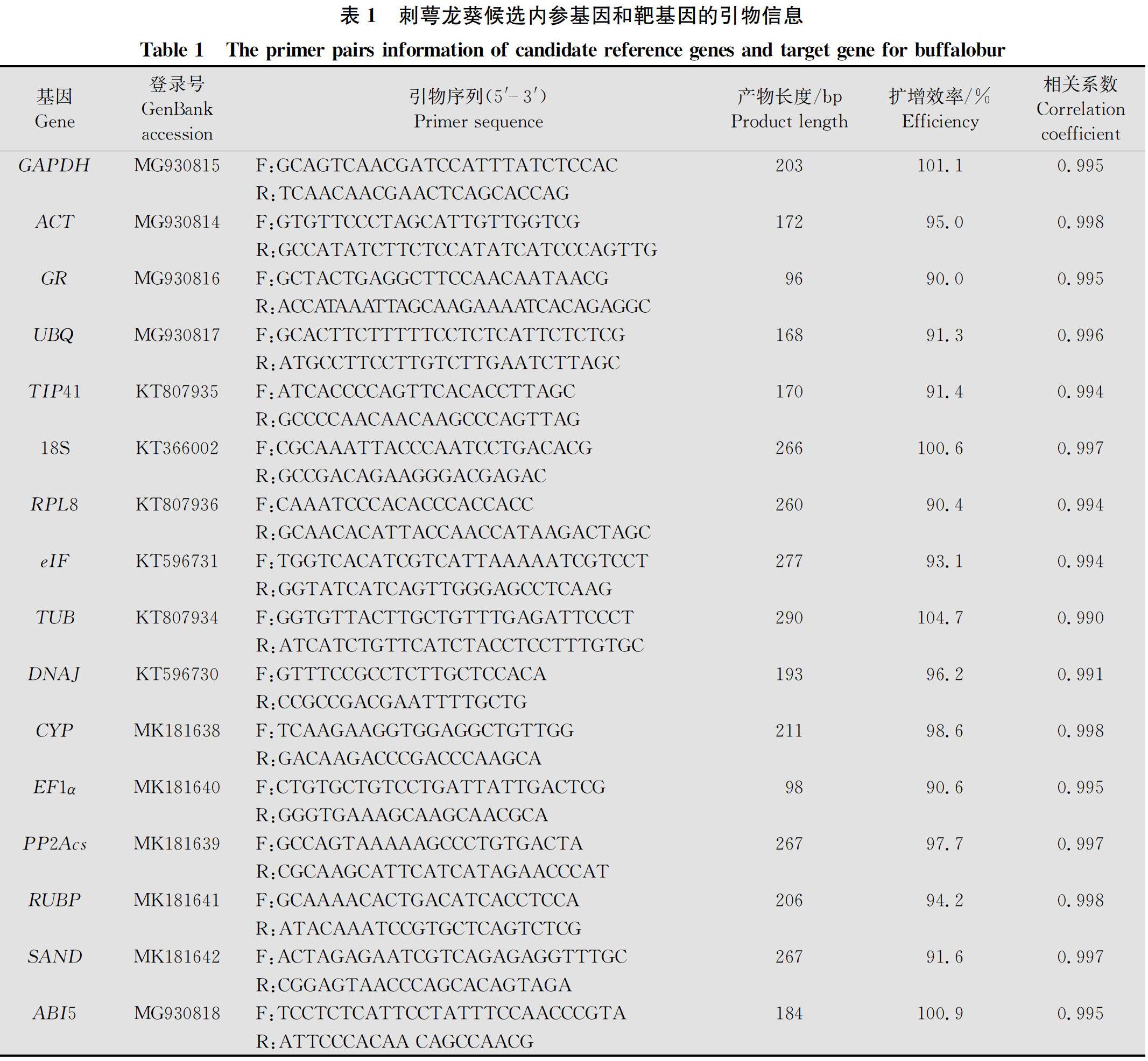
根据本实验室建立的刺萼龙葵转录组数据库以及文献中报道的常见内参基因,选择了15个刺萼龙葵的内参基因(GAPDH,ACT,GR,UBQ,TIP41,18S,RPL8,eIF,TUB,DNAJ,CYP,EF1α,PP2Acs,RUBP和SAND)作为候选。使用Oligo 7软件设计用于qPCR的引物,具体信息如表1。引物由上海生工生物工程股份有限公司合成。
1.4 常规PCR与qPCR反应
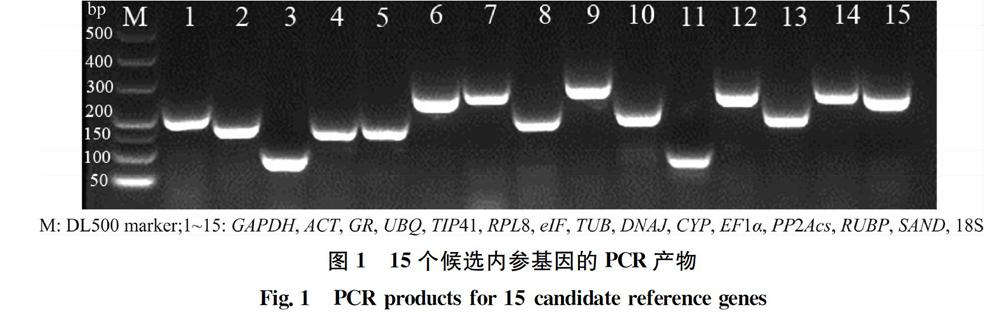
以刺萼龙葵种子的cDNA为模板,分别对15个候选内参基因进行常规PCR。反应体系为25 μL,包括:12.5 μL 2×Taq Master Mix,1 μL cDNA,2 μL上下游引物混合物(10 μmol/L),9.5 μL超纯水。反应程序为:94℃预变性5 min;94℃变性30 s,60℃退火30 s,72℃延伸15 s,35个循环;最后72℃延伸10 min。反应完成后,取5 μL扩增产物用2%的琼脂糖凝胶电泳进行检测,每对引物均扩增出了单一正确的产物,无可见的引物二聚体,说明引物的特异性良好(图1)。
2.2.4 RefFinder分析
RefFinder可以综合geNorm、NormFinder、BestKeeper和Delta-Ct 4种算法,列出15个候选内参基因的表达稳定性排名。对于赤霉素处理的种子,稳定性排名前三的内参基因是eIF、UBQ和EF1α;对于脱落酸处理的种子,最稳定的前三个内参基因是SAND、PP2Acs和GR;对于水处理的种子,最稳定的内参基因依次是ACT、SAND和PP2Acs;对所有样品进行分析,稳定性排名前三的内参基因分别是PP2Acs、SAND和eIF(表2)。
2.3 內参基因稳定性的验证
用不同处理的刺萼龙葵种子为材料,分别用筛选出的最稳定和最不稳定的基因为内参,检测了基因ABI5的表达。结果显示,种子经脱落酸处理后,用最稳定的基因作为内参,发现ABI5的表达水平没有显著差异;而用最不稳定的内参时,得出的结果是ABI5表达水平在24 h显著升高,后又显著降低(图4a)。对于赤霉素处理的种子,当用最稳定的内参基因时,ABI5的相对表达水平在0.5~1倍之间;当用最不稳定的内参基因时,ABI5在24 h时的表达量是0 h时的5倍(图4b)。对于水处理的种子,当用最稳定的内参基因时,ABI5的相对表达水平在0.8~1.6倍之间;当用最不稳定的内参基因时,ABI5的表达水平在处理后48 h之内显著升高,后又显著降低(图4c)。
3 讨论
qPCR具有灵敏度高、定量准确、特异性强等优点,是目前研究基因表达的常用方法。为了保证基因表达分析结果的准确性,除了规范操作流程之外,选择表达稳定的内参基因作为参照标准也非常重要。国内外大量研究表明,内参基因表达的稳定性会受到试验处理的影响,因此在进行基因表达分析之前筛选适合的内参基因有利于保证结果的准确性[15]。近年来,数字PCR(digital PCR,dPCR)技术发展迅速,虽然其较qPCR具有灵敏度更高、对抑制剂更耐受且不需要标准曲线与内参基因的优点,但易产生假阳性且成本较高;qPCR凭借更广的检测动态范围和更低的成本,目前仍有很广的应用范围,不可能完全被dPCR取代[16]。刺萼龙葵在中国的快速传播蔓延造成了严重的经济损失,由于化学防控与生物防控技术都存在一定的局限性,有学者利用刺萼龙葵种子具有复合休眠的特性,研究了其在不同胁迫处理下的萌发行为以及休眠相关基因的表达,力求找到更为高效的基于休眠萌发调控的防控措施[56]。为了筛选刺萼龙葵种子在激素处理下的最适内参基因,本研究使用4种算法对15个候选内参基因的表达稳定性进行了评估,不同软件得出的内参基因的稳定性排序有一定差异,这可能是因为每种软件的算法不同。geNorm的缺点在于其倾向选择具有相似表达谱的基因,例如最稳定基因可能具有同一类功能[10];NormFinder在计算标准化因子时考虑了组内和组间差异,但并不能排除样品制备过程中产生的系统误差[11];BestKeeper通过考虑单个样本的内在方差(InVar)克服了这一缺点,但是该软件至少需要3个内参基因的数据才能进行分析[12];RefFinder可以综合4种统计算法(geNorm、NormFinder、BestKeeper和Delta-Ct)来得出基因表达稳定性的综合排名。因此,使用多种评价软件来分析筛选内参基因,可以确保结果的可靠性。
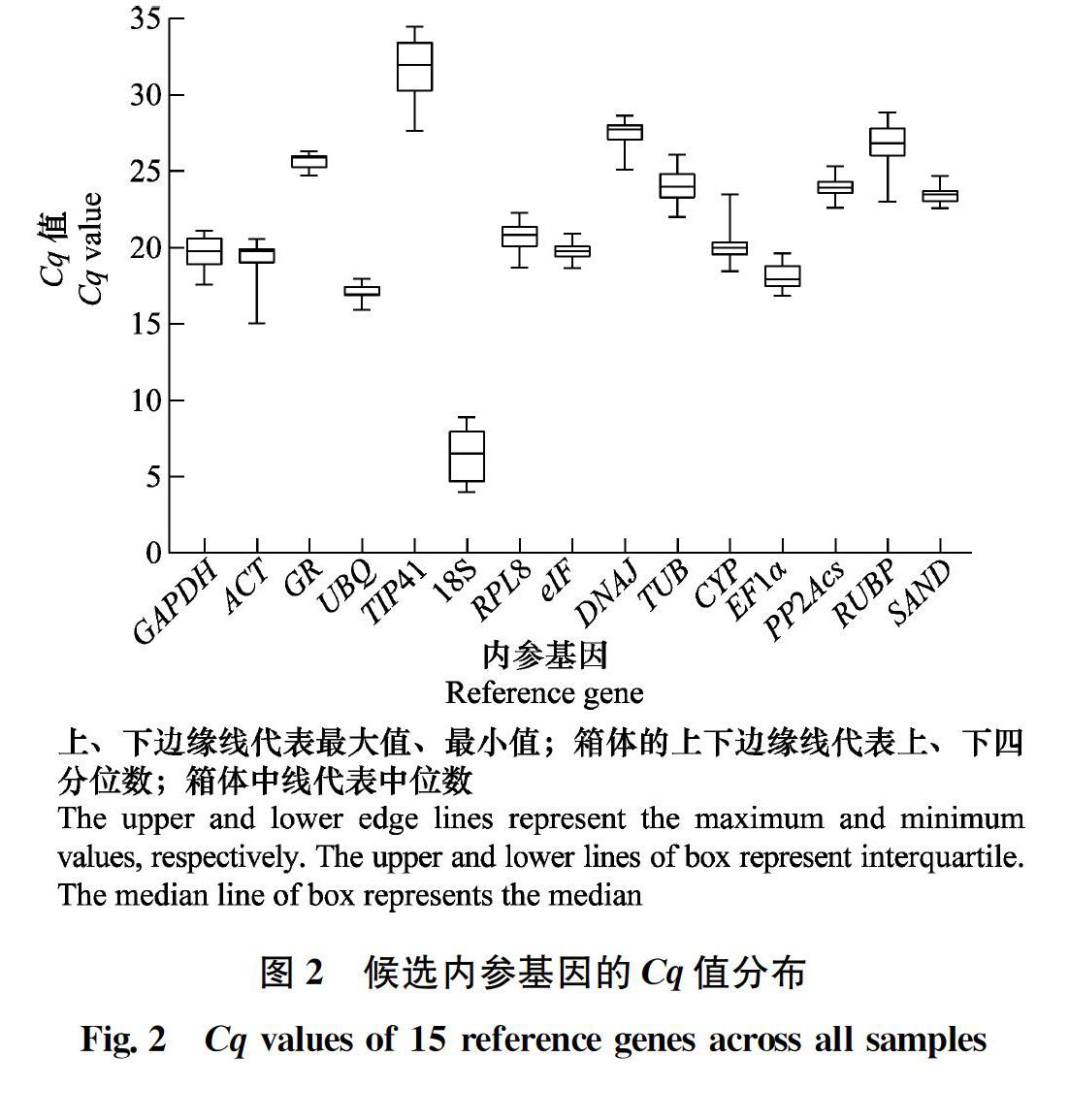
在本研究中,eIF在赤霉素处理的种子中表达稳定;同样的,eIF4A在亚麻Linum usitatissimum[17]以及干旱胁迫下的多花黑麦草Lolium multiflorum[18]中也表现出良好的稳定性。对于脱落酸处理的刺萼龙葵种子,内参基因SAND的表达最稳定;在不同组织的番茄[19]以及脱落酸胁迫处理下的狼毒Stellera chamaejasme[20]中,SAND同样稳定表达。在水处理过的刺萼龙葵种子中,稳定表达的基因为ACT,相似的结果也在成熟的花生Arachis hypogaea种子中发现[21];而在拟南芥Arabidopsis thaliana以及狗尾草Setaria viridis种子中,ACT的表达并不稳定[2223]。PP2Acs在刺萼龙葵所有种子样品中的表达最稳定;相似的结果在不同非生物胁迫下的西伯利亚剪股颖Agrostis stolonifera[24]以及不同激素作用下的御谷Pennisetum glaucum[25]中也被发现。候选内参基因TIP41和18S在试验中稳定性差,原因可能是其在刺萼龙葵种子中的表达水平过高或过低。由此可见,过高或过低的转录丰度不利于内参基因在样品中的稳定表达。理想的内参基因应具有高度或中度的表达水平,Cq值范围在15~30之间为宜。
ABI5主要参与种子成熟和萌发过程中ABA信号的传导过程[26]。在本研究中,用最佳内参基因组合与用单个最稳定内参基因标准化后得出的ABI5的表达水平相似,而这两者与用最不稳定内参基因得出的结果有显著差异,说明本研究所筛选出的内参基因是可靠的。
本研究评估了刺萼龙葵15个候选内参基因在赤霉素、脱落酸以及水处理的种子中的表达稳定性。在这些候选基因中,eIF在赤霉素处理的种子中表达最稳定,SAND在脱落酸处理的种子中最稳定,ACT在水处理的种子中最稳定,在所有样品中,PP2Acs是最稳定的内参基因。研究结果将为刺萼龙葵种子中相关功能基因的表达分析提供依据。
参考文献
[1] BASSETT I J, MUNRO D B. The biology of Canadian weeds. 78. Solanum carolinense L. and Solanum rostratum Dunal. [J]. Canadian Journal of Plant Science, 1986, 66(4): 977991.
[2] 关广清, 高东昌, 李文耀, 等. 刺萼龙葵—一种检疫性杂草[J]. 植物检疫, 1984, 4(11): 2528.
[3] 车晋滇, 刘全儒, 胡彬. 外来入侵杂草刺萼龙葵[J]. 杂草科学, 2006(3): 5860.
[4] 张少逸, 张朝贤, 杨连喜, 等. 茎叶除草剂对刺萼龙葵的防治效果评价[J]. 植物保护,2012,38(5):170173.
[5] WEI Shouhui, ZHANG Chaoxian, CHEN Xuezheng, et al. Rapid and effective methods for breaking seed dormancy in buffalobur (Solanum rostratum) [J]. Weed Science, 2010, 58(2): 141146.
[6] WEI Shouhui, ZHANG Chaoxian, LI Xiangju, et al. Factors affecting buffalobur (Solanum rostratum) seed germination and seedling emergence [J]. Weed Science, 2009, 57(5): 521525.
[7] 張少逸, 魏守辉, 张朝贤,等. 刺萼龙葵种子休眠和萌发特性研究进展[J]. 杂草科学, 2011, 29(2): 59.
[8] BUSTIN S, BENES V, NOLAN T, et al. Quantitative real-time RT-PCR——a perspective [J]. Journal of Molecular Endocrinology, 2005, 34(3): 597601.
[9] BUSTIN S A, BEAULIEU J F, HUGGETT J, et al. MIQE precis: Practical implementation of minimum standard guidelines for fluorescence-based quantitative real-time PCR experiments [J/OL]. BMC Molecular Biology, 2010, 11:74.
[10]VANDESOMPELE J, DE PRETER K, PATTYN F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes [J/OL]. Genome Biology, 2002, 3: research0034.1.
[11]ANDERSEN C L, JENSEN J L, RNTOFT T F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets [J]. Cancer Research, 2004, 64(15): 52455250.
[12]PFAFFL M W, TICHOPAD A, PRGOMET C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper——Excel-based tool using pair-wise correlations [J]. Biotechnology Letters, 2004, 26(6): 509515.
[13]XIE Fuliang, XIAO Peng, CHEN Dongliang, et al. miRDeepFinder: a miRNA analysis tool for deep sequencing of plant small RNAs [J]. Plant Molecular Biology, 2012, 80(1): 7584.
[14]LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method [J]. Methods, 2001, 25(4): 402408.
[15]BRUNNER A M, YAKOVLEV I A, STRAUSS S H. Validating internal controls for quantitative plant gene expression studies [J/OL]. BMC Plant Biology, 2004, 4(1): 14. DOI:10.1186/14712229414.
[16]冯兆民,舒跃龙. 数字PCR技术及其应用进展[J]. 病毒学报, 2017, 33(1): 103107.
[17]HUIS R, HAWKINS S, NEUTELINGS G. Selection of reference genes for quantitative gene expression normalization in flax (Linum usitatissimum L.)[J/OL]. BMC Plant Biology, 2010, 10(1): 71. DOI:10.1186/147122291071.
[18]LIU Qiuxu, QI Xiao, YAN Haidong, et al. Reference gene selection for quantitative real-time reverse-transcriptase PCR in annual ryegrass (Lolium multiflorum) subjected to various abiotic stresses [J/OL]. Molecules, 2018, 23(1): 172. DOI:10.3390/molecule.s23010172.
[19]EXPSITO-RODRGUEZ M, BORGES A A, BORGES-PREZ A, et al. Selection of internal control genes for quantitative real-time RT-PCR studies during tomato development process [J/OL]. BMC Plant Biology, 2008, 8(1): 131. DOI:10.1186/147122298131.
[20]LIU Xin, GUAN Huirui, SONG Min, et al. Reference gene selection for qRT-PCR assays in Stellera chamaejasme subjected to abiotic stresses and hormone treatments based on transcriptome datasets [J/OL]. PeerJ, 2018, 6: e4535. DOI:10.7717/peerj.4535.eCollection 2018.
[21]JIANG Shengjuan, SUN Yujun, WANG Songhua. Selection of reference genes in peanut seed by real-time quantitative polymerase chain reaction [J]. International Journal of Food Science & Technology, 2011, 46(10): 21912196.
[22]DEKKERS B J, WILLEMS L, BASSEL G W, et al. Identification of reference genes for RT-qPCR expression analysis in Arabidopsis and tomato seeds [J]. Plant and Cell Physiology, 2012, 53(1): 2837.
[23]MARTINS P K, MAFRA V, DE SOUZA W R, et al. Selection of reliable reference genes for RT-qPCR analysis during developmental stages and abiotic stress in Setaria viridis[J/OL]. Scientific Reports, 2016, 6: 28348. DOI:10.1038/srep28348.
[24]CHEN Yu, HU Baoyun, TAN Zhiqun, et al. Selection of reference genes for quantitative real-time PCR normalization in creeping bentgrass involved in four abiotic stresses [J]. Plant Cell Reports, 2015, 34(10): 18251834.
[25]SAHA P, BLUMWALD E. Assessing reference genes for accurate transcript normalization using quantitative real-time PCR in pearl millet [Pennisetum glaucum (L.) R. Br.] [J/OL]. PLoS ONE, 2014, 9(8): e106308. DOI:10.1371/journal.pone.0106308.eCollection 2014.
[26]DEKKERS B J, HE H, HANSON J, et al. The Arabidopsis delay of germination 1 gene affects ABSCISIC acid insensitive 5 (ABI5) expression and genetically interacts with ABI3 during Arabidopsis seed development [J]. The Plant Journal, 2016, 85(4): 451465.
(責任编辑:杨明丽)
收稿日期: 20190305 修订日期: 20190402
基金项目:国家自然科学基金(31572022);国家重点研发计划(2018YFD0200602);北京市自然科学基金(6182033)
致 谢: 参加本试验部分工作的还有江代礼、谭翰杰、张能和纪烨斌等同学,特此一并致谢。
通信作者E-mail:黄红娟hjhuang@ippcaas.cn;魏守辉shwei@ippcaas.cn
#为并列第一作者

