基于DNA折纸系统求解0-1整数规划问题的模型
严洋洋,殷志祥
(安徽理工大学数学与大数据学院,安徽淮南 232001)
0 引言
DNA纳米技术是一个多学科研究领域,能够在纳米级层次上进行计算.这一全新领域的开辟,为生物医学、生物制造、化工、材料等提供了很大的便利,如:药物传送[1,2]、纳米机器人[3,4].分子计算是一种具有巨大潜力的新兴技术,可以实现对纳米级物质结构的控制以及纳米计算.[5]其中,DNA链置换就是一种在分子水平上用于实现纳米级计算的简单而强大的自组装方法[6].由于DNA是一种具有稳定的规则的双螺旋结构的高分子化合物,具有精确的自组装能力、分子序列可编程性及良好的生物相容性[7],利用它的特殊的结构和碱基互补配对原则将其折叠成不同形状、不同尺寸的DNA纳米材料[8].
1982年,Seeman及其同事就提出了利用DNA分子构建不同简单结构的纳米器件的想法.几年后他终于利用DNA分子构建了三角形、方形等形状的结构.1991年和1993年Seeman成功的组装了三维的立方体结构和DX结构[8].随后,出现了越来越多的不同结构和尺寸的纳米结构和纳米器件.如Yan[9]等构建的的十字Tile以及具有设定尺寸的二维阵列图形[10].
在Seeman等人的基础上,加州理工学院的Rothemund在2006年开发了一种新的DNA自组装技术——DNA折纸术.相较于之前的“自下而上”的DNA自组装方法,不再是组装一些简单的具有规则的结构,而是一种“自上而下”的全新的策略,它能够构建出更多样更复杂的自组装纳米结构,而且结构的稳定性也大大的提高了,可控性更强了.DNA折纸是利用一条长单的DNA链(脚手架链)在特定的位置利用短单的DNA链(订书钉链)通过分子间碱基互补配对的原则,将长单的DNA链设计构造出不同形状、不同尺寸的自组装结构[10].Rothemund还提出了对单个DNA自组装结构进行编程以设计出更大的DNA纳米结构,并且利用六个三角形成功地组装了一个三角形六聚体,为后来的大结构组装提供了基础.可以说,DNA折纸使DNA纳米技术登上了一个新的台阶.2006年,钱璐璐[11]利用DNA折纸成功的“折”出了第一个非对称的纳米图形——中国地图.此后,在Rothemund的基础上,许多研究组对折纸进行了改进,设计了三维结构的DNA折纸[12]以及能够自动完成任务的计算机辅助程序等[13].2008年,Andersen[14]等进一步的发展了DNA折纸,不但成功的组装了一个海豚,而且还为海豚添加了一条可以摆动的尾巴.文献12-14的研究结果,说明了DNA折纸不仅可以折出非对称的纳米结构,而且还可以将DNA折纸应用到动态的纳米结构中,使得DNA纳米结构变得更加多样化、复杂化.文献15,基于DNA折纸设计了一个动态DNA系统,用于动态DNA纳米技术的集成开发环境.文献16介绍了一种在尺寸上能够达到微米级别具有可控性的分形组装的方法.2018年,Enzo Kopperger[17]等人设计了一个由电场控制的自组装纳米级机械臂,能够在创建的分子平台上进行移动.2019年,庄小威课题组创建了基于DNA折纸的转子成像追踪技术,在单分子成像方面取得重大的突破[18].1994年,Adleman借助一种基于DNA的多项式时间的方法解决了7顶点图的Hamilton路径问题.于是开始借助DNA的并行性解决一些NP问题(Non-deterministic Polynomial).0-1整数规划问题(0-1 Programming Problem)是一类特殊的整数规划问题,也是一个十分经典的NP问题.在很多方面都有着举足轻重的应用,诸如背包问题,图的最大团等也可以转化为0-1规划.常用穷举法、分支定界法等解决这一问题,可至今为止还没有一个通用的算法来完全解决.殷志祥等[19]在2003年,设计了一种基于表面的DNA计算荧光标记策略,用于解决0-1规划问题.文献20,提出了一种双重编码的方式的DNA计算模型来解决0-1问题.同年,杨静[21]也设计了一种基于三链DNA结构的算法解决此问题.文献22和文献23分别将DNA链置换和DNA折纸术应用于解决0-1规划问题.
本文主要根据链置换的组装方式设计了一个DNA折纸系统求解0-1规划问题,该折纸系统由DNA折纸基底和四种类型的辅助链组成.在加入输入链后,经过链置换,释放辅助链上金纳米颗粒,实现每一个约束条件,通过辅助链上金纳米颗粒接收和释放的情况得出问题可行解.
1 DNA折纸系统
该DNA折纸系统[24]主要有两部分组成,DNA折纸基底(图1a),四种类型的辅助链(DNA/AuNP共轭物)组成(图1b).该折纸基底(其折纸原理来源于Rothemund开发的折纸术,为了合成矩形DNA折纸,将1.6 nM的M13 mp18链与订书钉链在1×TAE / Mg2 +缓冲液中以1:10的比例混合,并从90°C退火至室温.)由一条约为7 000 bp的圆形单链M13 mp18和202条短订书钉链折叠而成的二维矩形折纸模板,尺寸为90 nm×60 nm×2 nm(图1a中的矩形).在折纸基底上延伸出四条辅链设计了四个金纳米颗粒的捕获位置,分别为a,b,c,d.
通过硫醇化后互补DNA链修饰金纳米颗粒,设计了四种类型的辅助链(图1b),即NP-A,NP-B,NP-C,NP-D,分别标记为a1,b1,c1,d1,并且每条辅助链的一端带有15 nm的金纳米颗粒(图1b 黄色用小圆圈表示).(矩形折纸模板和DNA/AuNP共轭物在反应中以1∶30比例混合,并从40 °C退火至室温)与四种类型的辅助链相对应的是四种类型输入链(图1c)a-L,b-L,c-L,d-L,能够识别识别折纸基底延伸的a,b,c,d的输入区,并且优先于置换出辅助链a1,b1,c1,d1.
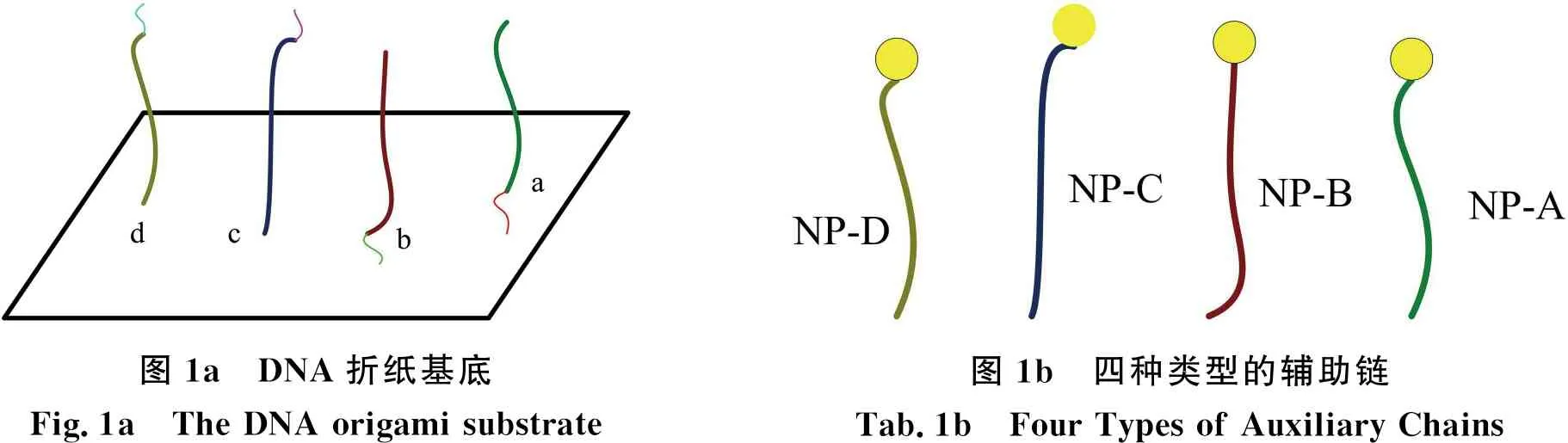
图1a DNA折纸基底Fig.1a The DNA origami substrate图1b 四种类型的辅助链Tab.1b Four Types of Auxiliary Chains

图1c 输入链a-L,b-L,c-L,d-LTab.1c The Input Chain a-L, b-L, c-L, d-L图1d DNA折纸系统 Tab.1d The DNA Origami System

图2 AuNPs 释放过程Tab.2 The Release Process of AuNPs
在初始的状态下,将DNA折纸基底与NP-A,NP-B,NP-C,NP-D进行反应,充分反应后,自组装成折纸系统.然后,再加入四种类型输入链 a-L,b-L,c-L,d-L,进行反应.接着,观察反应后的每个折纸基底上金纳米颗粒的释放情况,作出判断.即在该计算系统中,可行解的判定是通过DNA链置换在DNA折纸基底上选择性的释放金纳米颗粒来实现的.
2 0-1 整数规划问题的DNA折纸计算模型
0-1整数规划问题是一种特殊的整数规划问题,它的变量xi的取值仅为0或1.它在工程、市场分析、方案选择等方面都有着十分重要的应用.
它的一般形式为:
2.1 0-1整数规划问题的DNA折纸计算模型的基本算法:
步骤1:生成所求问题中变量0或1的所有可能性组合;
步骤2:利用每条约束条件删除非可行解,保留可行解;
步骤3:重复Step2,直至找到所有的可行解;
步骤4:得到各个可行解对应得目标函数值,并进行比较,获取最优解.
2.2 0-1整数规划问题的DNA折纸计算模型的生物算法:
对于一个含有n个变量x1,x2,…,xn和m个方程的0-1整数规划问题的方程组,


步骤3 经过前一个约束条件判断后不满足该约束条件的可能性组合,后一个约束条件不在进行判断.重复进行步骤2,直至m个约束条件,找出全部可行解的组合,判断结束.
步骤4 计算得到各个可行解对应的目标函数值,然后将得到的函数值进行比对,进而找到最优解.
3 具体实例
这里就一个简单的实例进行说明,该DNA折纸系统在0-1整数规划问题的应用:
步骤1 对各个变量进行指派,由于变量的取值只能是0或1,故在这个实例中共有8种不同的指派方式,分别为x1=1或x1=0,x2=1或x2=0,x3=1或x3=0,x4=1或x4=0所有的可能性组合为24=16种.下面将这8种不同的指派分为两组,前一组辅助链经过金纳米颗粒修饰,表示为x1=1,x2=1,x3=1,x4=1.前一组辅助链未经过金纳米颗粒修饰表示为x1=0,x2=0,x3=0,x4=0.取四种不同的辅助链与折纸基底组装成DNA折纸系统,分别用1(0 0 0 0), 2(1 0 0 0), 3(0 1 0 0), 4(0 0 1 0),5(0 0 0 1), 6(1 1 0 0), 7(1 0 1 0), 8(1 0 0 1),9(0 0 1 1),10(0 1 0 1),11(0 0 1 1), 12(1 1 1 0),13(1 1 0 1),14(1 0 1 1),15(0 1 1 1),16(1 1 1 1)表示.如(0 0 0 0)表示x1=0,x2=0,x3=0,x4=0,(0 0 1 1)表示x1=0,x2=0,x3=1,x4=1.
步骤2 DNA折纸系统组装完全后,加入辅助链a-L,b-L,c-L,d-L,进行链置换反应,单链a-L,b-L,c-L,d-L将单链a,b,c,d置换出,并与单链a1,b1,c1,d1互补形成双链.然后借助电镜观察.若是能够满足约束条件,则可以在DNA折纸系统中观察到金纳米颗粒的存在.若是不能满足约束条件,则观察不到金纳米颗粒的存在,即辅助链上金纳米颗粒被释放了.
针对第一个约束条件x1+x2+x3+x4≥2,经过DNA链置换后该折纸系统中能顾观察到的金纳米颗粒至少要有2个,满足条件的可能组合为:6(1 1 0 0), 7(1 0 1 0), 8(1 0 0 1),9(0 0 1 1),10(0 1 0 1),11(0 0 1 1), 12(1 1 1 0),13(1 1 0 1),14(1 0 1 1),15(0 1 1 1),16(1 1 1 1).

图3 可行解(1 1 0 0)示意图Tab.3 Diagram of the Feasible Solution (1, 1, 0, 0)
步骤3,重复进行步骤2,直至所有约束条件检测完全,找出所有满足约束条件的可能性组合.不满足前一个约束条件的可能性组合删除,即该组合不是可行解.
依次检测,针对第二个约束条件x1+x3+x41,满足该条件的可能组合为:6(1 1 0 0),9(0 0 1 1),10(0 1 0 1)
针对第三个约束条件x2+x3+x41,满足该条件的可能组合为:6(1 1 0 0).最后能够找到的可行解仅有6(1 1 0 0),即x1=1,x2=1,x3=0,x4=0.
最后在DNA折纸系统中的结果为:
步骤4 对于该实例,可行解只有一个(1 1 0 0),计算可得到目标函数:S=5
4 结论
在借助DNA自组装构建DNA纳米材料方面已经取得了很大的成就.2006年Rothemund开发的DNA折纸术更是在构建纳米结构方面向前迈出了一大步.文中主要通过DNA折纸建立一个折纸系统来解决0-1整数规划问题.借助DNA链置换有选择性的释放金纳米颗粒.结果的判断是通过电镜观察金纳米颗粒是否从折纸系统中释放来表示.这样设计不仅操作简单,结果也很容易观察和检测.不足的一点是,由于DNA折纸基底是由一条长单链折成的,于是基底的尺寸就受到了限制,由此也限制了折纸系统的规模.这样就只能解决一些变量比较少0-1整数规划问题.若变量过多就会单链之间发生反应导致错排,DNA链之间发生反应,同时也不利于结果的检测.但随着研究的不断进展,将来更大规模的DNA折纸结构也最终能够设计出来,也将会有更加广泛的应用.

