玉米大斑病菌StNPS6基因缺失突变体的转录组与蛋白质组差异表达分析
李宛泽, 刘 洋, 张艳华, 刘金亮, 潘洪玉, 张祥辉
(1.吉林农业科技学院 农学院, 吉林 吉林 132001; 2.吉林大学 植物科学学院, 长春 130012)
玉米大斑病(northern corn leaf blight)是玉米主要的真菌病害之一, 影响玉米的产量和品质.非核糖体肽合成酶基因6(StNPS6基因)是编码非核糖体肽合成酶家族(NRPS)基因中的一员, 其结构稳定, 是丝状真菌胞外铁载体生物合成酶的关键基因.NPS6基因在玉米小斑病菌、水稻胡麻叶斑病菌、禾谷镰孢菌以及链格孢菌中均与致病力有关: Lee等[1]对玉米小斑病菌中的12个NPS基因进行了敲除分析, 结果表明, 仅NPS6基因与致病力相关, 且对H2O2敏感性增加; 在柑橘病原菌链格孢菌中,NPS6基因缺失影响链格孢菌铁载体的产生和致病力[2]; 将稻瘟菌一个编码NRPS的基因SSM2敲除后, 可导致稻瘟菌不能产生任何铁载体、菌丝生长速率变慢及分生孢子产生数量变少, 且对氧胁迫更敏感[3].
为明确StNPS6基因在玉米大斑病菌中的作用, 本文构建玉米大斑病菌T-DNA插入突变体库, 获得StNPS6基因敲除突变体, 并利用蛋白质组和转录组差异表达分析对StNPS6基因相关调控网络进行分析, 为了解StNPS6基因与玉米大斑病菌致病力的关系提供理论依据.
1 材料与方法
1.1 材 料
用固体或液体马铃薯葡萄糖培养基(PD或PDA)培养玉米大斑病菌野生型菌株St28A及转化突变体菌株.用Luria-Bertani(LB)培养基培养农杆菌AGL-1.
1.2 方 法
1.2.1 构建玉米大斑病菌T-DNA插入突变体库 用农杆菌介导法进行玉米大斑病菌转化.先用热激法将质粒pBI-G3C转入农杆菌菌株AGL-1中, 将含有pBI-G3C的农杆菌AGL-1在添加卡那霉素(50 μg/mL)的基本(MM)培养基上28 ℃培养36 h.再用诱导培养基稀释农杆菌菌悬液, 并加入卡那霉素和乙酰丁香酮, 28 ℃震荡培养6 h后, 与玉米大斑病菌孢子悬浮液(106个/mL)混合.取400 μL混合液涂在铺有纤维素膜的诱导培养基上, 28 ℃培养48 h后, 将纤维素膜转移到共培养的培养基上.7 d后, 将候选转化子转移到新的含有潮霉素的肉汤(CM)培养基上, 并进行单孢分离.通过聚合酶链式反应(PCR)检测hph基因对候选转化子进行鉴定.
1.2.2 筛选致病力下降转化子 对具有潮霉素抗性的转化子进行致病力测定.将10 μL无菌水配制的孢子悬浮液(105个/mL)滴在玉米叶片上(品种: 先玉335), 7 d后观察发病情况.每次接种3个叶片, 每个实验重复3次.
1.2.3 T-DNA插入位置分析 根据T-DNA序列设计TAIL-PCR特异性引物, 三对特异性引物分别为
LB1: CTTGGTTGACGGCAATTTCGATGAT;
LB2: ATGATGCAGCTTGGGCGCAGGGTC;
LB3: CGTCCGAGGGCAAAGGAATAGAGTA;
RB1: CTTACAATTTCCATTCGCCATTCAG;
RB2: ATCGGTGCGGGCCTCTTCGCTATTA;
RB3: TGACTCCCTTAATTCTCCGCTCAT.
简并引物为
AD1: TG(A/T)GNAG(A/T)ANCA(G/C)AGA;
AD2: (A/T)AGTGNAG(A/T)ANCANAGA.
TAIL-PCR反应程序和反应参数参考文献[4].通过玉米大斑病菌基因组数据库(http://genome.jgi-psf.org/Settu1/Settu1.home.html)和NCBI数据库(http://www.ncbi.nlm.nih.gov/BLAST)做BlastN算法比对, 进行核苷酸同源性搜索, 确定插入位点.
1.2.4 目的基因敲除与突变体验证 用农杆菌介导的遗传转化敲除玉米大斑病菌StNPS6基因[5].设计特异性引物扩增StNPS6基因上下游各800 bp的片段, 并用PCR产物纯化试剂盒进行纯化.将上下游片段构建到入门载体pOSCAR上, 并进行验证, 将验证正确的载体命名为pOSCARNPS6.先用热激法将pOSCARNPS6转入农杆菌AGL-1中, 再将含有质粒pOSCARNPS6的AGL-1转化为玉米大斑病菌, 并用PCR方法对具有潮霉素抗性的转化子进行验证.
1.2.5 荧光定量分析 为进一步鉴定StNPS6基因的表达量, 分别从培养5 d的野生型和突变体菌丝中提取RNA, 并进行反转录合成cDNA, 用ABI7500荧光定量PCR仪测定StNPS6基因的表达量, 用2-ΔCt方法分析数据, 以Tub2和GAPDH基因为内参基因.
1.2.6 RNA提取 收集在添加2,2-联吡啶的MM培养基中培养的野生型与突变体菌丝, 液氮迅速冷冻后, 用RNA提取试剂盒提取RNA.用Nanodrop检测RNA的纯度和浓度.最后用质量分数为1%的琼脂糖凝胶电泳检测RNA, 由北京百迈克公司进行基因测序.
1.2.7 菌丝蛋白质提取 用TCA-丙酮法提取野生型玉米大斑病菌与突变体菌丝的蛋白质, 并进行定量分析.称取约1 g菌丝于液氮中研磨.将粉末转移至2 mL EP管中, 加入500 μL TCA-丙酮溶液, 充分混合后于-20 ℃沉淀过夜, 并于4 ℃, 20 000 r/min离心30 min, 弃上清液, 向沉淀中加入等体积的冷丙酮(含体积分数为0.07%的β-巯基乙醇), 充分混匀, 于4 ℃, 20 000 r/min离心15 min, 弃上清液, 室温干燥5 min, 加入300 μL裂解液, 室温溶解30 min, 保存备用.
1.2.8 蛋白质双向电泳 每个样品取200 μg进行双向电泳分析, 第一向电泳选择17 cm、pH3-10的非线性IPG胶条进行等点聚焦, 其程序为50 V 12 h,500 V 1 h,1 000 V 1 h,10 000 V 1 h,10 000 V 11 h.第二向电泳在质量分数为12%的SDS-PAGE分离胶进行.电泳结束后用考马斯亮蓝G-250进行染色, 将所得2-DE凝胶图像用PDquest 8.0软件分析.筛选差异表达量在2倍以上的蛋白质点进行下一步分析.
1.2.9 差异蛋白质的质谱鉴定 用MALDI-TOF-TOF/MS/MS分析差异蛋白质点.用Mascot 2.3软件分析质谱结果, 并与NCBInr数据库以及玉米大斑病菌基因组数据库(https://genome.jgi.doe.gov/Settu3/Settu3.home.html)进行比对, 对相关蛋白质点进行鉴定.
1.2.10 转录组数据分析 将测序得到的原始数据去除接头序列和低质量序列, 用TopHat v2.0.12 软件将所得序列与玉米大斑病菌基因组序列(http://genome.jgi-psf.org/Settu1/Settu1.home.html)进行对比.用FPKM(fragments per kilobase of exon per million fragments mapped)方法计算基因表达水平.以FDR≤0.001, 且表达量≥2倍为标准确定差异表达基因.将筛选获得的差异基因进行GO(gene ontology)功能注释和KEGG(Kyoto encyclopedia of genes and genomes)聚类分析.
2 结果与分析
2.1 致病力下降突变体相关基因的鉴定
以1×106个/mL的分生孢子浓度进行遗传转化, 潮霉素为抗性筛选标记, 建立玉米大斑病菌的突变体库.通过提取玉米大斑病菌转化子基因组DNA以及PCR方法证明带有hph抗性标记的T-DNA片段是否插入到玉米大斑病菌基因组内.对获得的约2 000个T-DNA插入突变体进行致病力分析, 结果如图1所示.由图1可见, 与野生型相比, 9株突变体菌株的致病力明显下降, 其中突变体AMT69的致病力下降最明显.通过分析T-DNA侧翼序列, 并与玉米大斑病菌基因组进行比对, 确定T-DNA在AMT69突变体中的插入位点为非核糖体肽合成酶6基因(StNPS6).

图1 转化子致病力检测Fig.1 Detection of pathogenicity of transformants
2.2 StNPS6敲除突变体的验证
用农杆菌介导法对玉米大斑病菌中的StNPS6基因进行敲除.通过对转化子进行PCR验证, 获得3株StNPS6基因敲除突变体(Δnps6), 结果如图2所示.
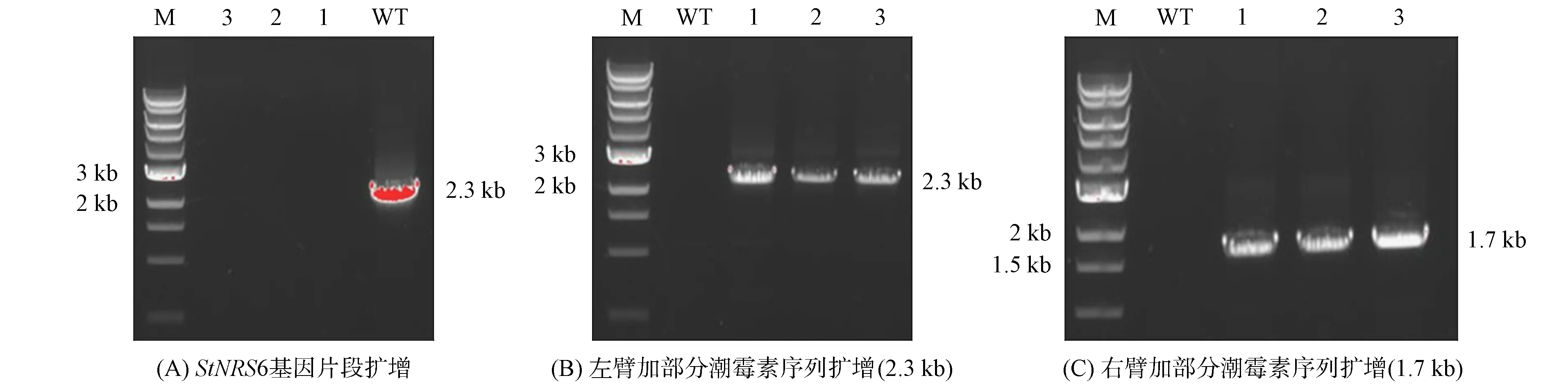
WT为野生型菌株; 1,2,3为突变体菌株.图2 StNPS6基因突变体PCR鉴定结果Fig.2 PCR identification results of StNPS6 gene mutants
2.3 StNPS6基因对致病力的影响

图3 野生型菌株和突变体菌株致病力差异比较Fig.3 Comparison of pathogenicity between wild type and mutant strains
为验证StNPS6基因敲除后对致病力的影响, 对生长14 d的玉米叶片进行接种, 7 d后观察, 结果如图3所示.由图3可见, 与野生型相比, T-DNA插入突变体与StNPS6敲除突变体的致病力均明显下降, 表明StNPS6基因对玉米大斑病菌的致病力影响较大.
2.4 差异蛋白质鉴定
为明确StNPS6基因敲除后对相关蛋白质表达的影响, 用双向电泳对野生型和StNPS6基因敲除突变体进行分析, 用MALDI-TOF-TOF/MS/MS鉴定的蛋白质数据进行Mascot(http://www.MatrixScience.com)分析.第一次选取在突变体中差异表达变化最显著的30个蛋白质点, 其中8个蛋白质上调表达, 22个下调表达, 结果列于表1.与转录组差异分析比较, 在30个鉴定的蛋白质中, 下调表达蛋白占比大于70%, 主要原因可能是StNPS6为次生代谢产物合成基因, 当其缺失后, 下游相关基因表达受到抑制, 导致下调表达蛋白质占比较大.
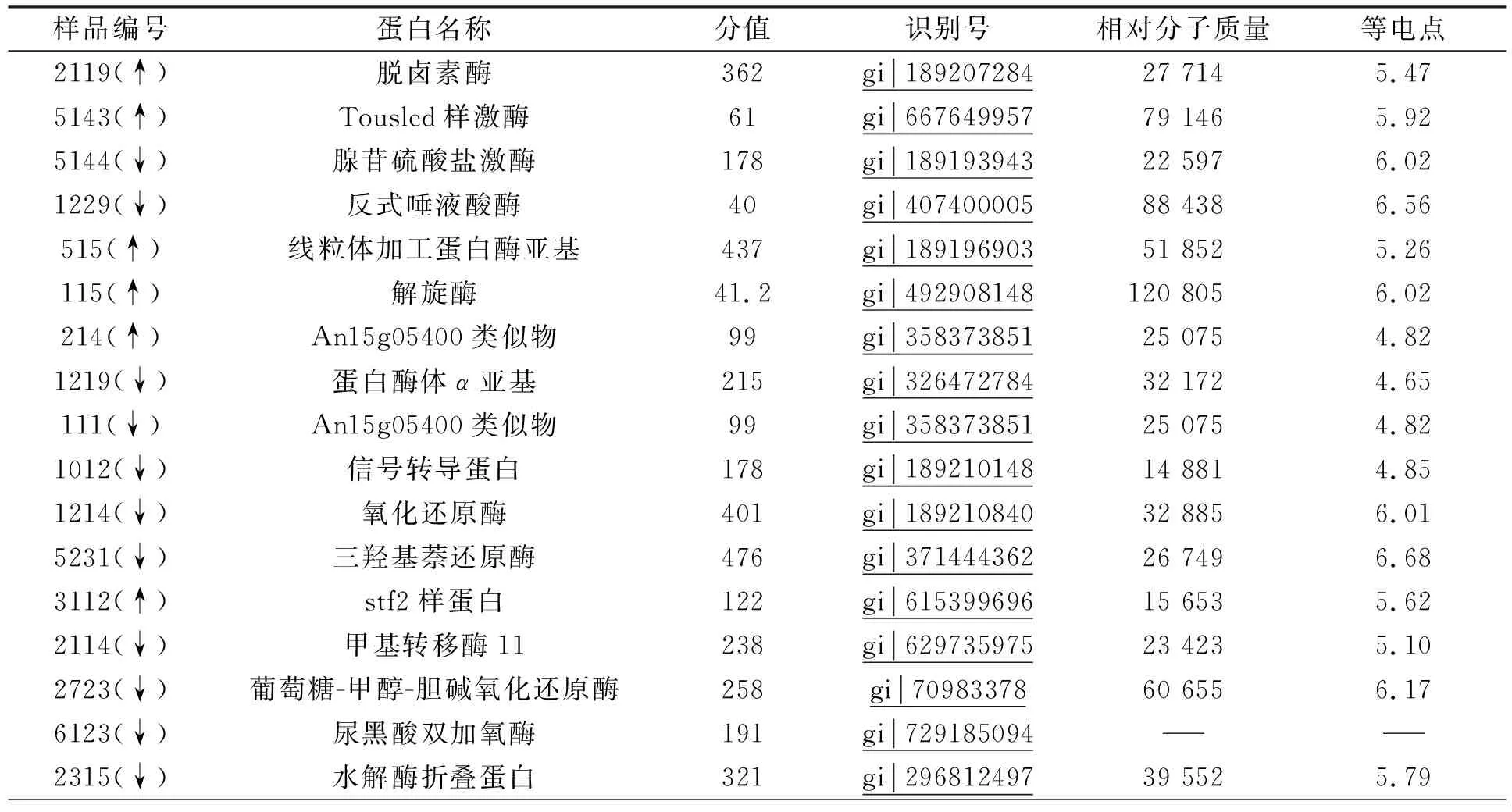
表1 差异蛋白鉴定结果
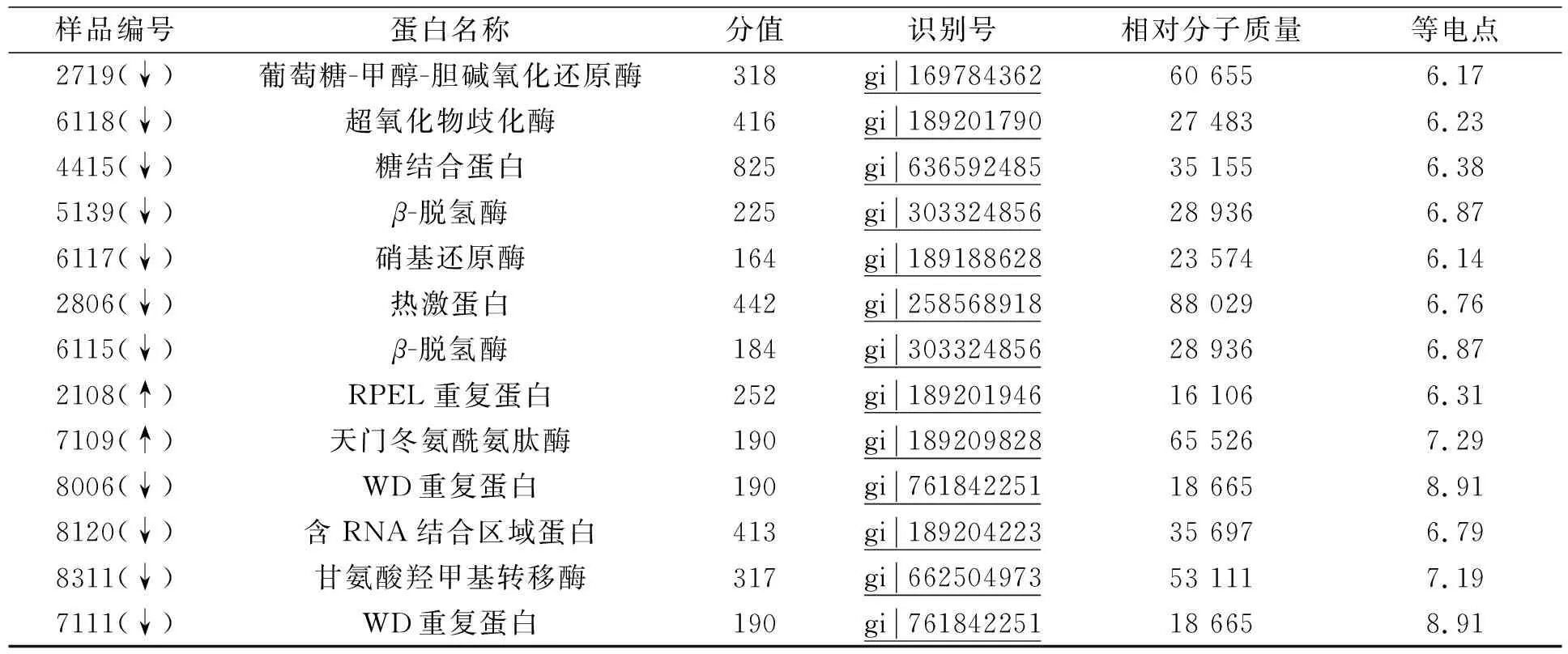
续表1
2.5 转录组测序数据与参考基因组比对分析
将野生型玉米大斑病菌及StNPS6基因突变体菌株转录组测序获得的reads与玉米大斑病菌基因组进行比对, 共有1.71×108个reads比对到参考基因组上, 约占总有效reads的80%(总有效reads共2.14×108个).其中, 80.82%比对到外显子区, 1.41%比对到内含子区, 17.77%比对到转录间隔区.
2.6 差异表达基因与功能注释
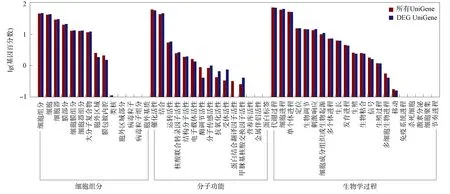
图4 差异表达基因的GO聚类分析结果Fig.4 Cluster analysis results of GO of differently expressed genes
通过对转录组测序后的基因注释及表达量分析, 共得到野生型菌株与突变体菌株差异表达基因1 767个, 其中上调表达基因903个, 下调表达基因864个.差异表达基因的GO富集聚类分析结果如图4所示, 共涉及50种生物功能, 其中生物学过程21个, 细胞组分14个, 分子功能15个.生物学过程主要包括代谢过程(25.8%)、细胞进程(23.7%)和单个体进程(19.0%); 细胞组分主要包括细胞(22.7%)、细胞器(15.9%)和细胞组分(24.5%); 分子功能主要包括催化活性(47.7%)和结合(38.8%).对1 767个差异表达基因进行KEGG通路富集聚类分析, 结果如图5所示.由图5可见, 342个差异表达基因比对到4条大的信号通路上.其中, 最主要的信号通路为代谢和遗传信息过程, 各占45.6%, 细胞过程和环境信息过程分别占7%和1.8%.将342个差异表达基因比对到50个信号途径上, 主要包括剪接体(27)、真核生物核糖体生物源(25)、RNA转运(22)、核糖体(17)、嘧啶代谢(16)和嘌呤代谢(15).
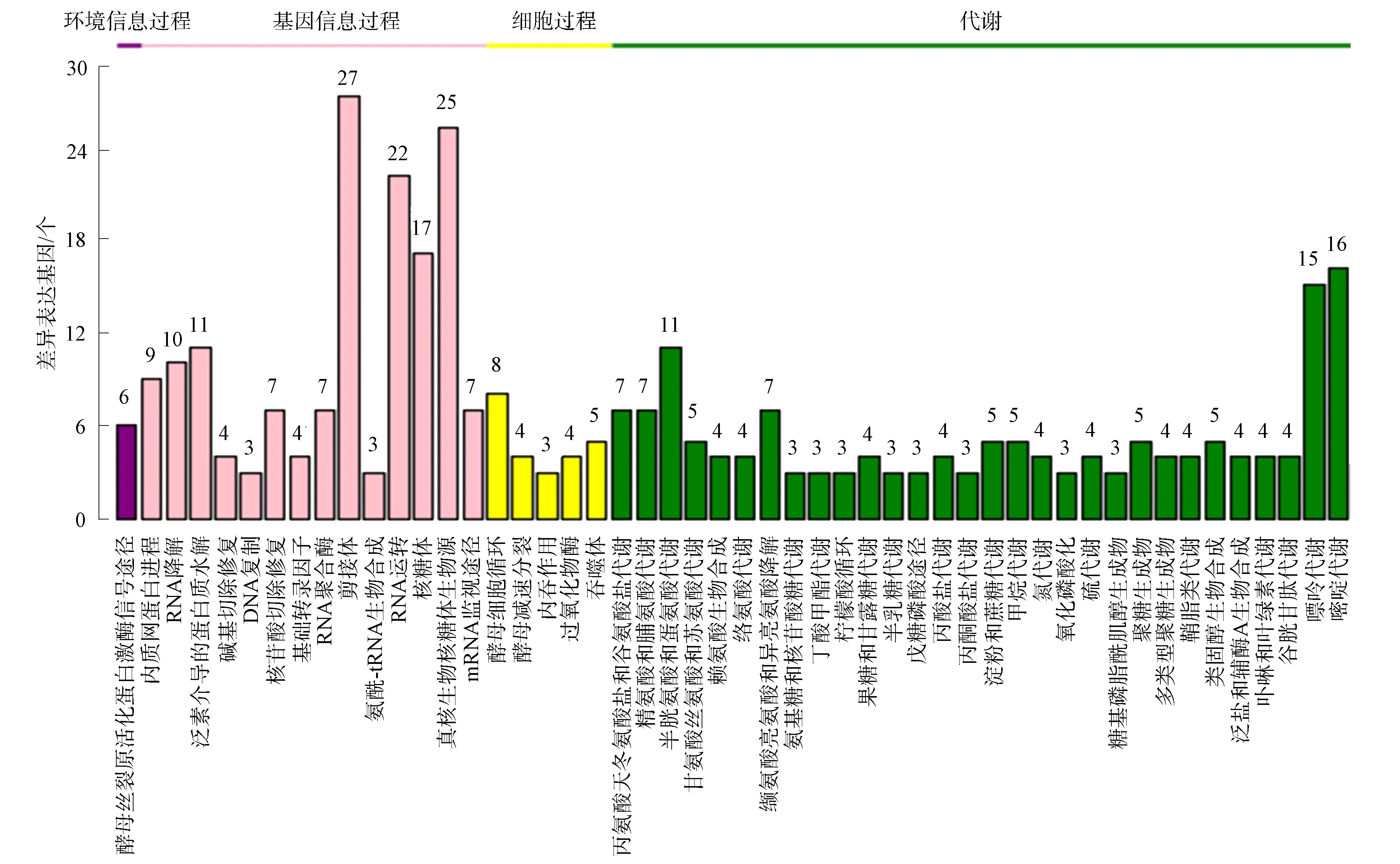
图5 差异表达基因的KEGG聚类分析结果Fig.5 Cluster analysis results of KEGG of differently expressed genes
用GO组分注释、GO功能注释和GO过程注释对差异表达基因进行划分, 结果分别列于表2~表4.
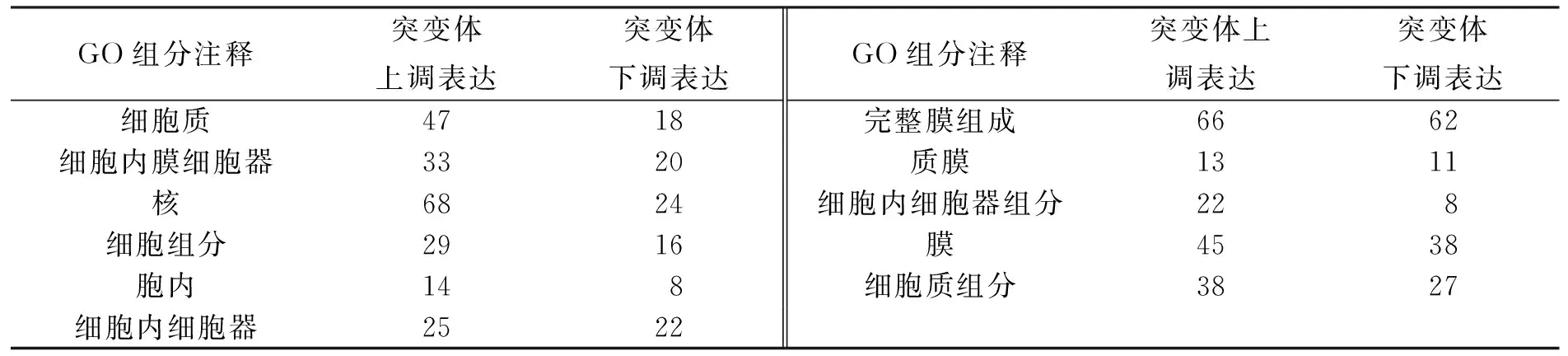
表2 差异表达基因的GO组分注释统计结果
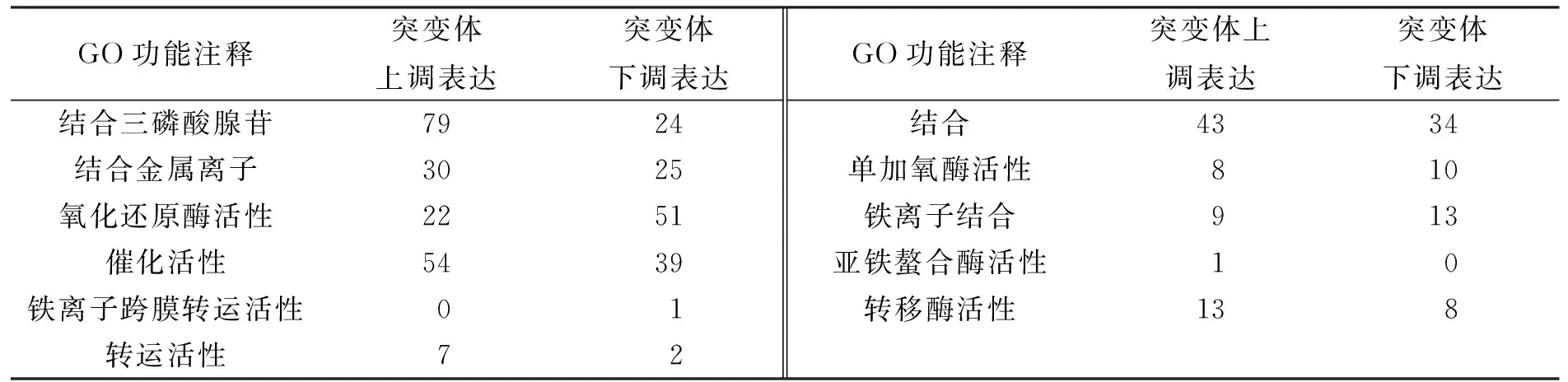
表3 差异表达基因的GO功能注释统计结果
由表2~表4可见, 某些差异表达基因在GO功能注释中明显上调表达, 如三磷酸腺苷结合、金属离子结合和运转活性等.在氧化还原酶活性和铁离子结合中的差异表达基因大多数呈下调表达.此外, 在逆境响应、氧化还原进程、致病力与寄主相互作用中的某些基因呈下调表达, 这可能与StNPS6突变体的致病力下降有关.其中, 与铁同化及铁氧化还原相关基因(基因68、基因2749、基因4360和基因10397)呈明显下调表达.这些基因与铁吸收和同化密切相关, 并间接影响致病力.在较多植物病原真菌中, 铁元素在病原菌的生长及致病力中具有重要作用[6].此外, 基因669被鉴定为三磷酸腺苷结合运转子(ABC运转子), 在突变体中下调表达11倍.研究表明, ABC 运转子在植物病原真菌中对杀菌剂的抗性及植物病原真菌的致病力均有影响.如在稻瘟菌中, 3个ABC运转子(MoABC5,MoABC6,MoABC7)对稻瘟菌致病力及非生物胁迫耐受力以及孢子产生均有显著影响[7].在灰霉病菌中, ABC运转子基因突变体的致病力明显下降[8].
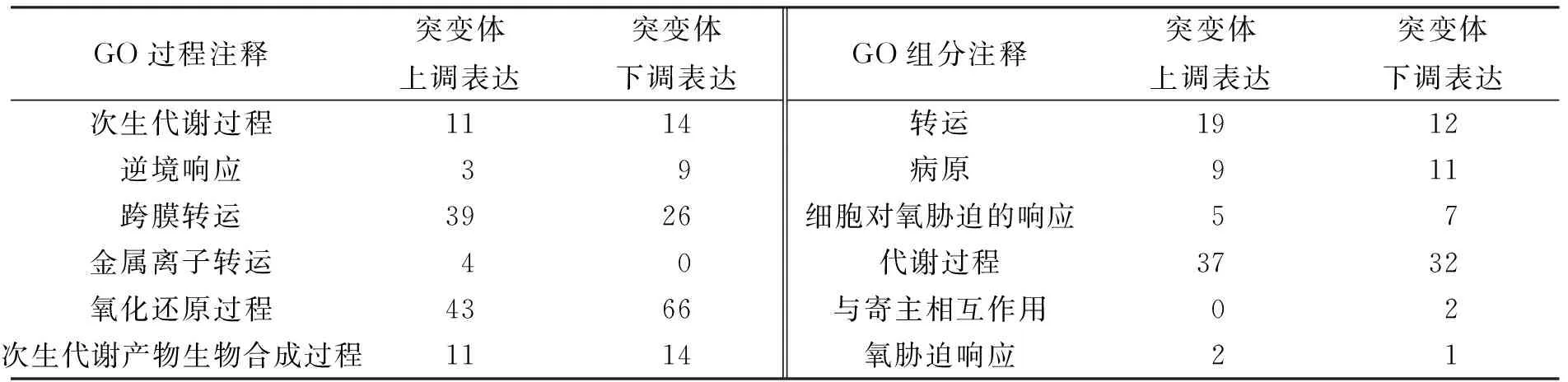
表4 差异表达基因的GO过程注释统计结果

