miR-924靶向TGM2抑制胃癌MGC803细胞增殖
周湘华,赵其辉,张志伟
(1.湖南环境生物职业技术学院,湖南 衡阳 421001;2.南华大学衡阳医学院肿瘤研究所,肿瘤细胞与分子病理学湖南省重点实验室,湖南 衡阳 421001)
胃癌(Gastric carcinoma,GC)是我国的最常见恶性肿瘤之一,其发病率与死亡率位列第二位,位列消化道恶性肿瘤第一位[1]。胃癌的有效防治取决于早期预防、早期诊断与早期治疗,而新方法与技术的发现对其防治具有十分重要的意义。课题组前期鉴定出243个胃黏膜癌变相关蛋白质[2],证实组织型转谷氨酰胺酶(Tissue Transglutaminase,TGM2)在胃癌组织中高表达,与肿瘤的发生相关[3]。许多研究发现,TGM2在多种肿瘤的发生、发展过程中发挥促进作用。本研究初步探讨miR-924对TGM2表达的调节和MGC803细胞增殖的影响,为阐明胃癌发病的分子机制提供理论依据。
1 材料与方法
1.1 材料
1.1.1 细胞系 人胃癌MGC803细胞购自中国医学科学院上海细胞生物研究所,由本实验室保存提供,用含10%小牛血清的RPMI 1640培养基常规培养。
1.1.2 引物序列 采用miRBase在线生物信息学软件(http://www.mirbase.org/)获取miR-924 mimics相应序列。通过TargetScan 6.2生物信息学软件(http://www.targetscan.org/),搜寻包含miR-924结合位点的TGM2 3′UTR序列片段,利用Premier 5分析序列,根据miR-924结合位点设计相应突变位点序列,设计qRT-PCR引物(TGM2引物:CCGAGGAGCTGGTCTTAGAG,TCTTAGTGGAAAAC GGGCCT;β-actin引物:GGGCACGAAGGCTCATCA TT,AGCGAGCATCCCCCAAAGTT)。
1.2 方法
1.2.1 预测调控TGM2基因表达的miRNAs 采用miRwalk、miRanda、microRNA.org、HOCTAR、TargetScan与EMBL-EBI等软件预测靶向调节TGM2基因表达的miRNAs,在10个软件中有5个或5个以上软件中预测到的miRNAs,以次数与不同软件网站中的得分排名为候选miRNAs。分析调控TGM2表达的micRNA及与TGM2基因3′UTR序列片段结合的位点。
1.2.2 细胞转染 将MGC803细胞接种于6孔板中,采用Lipofectamine 2000脂质体进行转染,分为MGC803细胞组、无关序列组与miR-924 mimics三组,扩大培养细胞,收集细胞进行后续实验。
1.2.3 qRT-PCR 提细胞总RNA,逆转录后进行PCR扩增,设置20 μL体系:miR-924或U6特异性引物0.4 μL、Taq DNA polymerase 0.2 μL、U6 RT product 2.0 μL、2×SYBR Mix 10 μL、无酶水7.4 μL,经real-time PCR仪PCR扩增(95.0 ℃ 3 min;95.0 ℃ 12 s;62.0 ℃ 35 s,40 cycles)。熔解曲线检测从62.0 ℃至95.0 ℃,计算不同细胞的CT值,miR-924表达用ΔCT进行评价。
1.2.4 荧光素酶报告基因活性分析 将MGC803细胞接种于6孔板中,将miR-924mimics、pMIR-report载体及0.5 μg β-半乳糖苷酶表达质粒(对照)共转染48 h后,采用单光子检测仪检测细胞的荧光素酶活性。同时检测细胞中β-半乳糖苷酶活性,计算荧光素酶报告基因活性相对值(荧光素酶报告基因活性值/β-半乳糖苷酶活性)。
1.2.5 细胞生长曲线 将细胞接种于96孔板中,采用Lipofectamine 2000脂质体进行转染,分为MGC803细胞组、无关序列组与miR-924 mimics三组,将96孔板放置培养箱中培养24 h,每孔加入5 mg/mL MTT 20 μL,继续培养4 h后,加入DMSO溶解紫蓝色结晶,置于ELX800酶标仪中检测每孔吸光值(检测波长为490 nm),重复检测96孔板中每孔细胞的吸光度值(OD值),每天检测细胞一次,将每孔检测值与第1天检测值比较值代表检测校正值,连续检测7天,将校正值连线绘制细胞生长曲线图。
1.2.6 平皿克隆形成实验 将细胞接种于6孔板中,采用Lipofectamine 2000脂质体进行转染,分为MGC803细胞组、无关序列组与miR-924 mimics三组,置于培养箱中培养1周左右,结晶紫进行细胞克隆染色,倒置显微镜或肉眼观察细胞克隆形成情况(肉眼可视或显微镜下细胞大于50个为克隆),计算克隆形成率(克隆数/接种细胞数×100%)。
1.2.7 软琼脂集落形成实验 用培养基与琼脂糖加热混合调整浓度为0.6%,加入6孔板每孔中冷却制备底层琼脂;用培养基与琼脂糖加热混合调整浓度为0.3%,分别加入细胞(5×103个/孔),加入底层琼脂上制备顶层琼脂,分为MGC803细胞组、无关序列组与miR-924 mimics三组,置于培养箱中培养2周左右,倒置显微镜下观察细胞集落形成情况(大于50细胞为集落),每孔随机选取10个低倍镜视野,计数集落形成率(集落形成数/接种细胞数)×100%。
1.2.8 Western blot检测 提取细胞总蛋白质,用10%不连续SDS聚丙烯酰胺凝胶电泳分离蛋白质,将分离蛋白质转移至PVDF膜上,5%脱脂牛奶漂洗PVDF膜,非特异性封闭2 h,用1∶1 000稀释一抗与1∶2 000稀释β-actin单抗4 ℃孵育过夜,TBST漂洗PVDF膜,用1∶1 000稀释羊抗鼠二抗,室温孵育2 h,TBST漂洗PVDF膜,经发光、压片、显影和定影后,经灰度扫描后计算蛋白质表达的灰度值,分析蛋白质表达的相对表达强度。
1.3 统计学处理
采用SPSS 20.0统计学软件进行数据分析,计量资料数据采用均数±标准差表示,两组均数间比较采用t检验,三组间比较采用单因素方差分析(One way ANOVA),不同组间比较采用q检验,多样本均数间比较采用单因素方差分析,以P<0.05表示差异具有统计学意义。
2 结 果
2.1 调控TGM2表达的miRNAs分析
采用miRwalk在线软件(http://www.umm.uni-heidelberg.de/apps/zmf/mirwalk)预测和分析调控TGM2基因表达的miRNAs(图1)。10个软件中有5个或5个以上软件出现者为候选miRNA。通过miRanda(http://www.microrna.org/microrna/)进行热力学稳定性和序列保守性评分分析。结果显示miR-924与miR-450b-3p与TGM2基因靶向结合最佳(表1)。
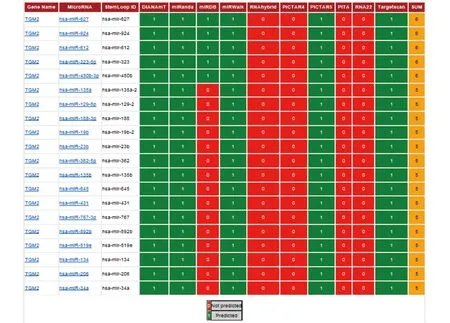
图1 生物信息学软件分析调控TGM2表达的miRNAs绿色代表该软件预测到miRNA与靶基因有结合,红色(0)代表该软件没有预测到结合,hsa是指人类的miRNA
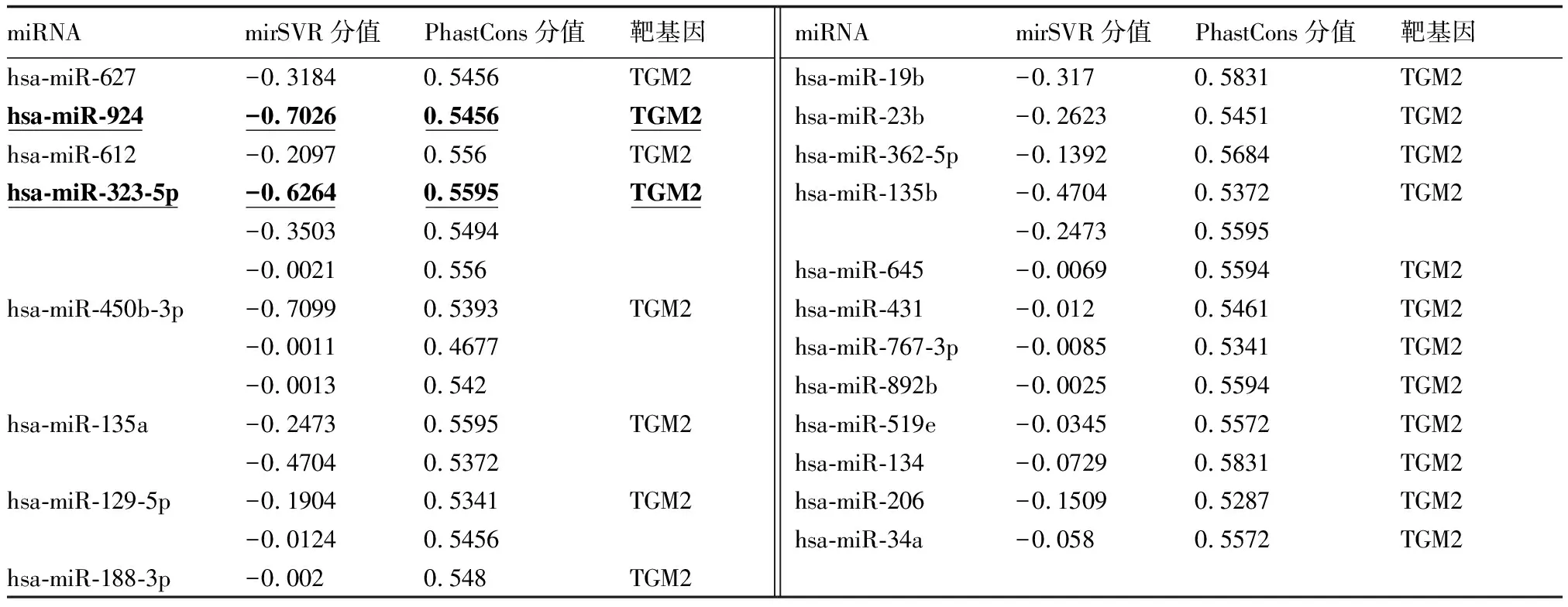
表1 调控TGM2基因表达的相关miRNAs
注:满足热力学稳定性评分(mirSVR)≤-0.1与序列保守性评分(PhastCons)在0.5~0.7之间者为有效miRNA,一个数值代表一个结合位点评分
2.2 miR-924与TGM2基因结合位点的预测
采用Targetscan在线生物信息学软件(http://www.targetscan.org/vert_72/)预测miR-924与TGM2基因3′-UTR结合位点。结果发现miR-924与TGM2基因3′UTR的864-871位核苷酸结合(图2)。因为miR-924与TGM2基因3′-UTR区域仅一个结合位点,所以选择其进行后续研究。
2.3 miR-924与TGM2基因3′UTR结合分析
通过qRT-PCR检测了胃腺癌MGC803、SGC7901、AGS细胞和永生化胃黏膜上皮细胞GES-1中miR-924的表达情况,发现miR-924在MGC803细胞中表达最低(图3,P<0.05)。然后通过荧光素酶报告基因活性检测miR-924对TGM2基因启动子活性的影响。结果表明,TGM2基因3′UTR突变序列与miR-924 mimics或miR-Control共转染MGC803细胞后荧光素酶活性强度基本一致,但TGM2基因3′UTR序列与miR-924 mimics或miR-Control共转染后,其中miR-924 mimics的荧光素酶活性明显降低(图4,P<0.05),说明miR-924可与TGM2基因3′UTR靶向结合。

图2 Targetscan预测miR-924与TGM2基因的靶向性结合
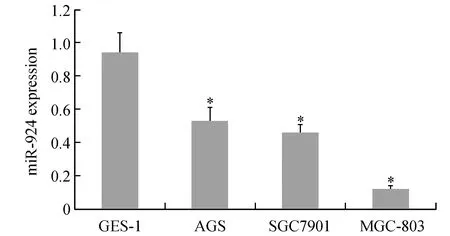
图3 qRT-PCR检测胃癌细胞中miR-924的表达与GES-1细胞比较,*P<0.05

图4 荧光素酶报告基因活性检测miR-924可与TGM2基因3′UTR的结合与miR-Control组比较,*P<0.05
2.4 miR-924对TGM2基因表达的影响
将miR-924 mimics和miR-Control分别转染MGC803细胞,通过qRT-PCR和Western blot检测TGM2的表达情况。结果显示,miR-924高表达后,MGC803细胞中TGM2基因mRNA及蛋白质均降低(图5,P<0.05),说明miR-924可抑制TGM2基因的表达。
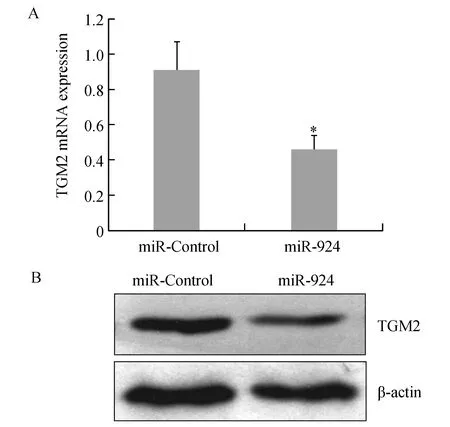
图5 miR-924对TGM2基因表达的影响A:qRT-PCR检测TGM2基因mRNA的表达,与MGC803组和miR-Control组比较,*P<0.05;B:Western blot检测TGM2蛋白质的表达
2.5 miR-924对MGC803细胞生长的影响
采用MTT法检测细胞,计算每组细胞的检测校正值,连续检测7天,绘制细胞生长曲线。结果显示,与MGC803组和miR-Control组比较,miR-924高表达后MGC803细胞的生长速度降低(图6,P<0.05)。
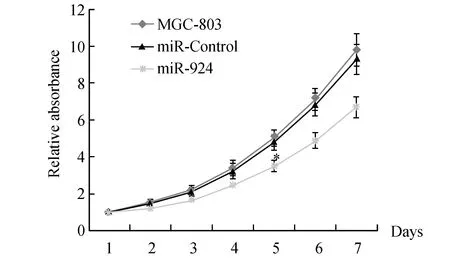
图6 生长曲线观察miR-924对MGC803细胞生长的影响与MGC803组和miR-Control组比较,*P<0.05
2.6 miR-924对MGC803细胞克隆形成的影响
采用平皿克隆观察miR-924高表达对细胞平皿克隆形成的影响。结果显示,MGC803、miR-Control和miR-924组细胞克隆数分别为(186.04±36.15)、(179.23±29.14)和(86.22±16.24),与MGC803细胞组和miR-Control组比较,miR-924高表达后细胞克隆体积变小,数目减少,细胞克隆形成率降低(表2,P<0.05)。

表2 miR-924对MGC803细胞克隆形成的统计分析
与MGC803组和miR-Control组比较,aP<0.05
2.7 miR-924对MGC803细胞集落形成的影响
采用软琼脂集落形成实验观察miR-924高表达对MGC803细胞集落形成的影响。结果显示,MGC803、miR-Control和miR-924组细胞集落数分别为(269.05±36.16)、(258.24±29.76)和(158.26±16.58),与MGC803细胞组和miR-Control组比较,miR-924高表达后细胞集落体积变小,数目减少,细胞集落形成率降低(表3,P<0.05)。
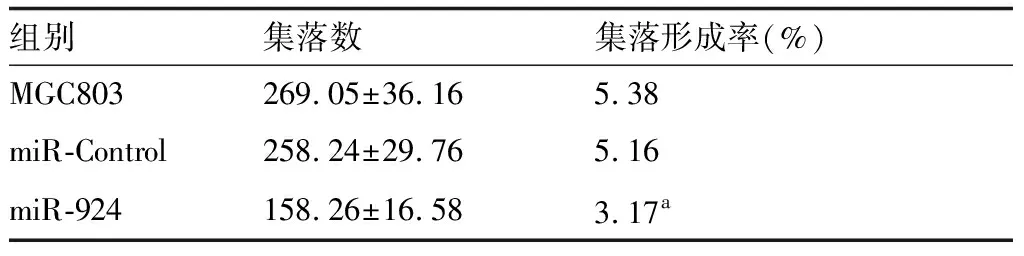
表3 miR-924对MGC803细胞集落形成的统计分析
与MGC803组和miR-Control组比较,aP<0.05
2.8 miR-924对JAK3/STAT3信号通路的影响
采用Western blot检测MGC803、miR-Control和miR-924组细胞中JAK3与STAT3蛋白表达及两者磷酸化情况,检测miR-924对JAK3/STAT3信号通路的影响。结果显示,miR-924高表达后MGC803细胞中JAK3、STAT3蛋白表达无明显差异,但JAK3、STAT3蛋白(Y705)的磷酸化水平降低(图7,P<0.05)。
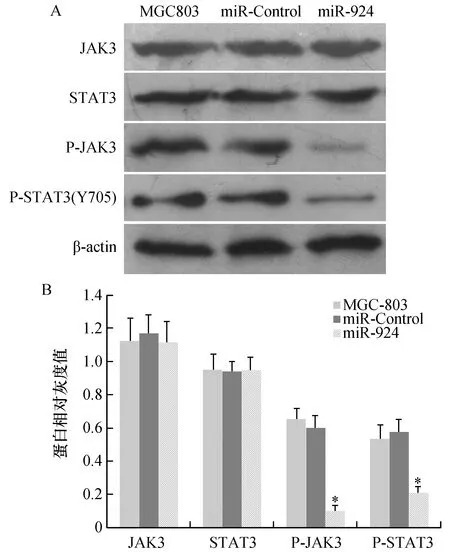
图7 miR-924对JAK3/STAT3信号通路的影响A:Western blot检测JAK3与STAT3蛋白表达及两者磷酸化情况;B:JAK3与STAT3蛋白表达及两者磷酸化的灰度统计分析图;与MGC803组和miR-Control组比较,*P<0.05
3 讨 论
TGM2属于转谷氨酰胺酶家族成员之一,普遍存在于组织中参与细胞内多种生物学过程,不同细胞中TGM2具有不同的生物学功能[4]。TGM2在肺癌、乳腺癌、卵巢癌、胰腺癌、恶性黑素瘤和神经胶质细胞瘤等肿瘤组织中均高表达[5]。TGM2表达后可激活FAK酪氨酸激酶,使PI3/AKT等抗凋亡信号通路活化,诱导细胞增殖[6]。干扰TGM2表达后,增强肿瘤细胞对化疗药物的敏感性,抑制细胞的迁移,使细胞的死亡增加[7]。有研究者报道,表皮生长因子(EGF)可诱导TGM2蛋白表达,增加肿瘤细胞产生耐药及下降细胞的凋亡[8]。本研究发现,TGM2在MGC803细胞中表达降低时,细胞增殖能力降低,说明TGM2表达可能参与了促进细胞增殖,与课题组前期及以往的研究结果基本一致。
目前发现miRNAs在胃癌细胞中参与调节细胞的增殖。miR-133a抑制胃癌细胞中TAGLN2基因表达,抑制细胞的增殖[9]。miR-374a抑制胃癌细胞中SRCIN1基因表达,促进细胞增殖、侵袭转移[10]。为了进一步了解TGM2促进MGC803细胞增殖的分子机制,本研究发现miR-924可与TGM2基因3′UTR的864-871位核苷酸结合,其高表达后TGM2表达降低,生长速度减慢,增殖能力也降低。结果说明miR-924可抑制MGC803细胞增殖,其可能通过抑制TGM2表达而抑制细胞增殖。在荧光素酶实验中,本对照组miR-924 inhibitor存在部分表达活性降低(结果未列出),可能存在实验设计问题或者影响其他miRNAs的作用,有待后续进一步研究证实其相互作用情况。miRNAs与基因表达间存在多基因调节关系,是否存在miR-924抑制TGM2表达同时,也抑制其他基因表达,有待后续的继续研究。
JAK/STAT信号通路是肿瘤细胞内非常重要的信号通过之一,其活化异常影响细胞的多种生物学行为[11]。有研究报道,STAT3与实体瘤发病密切相关,活化后参与细胞增殖、凋亡及侵袭转移等生物学行为。STAT3活化通过其蛋白磷酸化方式实现,具有Y705酪氨酸磷酸化位点与S727丝氨酸磷酸化位点[12]。JAKs对STAT3活化主要通过Y705位点酪氨酸磷酸化实现,影响细胞的生物学功能[13]。本文作者发现,miR-924高表达后抑制TGM2表达,MGC803细胞中JAK3与STAT3蛋白(Y705)的磷酸化水平降低,结果说明JAK3/STAT3通路活化受抑制,我们推测miR-924调节TGM2表达的同时,可能通过影响细胞内其他蛋白的表达,或TGM2参与下游相关信号通路的活化障碍,使JAK3/STAT3通路活化受阻,降低细的增殖能力。
总之,本研究初步证实了miR-924对TGM2基因表达的靶向抑制,参与MGC803细胞的增殖。另外,miR-924表达可下调JAK3/STAT3通路活化,抑制MGC803细胞的增殖。本文为阐明胃癌发病的分子机制提供了新的思路。

