副干酪乳杆菌SMN-LBK在乙醇 胁迫下的转录组分析
郭金凤,杨 婕,李宝坤,金 丹,黎 旭,卢士玲,王庆玲,姬 华,董 娟,李应彪,蒋彩虹
(石河子大学食品学院,新疆植物药资源利用教育部重点实验室,新疆 石河子 832003)
副干酪乳杆菌(Lactobacillus paracasei)是一种兼性厌氧、不运动、不产芽孢、发酵葡萄糖主要产生L-乳酸的杆状或长杆状革兰氏阳性菌[1]。副干酪乳杆菌能促进人体内微生物菌群的平衡以及酶的平衡[2],同时还可以刺激特异性和非特异性的免疫机制,具有预防某些疾病、促进发育、增强体质、延缓衰老和延长寿命的益生功效[3],是近年来国外研究颇多的益生乳酸细菌。
副干酪乳杆菌广泛存在于马奶酒等传统发酵乳制品及肠道中,还有少量存在于一些低度清酒[4]和白兰地中[5]。因此,副干酪乳杆菌易遭受乙醇胁迫。研究表明,在乙醇胁迫下细胞膜首先受到攻击,乳酸菌通过改变不饱和脂肪酸的比例[6],提高细胞膜的流动性[7],造成胞内物质流失,致使正常生理功能及代谢调控失常。因此,提高菌株的乙醇适应能力对其在食品工业的应用具有重要作用。
近年来,随着分子生物技术、转录组学及生物信息学的发展,在胁迫研究方面,科研人员正在通过转录组学,进行乳酸菌在不同因素胁迫后差异基因的分析,进而研究乳酸菌的胁迫机制。因此,转录组学是研究生物耐受机制的重要手段。目前虽有关于乙醇耐受性的报道,但是对乳酸菌的乙醇胁迫应答机制却缺乏了解。本研究以1 株分离于新疆塔城地区马奶酒中的耐乙醇菌株副干酪乳杆菌SMN-LBK为研究对象,分别以4%、6%、8%、10%乙醇进行胁迫处理,测定其存活率,并以10%乙醇为对其进行胁迫处理,对其基因表达情况进行研究,旨在从转录水平上揭示副干酪乳杆菌SMN-LBK的乙醇胁迫应答机制,并为进一步阐明乳酸杆菌的耐受机制奠定基础。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与培养基
副干酪乳杆菌SMN-LBK是从新疆塔城地区马奶酒样品中分离筛选出的1 株耐乙醇乳酸菌。
液体MRS培养基:蛋白胨10 g、牛肉膏10 g、酵母浸粉5 g、无水乙酸钠5 g、葡萄糖20 g、柠檬酸三铵2 g、磷酸氢二钾2 g、硫酸镁0.58 g、硫酸锰0.25 g、 吐温80 1 mL,蒸馏水1 000 mL,调节pH 6.2~6.4,121 ℃灭菌20 min。
固体MRS培养基:在液体MRS培养基内添加18 g/L琼脂粉,于121 ℃灭菌20 min。
含乙醇MRS培养基:已灭菌液体MRS培养基按不同体积比添加无水乙醇,配制成4%、6%、8%、10%(体积分数)乙醇培养基,以不添加乙醇的培养基作为对照。
1.1.2 试剂
焦炭酸二乙酯 西格玛奥德里奇(上海)贸易有限公司;TaKaRa逆转录试剂盒 宝生物工程(大连)有限公司;UltraSYBR Mixture 康为世纪生物科技有限公司;Trizol试剂、Qubit®RNA检测试剂盒 美国Life公司;RNA Nano 6000检测试剂盒 美国安捷伦公司;NEBNext®Ultra™ Directional RNA Library Prep Kit for Illumina®美国NEB公司;琼脂糖 北京天根生物科技有限公司;异丙醇、三氯甲烷、无水乙醇 天津市福晨化学试剂有限公司;其余均为分析纯。
1.2 仪器与设备
SW-CJ-1CU双人单面超净工作台 苏州苏洁净化设备有限公司;SPX-150B-Z型生化培养箱 上海博迅实业有限公司医疗设备厂;Centrifuge 5417R高速冷冻 离心机 德国Eppendorf公司;LDZX-30KBS立式压力蒸汽灭菌器 上海申安医疗器械厂;ND2000C微量核酸蛋白分析仪 美国赛默飞世尔科技有限公司;GeI Doc XR+ 凝胶成像系统 美国Bio-Rad公司;MX3000P实时荧光定量聚合酶链式反应(real-time quantitative polymerase chain reaction,real-time PCR)分析仪、2100生物分析仪 美国Agilent公司;HiSeq2500测序仪 美国Illumina公司。
1.3 方法
1.3.1 菌株培养
取-80 ℃甘油贮存液以2%接种量接种于5 mL已灭菌MRS液体培养基中,于37 ℃生化培养箱中培养24 h,获得活化种子液。以2%接种量接种种子液于5 mL已灭菌的液体MRS培养基中,于37 ℃生化培养箱中培养48 h,获得活化菌株。
1.3.2 乙醇胁迫
如图1所示,S10为空白对照,SP10为10%乙醇胁迫处理。将活化副干酪乳杆菌SMN-LBK以2%接种于已灭菌的液体MRS培养基,于37 ℃培养至OD600nm为0.8,吸取2%上述菌液分别接种至不含乙醇MRS培养基及含4%、6%、8%、10%乙醇MRS培养基内,37 ℃培养3 h。

图 1 乙醇胁迫处理示意图Fig. 1 Schematic illustration of ethanol stress treatment
1.3.3 存活率测定
将经过乙醇胁迫处理的菌液以无菌0.85%生理盐水清洗2 次,并同体积进行重悬,吸取1 mL重悬菌液于无菌0.85%生理盐水中梯度稀释,于固体MRS培养基上涂布,37 ℃条件下培养48 h,测定存活率,实验重复3 次,每次3 个平行样品。存活率计算公式如下:
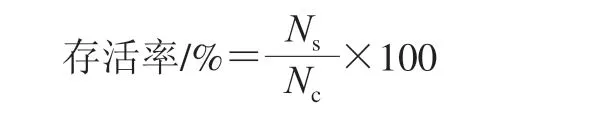
式中:Nc为未经乙醇胁迫活菌数;Ns为乙醇胁迫后活菌数。
1.3.4 转录组测序
采用焦炭酸二乙酯处理所有实验相关耗材,按照Trizol试剂说明书提取菌株总RNA,采用琼脂糖凝胶电泳分析RNA降解程度以及是否有污染;采用Nanodrop检测RNA的纯度(OD260nm/OD280nm);按照Qubit®RNA检测试剂盒说明书对RNA浓度进行精确定量;通过Agilent 2100生物分析仪精确检测RNA的完整性。运用NEBNext®Ultra™ Directional RNA Library Prep Kit for Illumina®进行文库构建,并用AMPure XP beads纯化PCR产物。采用Qubit 2.0进行初步定量,稀释文库至1 ng/μL后,采用2100生物分析仪对文库的插入片段长度进行检测。将合格的不同文库按照有效浓度及目标下机数据量的需求pooling后进行HiSeq 2500测序。
1.3.5 real-time PCR对转录组结果验证
按照TaKaRa逆转录试剂盒说明书合成第1链cDNA,real-time PCR所用的引物见表1,其中16S rRNA作为内参基因,采用ddH2O作为阴性对照。按照UltraSYBR Mixture说明书进行real-time PCR。real-time PCR体系(20 μL):荧光染料混合液10 μL、正向引物0.4 μL、反向引物0.4 μL、cDNA模板0.8 μL、用ddH2O补足至20 μL。real-time PCR程序:95 ℃预变性10 min,95 ℃变性15 s,60 ℃退火30 s,72 ℃延伸32 s,循环40 次;溶解曲线分析:95 ℃变性15 s,60 ℃退火1 min,95 ℃荧光信号采集15 s,60 ℃冷却15 s。每个样品设置3 次平行。利用2-ΔΔCt法分析基因的相对表达量,使用SPSS 19.0进行统计学差异的显著性分析。
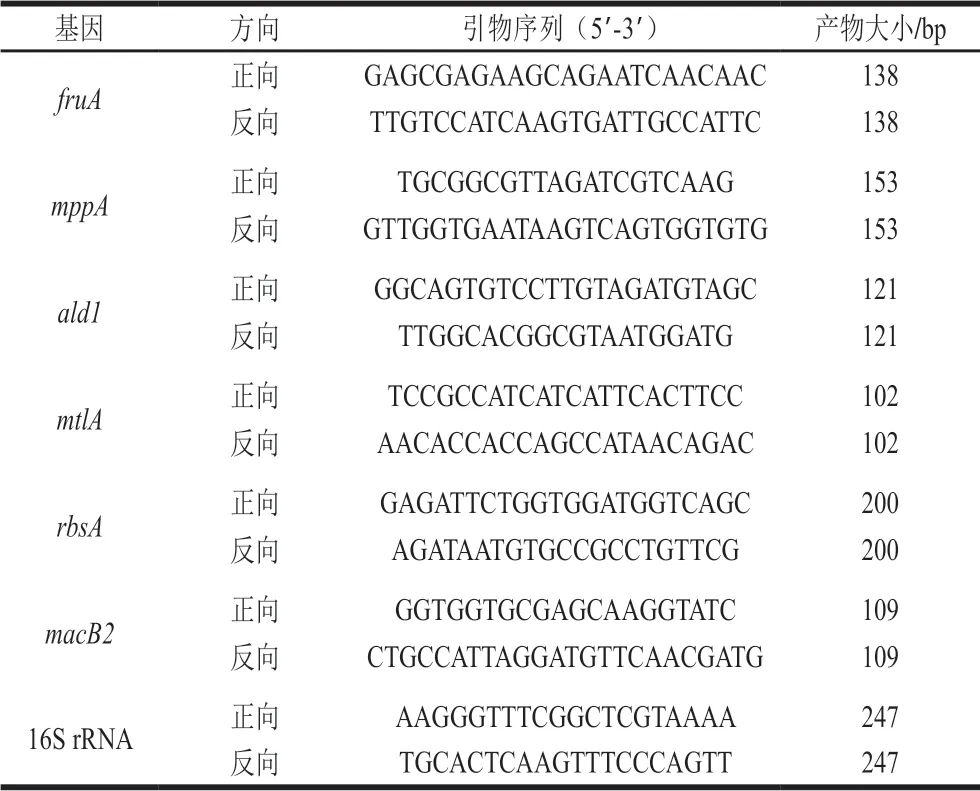
表 1 real-time PCR引物Table 1 Sequences of primers used for real-time PCR
1.4 数据分析
1.4.1 质量控制
测序得到的原始测序序列,对raw reads进行过滤,去除带接头的、低质量的reads,得到clean reads,后续分析都基于clean reads,同时计算clean reads的GC含量(碱基G和C数量总和占总碱基数量的百分比)、Q20、Q30含量(Phred数值大于20、30的碱基占总体碱基百分比)。
1.4.2 基因表达水平分析
采用HTSeq软件对各样品进行基因表达水平分析[8], 根据基因的长度计算每百万片段中来自某一基因每千碱基长度的片段(fragments per kilobase per million,FPKM)数目[9]。本研究以FPKM=1作为判断基因是否表达的阈值标准[10]。
1.4.3 差异基因表达分析
使用DESeq R软件包(1.18.0)进行两组的差异表达分析,基于负二项式分布的模型确定数字基因表达数据中的差异表达。使用Chen Rongjun等[11]方法调整所 得P 值以控制错误发现率。通过D E S e q 发现调整的P <0.0 5 的基因为差异基因。对检测结果以 log2|Fold Change|>1、P<0.05作为标准进行筛选,对差异基因表达情况进行具体分析。
1.4.4 差异基因功能富集分析
采用GOSeq R软件包对差异基因进行基因本体(Gene Ontology,GO)富集分析,其中对基因长度偏差进行校正,具有校正的P<0.05的GO term是显著富集的差异基因。京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)是一个数据库资源,用于从基因组测序产生的大规模分子数据集和其他高通量数据库中了解生物系统的高级功能和效用(http://www.genome.jp/kegg/)。采用KOBAS软件测试KEGG途径中差异基因的富集[12]。
2 结果与分析
2.1 乙醇胁迫对副干酪乳杆菌SMN-LBK存活率的影响
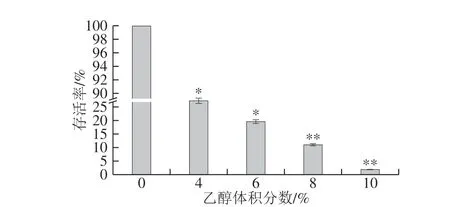
图 2 不同体积分数乙醇胁迫下副干酪乳杆菌SMN-LBK的存活率Fig. 2 Survival rates of L. paracasei SMN-LBK under ethanol stress at different concentrations
由图2可知,以乙醇体积分数0%作为对照组,以4%、6%、8%、10%乙醇对副干酪乳杆菌的SMN-LBK进行胁迫处理,其存活率依次为25.84%、18.39%、10.86%、1.67%,副干酪乳杆菌SMN-LBK的最高耐乙醇胁迫能力为10%。
2.2 测序数据质量分析
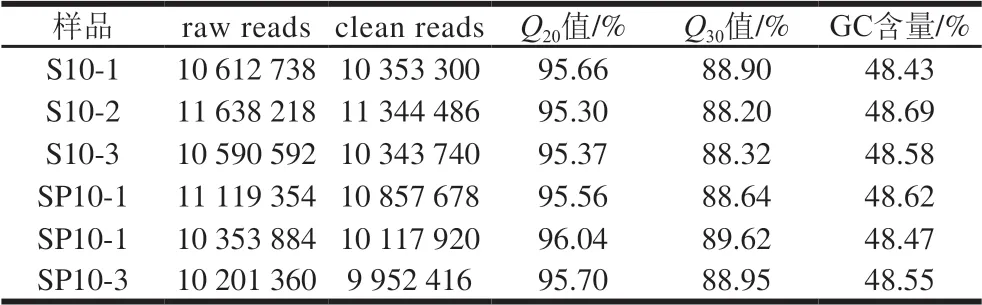
表 2 数据质量分析 Table 2 Quality analysis of transcriptomic sequencing data
由表2可知,两组处理的Q20值均大于95%,Q30值均大于88%,GC含量均在50%左右。结果表明转录组测序数据质量好,准确性高。
2.3 基因表达水平分析
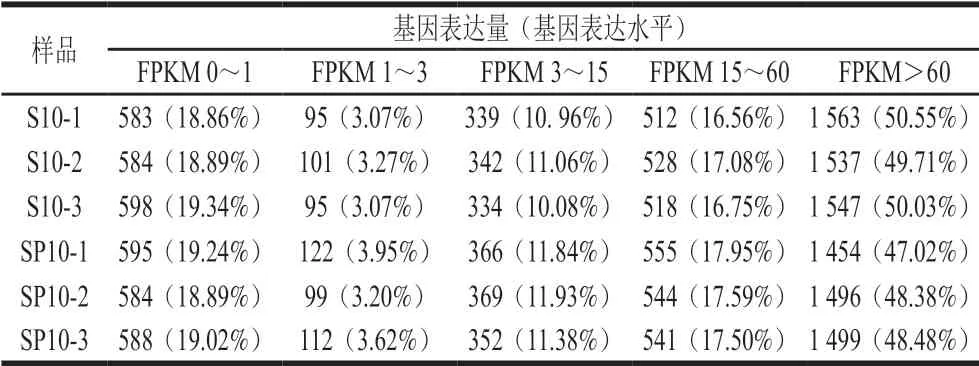
表 3 不同表达水平区间的基因数量Table 3 Statistics of expression levels of differentially expressed genes
由表3可知,两组处理的基因表达量均在PFKM 1~3区间最低,每个基因的表达水平在3.07%~3.95%范围内;基因表达量均在FPKM>60区间最高,每个基因的表达水平在47.02%~50.55%范围内,说明多数基因以高水平表达。
2.4 差异基因表达分析
2.4.1 差异基因筛选
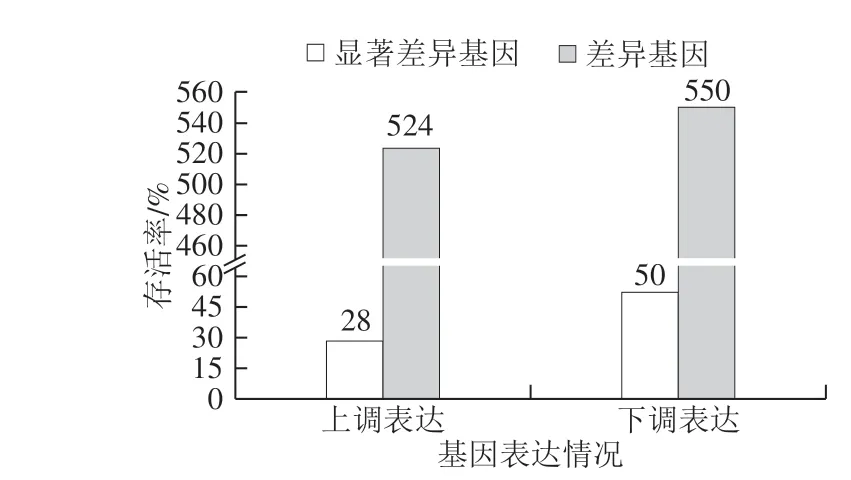
图 3 差异基因上下调情况Fig. 3 Statistics of up-regulated and down-regulated genes
由图3可知,在SP10与S10组内上调表达差异基因524 个,下调表达差异基因550 个;上调表达显著差异基因28 个,下调表达显著差异基因50 个(log2|Fold Change|>1,P<0.05)。
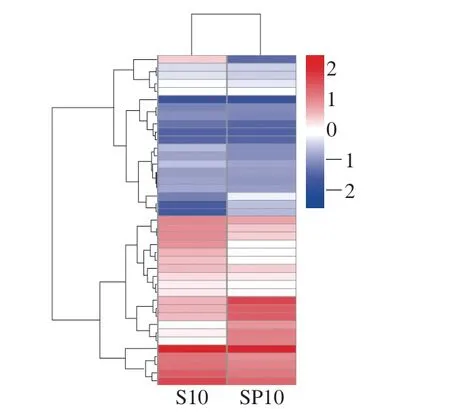
图 4 乙醇不同处理下差异基因聚类分析Fig. 4 Clustering analysis of differentially expressed genes under ethanol stress at different concentrations
2.4.2 差异基因表达聚类分析
采用差异基因火山图推断其整体分布情况,聚类分析判断差异基因在乙醇胁迫的表达模式。聚类分析以差异基因表达变化1 倍以上且错误发现率小于0.05进行绘制。由图4可知,有4 类表达模式相同或相近的基因聚集成类,它们可能具有相似的功能或参与相同的生物学过程。
2.4.3 real-time PCR验证分析
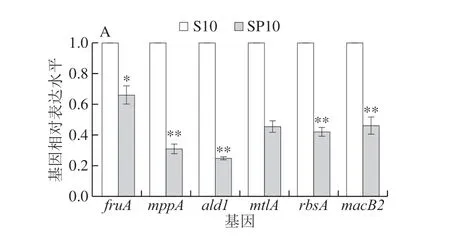
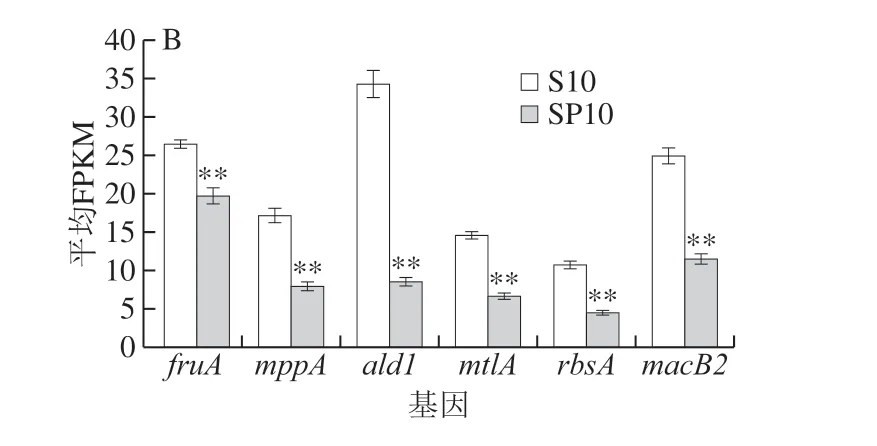
图 5 real-time PCR验证转录组测序Fig. 5 Real-time PCR validation of transcriptomic sequencing data
对差异显著的基因进行real-time PCR验证,如图5所示,real-time PCR结果与转录组结果具有一致性。因此,充分验证了转录组数据的正确性与可靠性。
2.5 差异基因功能富集分析
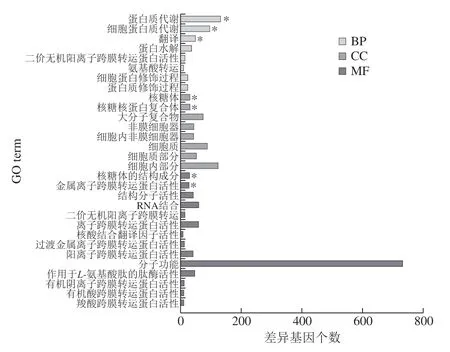
图 6 乙醇不同处理下差异基因GO富集分析Fig. 6 GO enrichment analysis of differentially expressed genes under ethanol stress at different concentrations
GO富集分析以阈值(P<0.05)为依据评价差异基因的主要生物学功能,其主要包括细胞组分(cellcular component,CC)、分子功能(molecular function,MF)和生物过程(biological process,BP)三类。由图6 可知,SP10与S10相比,BP组蛋白代谢过程、细胞内蛋白代谢过程、翻译过程为显著富集GO term;CC组核糖体、核糖核蛋白复合物为显著富集GO term;MF组核糖体的结构成分、金属离子跨膜转运蛋白活性为显著富集GO term。KEGG是一个综合数据库,整合了基因组信息、化学信息和生化系统功能信息。KEGG Orthology(KO)是KEGG直系同源数据库,将各个KEGG注释系统联系在一起,将分子网络和基因组信息联系起来,根据直系同源关系,实现跨物种的基因组或转录组的功能注释。结合KEGG数据库对差异显著基因参与的相关代谢通路进行了分析,由表4可知,差异显著基因主要参与了丙氨酸、天冬氨酸和谷氨酸代谢、脂肪酸生物合成、ATP结合盒式蛋白(ATP-bindingcassette,ABC)转运体、磷酸转移酶系统(phosphotransferase system,PTS)。
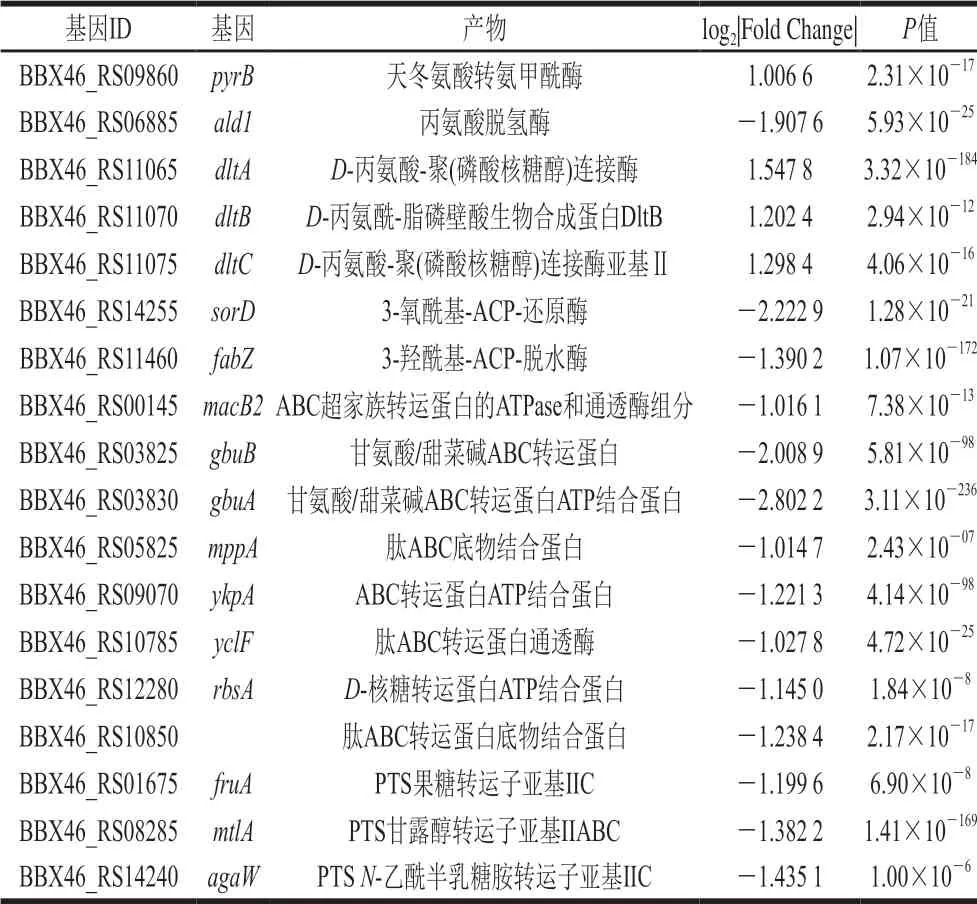
表 4 乙醇胁迫下差异显著基因Table 4 Genes that showed significantly different expression levels under ethanol stress
2.5.1 乙醇对参与丙氨酸、天冬氨酸和谷氨酸代谢的基因表达情况的影响
由表4可知,pyrB SP10相对S10显著上调。ald1显著下调表达,dltA、dltB、dltC SP10相对S10均显著上调。PyrB是包括大肠杆菌在内的所有原核生物嘧啶核苷酸从头合成中很重要的一个限速酶,它催化的是天冬氨酸与氨甲酰磷酸生成氨甲酰天冬氨酸和无机磷的反应[13]。Gerhart等[14]发现PyrB的一个最有效抑制剂是代谢途径的终产物胞嘧啶三磷酸(cytosine triphosphate,CTP),当CTP水平高时,CTP与PyrB结合,降低CTP合成的速度,反之当细胞内CTP水平低时,CTP从PyrB上解离,加快CTP合成速度,研究还发现ATP是酶的别构激活剂。本研究发现pyrB显著上调,这说明副干酪乳杆菌SMN-LBK在乙醇胁迫下,pyrB被激活,天冬氨酸转氨甲酰酶催化天冬氨酸与氨甲酰磷酸生成氨甲酰天冬氨酸和无机磷,此时细胞内CTP水平低,加速了pyrB作用,这也是副干酪乳杆菌自我保护的一种方式。
赵鹏[15]研究表明ald基因在氧化胁迫情况下显著下调表达,胞内丙氨酸的代谢受到影响。Ald是一种双向反应酶,正向以丙氨酸为底物氧化脱氨生成丙酮酸、NADH,并释放NH3[16]。逆向由丙酮酸氨化生成丙氨酸,消耗一分子铵和NADH[17]。本研究ald1 SP10相对S10显著下调表达,说明副干酪乳杆菌SMN-LBK在乙醇胁迫下,Ald催化丙酮酸生成丙氨酸的活性降低,而丙氨酸广泛存在于已知蛋白中,此外,它还在组成细胞壁肽聚糖骨架具有重要作用,所以ald可能参与并调控了副干酪乳杆菌S10的乙醇胁迫调控,并且在其细胞质的蛋白质以及细胞壁生物合成中受到了抑制。DltB是革兰氏阳性细菌的一种dlt操纵子,其催化D-丙氨酸残基并入脂磷壁酸中[18]。 脂磷壁酸是一种革兰氏阳性(G+)菌细胞壁特殊组分,由核糖醇或甘油残基经由磷酸二键互相连接而成的多聚物,其可以跨过肽聚糖层,以其末端磷酸共价连接于质膜中糖脂(例如二葡糖基二酰基甘油)的寡糖基部分。本研究发现dltA、dltB和dltC SP10相对S10均显著上调,说明副干酪乳杆菌SMN-LBK通过增强与细胞壁合成相关基因的表达抵抗乙醇的侵害。
2.5.2 乙醇对参与脂肪酸生物合成基因表达情况的影响
由表4可知,fabG和fabZ基因SP10相对S10显著下调表达。fabG催化细菌脂肪酸合成途径中催化3-氧酰基-ACP还原为3-羟酰基-ACP[19],消耗NADPH或NADH,是必需的还原步骤[20]。Filip’echev等[21]研究表明,用于假定fabG质粒AZOBR_p1-携带的fabG基因,对于在Azospirillum brasilenseSp245中双鞭毛系统的正确装配和工作是必需的。Putim等[22]基于STITCH数据库的分析预测fabG与结核分枝杆菌抗药性有关。Tan等[23]研究发现增加fabZ的用量可使大肠杆菌辛酸效价的最高增加45%。Kastaniotis等[24]发现YHR067w编码一种新的线粒体fabZ,线粒体FAS途径的功能与硫辛酸或磷脂生物合成、蛋白质膜插入或内膜氧化损伤修复相关。本研究中fabG和fabZ SP10相对S10显著下调表达,说明在乙醇胁迫下,副干酪乳杆菌SMN-LBK的脂肪酸合成受阻,细胞膜内膜受损,从而对乙醇的抵抗能力下降。
2.5.3 乙醇对ABC转运体基因表达情况的影响
由表4可知,gbuB、gbuA、macB2、肽ABC转运蛋白底物结合蛋白基因、mppA、ykpA、yclF和rbsA SP10相对S10均显著下调表达。ABC转运系统是一个与结构相关的吸收和外运系统的超家族[25]。ABC转运蛋白通常由多个亚基组成,其中1 个或2 个是跨膜蛋白,还有膜相关ATPase。ATPase亚基利用ATP结合和水解的能量促进各种底物跨膜转运,以吸收或输出底物。肽ABC转运蛋白底物结合蛋白基因、mppA、yclF都是与肽转运蛋白相关的基因,细胞膜上的肽转运蛋白不仅能够转运小肽进入细胞提供营养,并且还参与了信号转导、趋化性等重要生理活动[26]。王艳红等[27]从中度嗜盐菌喜盐芽孢杆菌Halobacillus Y5中成功克隆了1 个新的耐盐碱基因(HY5_opu D),该基因是1 个新的甘氨酸甜菜碱转运蛋白基因,能够使大肠杆菌KNabc在0.2 mol/L NaCl、5 mmol/L LiCl及碱性(pH 8.0)环境下生长。Park等[28]发现大肠杆菌高亲和力核糖转运蛋白由周质核糖结合蛋白(RBP或RbsB)、膜组分(RbsC)和ATP结合蛋白(RbsA)组成。本研究发现SP10相对S10下副干酪乳杆菌SMNLBK 8 个基因均显著下调表达。在乙醇作用下,细菌的细胞膜通透性增大,使跨膜蛋白肽健断裂,与膜相关ATPase活性受到抑制,使其正常的跨膜运动与物质运输遭受破坏。
2.5.4 乙醇对参与PTS的基因表达情况的影响
由表4可知,fruA、MtlA、agaW SP10相对S10均显著下调。糖PTS是许多乳酸杆菌中将外界碳源转运进细胞并将其进行磷酸化的一种重要工具[29]。PTS通过涉及PTS的不同组分的一系列步骤将磷酸烯醇式丙酮酸酯的磷酸盐基团转移到进入的糖中发挥作用。PTS由缺乏糖特异性的细胞质组分和膜相关酶构成,其对少数糖具有特异性。细胞质组分是酶I(EI)和组氨酸可磷酸化蛋白(HPr)。PTS系统的膜成分酶II(EII)由3~4 个亚基组成:IIA、IIB、IIC和有时IID[30]。本研究表明:副干酪乳杆菌SMN-LBK在乙醇胁迫下,其对果糖、甘露糖、乙酰半乳糖胺的转运能力下降,从而可能使其正常的碳代谢受到抑制而影响其代谢调控。
3 结 论
丙氨酸、天冬氨酸和谷氨酸代谢基因(ald1)、脂肪酸生物合成基因(fabG、fabZ)、ABC转运体(macB2、gbuB、gbuA、mppA、ykpA、yclF、rbsA、肽ABC转运蛋白底物结合蛋白基因)、PTS(fruA、mtlA、agaW)在乙醇胁迫下显著下调表达,丙氨酸、天冬氨酸和谷氨酸代谢基因(pyrB、dltA、dltB、dltC)在乙醇胁迫下显著上调表达,这说明副干酪乳杆菌SMN-LBK脂肪酸合成代谢受到抑制,物质跨膜运输受到抑制,而天冬氨酸和丙氨酸代谢相关基因与细胞壁主要成分肽聚糖、磷壁酸的生物合成相关,其可能通过提高这些基因表达抵御乙醇对自身的损害。副干酪乳杆菌面对高用量乙醇胁迫,其基因或者其编码的蛋白正常的结构受到破坏,从而使其正常功能不能够得以全部发挥,这些基因可能与副干酪乳杆菌SMN-LBK的乙醇胁迫机制密切相关,但其具体乙醇耐受机制还有待进一步研究。

