基于SRAP分子标记的贵州省棕榈种质资源遗传多样性分析
毛跃雄 陆跃堂 胡志姣 赵杨


摘要:【目的】研究貴州省棕榈资源的遗传多样性及遗传结构,揭示其分布格局与变异趋势,为贵州省棕榈种质资源保护及开发利用提供科学依据。【方法】基于SRAP分子标记对贵州省7个农业气候区的26个棕榈种源328份材料进行扩增,经Qsep 100全自动核酸蛋白分析仪电泳分析后,利用Popgene 1.32和NTSYS-pc 2.1计算主要遗传多样性参数及进行种源聚类分析。【结果】贵州省棕榈遗传多样性丰富,其种级水平上的多态性条带百分率(PPB)为97.31%,观测等位基因数(Na)为1.9731、有效等位基因数(Ne)为1.4717、Nei?s基因多样度(H)为0.2689、Shannon?s信息指数(I)为0.4212,但在种源水平上遗传多样性差异明显。贵州省26个棕榈种源间的遗传分化系数(Gst)为0.3305,即种源内变异是贵州省棕榈资源遗传变异的主要来源。UPGMA聚类分析结果显示,在阈值为0.91时,26个棕榈种源可分成四大类,其遗传分化与地理环境有一定相关性。Mantel检验结果显示,贵州省棕榈资源遗传分化与其地理距离呈正相关(r=0.2651),但相关性不显著(P>0.05)。贵州省7个农业气候区内的棕榈资源基因流(Nm)均大于贵州省棕榈种源Nm(1.0129),说明小区域内的棕榈资源基因交流频繁。【结论】贵州省棕榈种质资源总体遗传多样性高,种源间分化较明显,选择育种潜力巨大。为更好地开发利用棕榈资源,应结合其遗传结构特点,实行就地保护为主及建设种质资源库为补充的保护策略。
关键词: 棕榈;SRAP分子标志;遗传多样性;遗传分化;空间分布格局;贵州省
中图分类号: S792.91 文献标志码: A 文章编号:2095-1191(2020)01-0027-09
Abstract:【Objective】The study on genetic diversity and genetic structure of Trachycarpus fortunei(Hook.) H. Wendl resources in Guizhou, revealed its distribution pattern and variation trend,and provided scientific basis for the protection, development and utilization of T. fortunei germplasm resources in Guizhou. 【Method】Based on SRAP molecular markers, 328 materials from 26 T. fortunei provenances in 7 agroclimatic regions of Guizhou were amplified. After electrophoresis analysis by Qsep 100 automatic nucleic acid protein analyzer, Popgene 1.32 and NTSYS-pc 2.1 were used to calculate the main genetic diversity parameters and perform the provenances clustering analysis. 【Result】The genetic diversity of T. fortunei in Guizhou was rich, the percentage of polymorphic bands(PPB) was 97.31% at species level, the observed alleles(Na) was 1.9731, effective alleles(Ne) was 1.4717, Neis gene diversity index(H) was 0.2689, and Shannons information index(I) was 0.4212, but there were significant differences in genetic diversity at the provenance level. The genetic diversity(Dst) among the 26 T. fortunei provenances in Guizhou was 0.3305, which revealed that intra-provenance variation was the main source of genetic variation of T. fortunei resources in Guizhou. The UPGMA cluster analysis showed that the 26 T. fortunei provenances could be divided into four categories at the threshold of 0.91, and their genetic differentiation was related to the geographical environment. The Mantel test showed that there was a positive correlation between genetic differentiation and geographical distance(r=0.2651) in Guizhou,but the correlation was not significant(P>0.05). The gene flow(Nm) of T. fortunei resources in seven agroclimatic regions of Guizhou was larger than that of T. fortunei species in Guizhou(1.0129), indicated that gene exchange of T. fortunei resources was frequent in small areas. 【Conclusion】 T. fortunei germplasm resources of Guizhou have high genetic diversity, large differentiation between provenances, and huge potential of selection and breeding. In order to exploit and utilize T. fortunei resources better, combi-ning with its genetic structure characteristics, the conservation strategy is put forward, which is mainly in situ conservation and supplemented by the construction of germplasm resource bank.
Key words: Trachycarpus fortunei(Hook.) H. Wendl;SRAP molecular marker; genetic diversity; genetic differentia-tion; spatial distribution pattern; Guizhou
Foundation item: Guizhou Major Scientific and Technological Project(Qiankehezhongdazhuanxiang〔2014〕6024-1)
0 引言
【研究意义】棕榈[Trachycarpus fortunei(Hook.) H. Wendl]是贵州省的常见乡土树种,隶属于棕榈科(Palmae)贝叶棕亚科(Coryphoideae)棕榈属(Trachycarpus),全树均可开发利用,其产品多元、用途广泛(舒迎澜,2004;董云发,2005;卫强和王燕红,2016),且具有浅根性及宜石灰土等特性,是改善石漠化生态环境的优良经济树种(但新球等,2003)。棕榈在贵州省分布广泛,但天然林破坏严重,居群呈零星分布,多为野生或半野生状态,利用效率不高。采用分子标记技术研究物种遗传多样性和遗传结构特征,可为其种质资源保护、遗传改良及开发利用提供科学依据(凌士鹏等,2018;冯建灿等,2019),因此,加强贵州省棕榈种质资源遗传多样性研究,对促进贵州省棕榈产业发展及石漠化生态修复树种选择均具有重要意义。【前人研究进展】至今,已有专家学者分别使用RAPD、AFLP、SRAP、ISSR和SSR等分子标记在多种棕榈科植物上开展了种质资源遗传多样性分析、遗传图谱构建及系统进化关系等相关研究工作(Ohtani et al.,2009;周丽霞和曹红星,2018),包括油棕(Hayati et al.,2004;丁灿等,2011;肖勇等,2013)、山棕(贾春媛,2009)、董棕(赵春磊,2009)、槟榔(任军方,2010)、椰子(Rajesh et al.,2015;Geethanjali et al.,2018)、蒲葵(Kondo et al.,2017)等,但针对棕榈遗传多样性的研究较少。杨华(2005)利用RAPD分子标记与表型测定对黄藤和单叶省藤天然种群进行遗传多样性分析,结果证实种群内变异是其遗传变异的主要原因。安琪等(2010)采用AFLP分子标记对云南省境内的11种棕榈藤亲缘关系进行分析,结果显示云南省棕榈藤遗传多样性丰富,各品种间的遗传分化程度较高,且发现原始省藤亚属和省藤亚属两个亚属物种互相交叉渗透。Rajesh等(2015)采用SCoT分子标记评估全球23个椰子种质遗传多样性,进一步证实了SCoT分子标记用于物种遗传多样性分析的可行性。Kondo等(2017)利用20对SSR分子标记对日本的6个蒲葵群体进行遗传多样性分析,其结果为揭示蒲葵偏远种群的起源提供了依据。Geethanjali等(2018)以48对SSR分子标记分析世界范围内79种椰子种质的遗传多样性和种群结构,结果发现其遗传多样性较高,分层聚类为两大聚类,与其地理起源相对应。在棕榈种质资源遗传多样性研究方面,李文表(2005)利用ISSR分子标记分析了云南、广西和湖南3个分布区的棕榈遗传多样性,胡志姣和赵杨(2018)從形态学角度对贵州省棕榈种源遗传多样性进行了研究。【本研究切入点】SRAP分子标记具有多态性高、重复性好、操作简便及成本较低等优点,已广泛应用于观赏植物(卢超等,2014;邱帅等,2018;马秀花等,2019)、药用植物(陈大霞等,2018;潘绿昌等,2018)、粮食作物(庞新华等,2019;张明飞等,2019;赵小庆等,2019)等植物种质资源鉴定评价,但至今未见应用于棕榈种质资源研究领域。【拟解决的关键问题】从已发表的不同植物SRAP分子标记引物中筛选出多态性引物,对贵州省26个棕榈种源328份材料进行扩增,经Qsep 100全自动核酸蛋白分析仪电泳分析后,利用Popgene 1.32和NTSYS-pc 2.1计算主要遗传多样性参数及进行种源聚类分析,旨在揭示贵州省棕榈的分布格局与变异趋势,为其种质资源保护及开发利用提供科学依据。
1 材料与方法
1. 1 试验材料
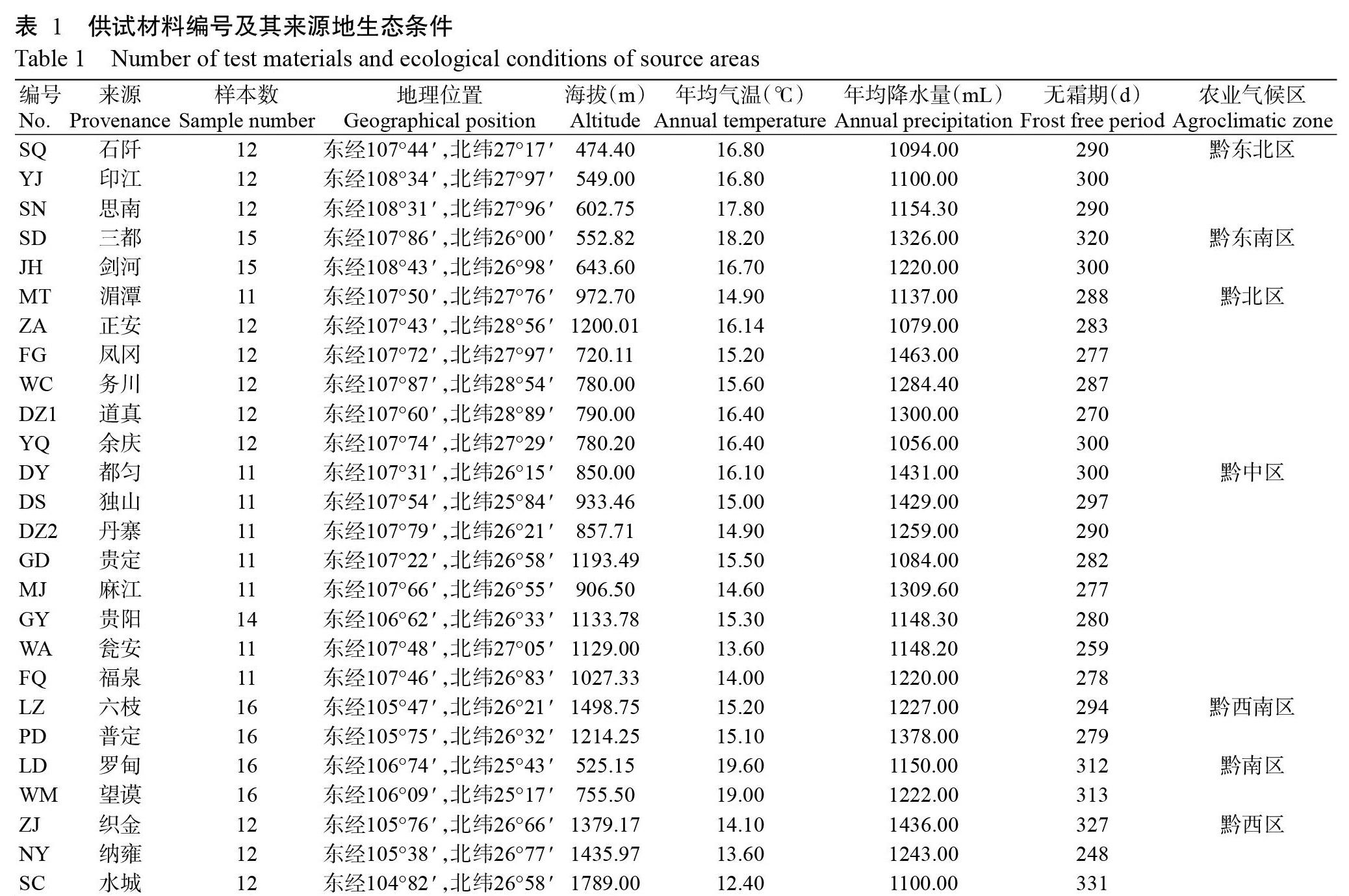
从黔东北部(温暖伏旱区)、黔东南部(温暖湿润区)、黔北部(温和伏旱区)、黔中部(温和湿润区)、黔西南部(温和春干夏雨区)、黔南部(温热春干区)及黔西部(温凉春干区)等7个农业气候区选取26个棕榈种源328份材料,所选样株均为野生资源,树龄在20年左右,单株距离在50 m以上。分别采集生长良好、无病虫害的当年生嫩叶,经硅胶干燥、液氮速冻后带回实验室,-80 ℃保存备用。利用GPS定位样品采集地的地理坐标,具体采样情况及种源地生态条件如表1所示。主要试剂:新型植物DNA提取试剂盒(DP320)、D2000 DNA Marker(MD114)和2×Taq PCR MasterMix(KT201)均购自天根生化科技(北京)有限公司。主要仪器设备:BioPhotometer Plus核酸蛋白定量检测仪(德国Eppendorf公司),BIO-RAD T100 PCR仪(美国Bio-Rad公司),Tanon 1600凝胶成像系统(上海天能科技有限公司),Qsep 100全自动核酸蛋白分析仪(中国台湾光鼎生物科技股份有限公司)。
1. 2 DNA提取
将采集的材料取出并迅速剪碎,置于盛有液氮的无菌研钵中研磨至粉末状,然后按新型植物DNA提取试剂盒说明提取棕榈总DNA,用1.0%琼脂糖凝胶电泳和BioPhotometer Plus核酸蛋白定量检测仪进行检测,合格的棕榈总DNA以TE稀释标定至100 ng/μL,-20 ℃保存备用。
1. 3 SRAP反应体系建立
从已发表的不同植物SRAP分子标记引物序列(李晓慧等,2007;李梅等,2009;刘倩,2013;林方养等,2014)中筛选出正向引物17条、反向引物18条,形成306对SRAP引物组合,委托北京全式金生物技术有限公司合成。利用4个种质材料对SRAP引物组合进行复筛,选出扩增条带清晰稳定、多态性丰富、重复性好的引物组合(表2)。优化后的SRAP反应体系20.0 ?L:DNA模板2.0 ?L,上、下游引物(10 ?mol/L)各0.5 ?L,2×Taq PCR MasterMix 10.0 ?L,ddH2O 7.0 ?L。参照林方养(2014)的SRAP扩增程序:94 ℃预变性3 min;94 ℃ 30 s,35 ℃ 45 s,72 ℃ 45 s,进行5个循环;94 ℃ 30 s,50 ℃ 45 s,72 ℃ 45 s,进行35个循环;72 ℃延伸10 min,4 ℃保存。
1. 4 扩增产物检测与分析
SRAP扩增产物直接利用Qsep 100全自动核酸蛋白分析仪进行分析,全自动进样,无须人工制胶、灌胶、上样、拍照,免去手工制胶的繁琐和人工上样的误差,产物长度(bp)可直接读出并根据需要选择呈现信号峰值图或模拟电泳图。根据模拟电泳结果,对不同材料在同一位点的扩增片段进行统计,条带清晰明亮记为“1”,无条带或模糊记为“0”,生成0-1矩阵。转换相应的数据格式后,用Popgene 1.32计算多态性条带百分率(PPB)、观测等位基因数(Na)、有效等位基因数(Ne)、Nei?s基因多样度(H)、Shannon?s信息指数(I)、遗传一致度(In)、遗传距离(D)、总基因多样性(Ht)、种源内基因多样性(Hs)、种源间的遗传分化系数(Gst)及基因流(Nm)等主要遗传多样性参数,并采用NTSYS-pc 2.1按照UPGMA法进行种源聚类分析。根据采样点地理坐标信息计算各样点间的地理距离,使用TFPGA 1.3对遗传距离和地理距离进行Mantel检验;利用PASW Statistics v18.0分析遗传多样性与种源地生态条件线性相关性,探索遗传多样性空间分布格局。
2 结果与分析
2. 1 SRAP多态性分析结果
10对SRAP引物组合共扩增得到260条目的条带,且扩增条带清晰稳定(图1),其中多态性条带253条(平均25.3条),PPB为97.31%(表3)。以Me4-Em5引物组合的PPB最低,为88.00%;有7对SRAP引物组合(Me1-Em2、Me2-Em1、Me3-Em1、Me3-Em4、Me4-Em4、Me5-Em2和Me6-Em5)的PPB達100.00%。说明这10对SRAP引物组合能稳定高效地检测棕榈资源遗传多样性,为核心种质筛选提供依据。
2. 2 棕榈遗传多样性评价及空间分布格局分析结果
26个棕榈种源的遗传多样性参数统计结果如表4所示。其中,PPB在31.92%(YJ)~79.62%(NY),平均为61.15%;Na在1.3192(YJ)~1.7962(NY),平均为1.6112;Ne在1.1809(SQ)~1.3678(NY),平均为1.2975;H在0.1136(SQ)~0.2292(NY),平均为0.1809;I在0.1741(YJ)~0.3571(NY),平均为0.2790。26个棕榈种源间的遗传多样性差异明显,纳雍(NY)、贵定(GD)、六枝(LZ)、贵阳(GY)和湄潭(MT)等种源的遗传多样性较丰富,而印江(YJ)、石阡(SQ)和剑河(JH)等种源的遗传多样性较低。贵州省棕榈种级水平的PPB为97.31%、Na为1.9731、Ne为1.4717、H为0.2689、I为0.4212,说明贵州省棕榈遗传多样性丰富。
由表5可知,各农业气候区棕榈居群的遗传多样性存在明显差异,其中,黔中(温和湿润)农业气候区的多态性条带最多(250条),PPB高达96.15%,对应的Na、Ne、H、I分别为1.9615、1.3946、0.2470和0.3895,均较高;黔东北(温暖伏旱)农业气候区的多态性条带最少(171条),PPB仅为65.77%,对应的Na、Ne、H、I分别为1.6577、1.2683、0.1697和0.2680,均最低。
棕榈主要遗传多样性参数与种源地生态条件的相关性分析结果(表6)显示,PPB、H和I与年均气温、无霜期均呈负相关,与年均降水量呈正相关,但相关性均不显著(P>0.05,下同)。PPB与海拔呈正相关,但相关性不显著;I和H与海拔呈显著正相关(P<0.05,下同),表明海拔对贵州省棕榈的遗传多样性分布有显著影响。
2. 3 棕榈居群的聚类分析结果
基于遗传一致性的UPGMA聚类分析结果(图2)显示,在阈值为0.91时,26个棕榈种源可分成四大类,第Ⅰ类只有都匀(DY)1个种源;第Ⅱ类10个种源,包括贵定(GD)、三都(SD)、湄潭(MT)、凤冈(FG)、务川(WC)、道真(DZ1)、纳雍(NY)、独山(DS)、丹寨(DZ2)和麻江(MJ);第Ⅲ类6个种源,包括水城(SC)、普定(PD)、织金(ZJ)、六枝(LZ)、罗甸(LD)和望谟(WM);第Ⅳ类9个种源,包括贵阳(GY)、瓮安(WA)、剑河(JH)、思南(SN)、福泉(FQ)、余庆(YQ)、正安(ZA)、石阡(SQ)和印江(YJ)。可见,属于同一农业气候区的棕榈种源大多数能聚在同一类,即贵州省棕榈资源的遗传分化与其地理环境有一定相关性。Mantel检验结果(图2)显示,贵州省棕榈资源遗传分化与其地理距离呈正相关(r=0.2651),但相关性不显著,说明地理隔离对棕榈遗传分化有一定影响,但并非主要因素。
2. 4 棕榈居群的遗传分化情况
由表7可知,贵州省棕榈资源种级水平的Ht为0.2702,Hs为0.1809,Gst为0.3305,说明贵州省棕榈资源遗传变异的66.95%来自种源内,而33.05%来自种源间,即种源内变异是贵州省棕榈资源遗传变异的主要来源。黔东南区棕榈种源间的遗传变异最大,占该区总遗传变异的30.19%;黔南区棕榈种源间的遗传变异最小,为7.40%。贵州省棕榈资源种级水平的Nm为1.0129,大于1.0000,说明贵州省各棕榈种源间存在较低的基因流。此外,7个农业气候区内的棕榈资源基因流均大于贵州省内棕榈种源基因流,说明小区域内的棕榈资源基因交流频繁。
3 讨论
PPB、H和I 是衡量种群遗传多样性的3个重要参数。其中,PPB只是一种粗略估计,尚无法确定各条带在频率上的均匀程度,且受样本大小、条带多少及筛选方法的影响(红雨等,2006);而H 和I 充分考虑了群体中的基因数目及其分布均匀度,三者结合可有效反映遗传多样性水平(黄勇,2013)。本研究结果表明,贵州省棕榈物种水平上的PPB为97.31%、H为0.2689、I为0.4212,明显高于蒙古栎(李文英,2003)、厚朴(郑志雷,2010)、杏(马丽娟,2013)、紫椴(王东升,2013)及玉铃花(王萱,2016)等常见温带多年生木本植物,也明显高于银杏(葛永奇等,2003)、构树(廖声熙等,2013)及杜仲(于靖,2015)等雌雄异株的木本植物,但在棕榈科植物中略低于乔木形的董棕(赵春磊,2009)、槟榔(任军方,2010)及丛生灌木形的山棕(贾春媛,2009),高于黄藤和单叶省藤等木质藤本(杨华,2005)。说明从地理分布、交配系统及生活型等多方面观察均证实贵州省棕榈具有丰富的遗传多样性。遗传多样性与种源地生态条件的相关性分析结果表明,海拔对贵州省棕榈资源遗传多样性分布有显著影响,可能是由于贵州高海拔山区地势复杂,交通不便,人为破坏少,能保存较高的遗传多样性。棕榈作为棕榈科植物中最抗寒的一个种,广泛的地理分布说明其拥有较大的基因库及生境异质性,多世代重叠有效保留了重组和突变;而严格的雌雄异株异交、风媒花保证了高频的远系杂交能力和异交率(朱华晨,2000)。这些特征都可能是棕榈仍维持较高遗传多样的重要原因。
本研究还发现,贵州省棕榈物种水平上的Gst为0.3305,明显高于木本植物(Gst=0.2100)及传粉植物(Gst=0.2300)的平均值(Nybom,2004),说明贵州省棕榈各种源间已有一定程度的分化。Mantel检验结果显示,贵州省棕榈资源的遗传分化与地理隔离呈较弱正相关,仅有不到0.1%的遗传变异可由地理距离解释,说明地理隔离不是棕榈遗传分化的主要因素。UPGMA聚类分析结果显示,贵州省棕榈资源的遗传分化与地理环境有一定相关性,同一农业气候区的棕榈种源大多数聚在同一类,可能是在自然选择压力下,具有相同基因型的个体共同聚集在较适应的生境中,从而产生遗传分化(王玉山等,2011;黄勇,2013;马少薇等,2017)。这种由生境异质性下选择压力及遗传漂移所形成的遗传分化大于地理距离所带来的遗传分化,但地理环境影响种源分化的具体进程有待进一步探究。此外,贵州省棕榈资源种级水平的Nm为1.0129,大于1.0000,即贵州省各棕榈种源间基因流动很少,故推测低水平的基因流动是造成遗传变异以种源内变异为主、种源间遗传分化明显的主要原因。各农业气候区内棕榈资源的Nm大于各棕榈种源间的Nm,表明棕榈的基因流动受限于小区域范围内。花粉与种子传播是棕榈主要的基因流动形式,二者均受自身特点与环境条件的影响。棕榈树体高大,花序抽生于树顶端、花量巨多、开花集中,使得花粉能借助风力广泛传播,但花期时贵州境内多阴雨天气,不利于花粉大范围传播;棕榈种子较大,果熟期天气寒冷,鸟兽活动相对较少,故其种子传播距离有限。这些因素是导致贵州省棕榈基因流动较低且较窄的主要原因。
棕榈作为贵州省内重要的生态与经济树种,其丰富的遗传多样性与种源变异是遗传改良的宝贵材料,拥有极高的种质资源保育价值。因此,在今后的种质收集与保存工作中,应将黔中区、黔北区和黔西区等棕榈资源遗传多样性相对较丰富的地区划为核心种源区,设立多个保护点,并尽可能收集多个种群的种子或幼苗,其他地区则应多选择一些优良单株的家系苗。在此基础上,结合高通量测序技术与生物信息学分析手段,揭示棕片产量等重要农业性状的形成机理,或挖掘与之紧密连锁的分子标记,并综合筛选出一批特异育种材料,为贵州棕榈资源的开发利用打下基础。
4 结论
贵州省棕榈种质资源总体遗传多样性高,种源间分化较明显,选择育种潜力巨大。为更好地开发利用棕榈资源,应结合其遗传结构特点,实行就地保护为主及建设种质资源库为补充的保护策略。
参考文献:
安琪,张国学,何承忠,王娟,杨宇明. 2010. 省藤属11种棕榈藤亲缘关系的AFLP分析[J]. 西北植物学报,30(6):1117-1122. [An Q,Zhang G X,He C Z,Wang J,Yang Y M. 2010. Analysis of phylogenetic relationship on 11 Calamus species(Palmae) by AFLP markers[J]. Acta Botanica Boreali-Occidentalia Sinica,30(6):1117-1122.]
陳大霞,张雪,王钰,潘媛,李隆云,易思荣,张泽. 2018. 基于ISSR、SCoT、SRAP标记的川续断属药用植物亲缘关系研究[J]. 中草药,49(4):903-909. [Chen D X,Zhang X,Wang Y,Pan Y,Li L Y,Yi S R,Zhang Z. 2018. Analysis on genetic relationship of medicinal species of Dipsacus marked by ISSR,ScoT,and SRAP[J]. Chinese Traditional and Herbal Drugs,49(4):903-909.]
但新球,喻甦,吴协保,宁小斌. 2003. 我国石漠化区域划分及造林树种选择探讨[J]. 中南林业调查规划,22(4):20-23. [Dan X Q,Yu S,Wu X B,Ning X B. 2003. Discussion on the regional division of rocky desertification and the selection of afforestation tree species in China[J]. Central South Forest Inventory and Planning,22(4):20-23.]
丁灿,郎南军,林位夫. 2011. 中国油棕种质材料的遗传多样性研究[J]. 中国农学通报,27(21):131-137. [Ding C,Lang N J,Lin W F. 2011. Study on genetic diversity of oil palm germplasm in China[J]. Chinese Agricultural Science Bulletin,27(21):131-137.]
董云发. 2005. 棕榈的综合利用和开发[J]. 中国野生植物资源,24(1):25-27. [Dong Y F. 2005. The many uses and exploitation of Trachycarpus fortunei[J]. Chinese Wild Plant Resources,24(1):25-27.]
冯建灿,张梦洋,李敏,程钧,方伟超,牛良,郑先波,叶霞,谭彬. 2019. 桃果皮茸毛性状IndelG标记基因分型与应用[J]. 河南农业大学学报,53(1):64-72. [Feng J C,Zhang M Y,Li M,Cheng J,Fang W C,Niu L,Zheng X B,Ye X,Tan B. 2019. Analysis and application of genotypes on peel fuzz trait in peach resources with IndelG molecular marker[J]. Journal of Henan Agricultural University,53(1):64-72.]
葛永奇,邱英雄,丁炳扬,傅承新. 2003. 孑遗植物银杏群体遗传多样性的ISSR分析[J]. 生物多样性,11(4):276-287. [Ge Y Q,Qiu Y X,Ding B Y,Fu C X. 2003. An ISSR analysis on population genetic diversity of the relict plant Ginkgo biloba[J]. Biodiversity Science,11(4):276-287.]
红雨,王林和,梁小荣. 2006. 不同生境臭柏种群的遗传多样性分析及其与环境因子的相关性[J]. 干旱区资源与环境,20(3):184-187. [Hong Y,Wang L H,Liang X R. 2006. Analysis on the genetic diversity of Sabina vulgaris at different habitats and the influence of the environmental factors[J]. Journal of Arid Land Resources and Environment,20(3):184-187.]
胡志姣,赵杨. 2018. 贵州省棕榈种源地理变异研究[J]. 西部林业科学,47(2):7-14. [Hu Z J,Zhao Y. 2018. Geographic variation of Trachycarpus fortunei in Guizhou Province[J]. Journal of West China Forestry Science,47(2):7-14.]
黄勇. 2013. 基于SRAP分子标记的小果油茶遗传多样性分析[J]. 林业科学,49(3):43-50. [Huang Y. 2013. Analysis of Camellia meiocarpa genetic diversity based on SRAP markers[J]. Scientia Silvae Sinicae,49(3):43-50.]
贾春媛. 2009. 中国原生香棕(Arenga engleri Becc.)种群的遗传多样性研究[D]. 厦门:厦门大学. [Jia C Y. 2009. Study on genetic diversity of native Arenga engleri Becc. populations in China[D]. Xiamen:Xiamen University.]
李梅,侯喜林,郝日明. 2009. 基于SRAP分子标记的桂花品种亲缘关系研究[J]. 园艺学报,36(11):1667-1675. [Li M,Hou X L,Hao R M. 2009. Analysis of genetic relationships of Osmanthus fragrans based on SRAP markers[J]. Acta Horticulturae Sinica,36(11):1667-1675.]
李文表. 2005. 棕櫚(Trachycarpus fortunei)的遗传多样性研究[D]. 广州:华南师范大学. [Li W B. 2005. Study on genetic diversity of Trachycarpus fortunei[D]. Guangzhou:South China Normal University.]
李文英. 2003. 蒙古栎天然群体遗传多样性研究[D]. 北京:北京林业大学. [Li W Y. 2003. Study on genetic diversity of natural population in Quercus mongolica[D]. Beijing:Beijing Forestry University.]
李晓慧,田朝阳,王从彦. 2007. SRAP分子标记分析西瓜遗传多态性[J]. 生物技术,17(3):23-26. [Li X H,Tian C Y,Wang C Y. 2007. Genetic polymorphism analysis by sequence-related amplified polymorphism markers in Citrullus lanatus[J]. Biotechnology,17(3):23-26.]
廖声熙,崔凯,周敏,何承忠,杨文云,李昆. 2013. 云南河谷构树的遗传多样性研究[J]. 林业科学研究,26(5):588-592. [Liao S X,Cui K,Zhou M,He C Z,Yang W Y,Li K. 2013. Analysis on genetic diversity of Broussonetia papyrifera distributed in river basin of Yunnan Province[J]. Forest Research,26(5):588-592.]
林方养,杨耀东,万婕,唐嘉,马子龙,吴翼. 2014. 棕榈科植物DNA的提取及SRAP引物筛選[J]. 江西农业学报,26(7):115-117. [Lin F Y,Yang Y D,Wan J,Tang J,Ma Z L,Wu J. 2014. Extraction of genomic DNA from Palm plants and screening of SRAP primers[J]. Acta Agriculturae Jiangxi,26(7):115-117.]
凌士鹏,孙萍,林贤锐,沈建生. 2018. 基于SSR标记的桃种质资源遗传多样性研究[J]. 江西农业学报,30(11):14-18. [Ling S P,Sun P,Lin X R,Shen J S. 2018. A study on genetic diversity of peach germplasm resources based on SSR markers[J]. Acta Agriculturae Jiangxi,30(11):14-18.]
刘倩. 2013. 应用SRAP分子标记构建红麻种质资源分子身份证[D]. 北京:中国农业科学院. [Liu Q. 2013. Establishment of molecular identity for kenaf germplasm using SRAP markers[D]. Beijing:Chinese Academy of Agricultural Sciences.]
卢超,张美德,何银生,刘海华,艾伦强. 2014. SRAP技术在植物遗传育种中的应用[J]. 现代农业科技,(16):18-19. [Lu C,Zhang M D,He Y S,Liu H H,Ai L Q. 2014. Application of SRAP technology in plant genetics and breeding[J]. Modern Agricultural Science and Technology,(16):18-19.]
马丽娟. 2013. 东北野生杏居群遗传多样性及遗传结构分析[D]. 长春:吉林农业大学. [Ma L J. 2013. Genetic diversity and genetic structure analysis of wild apricot populations in Northeast China[D]. Changchun:Jilin Agricultural University.]
马少薇,董建芳,刘果厚. 2017. 内蒙古地区黄柳遗传多样性的RAPD分析[J]. 干旱区资源与环境,31(8):175-180. [Ma S W,Dong J F,Liu G H. 2017. RAPD analysis of genetic diversity of Salix gordejevii in Inner Mongolia[J]. Journal of Arid Land Resources and Environment,31(8):175-180.]
马秀花,唐道城,唐楠. 2019. 万寿菊育性相关基因SRAP分子标记开发[J]. 分子植物育种,17(20):6718-6723. [Ma X H,Tang D C,Tang N. 2019. Development of SRAP molecular marker for fertility related genes of marigold[J]. Molecular Plant Breeding,17(20):6718-6723. ]
潘绿昌,赵致,何云,李园园,刘红昌,俸婷婷,周英. 2018. 基于SRAP分子标记的贵州5种忍冬属药用植物遗传多样性分析[J]. 南方农业学报,49(12):2349-2355. [Pan L C,Zhao Z,He Y,Li Y Y,Liu H C,Feng T T,Zhou Y. 2018. Genetic diversity among five species of medicinal plants of Lonicera L. in Guizhou Province by SRAP molecular markers analysis[J]. Journal of Southern Agriculture,49(12):2349-2355.]
庞新华,檀小辉,韦丽君,梁芳,张继,吕平,程琴,黄秋伟,周全光. 2019. 35份甘蔗种质的遗传多样性分析及DNA指纹图谱构建[J]. 南方农业学报,50(6):1157-1164. [Pang X H,Tan X H,Wei L J,Liang F,Zhang J,Lü P,Cheng Q,Huang Q W,Zhou Q G. 2019. Genetic diversity analysis for 35 sugarcane germanium resources and construction of DNA fingerprint[J]. Journal of Southern Agriculture,50(6):1157-1164.]
邱帅,沈柏春,李婷婷,郭娟,王霁,孙丽娜,陈徐平,胡绍庆. 2018. 基于随机森林算法和SRAP分子标记的桂花品种鉴定方法[J]. 林业科学,54(1):32-45. [Qiu S,Shen B C,Li T T,Guo J,Wang J,Sun L N,Chen X P,Hu S Q. 2018. A method of Osmanthus fragrans cultivars identification based on random forest algorithms and SRAP molecular markers[J]. Scientia Silvae Sinicae,54(1):32-45.]
任軍方. 2010. 利用ISSR标记对海南保亭槟榔遗传多样性的研究[D]. 海口:海南大学. [Ren J F. 2010. Genetic diversity of Areca catechu L. from Baoting of Hainan Pro-vince detected with ISSR markers[D]. Haikou:Hainan University.]
舒迎澜. 2004. 古代棕榈及其利用[J]. 园林,(7):44-45. [Shu Y L. 2004. Ancient palms and their utilization[J]. Gardens,(7):44-45.]
王东升. 2013. 山东省紫椴和野核桃AFLP遗传多样性研究[D]. 泰安:山东农业大学. [Wang D S. 2013. Genetic diversity study of Tilia amurensis ruprecht and Juglans cathayensis dode from Shandong Province by AFLP mar-kers[D]. Tai?an:Shandong Agricultural University.]
王萱. 2016. 玉铃花遗传多样性的AFLP分析[D]. 泰安:山东农业大学. [Wang X. 2016. Genetic diversity of Styrax obassia Sieb. et Zucc based on AFLP markers[D]. Taian:Shandong Agricultural University.]
王玉山,邢世岩,唐海霞,冯殿齐. 2011. 侧柏种源遗传多样性分析[J]. 林业科学,47(7):91-96. [Wang Y S,Xing S Y,Tang H X,Feng D Q. 2011. Genetic diversity of Platycladus orientalis provenances[J]. Scientia Silvae Sinicae,47(7):91-96.]
卫强,王燕红. 2016. 棕榈花、叶、茎挥发油成分及抑菌活性研究[J]. 浙江农业学报,28(5):875-884. [Wei Q,Wang Y H. 2016. Study on chemical components of the essential oils in flower,leaf and stem of Trachycarpus fortunei (Hook) H. Wendl and their antimicrobial activities[J]. Acta Agriculturae Zhejiangensis,28(5):875-884.]
肖勇,杨耀东,夏薇,沈雁,雷新涛,马子龙. 2013. 25个油棕SSR标记的开发及应用这些标记评估油棕资源的遗传多样性[J]. 江西农业学报,25(12):27-31. [Xiao Y,Yang Y D,Xia W,Shen Y,Lei X T,Ma Z L. 2013. Development of 25 SSR markers and their uses in evaluating genetic diversity of oil palm germplasm resources[J]. Acta Agriculturae Jiangxi, 25(12):27-31.]
杨华. 2005. 黄藤和单叶省藤遗传多样性与早期性别鉴定的研究[D]. 北京:中国林业科学研究院. [Yang H. 2005. Study on genetic diversity and sex identification of Daemonorops margaritae & Calamus simplificifolius[D]. Beijing:Chinese Academy of Forestry.]
于靖. 2015. 杜仲种质资源及其果实质量评价[D]. 杨凌:西北农林科技大学. [Yu J. 2015. Germplasm resource of Eucommia ulmoides oliver and fruits quality evaluation[D]. Yangling:Northwest Agriculture and Forestry University.]
张明飞,于卓,于肖夏,李景伟,马艳红,吴国芳,王颖. 2019. 四倍体马铃薯SRAP分子遗传连锁图谱的构建[J]. 草业学报,28(8):190-199. [Zhang M F,Yu Z,Yu X X,Li J W,Ma Y H,Wu G F,Wang Y. 2019. Construction of a genetic linkage map for tetraploid potato based on SRAP markers[J]. Acta Prataculturae Sinica,28(8):190-199.]
赵春磊. 2009. 利用ISSR分析中国原生董棕植物遗传多样性[D]. 厦门:厦门大学. [Zhao C L. 2009. Analyzed the Chinese primary Caryota urens Linn heredity multiplicity using ISSR[D]. Xiamen:Xiamen University.]
赵小庆,史功赋,张向前,方静,叶君,王建国,伊六喜,路战远. 2019. 内蒙古地区典型春小麦亲缘关系的SRAP分析[J/OL]. 分子植物育种. http://kns.cnki.net/kcms/detail/46.1068.s.20190424.1511.018.html. [Zhao X Q,Shi G F,Zhang X Q,Fang J,Ye J,Wang J G,Yi L X,Lu Z Y. 2019. Genetic relations of spring wheat varieties in Inner Mongolia region by SRAP markers[J/OL]. Molecular Plant Breeding. http://kns.cnki.net/kcms/detail/46.1068.s.2019 0424.1511.018.html.]
郑志雷. 2010. 厚朴遗传多样性研究及指纹图谱的构建[D]. 福州:福建农林大学. [Zheng Z L. 2010. Studies on genetic diversity and construction of fingerprinting of Magnolia officinalis[D]. Fuzhou:Fujian Agriculture and Fo-restry University.]
周丽霞,曹红星. 2018. 椰子种质资源遗传多样性的SSR分析[J]. 南方农业学报,49(9):1683-1690. [Zhou L X,Cao H X. 2018. SSR analysis on genetic diversity in coconut germplasm resource[J]. Journal of Southern Agriculture,49(9):1683-1690.]
朱华晨. 2000. 高等植物单性花的性别决定[J]. 中山大学研究生学刊(自然科学版),21(2):42-46. [Zhu H C. 2000. The sex determination in the higher plants unisexual flowers[J]. Journal of the Graduates Sun Yat-Sen University(Na-tural Sciences),21(2):42-46.]
Geethanjali S,Anitha Rukmani J,Rajakumar D, Kadirvel P. 2018. Genetic diversity,population structure and association analysis in coconut(Cocos nucifera L.) germplasm using SSR markers[J]. Plant Genetic Resources,16(2):156-168.
Hayati A,Wickneswari R,Maizura I,Rajanaidu N. 2004. Genetic diversity of oil palm(Elaeis guineensis Jacq.) germplasm collections from Africa:Implications for improvement and conservation of genetic resources[J]. Theoretical and Applied Genetics,108(7):1274-1284.
Kondo T,Kajita R,Yamada T. 2017. Development and characterization of 20 polymorphic microsatellite markers for Livistona chinensis R. Br. var. subglobosa Becc.,a fan palm in the Ryukyu Archipelago,Japan[J]. Plant Species Biology,32(4):477-481.
Nybom H. 2004. Comparison of different nuclear DNA mar-kers for estimating intraspecific genetic diversity in plants[J]. Molecular ecology,13(5):1143-1155.
Ohtani M,Tani N,Yoshimaru H. 2009. Isolation of polymorphic microsatellite loci in Livistona chinensis(Jacq.) R. Br. ex Mart. var. boninensis Becc.,an endemic palm species of the oceanic Bonin Islands,Japan[J]. Conservation Genetics,10(4):997-999.
Rajesh M K,Sabana A A,Rachana K E,Rahman S,Jerard B A,Karun A. 2015. Genetic relationship and diversity among coconut(Cocos nucifera L.) accessions revealed through SCoT analysis[J]. 3 Biotech,5(6):999-1006.
(責任编辑 兰宗宝)

