陆地棉SPL基因家族的全基因组鉴定及表达分析①
钟子达,钟晓真,沈超
( 广东石油化工学院 生物与食品工程学院,广东 茂名 525000)
鳞状启动子结合蛋白样(SPL)是植物特有的一类转录因子,在绿色植物中广泛存在[1]。SPL含有一个高度保守的SBP-box的DNA 结合结构域[2],SBP结构域专一识别并结合一段以GTAC为核心元件的回文序列[3],具有2个均含有8个保守的半胱氨酸(Cys)或组氨酸(His)残基的独立锌指结构[4]。
SPL转录因子影响植物的顶芽、花序发育以及开花时间[5]。SPL最初是从金鱼草[6]中发现的,它能识别并结合到SQUAMOSA (SQ-UA)的启动子上,进而参与调控金鱼草早期的花发育和开花过程[7]。研究发现,SPL对植物花和果实的发育、胁迫应答、孢子形成、激素信号传导以及植物阶段转变[8-17]等多种生理生化过程都有一定的调控作用,在植物的生长发育过程中发挥着重要的作用。
棉花是世界上重要的经济作物,也是天然纤维的主要来源。耕地面积的日益减少加剧了粮棉争地的矛盾,培育短季棉品种成为当前一个重要育种目标[18]。开花提前可以缩短生育期,因此研究SPL基因家族,对短季棉育种具有重要促进作用。本研究利用生物信息学方法,对棉花主要栽培种陆地棉中SPL基因家族成员进行鉴定及分析,从全基因组水平解析其基本理化性质、基因数目、基因结构与Motif变化、基因的进化和染色体定位等方面,为进一步探究SPL基因在棉花的成花转变、花发育中的功能和短季棉育种研究提供一定的理论基础。
1 材料与方法
1.1 SPL基因的鉴定
在Cotton FGD数据库(https://cottonfgd.org)中下载陆地棉(AD1, HAU)的基因组数据。在Pfam数据库(http://pfam.xfam.org/)下载SPL蛋白的hmm文件,标号为PF03110。使用 HMMER 3.0 软件(http://hmmer.org/)和BLASTP 程序比对含有SBP蛋白结构域的序列。之后,用SMART (http://smart.embl-heidelberg.de)和保守结构域数据库(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)进行比对验证。
1.2 陆地棉SPL基因家族成员的特性、基因结构与染色体定位分析
利用ExPASy的在线工具ProtParam(https://web.expasy.org/protparam)对陆地棉SPL基因家族成员的氨基酸长度、相对分子质量、等电点、不稳定系数等理化性质进行分析。使用ProtComp9.0 (http://www.softberry.com/berry.phtml?topic=protcomp-pl&group=programs&subgroup=proloc)预测其亚细胞定位。利用GSDS v2.0 (http://gsds.cbi.pku.edu.cn/)分析绘制其基因结构图。利用MEME (http://meme-suite.org/) 对SPL蛋白保守结构域进行分析,基序最大发现数量设置为10,其他参数为默认值。利用NPSA (https://npsa-prabi.ibcp.fr/cgi-bin/secpred_sopma.pl)预测蛋白质的二级结构。利用TBtools v0.6731 (https://github.com/CJ-Chen/TBtools-Manual) 定位基因在染色体上的位置。
1.3 多序列比对与系统进化树的构建
利用MEGA 7.0[19]软件对获得的SPL序列进行多重序列比对。基于多序列比对采用最大似然法构建进化树,执行参数 Bootstrap 1000次重复,其他参数为系统默认值。
1.4 陆地棉SPL家族基因的表达模式分析
基于陆地棉RNA-seq数据[20]分析陆地棉SPL基因的表达模式,包括棉花的根、茎、叶、花瓣、花药、柱头和开花后5,10,15,20,25 d的纤维及0,1,3,10,20 d的胚珠等组织。
2 结果与分析
陆地棉SPL基因家族的生物信息学分析主要包括理化分析、系统进化分析等内容。
2.1 SPL家族成员的鉴定和理化分析
最终在陆地棉中鉴定到30个SPL基因(表1)。在陆地棉中,基因的长度为1125~8132 bp,其中Ghir_D13G016090基因最短,为1125 bp,Ghir_A12G013050基因最长,为8132 bp。理化性质分析发现:陆地棉中SPL编码氨基酸的数量为141 ~ 1081,Ghir_A10G002400的氨基酸数量最少,为141,Ghir_A01G009280和Ghir_D01G009680的氨基酸数量最多,为1081,平均为527.9;相对分子质量介于16.11 ~ 119.59 kDa,等电点为5.25~9.88,亚细胞定位表明有6个基因定位到细胞质和细胞核(表1)。

表1 陆地棉中SPL特征

续表
2.2 SPL的系统进化分析
系统发育分析表明:SPL基因家族可以分为6个亚群,同一分支的进化程度相近,亲缘性高,不同亚群中的基因数目不等(图1)。
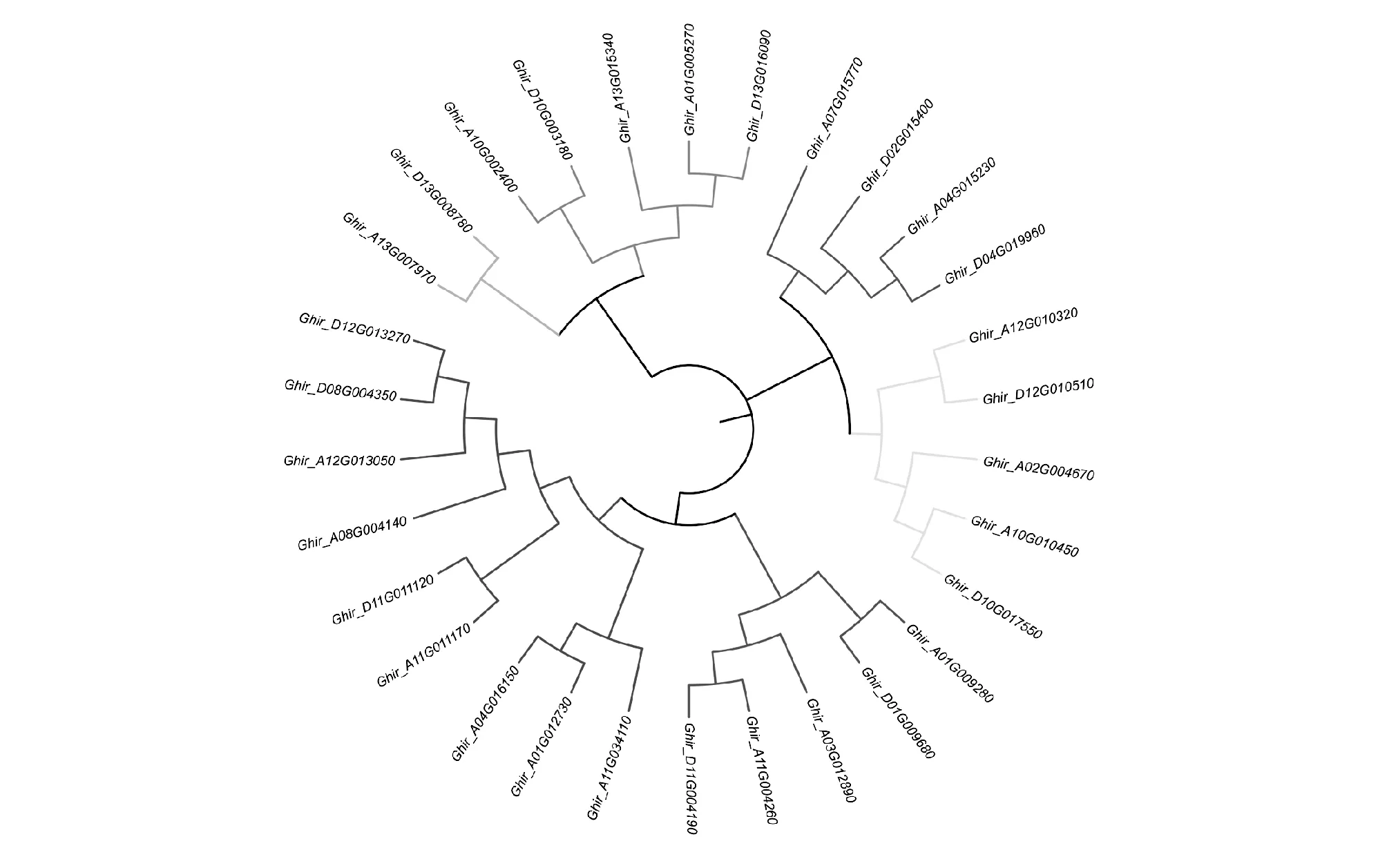
图1 陆地棉SPL家族基因的进化分析
2.3 SPL基因家族的结构
陆地棉SPL基因家族成员结构的外显子数目为2 ~ 12个,其中Ghir_A10G010450的外显子数目最多,为12个(图2),进一步分析得到10种保守基序,命名为Motif1—Motif10,每个成员含有3~10个Motif(图3)。系统发育分析发现,同一组内具有相似的外显子数目且具有相似的基因序列结构。

图2 陆地棉SPL的结构分析 图3 陆地棉SPL家族保守基序分析
2.4 SPL基因在染色体上的定位
SPL基因分布在陆地棉18条染色体上(图4)。每条染色体上基因的分布数量不等,A亚基因组的A02、A03、A04、A07和A08以及 D亚基因组的D01、D02、D04和D08染色体上各有1个基因;A亚基因组的A10、A12、A13和D亚基因组的D10、D12、D13染色体上分别有2个基因;A亚基因组的A11染色体有3个基因(图4)。
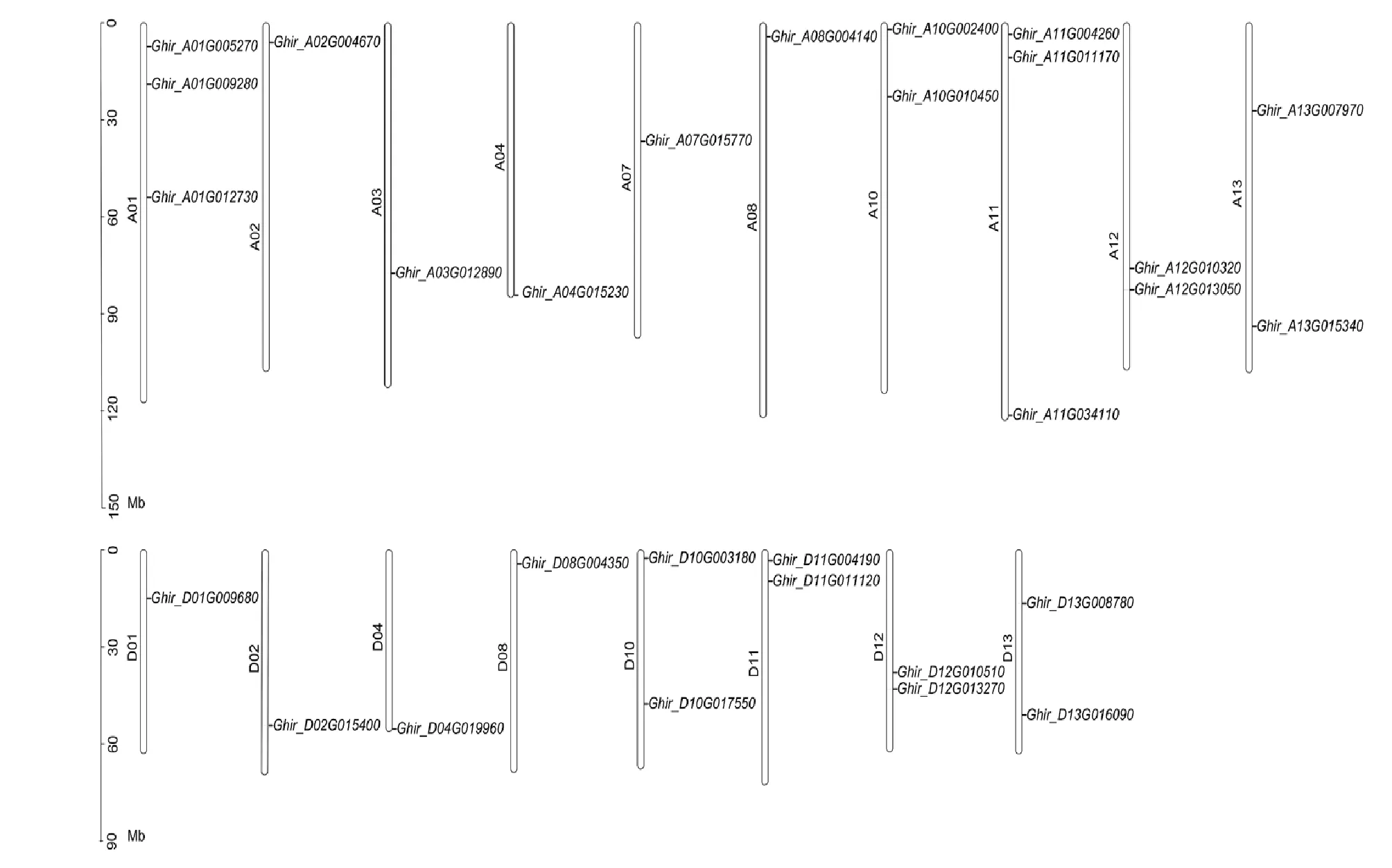
图4 陆地棉SPL基因家族染色体定位
2.5 SPL的二级结构分析
对陆地棉SPL蛋白的二级结构分析,如表1所示。发现SPL基因家族均含有α螺旋、β折叠、延伸链和无规则卷曲等二级结构原件。无规卷曲的占比最高(除基因Ghir_A01G005270,Ghir_A10G002400,Ghir_D10G003180外),其次是α螺旋、延伸链和β折叠(表1)。
2.6 SPL基因的表达模式分析
通过对陆地棉SPL在棉花的根、茎、叶、花瓣、花药、柱头和开花后5,10,15,20,25 d的纤维及开花后 0,1,3,10,20 d的胚珠中的表达模式进行分析,分析发现Ghir_D08G004350、Ghir_A08G004140、Ghir_A12G013050、Ghir_D12G013270、Ghir_D01G009680、Ghir_A01G009280、Ghir_A11G011170、Ghir_D11G011120、Ghir_D10G017550这9个基因在组织中的表达量很高(图5),例如:Ghir_D08G004350在花瓣的表达最高,表明该基因可能参与调控花瓣的生长发育。有些基因的表达量非常低,例如:Ghir_A01G005270除了在根和柱头表达量较高外,在其他组织的表达量很低,甚至不表达。

图5 陆地棉SPL基因家族的组织表达分析
3 结论与讨论
基因组测序技术的发展对基因组研究、转录因子的鉴定与表达分析起到了极大的促进作用。SPL基因家族在许多物种中都得到了深入的研究,表明它们在植物的生长发育过程中发挥了重要作用,并且不同物种的SPL数量也不同。
随着棉花基因组测序的完成与更新,本研究在陆地棉中鉴定30个SPL基因并进一步预测了其理化性质、二级结构及亲缘关系。研究结果表明,陆地棉中SPL相对分子质量为16.11 ~ 119.59 kDa,等电点介于5.25 ~ 9.88。二级结构主要元件为无规则卷曲。聚类分析显示,SPL基因可以分为6个亚组,同一分支的进化程度相近,亲缘性高。结合基因结构和保守基序分析发现,同一组内的SPL基因具有相似的外显子、内含子数目和保守基序,推测其结构域进化和基因的结构多样性相关。基因的染色体定位发现SPL基因并非出现在所有的染色体上。
进一步分析SPL基因的组织表达模式发现,在陆地棉中,Ghir_D08G004350基因可能参与调控花瓣的生长发育。Ghir_A01G005270基因可能与陆地棉的根和柱头的生长发育有关。本研究全面分析了陆地棉SPL的性质特征,表明其在棉花的发育过程中具有重要作用,可为SPL基因后续功能研究提供借鉴。

