新型冠状病毒3C样蛋白酶结构和功能特征分析
戴姿薇,唐 标
(湖南中医药大学医学院,湖南 长沙 410208)
2020年上半年,由新型冠状病毒(Severe acute respiratory syndrome coronavirus 2, SARS-CoV-2)引起的新型冠状病毒肺炎(Corona Virus Disease 2019, COVID-19)以数月时间在全世界引起了大流行,对全球居民健康构成了严重威胁。SARS-CoV-2是基因组为单股正链RNA且隶属于β冠状病毒组的新型冠状病毒类型,其结构蛋白主要由刺突蛋白(Spike Protein, S蛋白)、包膜糖蛋白、膜糖蛋白、核衣壳蛋白组成。在SARS-CoV-2入侵宿主时,表面S蛋白被跨膜丝氨酸蛋白酶2(transmembrane protease serines,TMPRSS2)剪切为S1亚基与S2亚基,S1亚基的受体结合区与宿主细胞表面血管紧张素转换酶2(Angiotensin-Converting Enzyme 2,ACE2)受体结合引起S2亚基构象变化,形成病毒与细胞膜融合物,从而释放病毒基因组至宿主细胞质中。此后病毒便利用宿主胞内物质合成自身复制所必需的两条复制酶多肽pp1a和pp1ab,同时也开始编码能精确剪切复制酶多肽的剪切酶:木瓜蛋白酶样蛋白酶(papain-like protease, PLpro)和3C样蛋白酶(3-chymotrypsin-like protease, 3CLpro)。PLpro和3CLpro剪切pp1a和pp1ab生成16个成熟非结构蛋白,非结构蛋白参与RNA复制酶-转录酶复合物(replicase-transcriptase complex, RTC)的形成,RTC形成后指导RNA复制和亚基因组RNA的转录,转录成功后,子代基因组与病毒结构蛋白组装为成熟的子代病毒,经胞吐作用释放后感染更多新的细胞[1-4]。已有研究揭示3CLpro是由两个彼此近似垂直排列的单体组成的二聚体,每个单体包含三个结构域:由β桶状结构组成的结构域Ⅰ与结构域Ⅱ,由α螺旋结构组成的结构域Ⅲ。其中,两个单体的N端通过与另一个单体上的Glu166位点相互作用来帮助定位底物结合位点S1亚基。目前在3CLpro氨基酸序列公布后,较多研究从已有序列信息结合其结构特点的角度筛选药物靶点:针对3CLpro进行临床试验的蛋白酶类抑制药物主要为洛皮纳韦和利托那韦,研究证实两者通过抑制CEP_C30的功能可阻止SARS-CoV-2复制周期的进行,以往分子动力学模拟分析也表明两者与SARS-CoV-3CLpro复合物结合亲和力相等,且在药物与复合物形成后分别可检测到6个与7个氢键;槲皮素作为一种中药类药物在毕赤酵母中对SARS-CoV-3CLpro复合物表达的抑制率可达82%,体外酶实验中也表现出对复合物的抑制活性[5-7]。3CLpro在病毒的复制中发挥了重要作用,抑制3CLpro功能能阻断SARS-CoV-2在宿主体内的复制,3CLpro成为了抗SARS-CoV-2药物的重要潜在靶点,但3CLpro的结构和功能特征还有待进一步阐明。本研究结合生物信息学方法,通过利用ProtParam、ProtScale、Bioedit服务器对3CLpro进行一级结构如氨基酸理化性质、疏水性的预测分析;利用COILS Server、SignalP、TMPred、TargetP Server、NetPhos Server、NetNGlyc Server服务器对3CLpro功能结构如卷曲螺旋区、信号肽、跨膜结构域、亚细胞定位、磷酸化位点、糖基化位点的预测分析;利用SOPMA、SWISS-MODEL服务器对3CLpro进行二级结构、三级结构的预测分析;利用IEBD对3CLpro进行B细胞表位的预测分析,为基于3CLpro的抗SARS-CoV-2药物研发提供参考。
1 材料与方法
1.1 材料
1.1.1 数据来源 登录美国国家生物信息中心NCBI网站(NCBI网址:http://www.ncbi.nlm.nih.gov/)获取SARS-CoV-2 3CLpro氨基酸序列信息,并下载其氨基酸FASTA序列(PDB ID:6LU7)。
1.1.2 主要数据库 通过ProtParam预测分析3CLpro等电点、半衰期、脂肪系数等理化性质;通过ProtScale和Bioedit双重预测分析3CLpro疏水性;通过COILS Server预测分析3CLpro卷曲螺旋区;通过SignalP预测分析3CLpro信号肽;通过TMPred预测分析3CLpro跨膜结构域;通过TargetP Server预测分析3CLpro亚细胞定位;通过NetPhos Server预测分析3CLpro磷酸化位点;通过NetNGlyc Server预测分析3CLpro糖基化位点;通过SOPMA预测分析3CLpro二级结构;通过SWISS-MODEL同源建模3CLpro三级结构;通过IEBD预测分析3CLproB细胞表位。
1.2 方法
1.2.1 3CLpro一级结构预测分析 通过ProtParam网页输入3CLproFASTA格式的氨基酸序列,点击“Compute parameters”运行结果,呈现3CLpro氨基酸数量、分子质量、理论等电点、正负电荷残基总数、分子式、原子总数、脂肪系数、总平均亲水性等理化性质结果;通过ProtScale网页上输入3CLproFASTA格式的氨基酸序列,选择“Hphob./Kyte & Doolittle”方法和“linear”权重变化模型,点击“Submit”运行结果,呈现3CLpro疏水性预测结果;同时运行Bioedit软件,导入序列后点击“Sequence-Protein”,选择“Kyte & Doolittle Mean Hydrophobicity Profile”方法,双重预测3CLpro疏水性结果[8-10]。
1.2.2 3CLpro结构预测分析 通过COILS Server网页选择默认矩阵MTIDK,分别赋予无权重与有权重选项,输入3CLproFASTA格式的氨基酸序列,呈现3CLpro卷曲螺旋区预测结果;通过SignalP网页上选择“Eukaryotes”训练集,D-cutoff values选择“Default”默认值,Method选择“Input sequences may include TM regions”,以“standard”模式输出,呈现3CLpro信号肽预测结果;通过TMPred网页上选择17~33个氨基酸作为跨膜螺旋疏水部分长度后输入3CLproFASTA格式的氨基酸序列,随即点击“Run TMPred”输出,呈现3CLpro跨膜结构域预测结果;通过TargetP Server网页上选择“non-plant”的生物类别,以“Long output”形式输出,呈现3CLpro亚细胞定位预测结果;通过NetPhos Server网页上选择“all three”的预测范围和“classic”的输出形式,勾选生成图形,呈现3CLpro磷酸化位点预测结果;通过NetNGlyc Server网页上输入3CLproFASTA格式的氨基酸序列,选择“Generate graphics”,点击“Submit”输出,呈现3CLpro糖基化位点预测结果[11-13]。
1.2.3 3CLpro高级结构预测分析 通过SOPMA网页输入3CLproFASTA格式的氨基酸序列,选择“4(Helix,Sheet,Turn,Coil)”选项,Similarity threshold填入“8”,点击“SUBMIT”输出,呈现3CLpro二级结构预测结果;于SWISS-MODEL网页上输入3CLproFASTA格式的氨基酸序列后选择“Build Model”选项同源建模,选择相似度最高模型输出,呈现3CLpro三级结构预测结果[14-16]。
1.2.4 3CLproB细胞抗原预测分析 通过IEBD网页选择“B Cell Epitope Prediction”后点击“Prediction of linear epitopes from protein sequence”输入3CLproFASTA格式的氨基酸序列,选择“Bepipred Linear Epitope Prediction”模式输出,呈现3CLproB细胞表位预测结果[17]。
2 结果与分析
2.1 3CLpro氨基酸理化性质特性分析
利用ProtParam服务器在线分析3CLpro氨基酸理化性质,结果如表1所示。3CLpro由306个氨基酸组成,其中亮氨酸(Leu)占比最高,其次为甘氨酸(Gly),没有吡咯赖氨酸(Pyl)和硒代胱氨酸(Sec);相对分子质量为33 796.64,理论等电点值为5.95;正电荷残基数为22个,负电荷残基数为26个;半衰期为1.9 h;不稳定系数为27.65;脂肪系数为82.12。

表1 3CLpro氨基酸理化性质部分分析结果
2.2 3CLpro氨基酸序列的疏水性分析
蛋白质肽链中残基侧链对溶剂的相对亲水性是一个重要的蛋白质特征参量,且疏水性的变化规律对维系蛋白质超二级结构与三级结构有一定的意义。利用ProtScale服务器在线分析3CLpro氨基酸疏水性,结果如图1所示。其中评分最高的为位于第206位的丙氨酸和第207位的色氨酸,均为1.823,表明该位点的氨基酸疏水性最强;评分最低的为位于96位的脯氨酸,为-1.731,表明该位点的氨基酸亲水性最强。亲水性区域一般是集中于蛋白质的卷曲结构部位,其表面通常富集亲水性氨基酸,同时也是蛋白质进化过程中氨基酸插入的主要位点。以上结果表明,3CLpro亲水性较高,且利用BioEdit双重预测,结果一致(图2)。
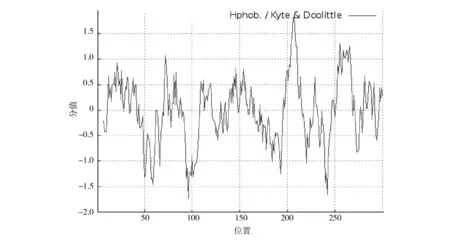
图1 ProtScale预测3CLpro疏水性Fig.1 Hydrophobic characteristics analysis results of 3CLpro via ProtScal

图2 BioEdit预测3CLpro疏水性Fig.2 Hydrophobic characteristics analysis results of 3CLpro via BioEdit
2.3 3CLpro卷曲螺旋区分析
卷曲螺旋是存在于多种天然蛋白质中一类由2股或2股以上右手α-螺旋相互缠绕而形成的平行或反平行左手超螺旋结构的总称。利用COIL Server服务器在线预测3CLpro卷曲螺旋结构。本研究在有权重和无权重的情况下进行分析得知,3CLpro并未预测到具有卷曲螺旋结构。
2.4 3CLpro信号肽分析
信号肽是指引导新合成的蛋白质向分泌通路转移的短肽肽链,位于分泌蛋白的N端。其由三个区组成:一个为带负电荷的C末端,一个为以中性氨基酸为主的中间疏水序列,一个为带正电的N末端。中间疏水序列为信号肽的主要功能区,C末端为信号序列的加工区。利用signalP服务器在线预测3CLpro信号肽区段,结果如图3所示。图3中C分值是指剪切位点分值,S分值表示信号肽分值,Y分值表示综合剪切点分值。本次预测结果显示,3CLpro信号肽预测得分较低,剪切位点得分也较低,因此3CLpro可能不具有信号肽的特点。
2.5 3CLpro跨膜结构域分析
跨膜结构域一般由跨膜蛋白组成的效应区域所展现,跨膜蛋白即跨越膜两端的蛋白质,跨膜部分形成的α螺旋或β桶结构对认识蛋白质在细胞中的定位与作用具有指示意义。利用TMPred预测3CLpro跨膜结构域,得出其有1个跨膜螺旋区,其位置在193~215位,结果如图4所示。
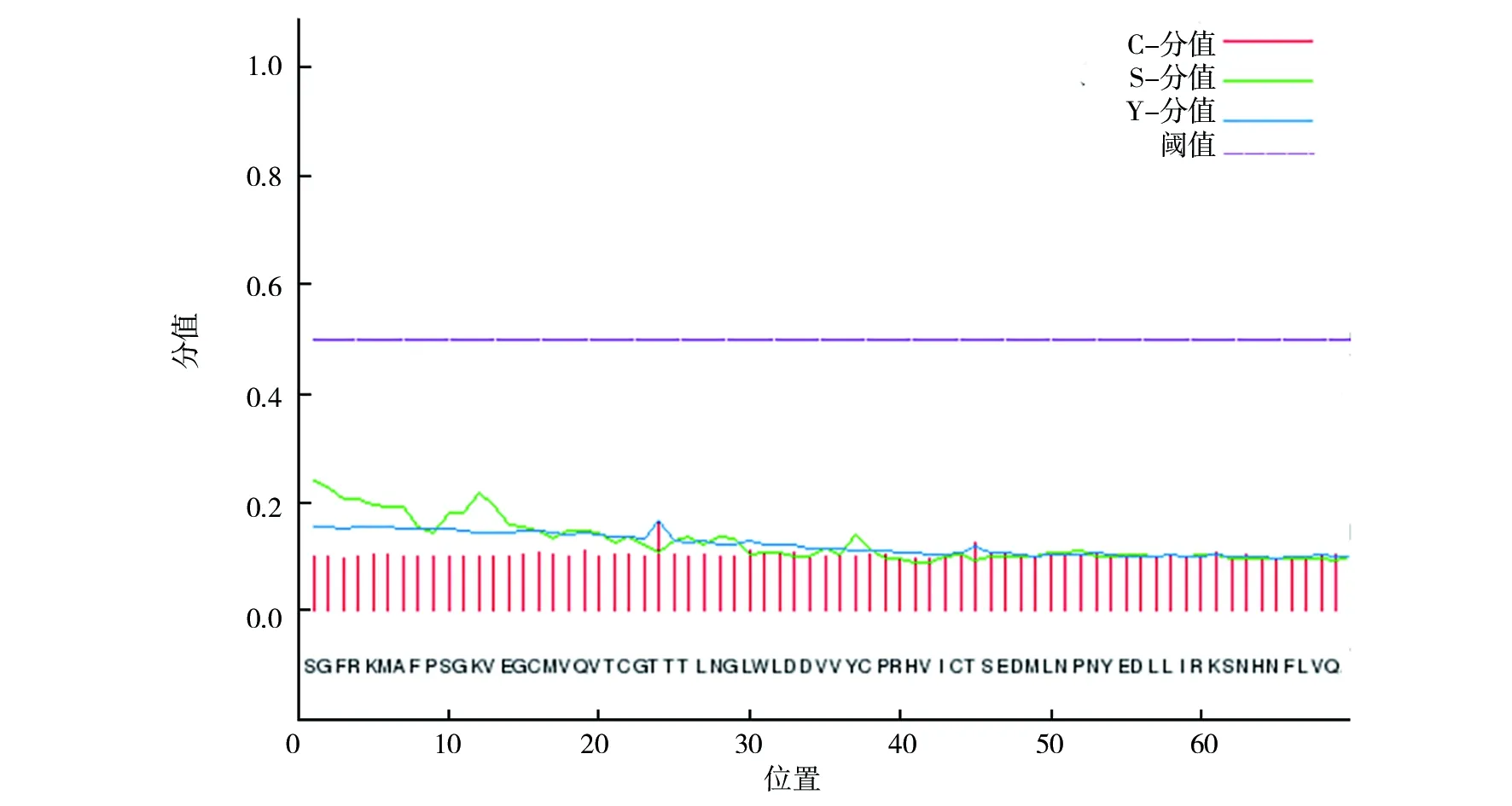
图3 SignalP预测3CLpro信号肽Fig.3 Signal peptide analysis results of 3CLpro via SignalP
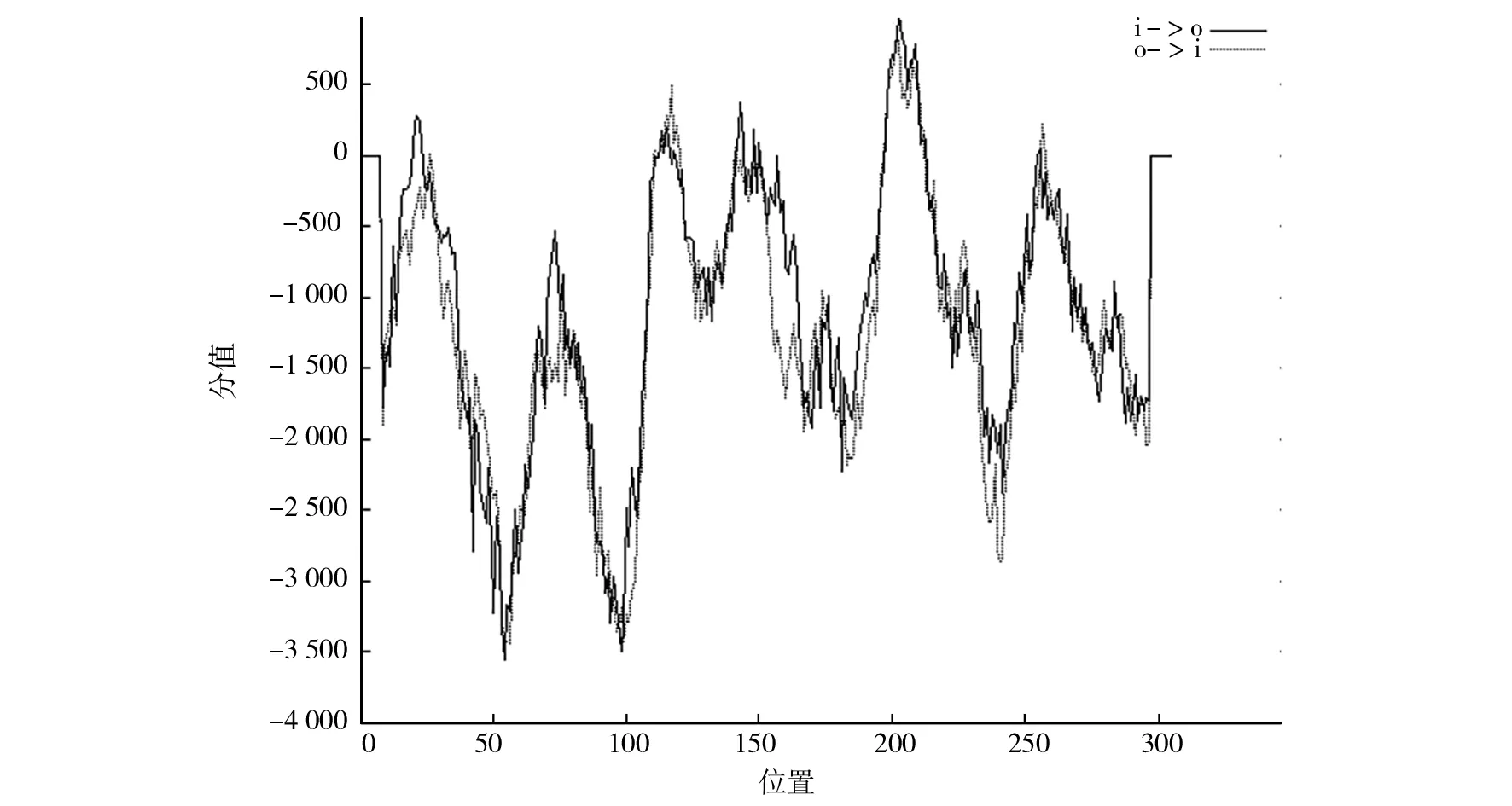
图4 TMPred预测3CLpro跨膜结构域Fig.4 Transmembrane domain analysis results of 3CLpro via SignalP
2.6 3CLpro亚细胞定位分析
亚细胞定位是指某种蛋白或其产物在细胞内的具体位置,由于蛋白质表面直接暴露于细胞器环境中,它由序列折叠过程决定,而后者取决于氨基酸组成。利用TargetP Server服务器在线预测3CLpro亚细胞定位,结果如表2所示。该序列指向定位于线粒体分值为0.199,指向具有分泌途径即存在信号肽可能性的分值为0.117,可靠性等级为3,因此推测其序列不是定位于线粒体,无信号肽。
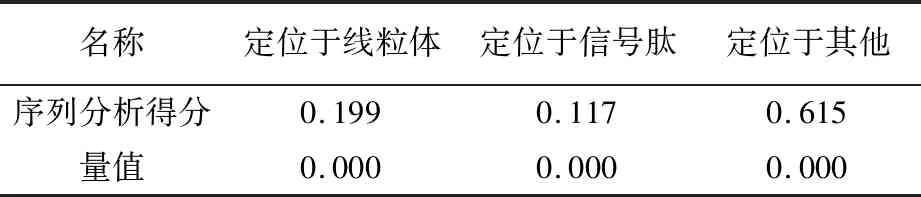
表2 TargetP Server预测3CLpro亚细胞定位
2.7 3CLpro磷酸化位点分析
蛋白质磷酸化是指蛋白质激酶将磷酸基团转移到底物蛋白质氨基酸残基(丝氨酸、苏氨酸、酪氨酸)的过程,是一种体内调节蛋白质功能和参与细胞信号转导的基本机制。利用NetPhos Server服务器在线预测3CLpro的磷酸化位点,结果如图5所示。分析其可能的1个丝氨酸磷酸化位点为18,1个苏氨酸磷酸化位点为7,2个酪氨酸磷酸化位点为19和23,以超过阈值0.5判定为潜在的磷酸化位点。

图5 NetPhos Server预测3CLpro磷酸化位点Fig.5 Phosphorylation sites analysis results of 3CLpro via NetPhos Server
2.8 3CLpro糖基化位点分析
蛋白质糖基化是指在糖基转移酶作用下将糖转移到蛋白质并与其氨基酸残基形成糖苷键的过程,对蛋白质具有重要的修饰与调节作用。利用NetNGlyc Server服务器在线预测3CLpro的糖基化位点,结果如图6所示。以0.5为阈值得出位于133位点与142位点有可能发生糖基化修饰。
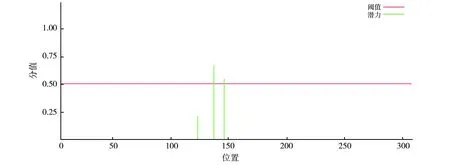
图6 NetNGlyc Server预测3CLpro糖基化位点Fig.6 Glycosylation sites analysis results of 3CLpro via NetNGlyc Server
2.9 3CLpro二级结构分析
蛋白质二级结构是指其主链骨架原子沿一定的轨迹盘旋或折叠成特定的构象,主要形式包括α-螺旋、β-折叠、β-转角和无规卷曲,并不涉及氨基酸残基侧链。利用SOPMA服务器在线预测3CLpro的二级结构,结果如图7所示。α-螺旋结构占比29.08%;β-折叠结构占比27.12%,β-转角结构占比11.44%,无规则卷曲结构占比32.35%。

图7 SOPMA预测3CLpro二级结构Fig.7 Secondary structure prediction of 3CLpro via SOPMA
2.10 3CLpro三级结构分析
蛋白质三级结构是指蛋白质在二级结构的基础上通过侧链基团相互作用进一步卷曲折叠,依靠分子内和分子间作用力维系形成的特定空间结构。利用SWISS-MODEL服务器在线预测3CLpro的三级结构,3CLpro蛋白酶的1~301位氨基酸与序列相似度为100%的6y2g.1(SMTL ID)能够进行同源建模,建模结果如图8所示。模型GMQE评分为0.99,说明模型质量较好;QMEAN评分为0.45,说明3CLpro与模板蛋白匹配度较高。匹配预测的寡聚态(Oligomeric state)为Homo-dimer。
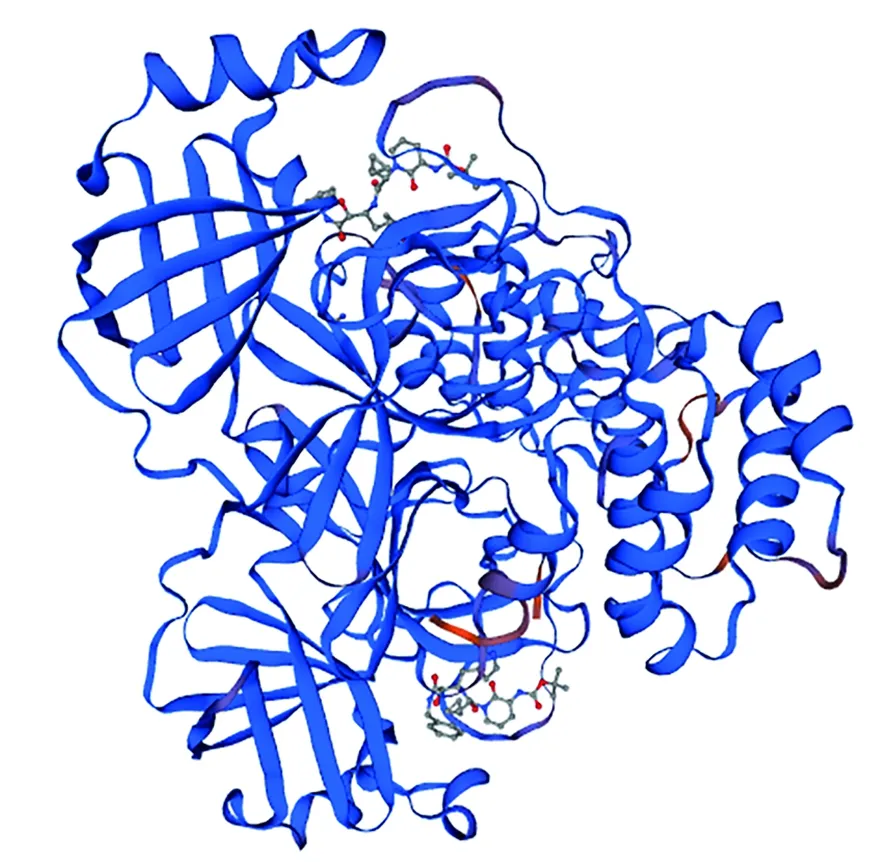
图8 SWISS-MODEL同源建模3CLpro三级结构Fig.8 Tertiary structure prediction of 3CLpro via SWISS-MODEL
2.11 3CLproB细胞表位预测
B细胞表位是指易于接近并被B细胞受体和抗体分子识别,由3~5个在空间中连续或不连续氨基酸组成的亲水性氨基酸区域。利用IEBD服务器在线预测3CLpro的B细胞表位,结果如图9所示。得分最高的为位于92~101位的氨基酸,分数为2.020;其次为186~199位的氨基酸、119~125位的氨基酸、170~180位的氨基酸,以0.350为阈值判定结果。
3 讨 论
本研究采用生物信息学的分析方法对3CLpro氨基酸的理化性质、疏水性、卷曲螺旋区、信号肽、跨膜结构域、亚细胞定位、磷酸化位点、糖基化位点、二级结构、三级结构、B细胞表位进行了预测与分析。
从其理化性质预测结果来看,3CLpro全长306个氨基酸,亮氨酸占比最高,理论等电点为5.95,有较高的亲水性。研究表明在冠状病毒3CLpro的水解位点上,P1位偏好谷氨酰胺,P2、P3、P4位分别偏好亮氨酸、碱性残基和小疏水残基,P30位并未表现出强烈偏好[18],冠状病毒入侵宿主的过程也是一种依赖pH环境和受体内吞介导的入侵方式,这与其特定的理化性质相关。因此3CLpro理化性质的阐明有利于对了解病毒入侵中3CLpro发挥水解剪切的具体作用机制提供帮助。

图9 IEBD预测3CLproB细胞表位Fig.9 B cell epitopes analysis results of 3CLpro via IEBD
从其信号肽与跨膜区预测结果来看,3CLpro属于非分泌蛋白,因此无需考虑该蛋白外泌问题[19]。从其磷酸化位点与糖基化位点预测结果来看,3CLpro具有明确的上述位点,推测其可能具有较高的生物活性。蛋白质在翻译后形成成熟蛋白质的过程中通常需要适当修饰,磷酸化与糖基化被认为是自然界最为重要的翻译后修饰形式[20]。
从其二级结构预测结果来看,α-螺旋与β-折叠化学键能较高,作为中心支架较难与抗体嵌合,但其占比也较高,提示3CLpro具有一定程度的稳定性,可为筛选蛋白提供靶点提供帮助;而3CLpro二级结构中占主导地位的无规则卷曲结构则常位于分子表面,相比之下更利于与抗体结合,其虽为无法被归入明确二级结构的多肽片段,但其具有一定的结构且受侧链相互作用影响,因此推测此类占比32.35%的非重复性无规则卷曲结构有可能会有效促进机体产生免疫反应并为3CLpro活性和功能区域提供构象支持。
从其三级结构预测结果来看,三级结构同源建模显示其配体为1xGLY与2xO6K。3CLpro三级结构中N-端部分氨基酸对其二聚化和活性位点的形成具有重要作用,同时位于结构域Ⅰ与Ⅱ间的氨基酸残基区域构成了底物的结合位点,针对3CLpro二聚化的肽基抑制剂组合设计成为了一种潜在的治疗策略。有文献指出[21],通过建立配体-蛋白质相互作用网络可得出SARS-CoV和SARS-CoV-2 3CLpro中的配体结合极为相似,并发现最有效的对接配体与芳香基团具有共同的结合模式——通过可旋转键以假线性形式连接,并得出结合能负值较大的生物碱和萜类化合物与3CLpro亲和力较高,这种结合可能是通过活性残基的硫酸盐阴离子的共价改变实现的,因此提示3CLpro配体结构和同源建模其三级结构为阻断SARS-CoV-2与ACE2受体结合后发挥生理功能与疫苗药物研究提供了潜在靶点。此外,抗原优势表位是临床应用与疫苗研制的重要依据。目前广泛运用的抗原表位鉴定方法通常为通过预测蛋白质抗原优势表位后通过体外合成进行验证其可靠性。本研究发现了四个可能性较高的表位区域,说明3CLpro可能具有较好的抗原性。除此之外,疫苗肽的研制还需要3CLpro免疫原性细胞毒性T细胞和辅助T细胞等信息,因此进一步确认3CLpro抗原表位细节更有利于疫苗靶点的确定。
目前人们在病毒学、流行病学、临床试验方面对SARS-CoV-2逐步有了更多的认识。然而,由于缺乏足够的理论依据,尚未有正式批准使用的抗SARS-CoV-2病毒的药物和疫苗,因此新型冠状病毒肺炎的治疗在现阶段主要依靠临床医生治疗经验进行。当前已有研究结合中国上海科技大学课题组测定发表的3CLpro高分率晶体结构预测药物靶点,大多从建立分子模型进行药物对接虚拟筛查的角度理解3CLpro的功能机制,如利用对应数据库进行不同抑制剂的结合亲和力对接计算后聚类排序再观察其之间的分子相互作用[22-23],但对于3CLpro基础理化性质与功能结构如疏水性、卷曲螺旋区、信号肽、跨膜结构域、亚细胞定位、磷酸化位点、糖基化位点等详细阐述较少。因此本文结合生物信息学方法较全面地分析3CLpro功能与结构特征,为抗SARS-CoV-2药物的相关研究提供参考。