基于粪便DNA技术的鸟类物种鉴定体系研究
秦 啼 王东权 曲宁新 刘帅齐 李 波,2* 柴洪亮
(1.东北林业大学野生动物与自然保护地学院,哈尔滨,150040;2.国家林业和草原局野生动植物检测中心,哈尔滨,150040)
利用粪便DNA技术对鸟类进行物种鉴定,对了解鸟类在繁殖地和越冬地的种类分布、研究其迁徙路线等具有重要意义[1]。鸟类粪便可以通过非损伤性采样技术获得,其DNA含量大,是分子生态学中广泛应用的检材[2-3]。物种鉴定中常用的分子标记有限制性片段长度多态(RFLP)、扩增片段限制性长度多态(PCR-RFLP)、扩增片段长度多态(AFLP)、随机扩增片段长度多态(RAPD)和物种特异性引物扩增[4]。近年来,DNA条码技术因原理简单、操作便捷等优点被广泛关注和使用[5-8]。基于鸟类粪便的物种鉴定体系通常如下:首先,从单一的粪便样本中提取总DNA,并扩增目标物种的Cytb基因、COI基因或D-loop片段等特定序列片段,而后测序;其次,将测得序列与已知的同源序列进行比对,即利用BLAST比对法确定来源物种[1]。由于鸟类中近缘物种DNA序列的高度相似性,时常导致不能得到单一物种的鉴定结论[9]。在哺乳动物、无脊椎动物的物种鉴定中,常用的序列分析方法还包括:遗传距离法、ABGD划分法及系统发育树法[10]。因此,本研究尝试在鸟类粪便DNA分析的基础上结合多种序列分析方法,构建一套完整的、标准明确的鸟类物种鉴定体系。
本研究以野外雁形目(Anseriformes)鸟类的粪便为对象,通过扩增、测定粪便DNA D-loop区序列,并与NCBI基因库中同源性序列进行比对分析。在此基础上,比较了常用序列分析方法的鉴定标准、分析了基于不同的碱基替换类型在计算遗传距离及构建系统发育树时的优劣性,以期得到高效、可信的鸟类物种鉴定体系。
1 材料与方法
1.1 材料
于青海湖附近采集76份雁形目鸟类的粪便,储存于国家林业与草原局动植物检测中心。
1.2 基因组DNA提取、PCR扩增与测序
利用粪便DNA基因组提取试剂盒(QIAamp DNA Stool Mini Kit,Germany)提取鸟类粪便基因组DNA。选取引物L438:5′-TCACGTGAAATCAGCAACCC-3′,H1248:5′-CATCTTCAGTGCCATGCTTT-3′[11]扩增mtDNA D-loop区部分片段。利用梯度PCR仪(Effendorf,Germany)筛选引物的最佳退火温度。PCR反应在9700型PCR仪(GeneAmp,USA)中完成。扩增反应体系(50 μl)是:2×EasyTaqPCR SuperMix 25 μl,10 μM引物各0.5 μl,DNA模板10 μl,加灭菌去离子水至50 μl。扩增程序为:94℃/3 min,(94℃/30 s,59℃/30 s,72℃30 s)35 cycles,72℃/10 min。PCR产物采用1%琼脂糖凝胶电泳检测,然后送至北京擎科生物有限公司哈尔滨分公司进行纯化回收和双向测序。
1.3 数据处理
利用DNAstar软件包的SeqMan对测得序列核对、拼接,删除两端引物得到目的片段。采用GenBank数据库 BLAST工具搜寻与粪便样品D-loop序列相似度高的同源序列。下载相似度98%以上的同源序列,涉及物种和登录号分别为灰雁(Anseranser)(EU601724、EU601725、EU601729、EU601732、EU601728);鸿雁(Ansercygnoid)(KT427472、KT427463、KJ124555、KJ778677);白额雁(Anseralbifrons)(EU601720、EU601721);小天鹅(Cygnuscolumbianus)(JQ282800、KF800698);大天鹅(Cygnuscygnus)(KR821135、KP981363);凤头潜鸭(Aythyafuligula)(JQ422475);针尾鸭(Anasacuta)(HM063478);琵嘴鸭(Spatulaclypeata)(HM063479);斑嘴鸭(Anaszonorhyncha)(KF751616);绿头鸭(Anasplatyrhynchos)(KJ778676);绿翅鸭(Anascrecca)(JX138088)(表1)。利用Mega 7.0中的Clustal W对测得和下载序列进行排列,并使用Gblock软件(http://www.phylogeny.fr/one_task. cgi?task_type=gblocks)选择保守区,参数设置为系统默认。采用DNAsp5分析单倍型及变异位点,利用Mega 7.0基于不同的碱基替换类型(transition,Ts、transversion,Tv、Ts+Tv)计算种内、种间的Kimura双参数(K2P)遗传距离。将上述序列K2P遗传距离矩阵提交到ABGD网站(https://bioinfo.mnhn.fr/abi/public/abgd/abgdweb.html),参数P(prior intraspecific divergence)设为0.005—0.01,参数X(minimum relative gap width)为1.0。以家鸡同源序列(AP003318、AP003317、AP003580)作为外群,基于Ts+Tv利用K2P模型采用邻接法(neighbor-joining,NJ)构建系统发育树,系统树分支的置信度用自展法(bootstrap analysis,BP)进行1 000次重复检验。然后比较系统发育树与ABGD划分结果。
表1 控制区单倍型 和Blast比对结果
2 结果
2.1 序列比对
测序获得雁形目鸟类粪便样本D-loop序列76条(683—757 bp),包含236个变异位点、其中简约信息位点209个,可划分为20个单倍型(表1)。与GenBank数据库中已知同源序列的相似度为98.07%—100%,涉及11种雁形目鸟类。其中,单倍型H1—H7、H13—H20检测为单一物种,而单倍型H8—H12检测为非单一物种、与已知灰雁、鸿雁同源序列都有着较高相似度(98.11%—99.60%)。
2.2 遗传距离、系统发育分析与ABGD的划分
利用Gblock选择的保守区长度为473 bp。基于Ts、Ts+Tv计算K2P种内和种间遗传距离(表2,表3),种内遗传距离分别为0—0.002、0—0.015,种间遗传距离分别为0.002—0.628、0.023—1.602,平均遗传距离为0.291、0.681。根据GenBank数据库相似度检测为单一物种的单倍型,其基于Ts+Tv计算的种间遗传距离明显大于基于Ts种间遗传距离。单倍型H16—H18与已知大天鹅和小天鹅的Ts遗传距离分别为0.002、0.003,而Ts+Tv遗传距离分别为0.037、0.004。前者难以作为物种鉴别依据;后者种间遗传距离与种内遗传距离差异明显,可以得到可信的鉴定结果。根据GenBank数据库相似度检测为非单一物种的单倍型也显示出类似的结果。单倍型H8—H12与已知灰雁和鸿雁的Ts遗传距离差异都极小(0 —0.001),而单倍型H8—H10的Ts+Tv遗传距离分别为0.023、0.015,单倍型H11—H12的为0.013、0.029。这表明单倍型H8—H10属于鸿雁,单倍型H11—H12属于灰雁。
基于D-loop单倍型构建的雁形目鸟类NJ系统发育树呈现出清晰的拓扑结构:包括外群的44条序列聚类后没有出现交叉、嵌套现象(图1)。待鉴别的单倍型分别与11种已知雁形目鸟类构成单系。利用ABGD软件以0.005—0.01的先验值P区间对包括外群的44个序列进行划分,结果包括了初始划分和递归划分两种情况(图2)。其中,初始划分较为稳定,包括外群的44个序列均被划分12个组,递归划分将样本划分成8—14组,波动较大。因此选择比较稳定的初始划分与系统发育树的聚类结果进行比较,表明系统发育树的聚类结果与每个分组呈逐一对应关系(图1)。
3 讨论
D-loop片段为线粒体DNA的非编码区,进化速率较快,适用于亲缘关系较近的物种鉴别[12-13]。本研究利用GenBank数据库BLAST工具分析了雁形目鸟类粪便D-loop序列单倍型,结果包括5个单倍型(25%)为非单一物种。它们与已知雁形目鸟类同源序列的相似度皆大于98%,高于95%的相似度鉴定标准[14]。由于分子标记的序列长度有限及近缘物种的序列相似性很高,使得部分结果为非单一物种、无法得出明确的鉴定结论。因此,BLAST比对法的序列分析结果对物种鉴定有一定的参考价值,但涉及近缘物种时需要结合其他分析方法得出鉴定结论。
表2 基于Ts计算的控制区单倍型K2P遗传距离

Tab.2 K2P-distances for haplotypes of D-loop computed based on Ts
表3 基于Ts+Tv计算的控制区单倍型K2P遗传距离

Tab.3 K2P-distances for haplotypes of D-loop computed based on Ts+Tv
物种鉴定中常用的遗传距离法是依据种间最小遗传距离大于种内最大遗传距离的原则,利用阈值法或最小距离法得到鉴别结果[15]。计算遗传距离时,需要考虑不同的碱基替代类型,包括Ts、Tv、Ts+Tv、Ts/Tv比率等[16-17]。对于特定序列,使用不同替换类型计算的遗传距离会有差异[18]。利用COI和Cytb基因鉴别雀形目(Passeriformes)鸟类时,Ts是最有效的碱基替代类型[18]。本研究比较Ts、Ts+Tv计算K2P种内和种间遗传距离,结果表明基于Ts+Tv计算遗传距离能更好地实现物种分辨。这种差异主要是源于分子标记的不同。COI和Cytb基因作为编码基因受到自然选择压力影响,经过长期的适应性进化后Tv常被自然选择所淘汰,而Ts则容易保留下来。D-loop区作为非编码基因不受自然选择压力影响,Ts和Tv保留的几率是相同的。因此,基于D-loop计算遗传距离时,Ts+Tv是最佳的碱基替换类型。而且,NJ系统发育树呈现出清晰的拓扑结构,待鉴别的单倍型与11种已知雁形目鸟类分别构成单系。这符合系统发育树法的判别标准:同种个体聚成单系分支[19]。另外,ABGD方法是基于遗传距离在先验值P的一定范围自动检测序列间的“barcoding gap”[20],某种程度上减低了阈值法或最小距离法界定的人为因素,提高了遗传距离法的可信度[21-22]。本研究中,ABGD划分结果与系统发育树的聚类结果完全吻合,因此,ABGD划分法可作为遗传距离法的补充或再处理。
综上所述,利用粪便DNA技术鉴别鸟类时,需根据物种间亲缘关系远近选择合适的分子标记进行扩增和测序;其次,在序列分析方法上,BLAST比对法作为最基本的序列分析方法,可以为物种鉴定提供参考。遗传距离法、ABGD划分法和系统发育树法可进一步检验BLAST比对结果,并鉴别亲缘关系较近的物种。以此为基础构建了一套标准明确的鸟类物种鉴定体系(图3)。相比以前单一的BLAST比对法,该鉴定体系集成遗传距离法和系统发育树法等多种序列分析方法,可以得到更加可信的鉴定结果。该体系使得鸟类物种鉴定体系更具系统性、规范性,可以推广应用于其他物种鉴定领域。
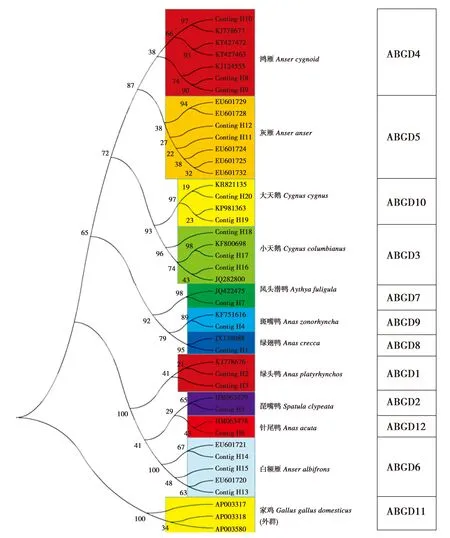
图1 基于控制区单倍型构建的雁形目鸟类邻接系统发育树Fig.1 Neighbor-Joining phylogenetic trees for Anserimormes birds based on haplotypes of D-loop注:左侧为NJ系统发育树,右侧为ABGD软件的初始划分结果Note:The Neighbor-Joining phylogenetic tree for Anseriformes birds is on the left.The results of initial partition by ABGD soft are on the right

图2 ABGD软件对雁形目鸟类的划分结果Fig.2 The automatic partition results of Anseriformes birds ABGD soft
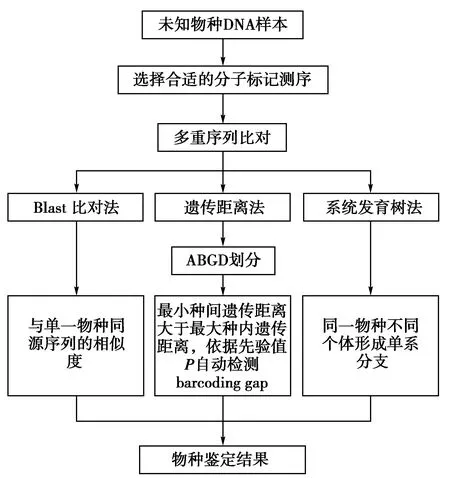
图3 物种鉴定体系Fig.3 The system of species identification

