基于多尺度多模式图像的肺结节分类对比研究
汤 宁,卫泽良,张 瑞,易 东,伍亚舟
陆军军医大学 军事预防医学系 军队卫生统计学教研室,重庆400038
1 引言
肺癌是全球癌症相关死亡的最主要原因[1]。2016年,美国癌症协会发布数据[2],早期肺癌的5年生存率是56%,而晚期肺癌的5年生存率仅有5%。因此肺癌的早期诊断是提高肺癌患者生存率的关键[3-4],通过低剂量CT筛查,可使高危人群肺癌死亡率降低14%~20%[5-6]。然而,对于目前的CT临床评估,主要是依赖放射科医师阅片,这是一个非常繁琐且缓慢的过程;另一方面,放射科医师的评估也缺乏一定的稳定性和客观性。因此,计算机辅助诊断(Computer-Aided Diagnosis,CAD)具有重要的临床意义,它能有效减少放射科医师的负担,增加临床评估效率;更重要的是,它可能发现一些人类肉眼无法识别的影像学特征[7]。
目前对于肺结节的自动分类主要包括两种方法:影像组学方法(Radiomics)和深度学习(Deep Learning,DL)方法。影像组学[8-9]主要分为以下几个步骤:首先,精确勾勒肺结节轮廓,提取感兴趣区域(Region of interest,ROI);对ROI提取体积、形状、纹理、密度等量化特征[10];然后对这些特征进行筛选、降维;再将这些特征输入诸如K邻近算法(K-Nearest Neighbor,K NN)、支持向量机(Support Vector Machine,SVM)、随机森林(Random Forests,RF)等机器学习分类器得到结节分类结果。影像组学方法是早期计算机辅助诊断的主要方法,El-Baz等人[11]通过分析肺结节的形状特征对其良恶性进行诊断;Chen等人[12]将肺结节的纹理、形状、强度、小波特征输入到一个SVM分类器来对肺结节进行分类。影像组学方法为计算机辅助诊断提供了一个思路,但是该方法需要精确分割感兴趣区域ROI之后[9],才能提取特征,而这个过程往往需要人工完成,极大地影响了该方法的效率和稳定性。
第二种方法是基于卷积神经网络(Convolutional Neural Network,CNN)的深度学习方法,CNN最大的特点是不需要对图像进行精准的分割,可以将图像直接输入模型,因此它又被认为是一种端到端(End to End)的模型。深度卷积神经网络最开始主要应用于自然图像识别领域,后来人们才开始将其应用于医学影像领域,并取得了一些成果,如Gulshan等人[13]将其应用于糖尿病视网膜病变的检测,Esteva等人[14]将其应用于皮肤癌的检测等,据报道,他们的自动化诊断系统已经到达甚至超越了人类医师的水准。近年来,许多针对肺结节分类的研究也都利用了深度卷积神经网络,Dey等人[15]用一个3D CNN对肺结节良恶性进行分类;Shen等人[16]提出了一个多尺度卷积神经网络(Multi-Scale Convolutional Neural Networks,MS-CNN),通过从不同尺度的肺结节图像中提取鉴别特征来捕获良恶结节之间的异质性;而后,他们又对该模型进行改造,提出MC-CNN[17](Multi-Crop Convolutional Neural Network),通过从卷积特征图中裁剪不同区域,以此来获取结节的主要信息;Causey等人[7]则将影像组学和深度学习方法联合,将结节的放射组学特征和CNN提取的高级特征融合后输入到一个RF分类器来对结节进行分类;Liu等人[18]提出一个多视图多尺度卷积神经网络(Multi-View Multi-Scale Convolutional Neural Network,MVMS-CNN),对肺结节的解剖学类型进行了分类。
目前的肺结节自动分类主要是基于卷积神经网络的深度学习方法,而这方面的绝大多数研究都只针对CNN模型进行改进或改造,很少有研究对深度学习的特征工程(feature engineering)进行探讨。尽管卷积神经网络可以从原始图像中提取有用的表示,但这并不意味着特征工程对深度学习模型不重要,良好的特征表示可以使模型用更少的资源更高效地解决问题[19]。
本研究针对该问题,对不同尺度不同模式肺结节图像对卷积神经网络模型分类性能的影响进行了探讨,并提出一种2D多视图融合(two Dimensional Multi-View Fusion,2D MVF)的肺结节表示方式,该方式在利用更多肺结节信息的同时又不会引入太多干扰性物质。利用肺癌CT数据集LIDC-IDRI[20-21](the Lung Image Database Consortium and Image Database Resource Initiative)和LUNA16[22](the LUng Nodule Analysis 2016)获取了三种不同尺度、四种不同模式的肺结节图像数据,构建了相应的卷积神经网络模型,并对它们的分类表现进行了比较。
2 方法
2.1 数据集及预处理
2.1.1 数据集
LIDC-IDRI数据集包含了1 010个病人的1 018套胸部CT扫描图像,由四名放射科医师独立地对三类病变进行标记,包括大于等于3 mm的结节、小于3 mm的结节以及非结节(无论大于还是小于3 mm)。对于非结节和小于3 mm的结节,四名放射科医师只需大致标记其中心坐标,而对于大于等于3 mm的结节,需标注出结节三维轮廓的坐标,且要对其影像学特征进行评估,其中恶性得分malignancy在1~5分之间,分值越高表示越有可能是恶性。由于一名患者的CT图像是由四名医师共同标注,所以同一个结节可能会被标注多次,因此同一个结节有可能产生最多四个不同的恶性得分。关于LIDC-IDRI数据集的更多细节可以参考文献[20-21,23]。
LUNA16数据集是LIDC-IDRI的子集。为了减少LIDC-IDRI图像的异质性,LUNA16舍弃了切片厚度大于等于3 mm和一些有缺失的CT扫描图像,最终从LIDC-IDRI的1 018套CT图像中筛选出了888套CT图像。由于在LIDC-IDRI中一个病灶可能会被标记多次,因此LUNA16将那些中心距离小于半径之和的标记进行了合并。并且,LUNA16只保留了由三或四名医师都标注了的直径大于等于3 mm结节,最终得到共计1 186个结节。关于LUNA16的其他细节可以参考文献[22]。
2.1.2 数据集预处理
利用LUNA16数据集和LIDC-IDRI数据集制作了三种不同尺度四种不同模式的肺结节图像数据,以此来探索不同尺度不同模式的图像对肺结节分类表现的影响。该过程主要包括肺结节图像数据制作和标签提取两个步骤。
2.1.2.1 制作多尺度多模式肺结节图像数据
(1)肺结节2D图像数据制作
2D图像模式即在结节中心坐标周围截取一张二维图片,这是大多数肺结节分类研究所采取的方式。LUNA16中提供了1 186个结节的中心坐标信息,但由于CT扫描图像分辨率各异,因此首先将它们重采样到1×1×1 mm3/voxel,然后根据结节中心坐标信息截取周围16×16、25×25、36×36三种不同像素的2D图像,如图1,该图展示了20个不同结节的2D图像在不同尺度下的视图,为了便于比较,已将其缩放到同一尺寸,红色箭头标示了某一个结节在三种不同尺度下的视图。

图1 三种尺度下的2D肺结节视图
(2)肺结节3D图像数据制作
同样的,根据LUNA16所提供的中心坐标信息截取周围16×16×16、25×25×25、36×36×36三种不同尺度3D图像,如图2。由于篇幅限制,图2仅展示了四个不同结节在不同尺度下的视图,红色箭头标示了同一结节在三种不同尺度下的视图。可以看到在较大尺度时结节周围有大量的血管组织,这会对结节的分类造成较大的干扰。
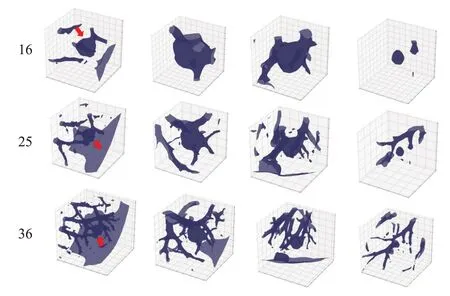
图2 三种尺度下的3D肺结节视图
(3)肺结节2D全视图融合图像数据制作
2D全视图融合(two Dimensional Full-View Fusion,2D FVF)图像就是将一个3D结节铺展开并拼接成2D图像。对于3D图像而言,可以将其视为2D图像的堆叠,将这多张2D图像拼接就可以得到2D FVF模式图像,如图3,该图展示了五个不同结节在三种不同尺度下的2D FVF视图。图3左侧16、25、36分别表示16×16×16、25×25×25、36×36×36三种不同尺度的3D结节铺展开的2D FVF图像。例如,对于16×16×16的3D图像,可以将其视为16张16×16的2D图像的堆叠,将这16张2D图像从上到下从左到右依次拼接,就得到一张64×64的2D FVF图像,如图3第一行所示。类似的,25、36尺度下的3D结节可以分别得到125×125、216×216像素的2D FVF图像。红色方框标示同一结节在三种不同尺度2D FVF视图中的结节部分,可以看到,在大尺度下结节的切片只占很小一部分,而大多数组织是一些血管、胸腔壁等组织。

图3 三种尺度下的2D FVF肺结节视图
(4)肺结节2D多视图融合图像数据制作
2D多视图融合图像(two Dimensional Multi-View Fusion,2D MVF)就是把3D结节最中间的4张2D切片拼接成一张更大的2D图像,这样做的目的是,这样可以比单纯的一张2D切片获取更多的结节信息,但同时又能比3D、2D FVF模式图像纳入更少的干扰性组织。制作方法和2D FVF类似,即利用3D结节图像,取其中间的四张切片,然后将其拼接。从16、25、36三种不同尺度3D结节分别可以得到32×32、50×50、72×72像素的2D MVF图像,如图4。该图展示了10个结节在三种不同尺度下的2D MVF视图,为了便于比较,图中已经将其缩放到了同一尺度。

图4 三种尺度下的2D MVF肺结节视图
对比不同尺度图像的差别,从图1~4中可以发现,尺度越大,结节周围的非结节组织越多,对结节分类造成的干扰就越大;对于不同模式图像而言,简单的2D模式图像浪费了大量CT扫描所提供的结节信息,而3D、2D FVF模式图像在获取较多结节信息同时又引入大量与结节无关的干扰组织,2D MVF模式图像则可以在两者间取得一个平衡。
2.1.2.2 标签数据制作
本研究将一个结节多个恶性评分的均值作为结节的标签。根据LUNA16提供的结节中心坐标、直径以及患者ID号,从LIDC-IDRI的标注文件中寻找该结节的恶性评分。由于同一个患者的CT扫描是由三或四名放射科医师共同进行标注,因此理论上可以为每个结节找到三或四个恶性评分。得到结节相应的恶性评分后,将这多个评分求均值后四舍五入取整作为其最终得分。由于LUNA16中的结节中心坐标是根据LIDC标注信息重新计算而来,因此会有少许的偏差,最终为1 183个结节找到了恶性评分。
最后,根据结节的恶性程度评分,将它们标记为M1、M2、M3、M4、M5共五组,不同组分别有88、266、499、282、48个结节,共计1 183个结节。考虑到数据的平衡性,设计了M12 vs M45的分类任务,即把恶性评分小于3分的记为“良性”,大于3分的记为“恶性”。这样每一种模式(四种模式)、每一种尺度(三种尺度)图像下都包含354例“良性”样本,330例“恶性”样本,共684例样本。
2.1.3 数据增强
数据增强(Data Augmentation)是一种有效防止模型过拟合的技术。一方面它可以增加模型泛化能力,另一方面可以为数据添加噪声,增加模型鲁棒性[24]。本次实验数据仅有684例样本,作为对比,数据库MNIST[25]拥有7万张手写数字图像,著名的VGG[26]、GoogleNet[27]等模型则是在百万级自然图像数据库ImageNet[28]上训练的。因此要训练一个性能良好的神经网络,庞大的数据是必要的支撑。数据增强是一种可以将数据量在一定程度上进行扩增的技术,主要方法包括对原始图像进行旋转、翻转、平移、添加噪声、裁剪等操作。
本研究所采取的数据增强方式不同于传统的静态的数据增强方式,即先将图像进行变换、保存后,再在模型训练时将其输入模型,本研究所采取的数据增强方式是一种实时的随机的增强方式,即在图像输入模型后,它会经过多个图像变换“关卡”。对于2D图像而言,这些“关卡”包括上下翻转,左右翻转,0°~360°的随机角度旋转,每种数据变换“关卡”都有50%概率发生。增强后的数据量与训练Epoch有关,理论上同一张图片在每一个Epoch都会不一样,本研究训练集原始数据有548例,验证集有136例,若训练Epoch设置为200,那么增强后训练集数据量为548×200=109 600,若训练Epoch设置为300,那么增强后训练集数据量为548×300=164 400。需要注意的是,增强后的数据集中存在较多相似性很强的样本,如对于旋转角度为90°和91°的样本,其差异其实不大。
而对于3D模式图像,只对其进行随机上下翻转、随机左右翻转以及三个轴方向上90°、180°、270°的随机旋转。由于3D模式图像旋转角度上的限制,其增强的倍数最多为2×2×23×3=96倍(上下翻转×左右翻转×三种不同角度旋转×三个轴方向),即增强后样本量理论上为548×96=52 608。对于验证集数据不作变换。
2.2 模型的构建
为了比较三种不同尺度、四种不同模式肺结节图像对分类性能的影响,搭建了不同的卷积神经网络模型。
2.2.1 2D CNN模型
针对16×16、25×25、36×36三种不同尺度的2D图像,构建了三个2D CNN,如图5。在Input(n×n)中,n×n表示输入图像的大小;Conv2D(n,m×m)代表一个卷积层,其中n表示卷积特征图的通道数,n×n表示卷积核的大小,所有卷积层的默认步长为1×1;Maxpooling(n,m)表示池化层,n表示池化核大小为n×n,m表示步长为m×m;Flatten()表示将特征图平铺为一个一维向量;Dropout(x)表示随机丢弃,是一种正则化技术,防止模型过拟合,x表示丢弃的比例;Softmax是一个分类器函数,它可以得出样本属于每一类别的概率。参数配置图右边Output表示数据向前传播时,经过每一层网络过后的张量维度,如(n×n,m)中,n×n表示特征图的大小,m表示特征图的通道数。
2.2.2 3D CNN模型
针对16×16×16、25×25×25、36×36×36三种不同尺度的3D图像,构建了3个3D CNN,如图6。在Input(n×n×n)中,n×n×n表示输入的3D结节的大小;Conv3D(n,m×m×m)代表一个卷积层,其中n表示卷积特征图的通道数,m×m×m表示卷积核的大小,所有卷积层的默认步长为1×1×1;Maxpooling(n,m)表示池化层,n表示池化核大小为n×n×n,m表示步长为m×m×m;Flatten()、Dropout(x)、Softmax的含义同2.2.1节所述。Output下面的式子同样表示数据向前传播时,经过每一层网络过后的张量维度,如(n×n×n,m)中,n×n×n表示特征图的大小,m表示特征图的通道数。

图5 2D CNN模型配置

图6 3D CNN模型配置
2.2.3 2D FVF-CNN模型
2D FVF-CNN模型的输入具有64×64、125×125、216×216三种尺度,因此也分别构建了三个不同的卷积神经网络,参数设置如图7,各个符号及数字的含义同2.2.1节所述。
2.2.4 2D MVF-CNN模型
2D MVF-CNN模型的输入同样具有三种不同的尺度,同样构建了三个不同的卷积神经网络,其参数设置如图8,其中的符号及数字含义同2.2.1节。
由于本研究的主要目的是探讨不同模式、不同尺度图像对肺结节分类表现的影响,因此未使用诸如VGG、GoogleNet等复杂模型,所构建的模型都是由卷积层和池化层交替连接而成的2D或3D卷积神经网络。各个模型之间最主要的区别是模型深度,针对不同尺度的图像,模型深度不同,图像尺寸越大,模型深度就越深;其他方面区别主要包括卷积层通道数、卷积核大小等,在不同模型调参过程中它们有略微调整。
2.3 模型的训练
构建好模型后,需要对其进行训练。所有的模型都对其进行5倍交叉验证(5-fold cross validation),为了减少结果的变异性,对每个模型都进行了5次5倍交叉验证,即最终模型的评估指标是由25次验证后取均值得到的。所有模型训练的Batch size设置为32,Epoch设置为200或300,使用的优化器为Rmsprop(Root Mean Square Prop),学习率都设置为0.001。模型的搭建使用Tensorflow版本的Keras,模型的训练是在NVIDIA GTX 2080 Ti显卡上运行。

图8 MVF-CNN模型配置
2.4 模型的评价指标
模型的评价指标采用了准确率(Accuracy,ACC)、敏感性(Sensitivity,SENS)、特异性(Specificity,SPEC)、ROC曲线下面积(Area Under the Curve,AUC)四个指标,相关计算公式如下所示:
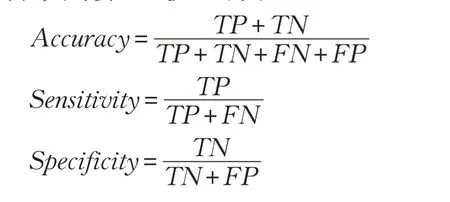
其中TP、TN、FP、FN含义如表1所示。

表1 二分类混淆矩阵
3 实验结果及分析
3.1 模型结果
通过对LIDC-IDRI和LUNA16数据集的预处理,获得了三种尺度四种不同模式的肺结节图像,图9展示了同一个肺结节在不同尺度及不同模式下的视图。针对不同尺度及不同类型的肺结节图像构建了不同的卷积神经网络模型,所有模型的训练结果如表2所示。

图9 三种尺度下的四类肺结节
3.2 三尺度四种模式下模型结果的两两比较
3.2.1 2D CNN与3D CNN模型结果比较
图10对比了2D CNN与3D CNN模型在不同尺度下的分类表现。2D和3D肺结节在不同尺度下的视图如图9所示。从图10可以看到,在16和25尺度下,2D CNN和3D CNN的分类表现差异不大,而在36尺度下时,3D CNN的分类表现明显低于2D CNN。尽管3D CNN能利用肺结节的全局上下文信息,但是在本次实验结果中可以发现,3D CNN的性能并不一定优于2D CNN。其原因可能是3D肺结节引入了较多的诸如血管、骨质等非结节组织,对肺结节的分类造成了干扰。对比不同尺度下2D CNN和3D CNN的分类表现,在16、25尺度下的分类表现明显优于在36尺度下的表现,其原因可能是因为在36尺度下的图像引入了更多的无关组织。

图10 2D CNN与3D CNN模型结果比较
3.2.2 2D CNN与FVF-CNN模型结果比较
图11对比了2D CNN和FVF-CNN模型的结果,2D和2D FVF模式肺结节图像在不同尺度下的视图如图9所示。
对比两种不同模式图像下的差异,从图11中可以看到,在16和25两种尺度下,2D CNN的分类表现略微优于FVF-CNN,而在36尺度下时,2D CNN的表现明显好于FVF-CNN。其原因与3D肺结节类似,2D FVF在获得更多肺结节信息的同时,也纳入更多的干扰物质,尤其是在截取较大尺度肺结节时。对比不同尺度下的分类表现,在16、25两种尺度下时,引入的干扰组织较少,此时2D CNN和FVF-CNN在两尺度下的分类表现差异不大,而在大尺度图像下时,干扰物质大量引入,造成分类性能明显下降,FVF模式的图像尤其明显。
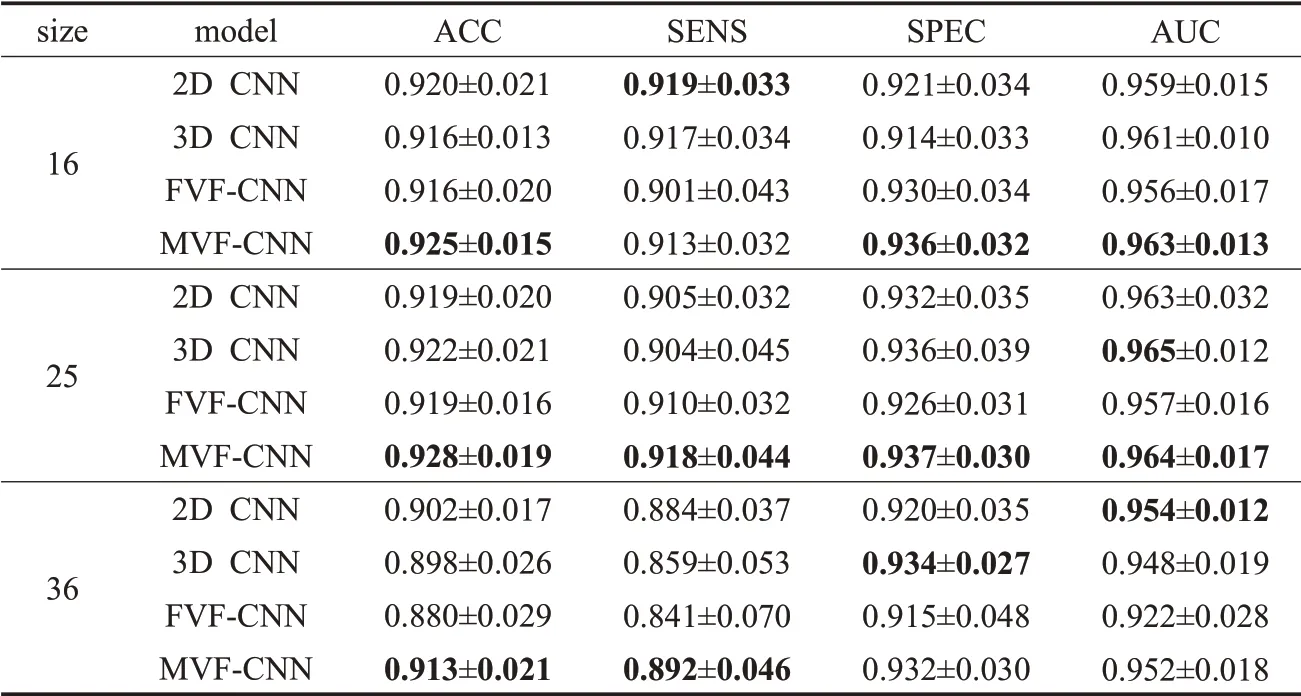
表2 三种尺度四种模式下所有模型的训练结果

图11 2D CNN与FVF-CNN模型结果比较
3.2.3 2D CNN与MVF-CNN模型结果比较
图12对比了2D CNN和MVF-CNN在不同尺度下的分类表现。对比不同模式图像下的分类表现,从图12可以看到,在三种尺度下MVF-CNN的性能都要优于2D CNN,这可能是因为2D MVF视图可以表示更多的结节信息,但又不会像3D或FVF模型图像那样引入大量的干扰组织。对比不同尺度下的分类表现,可以发现,在16和25尺度下两种模型的表现都要优于在36尺度下的分类表现,其原因也可能是因为在大尺度结节下引入更多干扰组织所致。

图12 2D CNN与MVF-CNN模型结果比较
3.2.4 3D CNN和FVF-CNN模型结果比较
图13比较了3D CNN和FVF-CNN的分类表现。对比两种模式图像下的分类表现,从图13可以看到,在16、25尺度下,3D CNN的分类表现略微优于FVF-CNN,在36尺度下,3D CNN的分类表现明显优于FVF-CNN。分析其原因,尽管两种模式的图像都尽可能展示了肺结节的信息,但是3D模式的图像可以捕获结节的空间上下文信息,这一点是2D FVF所不具备的,这可能是3D CNN的分类表现优于FVF-CNN的原因。对比不同尺度下的表现,可以看到在36尺度下,两种模型的表现都出现了下降。

图13 3D CNN与FVF-CNN模型结果比较
3.2.5 3D CNN和MVF-CNN模型结果比较

图14 3D CNN与MVF-CNN模型结果比较
图14比较了3D CNN和MVF-CNN在不同尺度下的分类表现。对比不同模式图像的分类表现差异,从图14中可以看到,在三种尺度下,MVF-CNN的分类表现都要优于3D CNN,在36尺度下两者差距尤为明显,其原因是3D模式结节图像比2D MVF图像含括更多的非结节组织,这可以从图9中直观地观察到,尤其在36尺度下时,这些干扰组织大量引入,使得其分类性能明显低于MVF-CNN。对比不同尺度下模型分类表现的差异,同样可以看到在36尺度下的分类表现要比在16、25尺度下较差。
3.2.6 FVF-CNN和MVF-CNN模型结果比较
图15比较了FVF-CNN和MVF-CNN的在不同尺度图像下的单分类表现。对比不同模式图像的差异,从图15中可以看到,在三种尺度下,FVF-CNN的各项指标均低于MVF-CNN,在36尺度时这种差异最大。其原因是2D FVF模式图像中大量的非结节组织对模型造成了干扰,而在36尺度下时这些干扰组织更多。对比两个模型在不同尺度下的表现,从图15中可以看到,仍然是在36尺度下两个模型的分类表现最差。

图15 FVF-CNN与MVF-CNN模型结果比较
3.3 肺结节图像的类激活图
从表2中可以发现,在36尺度2D FVF模式图像下的肺结分类表现最差,因此想利用类激活图[29](Class Activation Map,CAM)来观察这些误判的图像是如何影响模型判断的。类激活图是与特定输出类别相关的二维分数网格,它表示图像中每个位置对该类别的重要程度,具体实现方式参考文献[29]。图16展示了被错误判定为“恶性”(FP)及被正确判定为“恶性”(TP)的2D FVF原始图像及其类激活图,类激活图中越趋于红色的区域表示该区域对“恶性”的激活强度越高。从图16中可以观察到,容易被错误判定为“恶性”的结节(FP)主要是一些小结节,此时周围的组织容易对其造成干扰,而较大的结节不容易受这些组织的干扰,模型能准确对结节区域作出响应。因此要降低模型误判的概率,减少结节周围的干扰组织是一个关键,而不仅仅是针对深度学习模型进行改造。

图16 假阳性结节与真阳性结节的类激活图
3.4 与其他研究结果比较
为进一步验证本研究所提出的2D MVF的性能,将MVF-CNN与其他研究的模型进行了对比。这些研究使用的数据集都是LIDC-IDRI,且同样采用了M12 vs M45的分类策略,即把结节恶性评分小于3分的记为“良性”,大于3分的记为“恶性”,对比结果如表3。其中,Dhara等人[30]采用的是影像组学方法,他们通过分析结节的纹理和形状特征来鉴别良恶性结节;Xie等人[31]将结节的纹理、形状特征以及CNN提取的抽象特征进行融合来对肺结节进行分类;Shen等人[16]是利用卷积神经网络提取同一结节三种不同尺度的抽象特征,然后将这些特征融合后输入到一个SVM或者RF分类器来对结节进行分类;Han等人[32]则是通过分析结节的3D纹理特征,然后将其输入到一个SVM分类器来进行分类。从表3可以看到,在三种不同尺寸大小的结节下,MVFCNN模型都取得了非常有竞争力的表现。

表3 与其他肺结节分类研究结果比较
4 讨论
探讨了不同尺度不同模式肺结节图像对结节分类的影响,并提出了一种2D多视图融合的肺结节表示方式。通过对比研究发现,肺结节的呈现方式对模型有很大的影响,对于各种数据模式,对比三种不同尺度,36尺度下的分类表现最差,16、25尺度下的分类表现相当,其原因是在较大尺度时引入的干扰物质所致。对比不同模式图像的差异,无论在何种尺度下,2D MVF模式下的分类表现都最优,其原因是它比单纯地截取一张2D图像提供更多的肺结节信息,同时又不会像3D、2D FVF模式图像那样引入大量与肺结节无关的物质。对于2D、3D、2D FVF三种模式数据,在16、25尺度下时它们的分类表现相当,在36尺度下时三者差异较大,2D模式拥有最好的分类表现,其次是3D,最后是2D FVF,其原因是在大尺度下3D、2D FVF引入大量血管、骨质等干扰组织所致,而3D优于2D FVF是因为3D模式的数据可以表示肺结节空间上下文信息的能力。
5 结束语
当前,基于卷积神经网络的深度学习模型广泛应用于肺结节分类任务当中,但是绝大多数这方面的研究都关注于对模型的改进或改造,很少有研究对CT图像的处理方式进行探讨,Yan等人[33]也仅对2D和3D模式进行了比较。深度学习出现之前,特征工程显得非常重要,因为经典的浅层算法没有足够大的假设空间来学习有用的表示,因此将数据呈现给算法的方式对解决问题至关重要。而深度卷积神经网络出现后,看似大部分特征工程都是不需要的,因为它可以从原始图像中自动提取有用的特征,但这并不意味着特征工程不重要,因为良好的特征工程可以使模型用更少的数据更容易地学习到数据中的规律。
因此,本文没有对卷积神经网络模型的结构进行过多的探讨,而是重点针对不同尺度及不同模式的肺结节图像对模型分类结果的影响进行了深入分析,旨在说明在深度学习中,特征工程仍然具有其重要意义,良好的数据表示是模型学习其中规律的关键,而不仅仅关注模型的结构。

