茶尺蠖丝氨酸蛋白酶基因EoSP1的克隆、时空表达及对饥饿的表达响应
张新,陈成聪,杜琴,李喜旺,孙晓玲*
茶尺蠖丝氨酸蛋白酶基因的克隆、时空表达及对饥饿的表达响应
张新1,2,陈成聪3,杜琴1,2,李喜旺1,2,孙晓玲1,2*
1. 中国农业科学院茶叶研究所,浙江 杭州 310008;2. 农业农村部茶树生物学与资源利用重点实验室,浙江 杭州 310008;3. 国家茶叶质量安全工程技术研究中心,福建 泉州 362400
丝氨酸蛋白酶是鳞翅目昆虫消化系统中一类重要的蛋白酶。本研究从茶尺蠖()中克隆到一条丝氨酸蛋白酶基因并分析了其结构特征和表达特性。基因序列全长858 bp,编码285个氨基酸,预测蛋白分子量为29.53 kDa,等电点为5.44。经与其他丝氨酸蛋白酶比对,EoSP1含有保守的丝氨酸蛋白酶催化位点(H95,A161和S328)及蛋白互作结构域,与蓓带夜蛾()中SPs亲缘关系较近。进一步获得了与EoSP1-GST融合蛋白大小接近的目的蛋白。qRT-PCR分析发现,在幼虫期的表达显著高于成虫、蛹和卵期,并在幼虫中肠中特异性表达;饥饿处理后表达下降,恢复饲喂后表达量接近对照组。以上结果为茶尺蠖消化酶功能解析及抗虫新靶点的筛选提供了依据。
茶尺蠖;丝氨酸蛋白酶;克隆;表达分析
丝氨酸蛋白酶(Serine protease,SP)是一类以丝氨酸为活性中心的蛋白水解酶家族,催化结构域由组氨酸(His)、天冬氨酸(Asp)和丝氨酸(Ser)3个氨基酸残基组成[1-2]。根据蛋白结构域的不同,SP可分为单结构域和多结构域蛋白,单结构域蛋白主要发挥蛋白消化功能,而多结构域蛋白常作用于各种生物过程中的分子识别[3-4]。根据酶切位点的不同,SP又可主要分为胰蛋白酶(Trypsin)、胰凝乳蛋白酶(Chymotrypsin)、弹性蛋白酶(Elastase)及其他酶类等[5-6]。其中,胰蛋白酶除包含His-Asp-Ser保守活性三联体外,在蛋白序列的N端还含有保守氨基酸残基IVGG,可水解多肽链中赖氨酸和精氨酸残基中的羧基侧,是昆虫体内含量最丰富的消化酶[7];胰凝乳蛋白酶丰度仅次于胰蛋白酶,包含催化三联体及胰凝乳蛋白酶所特有的结合口袋氨基酸残基,可专一切割酪氨酸、色氨酸、苯丙氨酸等芳香族氨基酸形成的羧基端肽键[5]。目前,在动物、植物和微生物等中均发现SP参与消化、先天免疫和发育等多种生理活动[2,8-9]。
SP是鳞翅目昆虫消化系统中最重要的消化酶,占总消化活性的95%以上[2,10-12],由基因编码。目前,的cDNA序列已经从烟草天蛾()、棉铃虫()、斜纹夜蛾()、小菜蛾()、家蚕()和美国白蛾()等多种鳞翅目昆虫中克隆获得,并发现它们编码的蛋白主要为胰蛋白酶和胰凝乳蛋白酶[13-14]。大量研究结果表明,在鳞翅目昆虫体内具有组织表达特异性,其编码的蛋白参与昆虫对营养物质的消化与吸收、免疫以及对饥饿的耐受性等多个生理过程。例如,烟草夜蛾、斜纹夜蛾和家蚕的基因在中肠中特异表达,饥饿胁迫后会引起部分基因表达下调[15-17];美国白蛾在取食量较大的时期表达量显著高于取食量较小的时期,并且取食喜树后的表达量高于其他寄主植物[18];家蚕在病原微生物BmNPV入侵过程中参与免疫应答[19-20];东方粘虫()和主要表达于中肠,饥饿胁迫后二者表达均受抑制,但受蜕皮激素诱导上调,则主要响应苏云金芽孢杆菌的刺激[21]。有研究发现,沉默的大豆二化螟()幼虫体重增长显著变慢,死亡率显著升高[22]。类似地,植物源丝氨酸蛋白酶抑制剂通过与昆虫体内的SP发生反应使其失活,从而显著阻滞幼虫的生长发育,甚至降低成虫的繁殖能力[12, 23]。因此,作为潜在的防控靶标基因已经引起科研工作者的广泛关注。
茶尺蠖()属鳞翅目(Lepidoptera)尺蠖蛾科(Geometridae),是危害茶树()的主要害虫之一,主要以幼虫取食茶树叶片,发生严重时可造成树势衰败及茶叶绝收[24-25]。目前,对其有效的防治仍依赖于化学农药。然而,化学农药的大量使用导致了农药残留、害虫抗药性以及次期性害虫大发生等诸多问题[26]。因此,开发茶尺蠖的绿色防控技术已成为茶树植保工作的迫切需求。通过对茶尺蠖体内重要消化酶的研究将有助于理解茶尺蠖的营养利用机制,可以为茶尺蠖绿色防控技术的开发提供理论基础。遗憾的是,目前尚无茶尺蠖幼虫基因的相关报道。鉴于此,本研究从茶尺蠖幼虫的转录组数据库中获得基因片段的转录本序列,利用RACE技术对其全长进行克隆,并利用原核表达方法确定克隆到的基因能够编码大小一致的蛋白,继而采用荧光定量PCR(qRT-PCR)技术对在茶尺蠖不同发育时期、不同组织部位,以及饥饿和回补食物后的表达量变化情况进行了研究,拟为该基因的利用提供理论基础。
1 材料和方法
1.1 供试昆虫
供试的茶尺蠖幼虫采自浙江省杭州市西湖区中国农业科学院茶叶研究所试验茶园(N 30°10',E 120°5'),以新鲜茶树嫩梢饲养于人工气候箱内[(26±1)℃,(70±5)%,14L∶10D]。繁殖两代后用于试验。各处理方法如下:
1.1.1 不同发育时期
分别收集产卵后1 d的卵(200粒)、孵化第2天的1龄幼虫(200头)、蜕皮第2天的2龄幼虫(50头);蜕皮第2天的3龄幼虫(5头);蜕皮第2天的4、5龄幼虫(1头)、化蛹第2天的雌蛹和雄蛹(1个)、羽化第2天的雌、雄成虫(1头),液氮冷冻后于–80℃冰箱保存。每个样品8个生物学重复。
1.1.2 不同组织部位
实体显微镜(OLIMPUS)下解剖茶尺蠖4龄幼虫,分别采集80头茶尺蠖幼虫的中肠、唾液腺、脂肪体、表皮和血淋巴,液氮冷冻后于–80℃冰箱保存备用。每类组织样品取8个生物学重复。
1.1.3 饥饿处理
中肠组织:对照组为饲喂茶树叶片的4龄茶尺蠖幼虫,饥饿处理组同为4龄茶尺蠖幼虫但不饲叶片,饥饿24 h。饥饿24 h后再饲喂茶树叶片24 h的4龄幼虫为先饥饿-再饲喂组。每个处理设3个生物学重复。
虫体:对照组为饲喂茶树叶片的4龄茶尺蠖幼虫,饥饿处理组同为4龄茶尺蠖幼虫但不饲以叶片。分别于处理后的3、6、12、24 h和48 h后取样;饥饿24 h后再饲喂茶树叶片24 h的4龄幼虫为先饥饿-再饲喂组。每个处理设8个生物学重复。
1.2 RNA提取
参照Promega总RNA提取试剂盒说明书,对样品进行总RNA提取。利用琼脂糖凝胶电泳检测RNA质量,并使用Nano Drop ND-2000微量核酸蛋白测定仪(Thermo,美国)测定浓度。参照Prime Script RT Reagent(Takara,日本)反转录试剂盒说明书,取1 μg总RNA进行cDNA合成,用于后续基因克隆和表达分析。
1.3 EoSP1基因的克隆
从本实验室茶尺蠖幼虫转录组数据库中得到1个SP基因片段的转录本序列,根据此序列分别设计特异性RACE引物(表1),以茶尺蠖幼虫cDNA为模板,采用高保真酶PrimeSTAR®Max DNA Polymerase(TAKARA,大连)进行PCR扩增,获取基因全序列。PCR反应体系为:cDNA 1.0 µL、Max 25 µL、上下游引物各1.5 µL、ddH2O 21 µL。反应程序为:94℃预变性3 min;98℃变性10 s,55℃退火30 s,72℃延伸30 s,35个循环;72℃延伸10 min,4℃保存。采用1%琼脂糖凝胶检测RCR产物,将单一目标条带切胶回收,使用pClone007 Blunt Vector Kit(TSINGKE,北京)用于目的片段与克隆载体的构建,转化DH5α大肠杆菌,挑选单克隆进行菌液PCR,阳性克隆送测,引物合成和测序均由北京擎科生物科技有限公司(杭州)完成。
1.4 生物信息学分析
通过Softberry在线软件(http://softberry.com)的FGENESH功能对全长进行开放阅读框(Open reading frame,ORF)及氨基酸序列分析;使用ProtParam(https://web.expasy.org/protparam)在线预测EoSP1的氨基酸组成、分子量和等电点特征;通过PSIPRED(http://bioinf.cs.ucl.ac.uk/introduction)分析EoSP1的蛋白结构;在NCBI(https://www. ncbi.nlm.nih.gov)数据库中下载已登录其他昆虫的丝氨酸蛋白酶氨基酸序列,采用MEGA5.0软件中的邻接法(Neighbor-Joining tree)构建系统发育进化树。
1.5 原核表达载体构建及蛋白纯化
利用EoSP1-pGEX-F和EoSP1-pGEX-R一对引物对(表1)扩增获得包含BamH Ⅰ和Sma Ⅰ多克隆酶切位点的全长序列,通过酶切连接到表达载体上,连接产物转化大肠杆菌DH5α感受态细胞,筛选阳性克隆。将经测序验证的重组质粒转入大肠杆菌BL21(Rosetta)菌株中,挑选阳性克隆接种于液体LB培养基中,培养至OD值达到0.4~0.6,加入终浓度1 mmol·L-1IPTG诱导重组质粒表达,于37℃,200 r·min-1继续培养2 h,收集菌体。分别收集诱导前后各1 mL菌液,将菌体重悬于100 μL SDS上样缓冲液中,煮沸5 min变性,室温12 000 r·min-1离心5 min后,取25 μL上清液进行SDS-PAGE(10%浓度)检测蛋白表达情况。
将上述条件扩大培养至100 mL LB菌液,将诱导后的菌液于4℃下,转速12 000 r·min-1离心10 min收集至50 mL离心管中,加入15 mL 0.1 mol·L-1的Tris-HCl(pH=8.0)缓冲液重悬菌体。对重悬菌体进行超声破碎(超声5 s,停5 s,共15 min),结束后于4℃,12 000 r·min-1离心30 min。参考GST标签蛋白纯化试剂盒(GST-tag Protein Purification Kit,碧云天,上海)进行蛋白纯化,利用梯度透析法对纯化后的蛋白进行复性,对复性后的重组蛋白进行SDS-PAGE鉴定。
1.6 定量分析
1.7 数据分析
使用SPSS Statistics 20对结果进行统计分析。不同发育时期、不同组织部位以及饥饿后再饲喂样品中相对表达量的差异显著性采用单因素方差分析法(Tukey’s多重检验)进行比较;饥饿与对照组茶尺蠖样品中相对表达量的差异显著性采用Student'stest进行分析。
2 结果与分析
2.1 EoSP1全长克隆及蛋白结构预测
基因ORF全长为858 bp,编码285个氨基酸残基(图1);利用Protparam对EoSP1氨基酸序列的理化性质进行在线预测,结果表明,EoSP1推导氨基酸残基数为285,分子量为29.53 kDa,理论等电点pI为5.44,蛋白质不稳定指数为36.98,脂溶指数为91.68,平均疏水性指数为0.214。PSIPRED Server预测结果表明,EoSP1由6个α螺旋、16个β折叠和一些无规则卷曲构成。此外,发现EoSP1的N末端疏水区包含从第1位开始的18个氨基酸组成的信号肽,预测酶切位点为18、19位,表明EoSP1是分泌型蛋白(图2)。

表1 引物信息

图1 EoSP1核苷酸序列及其预测氨基酸序列
2.2 EoSP1蛋白结构及进化树分析
将EoSP1同其他已知昆虫的丝氨酸蛋白酶进行蛋白序列比对,发现EoSP1含有丝氨酸蛋白酶的3个保守催化活性位点(H95,A161,S328)。活化相关元件包含RIVGG,TAAHC和GDSGSAL序列,以及3对二硫键的半胱氨酸残基等也是保守存在于EoSP1中(图3)。进一步保守结构域分析发现EoSP1属于类胰蛋白酶类(Trypsin-like)的丝氨酸蛋白酶。
与其他已知昆虫丝氨酸蛋白酶序列进行进化树分析,结果表明,EoSP1与蓓带夜蛾和小地老虎()的丝氨酸蛋白酶家族成员亲缘关系较近,而与大红斑蝶()亲缘关系最远(图4)。
2.3 EoSP1原核表达及纯化
在37℃,0.1 mmol·L-1IPTG诱导条件下促使融合载体表达。经SDS-PAGE检测发现,与对照相比,诱导后泳道中有1条单一明显、分子质量略高于50 kDa的蛋白条带(图5,泳道2)。EoSP1蛋白分子量预测为29.53 kDa,融合27 kDa GST标签后约为57 kDa,诱导条带大小接近重组蛋白的预测分子量。超声后发现EoSP1-GST重组蛋白以包涵体形式存在(图5,泳道3—4),通过对沉淀中的包涵体进行蛋白质变性、复性,最终纯化到目的蛋白(图5,泳道5)。由此证明,所编码的蛋白与EoSP1蛋白的预测分子量相当。

图2 EoSP1蛋白结构预测
2.4 EoSP1在茶尺蠖不同发育时期的表达特征
在整个茶尺蠖生活史中,在1~4龄茶尺蠖幼虫中的相对表达量均显著高于5龄幼虫、蛹期、成虫期和卵期,2龄幼虫的相对表达量是卵期的2 600倍以上;在幼虫期,1~4龄幼虫体内的相对表达量显著高于5龄;在不同虫态中,预蛹中的表达量显著高于蛹期、成虫和卵等其他虫态(图6)。
2.5 EoSP1在不同组织中的表达特征
在中肠、脂肪体、唾液腺和血淋巴中均有表达,在中肠中的相对表达量显著高于其他4个组织;而在表皮、脂肪体、唾液腺和血淋巴中的表达量间不存在显著差异(图7)。
2.6 饥饿胁迫下EoSP1的表达响应
与对照相比,饥饿24 h幼虫中肠内的相对表达显著降低,恢复饲喂后中肠内的相对表达量与对照之间不存在显著差异(图8-A)。
饥饿12 h,处理组幼虫体内的相对表达量显著高于对照;饥饿24 h,处理组幼虫体内的相对表达量是对照组的2.7倍;饥饿48 h,饥饿处理的幼虫体内的相对表达量是对照的2.3倍,恢复饲喂组茶尺蠖幼虫体内的相对表达量与对照组幼虫体内的相对表达量基本相当,但是显著低于饥饿处理48 h茶尺蠖幼虫体内的相对表达量(图8-B)。
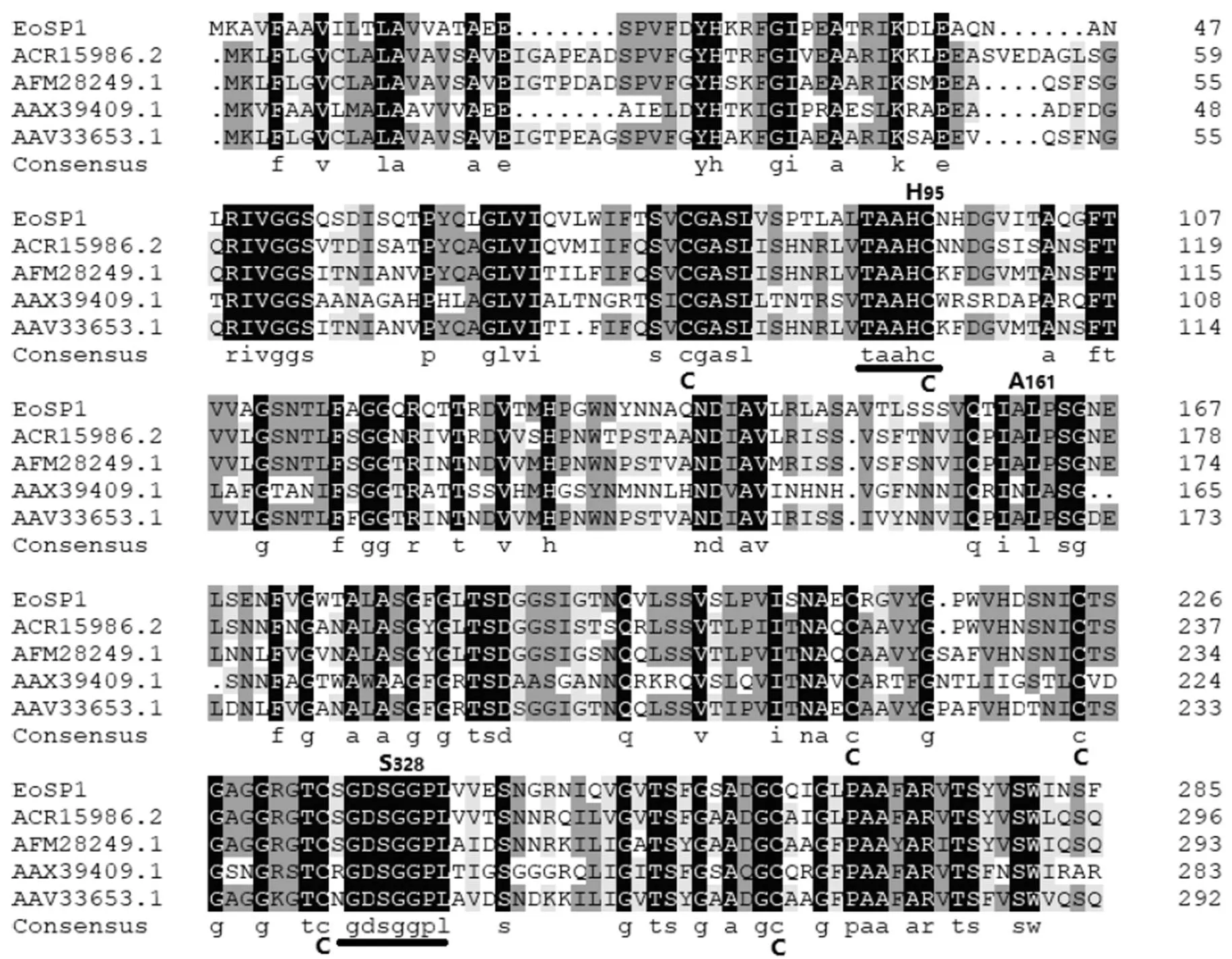
注:ACR15986.2:蓓带夜蛾;AFM28249.1:烟草夜蛾;AAX39409.1:家蚕;AAV33653.1:斑实夜蛾
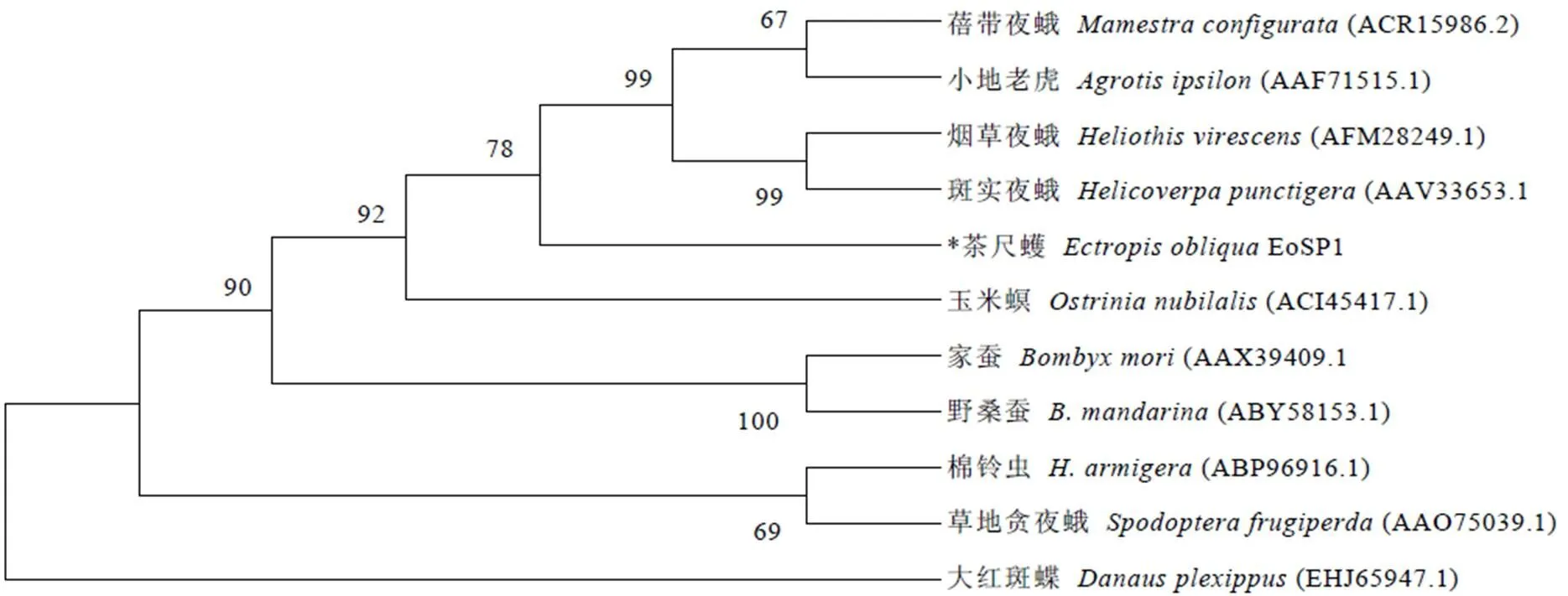
图4 EoSP1与其他昆虫SPs系统进化树分析
注:M:蛋白分子量标记;1:对照;2:IPTG诱导后;3:超声破碎上清;4:超声破碎沉淀;5:纯化目的蛋白
Note: M: Protein Marker. 1: Control. 2: Induced by IPTG. 3: Supernatant after sonification. 4: Sediment after sonification. 5: Purified protein
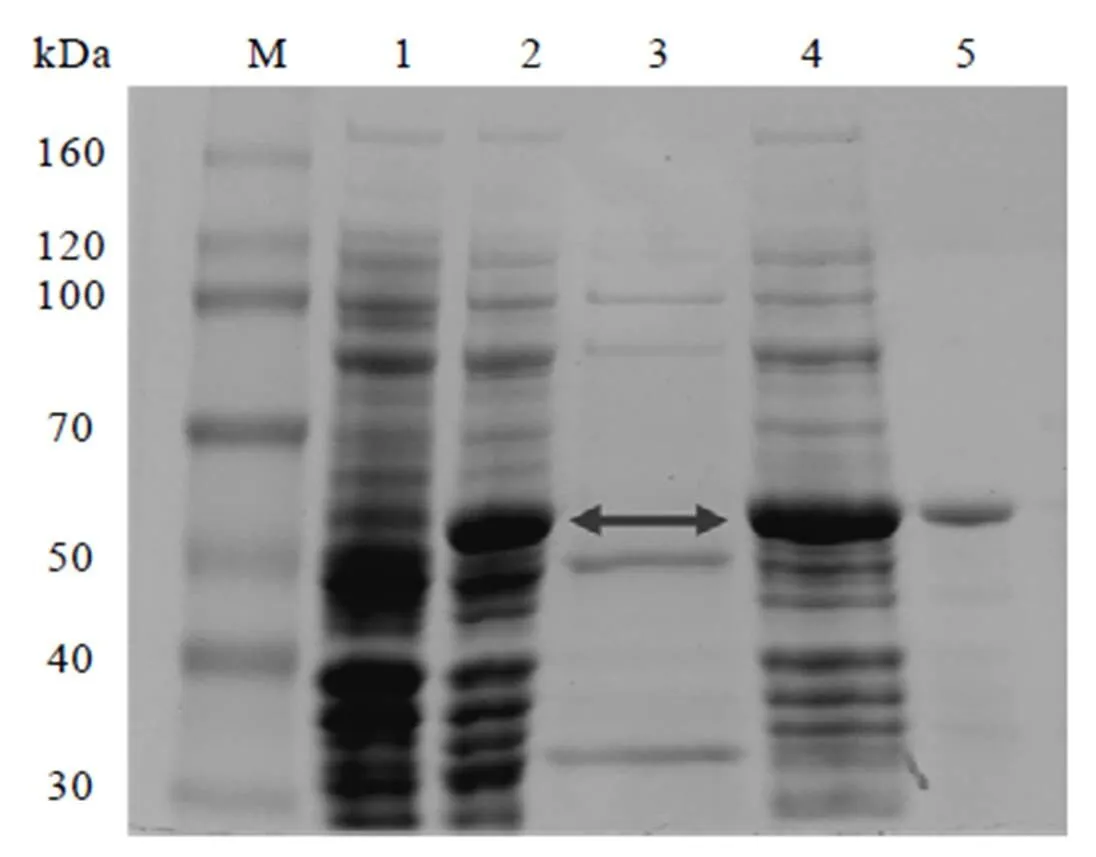
注:M:蛋白分子量标记;1:对照;2:IPTG 诱导后;3:超声破碎上清;4:超声破碎沉淀;5:纯化目的蛋白

注:数据为平均值±标准误,单因素方差分析(Tukey’s多重检验),不同大写字母代表EoSP1的相对表达量在茶尺蠖生活史的不同时期具有显著性差异(P<0.05);不同小写字母代表EoSP1的相对表达量在幼虫不同龄期(虚线左侧)或不同虫态之间(虚线右侧)具有显著性差异(P<0.05)
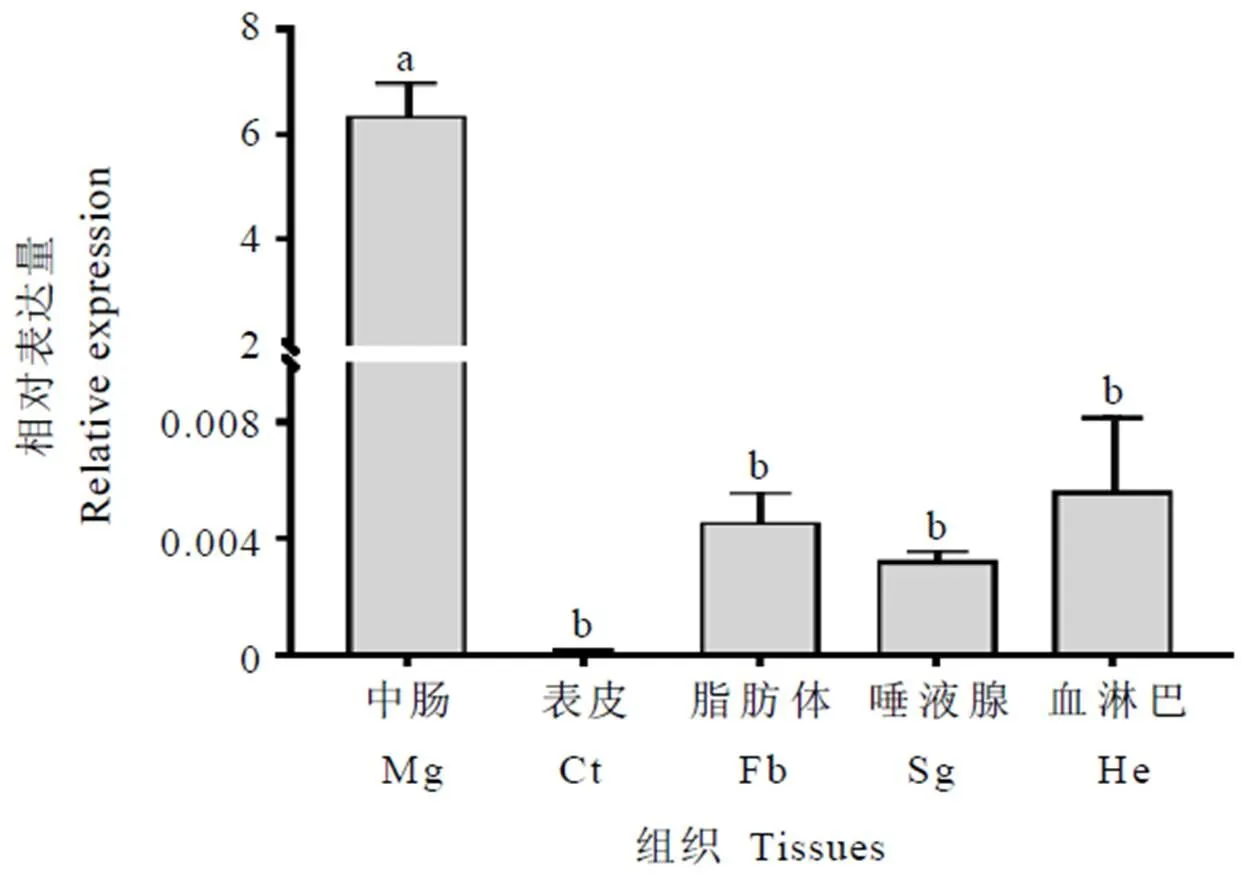
注:不同小写字母表示不同处理之间差异具有显著性(Tukey’s多重检验,P<0.05)
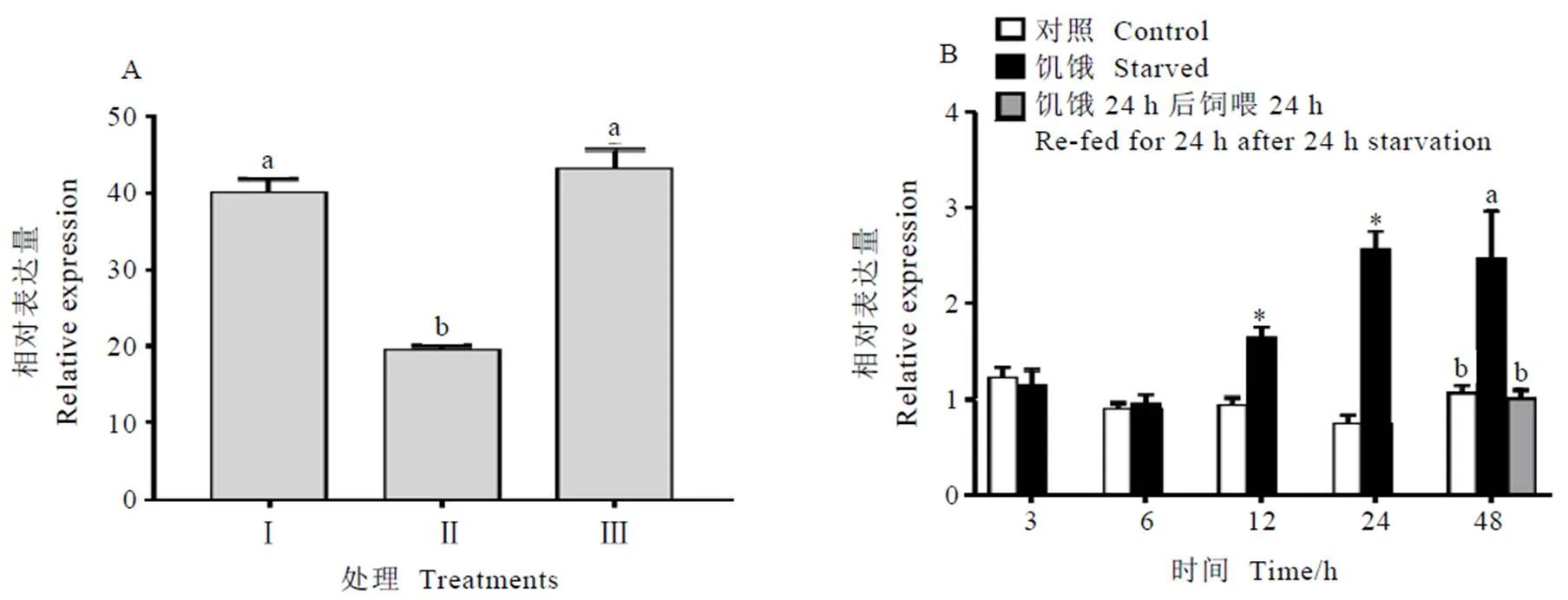
注:A:中肠;B:虫体。Ⅰ:对照;Ⅱ:饥饿24 h;Ⅲ:饥饿24 再饲喂24 h。*表示饥饿处理前后间差异显著(Student’s t检验,P<0.05);柱上字母表示不同处理之间差异具有显著性(Tukey’s多重检验,P <0.05)
3 讨论
丝氨酸蛋白酶作为鳞翅目昆虫消化系统中最重要的消化水解酶,不仅参与食物中蛋白质的水解和其他蛋白质的合成,还与昆虫生长、免疫、蜕皮和变态等生长发育过程密切相关[1]。例如,烟草天蛾卵中含有的多种与黑化和抗菌肽合成相关的丝氨酸蛋白酶参与抵御细菌感染,构成了烟草天蛾卵的先天免疫系统[27]。近年来,还有研究发现昆虫对产生抗性可能依赖于昆虫肠道中蛋白酶水平或类型的变化[2]。目前,科研工作者已从多种鳞翅目昆虫中发现了丝氨酸蛋白酶家族的存在,如甜菜夜蛾SeCT34、家蚕BmSP25和菜青虫PrCT1等,通过对该类蛋白酶的结构和功能分析,鉴定到SP家族的保守结构域和催化位点[18,20,22,28-30]。本文从茶尺蠖幼虫体内克隆到1条丝氨酸蛋白酶的cDNA全长序列(图1),并发现所编码的氨基酸N端含有1个由18个氨基酸残基组成的信号肽(图2),这与已报道的BmSP25,PrCT1等丝氨酸蛋白酶是分泌型蛋白的结构一致[28]。通过与蓓带夜蛾等已知昆虫丝氨酸蛋白酶的氨基酸序列比对,鉴定到EoSP1存在高度保守的H95-A161-S328催化三联活性位点,以及3对半胱氨酸残基(图3)。此外,EoSP1还包含胰蛋白酶蛋白N端保守氨基酸残基IVGG,表明其属于丝氨酸蛋白酶家族中的类胰蛋白酶亚族(图3)。系统进化树与序列比对结果一致,茶尺蠖EoSP1与蓓带夜蛾和小地老虎的SPs同源性较高(图4)。在原核系统中诱导表达了EoSP1-GST重组蛋白,其大小与预测的EoSP1蛋白分子量(约29.53 kDa)一致,表明已克隆获得的可以正常翻译和表达(图5)。综上所述,本文克隆得到的是编码丝氨酸蛋白酶家族基因成员之一,所编码的丝氨酸蛋白酶属于类胰蛋白酶亚族,为分泌型蛋白。
已有研究结果表明,丝氨酸蛋白酶在鳞翅目昆虫不同发育阶段和不同组织中的表达与其生物学功能紧密相关[10,16,31]。本研究发现,在茶尺蠖幼虫中肠中的表达量显著高于脂肪体、血淋巴和唾液腺等其他组织;并且,在1~4龄幼虫阶段的表达量最高,在5龄幼虫、预蛹和蛹期表达量显著下降,在成虫期及卵期几乎不表达(图6)。这一研究结果表明,所编码的蛋白酶参与了茶尺蠖幼虫对植物蛋白质的水解、消化和吸收过程,推测其在调控茶尺蠖的生长、发育和繁殖过程中具有重要作用。类似的研究结果在美国白蛾、小菜蛾丝氨酸中也有发现,但是二者高龄幼虫的表达量均显著高于低龄幼虫[18,32],与之不同的是,茶尺蠖5龄幼虫进入暴食期,但是的相对表达量却显著低于1~4龄幼虫(图6)。究其原因,这可能与茶尺蠖幼虫的取食特点和不同老嫩程度叶片中的蛋白或者其他代谢物含量改变相关[33]。茶尺蠖5龄幼虫进入暴食期,主要以茶树老叶为食。有研究表明,相较于嫩叶或成熟叶,茶树老叶中蛋白含量明显下降[34]。因此,5龄幼虫水解和消化蛋白的需求亦会降低,相应地,主要负责消化蛋白的丝氨酸蛋白酶的表达量也下降。此外,老叶与嫩叶之间生化成分的差异也可能会影响茶尺蠖幼虫体内丝氨酸蛋白酶的表达[35]。
如前所述,丝氨酸蛋白酶在鳞翅目昆虫对植物蛋白质的水解和吸收过程中发挥着重要作用[19,32]。基于此,众多科研工作者对饥饿处理后鳞翅目昆虫中肠内基因的表达响应特征进行了研究,并发现饥饿昆虫中肠中基因的相对表达量显著低于正常取食的昆虫;重新饲喂后,基因的表达量在有的昆虫中可以恢复至正常取食的幼虫水平,但也在个别昆虫则表现出不可逆的抑制作用[15,16,31]。例如,饥饿处理后,烟草天蛾丝氨酸蛋白酶的相对表达量显著下调,而同家族的和的表达量则不受影响,重新饲喂后,的表达量未恢复至正常取食的幼虫水平[15];斜纹夜蛾丝氨酸蛋白酶Slctlp1和Slctlp2的转录水平和蛋白水平在饥饿处理后皆显著下调,但在重新饲喂后,Slctlp2在转录和蛋白水平都恢复至正常取食的幼虫水平,而Slctlp1只在蛋白水平有所恢复[16,31]。本研究中,与对照相比,饥饿处理后茶尺蠖幼虫中肠内的相对表达量显著下降,恢复饲喂后中肠内的相对表达量与对照幼虫的表达量基本一致(图8),这与斜纹夜蛾的研究结果相似。这一结果进一步表明,的表达与茶尺蠖的进食量密切相关,从而推测其在茶尺蠖中肠中发挥着重要的消化作用。有趣的是,饥饿处理诱导了茶尺蠖幼虫整个虫体中相对表达量显著上调,并且恢复饲喂后茶尺蠖幼虫整体的表达量与对照组幼虫体内的表达量基本相当。这说明除了参与茶尺蠖幼虫对植物蛋白质的水解、消化和吸收以外,可能还参与虫体免疫调节等其他重要的生理过程。在今后的研究中,我们将利用RNA干扰技术对的生理功能进行研究。
[1] Jiang H B, Michael R Kanost. The clip-domain family of serine proteinases in arthropods [J]. Insect Biochemistry and Molecular Biology, 2000, 30(2): 95-105.
[2] Srinivasan A, Giri A P, Gupta V S. Structural and functional diversities in lepidopteran serine proteases [J]. Cell Mol Biol Lett, 2006, 11(1): 132-154.
[3] Jeremy Rossa, Michael R Kanostb, Wang Y. Serine proteases and their homologs in thegenome: an initial analysis of sequence conservation and phylogenetic relationships [J]. Gene, 2003, 304: 117-131.
[4] Veillard F, Troxler L, Reichhart J M.clip-domain serine proteases: Structure, function and regulation [J]. Biochimie, 2016, 122: 255-269.
[5] Colebatch G, Cooper P, East P. cDNA cloning of a salivary chymotrypsin-like protease and the identification of six additional cDNAs encoding putative digestive proteases from the green mirid,(Hemiptera: Miridae) [J]. Insect Biochemistry and Molecular Biology, 2002, 32(9): 1065-1075.
[6] 王镜岩, 朱圣庚, 徐长发. 生物化学[M]. 第三版. 北京: 高等教育出版社, 2002: 405.
[7] Joseph Kraut. Serine proteases: Structure and mechanism of catalysis [J]. Ann. Rev. Biochem, 1977, 46: 331-358.
[8] Page M J, Cera E Di. Serine peptidases: classification, structure and function [J]. Cell Mol Life Sci, 2008. 65(7/8): 1220-1236.
[9] Hedstrom L. Serine Protease Mechanism and Specificity [J]. Chemical Reviews, 2002, 102(12): 4501-4523.
[10] Lomate, P R, Mahajan N S, Kale, et al. Identification and expression profiling ofmicroRNAs and their possible role in the regulation of digestive protease genes [J]. Insect Biochem Mol Biol, 2014, 54: 129-137.
[11] Anisuzzaman, Islam M K, Alim M A, et al. Longistatin is an unconventional serine protease and induces protective immunity against tick infestation [J]. Mol Biochem Parasitol, 2012, 182(1/2): 45-53.
[12] Srinivasan A, Ashok P Giri1, Abhay M Harsulkar, et al. A Kunitz trypsin inhibitor from chickpea (L.) that exerts anti-metabolic effect on podborer () larvae [J]. Plant Mol Biol, 2005, 57(3): 359-374.
[13] 李莉莉, 周晓群, 赵奎军, 等. 苜蓿夜蛾丝氨酸蛋白酶基因cDNA序列的克隆与原核表达研究[J]. 应用昆虫学报, 2016, 53(4): 706-715.
[14] 周晓群, 高艳玲, 赵奎军, 等. 苜蓿夜蛾中肠丝氨酸蛋白酶cDNA 的克隆、序列分析及原核表达[J]. 昆虫学报, 2014, 57(9): 1008-1017.
[15] Broehan G, Kemper M, Driemeier D, et al. Cloning and expression analysis of midgut chymotrypsin-like proteinases in the tobacco hornworm [J]. J Insect Physiol, 2008, 54(8): 1243-1252.
[16] Zhang C, Zhou D H, Zheng S C, et al. A chymotrypsin-like serine protease cDNA involved in food protein digestion in the common cutworm,: Cloning, characterization, developmental and induced expression patterns, and localization [J]. J Insect Physiol, 2010, 56(7): 788-799.
[17] 冯一璐, 傅晓斌, 吴帆, 等.茶尺蠖信息素结合蛋白PBP2的基因克隆、原核表达及其结合功能[J]. 中国农业科学, 2017, 50(3): 504-512.
[18] 赵旭东, 孙宇航, 陈昌宇, 等. 美国白蛾丝氨酸蛋白酶基因的克隆、时空表达及对取食不同寄主植物的表达响应[J]. 昆虫学报, 2019, 62(2): 160-169.
[19] Wang R X, Tong X L, Gai T T, et al. A serine protease homologueinduces a short and fat body shape in silkworm [J]. Insect Mol Biol, 2018, 27(3): 319-332.
[20] 张永红, 朱峰, 唐芬芬, 等. 家蚕丝氨酸蛋白酶基因BmSP25转录分析及其免疫响应[J]. 南方农业学报, 2017, 48(6): 1093-1098.
[21] Liu Q Y, Li M G, Liu X F, et al. Characterization of trypsin-like and chymotrypsin-like serine proteases from midgut ofWalker [J]. Arch Insect Biochem Physiol, 2016, 92(3): 173-191.
[22] Fan L M, Rui X R, Yang L, et al. RNA interference mediated serine protease gene () knockdown affects growth and mortality in the soybean pod borer (Lepidoptera: Olethreutid) [J]. Florida Entomologist, 2017, 100(3): 607-615.
[23] Tamhane V A, Chougule N P, Giri A P, et al.andeffect ofproteinase inhibitors ongut proteinases [J]. Biochimica Et Biophysica Acta-General Subjects, 2005, 1722(2): 156-167.
[24] Yang Z W, Duan X N, Jin S, et al. Regurgitant derived from the tea geometridsuppresses wound-induced polyphenol oxidases activity in tea plants [J]. Journal of Chemical Ecology, 2013, 39(6): 744-751.
[25] Huang C, Zhang J, Zhang X, et al. Two new polyphenol oxidase genes of tea plant () respond differentially to the regurgitant of tea geometrid,[J]. Int J Mol Sci, 2018, 19(8): 2414. DOI: 10.3390/ijms19082414.
[26] Sun X L, Li X W, Xin Z J, et al. Development of synthetic volatile attractant for malemoths [J]. Journal of Integrative Agriculture, 2016, 15(7): 1532-1539.
[27] Gorman M J, Kankanala P, Kanost M R. Bacterial challenge stimulates innate immune responses in extra-embryonic tissues of tobacco hornworm eggs [J]. Insect Molecular Biology, 2004, 13(1): 19-24.
[28] 张娴, 李超林, 郑乔木, 等. 菜青虫胰凝乳蛋白酶基因PrCT1的克隆及表达分析[J]. 河南农业科学, 2019, 48(9): 74-81.
[29] Chen C X, Yan Y, Yang H, et al. Cloning of two clip-domain serine protease genes and their expression in response to exogenous hormone and immune challenge in(Coleoptera: Anobiidae) [J]. Acta Entomologica Sinica, 2019, 62(5): 535-546.
[30] He W Y, Zheng Y P, Tang L, et al. Cloning, expression and localization of a trypsin-like serine protease in the spruce budworm,[J]. Insect Science, 2009, 16(6): 455-464.
[31] Zhan Q L, Zheng S C, Feng Q L, et al. A midgut-specific chymotrypsin cDNA () from: cloning, characterization, localization and expression analysis [J]. Arch Insect Biochem Physiol, 2011, 76(3): 130-143.
[32] Shi M, Zhu N, Yi Y, et al. Four serine protease cDNAs from the midgut ofand their proteinase activity are influenced by the endoparasitoid,[J]. Arch Insect BiochemPhysiol, 2013, 83(2): 101-114.
[33] 高旭晖, 宛晓春, 杨云秋, 等. 茶尺蠖生物学习性研究[J]. 植物保护, 2007, 33(3): 110-113.
[34] 郭萧, 王晓庆, 彭萍, 等. 茶树不同成熟度叶片对茶尺蠖发育适合度的影响[J]. 茶叶科学, 2012, 32(3): 229-235.
[35] Fang R, Sally P Redfern, Don Kirkup, et al. Variation of theanine, phenolic, and methylxanthine compounds in 21 cultivars ofharvested in different seasons [J]. Food Chemistry, 2017, 220: 517-526.
Cloning and Expression Analysis of Serine Proteasein Tea Geometrid () and Its Response to Starvation
ZHANG Xin1,2, Chen Chengcong3, DU Qin1,2, LI Xiwang1,2, SUN Xiaoling1,2*
1. Tea Research Institute of the Chinese Academy of Agricultural Sciences, Hangzhou 310008, China; 2. Key Laboratory of Tea Biology and Resources Utilization, Ministry of Agriculture and Rural Affairs, Hangzhou 310008, China; 3. National Research Center of Engineering and Technology of Tea Quality and Safety, Quanzhou 362400, China
Serine protease plays an important role in the digestion process of Lepidoptera insects. In this study, we cloned a serine protease encoding genefromand analyzed its basic characteristics and expression patterns. The coding sequence ofis 858 bp, encoding 285 amino acid residues with deduced molecular weight of 29.53 kDa and isoelectric point of 5.44. Compared with other serine proteases, EoSP1 contains conserved serine protease catalytic sites (H95, A161and S328) and protein interaction domains, and shows the closest relationship with SPs from. Further, EoSP1-GST fusion protein similar to the predicted size was purified fromcells. qRT-PCR analysis showed that the expression level ofwas much higher in larvae than that in adults, pupae and eggs, and expressed in midgut of larvae specifically.was down-regulated by starvation treatment, and the expression level was change back to that of control group after re-feeding. The above results provide a basis for the function analysis of digestive enzyme and screening of new insect-resistance targets in.
, serine protease, cloning, expression pattern
S435.711;Q52
A
1000-369X(2019)06-669-12
2019-10-08
2019-10-23
国家自然科学基金项目(31272053)、中央级公益性科研院所基本科研业务费专项(1610212017017、1610212018004)国家茶叶质量安全工程技术中心开放课题基金项目(2018NTQS0102)
张新,助理研究员,主要从事茶树抗虫机理研究,xinzhang@tricaas.com。
xlsun1974@163.com

