基于3D深度残差网络与级联UNet的缺血性脑卒中病灶分割算法
王平 高琛 朱莉 赵俊 张晶 孔维铭
摘 要:为了解决人工勾画缺血性脑卒中病灶费时费力且易引入主观差异的问题,提出了一种基于三维(3D)深度残差网络与级联UNet的自动分割算法。首先,为了有效利用图像的3D上下文信息并改善类不平衡现象,将脑卒中核磁共振图像(MRI)采样成图像块作为网络输入;然后,利用基于3D深度残差网络与级联UNet的分割模型对图像块进行特征提取,获得粗分割结果;最后,对粗分割结果进行精分割处理。在ISLES数据集上的实验结果表明,该算法的Dice系数可达到0.81,精确度可达到0.81,灵敏度可达到0.81,平均对称表面距离(ASSD)距离系数为1.32,HD为22.67。所提算法与3D UNet算法、基于水平集算法、基于模糊C均值(FCM)算法和基于卷积神经网络(CNN)算法相比分割性能更好。
关键词:急性缺血性脑卒中;自动分割;三维;磁共振图像
中图分类号:TP391.7
文献标志码:A
Segmentation algorithm of ischemic stroke lesion based on
3D deep residual network and cascade UNet
WANG Ping1, GAO Chen1, ZHU Li1,ZHAO Jun1,ZHANG Jing1,KONG Weiming2
1.College of Information Engineering, Nanchang University, Nanchang Jiangxi 330031, China;
2.The 94th Hospital of the Chinese Peoples Liberation Army, Nanchang Jiangxi 330031, China
Abstract:
Artificial identification of ischemic stroke lesion is timeconsuming, laborious and easy be added subjective differences. To solve this problem, an automatic segmentation algorithm based on 3D deep residual network and cascade UNet was proposed. Firstly, in order to efficiently utilize 3D contextual information of the image and the solve class imbalance issue, the patches were extracted from the stroke Magnetic Resonance Image (MRI) and put into network. Then, a segmentation model based on 3D deep residual network and cascade UNet was used to extract features of the image patches, and the coarse segmentation result was obtained. Finally, the fine segmentation process was used to optimize the coarse segmentation result. The experiment results show that, on the dataset of Ischemic Stroke LEsion Segmentation (ISLES), for the proposed algorithm, the Dice similarity coefficient reached 0.81, the recall reached 0.81 and the precision reached 0.81, the distance coefficient Average Symmetric Surface Distance (ASSD) reached 1.32 and Hausdorff Distance (HD) reached 22.67. Compared with 3D UNet algorithm, level set algorithm, Fuzzy CMeans (FCM) algorithm and Convolutional Neural Network (CNN) algorithm, the proposed algorithm has better segmentation performance.
Key words:
acute ischemic stroke; automatic segmentation; threedimensional; Magnetic Resonance Image (MRI)
0 引言
腦卒中是世界上最常见的脑血管疾病,也是最常见的死亡和致残疾病之一。脑卒中可分为缺血性卒中和出血性卒中, 其中缺血性卒中的发病率远高于出血性卒中,占脑卒中总数的87%[1]。缺血性脑卒中是由脑供血障碍引起的脑组织缺氧,并因血流在接下来的几小时内中断太严重或中断时间过长导致的脑细胞逐渐坏死,最终形成不可逆的受损梗塞核心。如果血流恢复得足够快,脑组织可能不会永久损坏,这种围绕着缺血性损伤核心的潜在可逆性损伤的组织区域,被称为“缺血半影”[2]。根据脑卒中发病后经过的时间,可将病灶阶段细分为急性(0~24h)、亚急性(24h~2week)和慢性(超过2week)[3]。急性缺血性脑卒中的典型症状是局灶性神经功能缺损的突然发作,如失语、偏盲、感觉丧失等。这些症状可能发展成慢性疾病(例如痴呆、偏瘫等),在很大程度上影响了患者的生活并且医疗开销也会急剧增高。急性期的及时诊断和治疗对脑卒中患者的康复和预后至关重要,医生需要在卒中发作后的3h内快速定位并量化病灶。但人工描绘急性脑卒中病灶费时费力且会引入观察者间的主观差异性。自动分割算法可以准确、客观地评估病灶,且具有可重复、可扩展等优点,现已成为医学图像计算领域的一个主要研究热点。
以往有很多分割脑卒中病灶的方法,基于纹理的特征提取算法就常用于脑卒中病灶的分割中,比如Neethu等[4]先用Gabor滤波方法对图像进行边缘和轮廓的提取,再利用区域生长进行脑卒中区域的分割。另一种则是由Karthikeyan等[5]提出的,该算法先用灰度共生矩阵(Gray Level Cooccurrence Matrix, GLCM)从图像中提取特征,然后再用神经网络模型对提取的特征进行分类。但基于纹理的特征提取一般只能用来对图像的局部缺血性脑卒中进行分析;且这些方法用的都是较低级的图像特征(图像灰度信息或边缘信息),对于低信噪比且存在伪影的图像的分割缺乏鲁棒性。近年来也有许多基于随机森林的脑卒中病灶分割方法[6-8]结果较好,但基于随机森林的方法较依赖于人工提取特征,步骤相对复杂。当前基于深度学习的算法已广泛应用到医学图像处理工作中,其具有很强的模型能力,并且能够更好地提取图像特征,尤其在特征不明显的图像中更能体现其优势。在脑卒中分割中一些深度学习的方法获得了良好的分割效果,比如,Chen等[9]提出了一种融合了卷积网络、空洞卷积和全连接条件随机场的DeepLab算法,且该方法在ISLES(Ischemic Stroke LEsion Segmentation)大赛中获得了优异的成绩。Lucas等[10]提出了一种改进的U型全卷积神经网络架构。但这些方法都只考虑了二维切片信息,忽略了医学图像的三维上下文信息。为了能充分利用三维医学影像的深度信息,2017年,Kamnitsas等[11]提出了一种多尺度三维卷积神经网络(Threedimensional Convolutional Neural Network, 3D CNN)的方法实现了对亚急性缺血性脑卒中病灶的三维分割。
本文面向急性缺血性脑卒中的辅助性诊断,将深度学习的方法引入急性缺血性脑卒中核磁共振图像(Magnetic Resonance Images, MRI)的分析中,提出了一种基于3D深度残差网络与级联UNet的分割新算法。新算法将采样的MRI图像块作为网络的输入,可以避免医学图像中常出现的类不平衡现象并可得到更细致的分割。新算法在网络结构中加入了残差模块和级联思想,使其在避免梯度消失问题的同时也可更高精度地分割出病灶。用该算法对ISLES 2015的急性缺血性脑卒中数据进行分割,结果表明本算法可得到较高的分割精度,且具有速度快、无需参考专家先验知识等优势,对今后急性缺血性卒中的计算机辅助诊断研究有很大的参考价值。
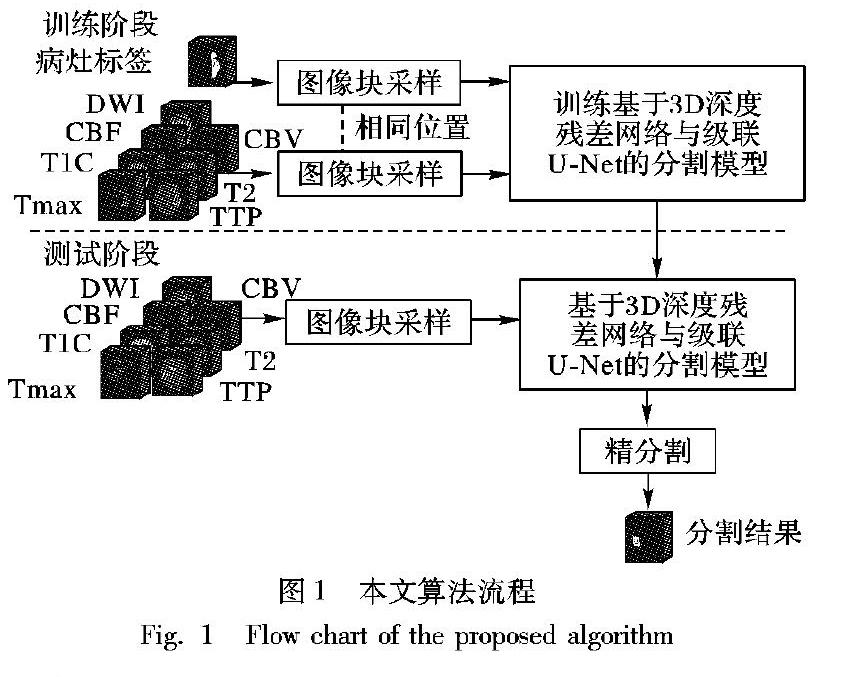
1 算法流程
本文用基于3D深度残差网络与级联UNet的分割新算法对急性缺血性脑卒中进行病灶分割,该研究方法的整个算法流程如图1所示, 其中T1c、T2、脑血流量(Cerebral Blood Flow, CBF)、脑血容量(Cerebral Blood Volume, CBV)、扩散加权成像(Diffusion Weighted Imaging, DWI)、血流达峰时间(Time maximum,Tmax)和对比剂峰值时间(Time To Peak, TTP)为七種不同模态的磁共振图像。
首先,为了有效地利用三维上下文信息并改善类不平衡现象,对训练集和测试集的MRI数据进行图像块采样操作;然后,将训练集图像块输入基于3D深度残差网络与级联UNet的分割模型中进行特征提取及训练,在固定分割模型参数后对测试集MRI数据进行分割;最后,为了优化粗分割结果降低假阳性,对分割结果进行进一步的精分割处理。
2 本文方法
2.1 图像预处理
2.1.1 数据集介绍
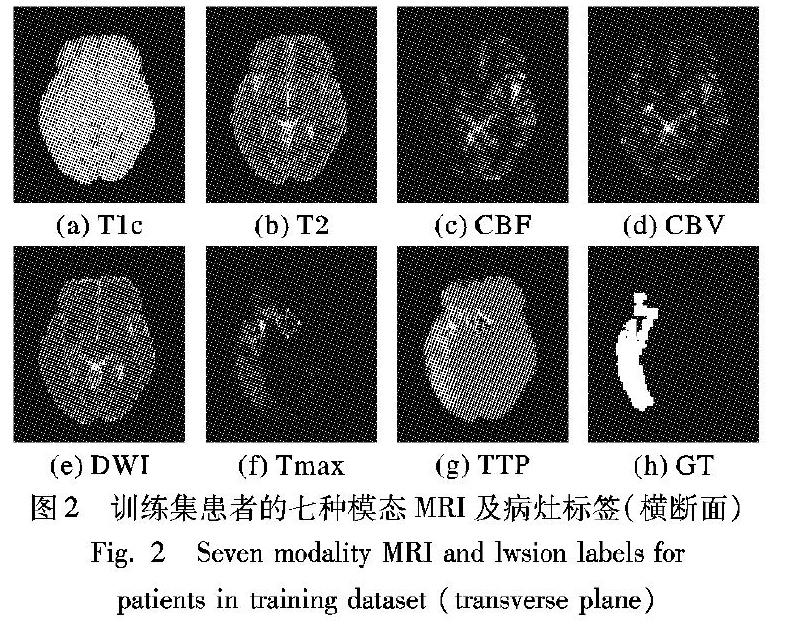
本文方法在缺血性脑卒中挑战赛ISLES2015(http://www.isleschallenge.org/ISLES2015/)所提供的数据集上进行了实验。该数据集提供亚急性脑卒中病变分割扫描影像(SubAcute Stroke Lesion Segmentation, SISS)和急性脑卒中病变分割扫描影像(Stroke Perfusion Estimation, SPES)。其中SPES数据集包含了从伯尔尼大学医院选取的50例急性缺血性患者的临床图像。医院用一台3T MR扫描仪(Siemens Magnetom Trio)和一台1.5T MR扫描仪(Siemens Magnetom Avanto)来获取DWI和灌注成像(Perfusion Imaging, PWI),最终每位患者都配有T1c、T2、CBF、CBV、DWI、Tmax和TTP七种模态的MRI,各模态间都已配准并进行了颅骨剥离处理,图像分辨率都为2×2×2。该数据集将30例MRI数据作为训练集并公开了由权威专家手动标注的病灶标签如图2所示,测试集则包含20位患者的七种模态MRI但没有公开病灶标签。
2.1.2 图像块提取
本文的图像块提取工作包括以下3个步骤:
1)强度归一化。
强度归一化就是在保留具有诊断价值的灰度差异的同时,为了减小并消除图像中灰度不一致的现象而进行的图像转换方法,以便计算机自动分析处理。通过减去图像灰度值的均值再除以方差,使其灰度分布达到均值为0、标准差为1的正态分布。
2)图像块采样。
在医学图像中一般健康组织占比远超过病灶,会出现医学图像分割中常见的类不平衡问题,为了解决该问题,本文用以病灶为中心的采样方式[12]将图像采样成小块作为网络的输入。这里用NPatch表示提取图像块的个数,在本实验中NPatch=10-000,NLesion表示病灶体素数,T表示采样图像块的中心间隔。训练集图像块提取步骤如下:
①找出训练集标签中的病灶区体素,计算体素个数NLesion并记录病灶体素坐标位置。
②为了使病灶大小不同的病例可以输入相同数量的采样图像块,本研究将采样方式分为以下两种情况。若NPatch
③在确定图像块中心体素之后,对这些中心体素进行随机偏移,随机移位Δx、Δy的范围为提取图像块大小的一半。最后,以24×24×8的大小提取图像块。
以上述方法提取训练集的图像块,可以保证输入的图像块都包含病灶,让模型可以学习病灶及病灶边沿区域的特征,同时避免了训练时出现负样本扰乱分割模型的情况。测试集图像块则以均匀采样的方式提取。确定测试集图像块中心后,以24×24×8的大小提取图像块。
3)數据增广。
数据增广目的是扩增更多的训练数据来提高网络性能并避免出现过拟合现象。通过对提取的图像块进行90°、180°、270°的旋转来使训练数据扩大。
2.2 分割网络架构
2.2.1 基于残差学习的特征提取
残差学习的基本思想是利用多层卷积拟合一种残差映射F(x),相对于直接学习目标近似恒等映射更易优化。假设输入变量为x,残差学习就是通过“捷径连接”(shortcut connection)直接把输入x传到输出作为初始结果,输出结果为:
H(x)=F(x)+x(1)
当F(x)=0时,H(x)=x,也就是所谓的恒等映射。这样残差的学习目标不再是一个完整的输出,而是目标值H(x)和x的差值,即:
F(x)=H(x)-x(2)
残差学习中的“捷径连接”可以跳过一个层或多个层,执行恒等映射,该方式可以避免因网络加深导致的梯度消失等问题,使网络更好地优化。因此,本文在卷积层中引入残差学习,残差块结构如图3所示。
残差块由两个核大小为3×3×3的卷积层和一个随机失活层(dropout)构成。因为PReLU(Parametric Rectified Linear Unit)激活函数可以使网络更快地过滤出不相关的信息,更适合其训练[13],所以在残差模块中,用PReLU作为卷积层的激活函数,PReLU激活函数如式(3)所示:
g(x)=max(0,x)+aimin(0,x)(3)
其中,ai表示控制激活函数负部分倾斜的系数。为了加快网络收敛,卷积层中还加入了批量归一化(Batch Normalization, BN)处理。在残差模块中引入随机失活层(失活概率设置为0.2),可以使部分神经元在训练中失活,避免参数过拟合,从而提高网络的泛化性能。
2.2.2 缺血性脑卒中分割网络模型
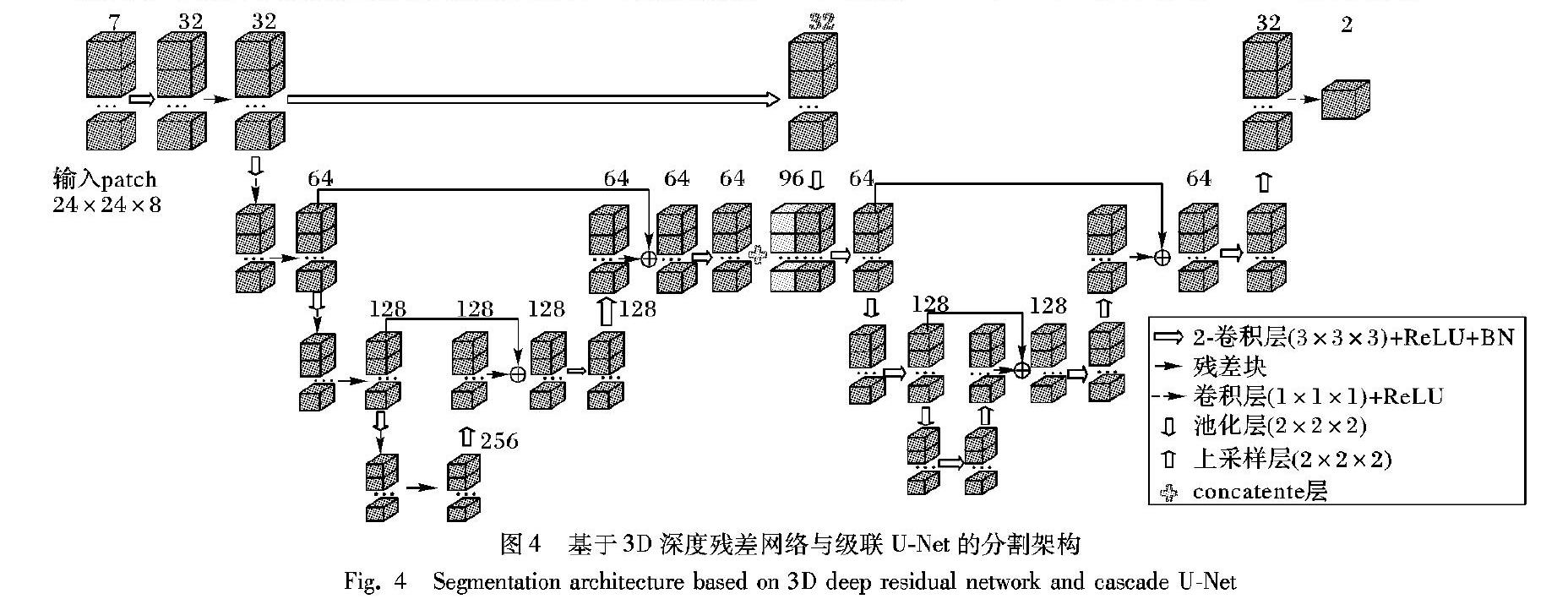
针对缺血性脑卒中病灶特征的复杂性(如病灶边缘模糊、形态各异),设计了基于3D深度残差网络与级联UNet分割模型,该模型引入了残差模块和级联思想,使网络在改善优化困难问题的同时进一步提高了分割精度。
本文算法的分割架构如图4所示,该架构由一个非对称型残差UNet和一个三层下采样UNet级联构成。
在一级非对称型残差UNet中,用残差模块进行特征提取,在每个残差模块后用一个步长为2、核大小为2×2×2的池化层进行下采样操作,本架构还在一级残差UNet的编码部分设置了Bottleneck层,即在每一个池化层后加入1×1×1的卷积层对输出特征层通道数进行加倍,帮助完成下一层的残差学习。一级非对称型残差UNet的解码部分则由卷积层和上采样层组成,其中上采样层是核大小为3×3×3、步长为2的反卷积层,通过上采样操作特征图的大小可恢复为原尺寸,但上采样不可恢复池化操作时丢失的空间信息,因此用一个跳跃连接将上采样的特征图与编码器中相同大小的特征图进行融合,待充分融合底层的空间信息和高层的特征信息后,输出特征图。二级UNet则是对一级非对称型残差UNet输出的特征图进行二次特征提取,即将一级非对称残差UNet解码器中经过两次上采样后的特征图与编码器中第一层输出的特征图级联到一起继续训练。在二级UNet中用3×3×3大小的卷积层和步长为2、核大小为2×2×2的池化层进行编码,然后用卷积和上采样进行解码,为了使网络参数减少,二级UNet中同样用像素级相加的融合操作进行跳跃连接。最后,用一个1×1×1的卷积层将多通道数映射为2,再将通道数为2的特征图送入Softmax分类器中,得到脑卒中病灶分割概率图(每一个值即代表该像素点属于病灶的概率值)即分割输出结果。
2.3 精分割
在得到各图像块粗分割结果后,将各图像块根据其中心坐标还原回原先位置。但由于图像块分割结果是对重合处取概率平均,且以类概率作为病灶区体素值的结果会导致假阳性提高,从而会产生误差。为了优化分割结果,除去伪缺血点进而降低假阳性,本方法用如下两个步骤提高分割精度:
1)设定类概率阈值h,将高于阈值处的体素值置为1,低于阈值处的体素置为0,即粗分割结果中会出现一些类概率很低的体素值,而该区域并不属于病灶区,需设定阈值将其设为健康区域。
2)分割结果中会出现一些小的伪病灶点,所以在步骤1)后可通过计算三维连通域体积来进一步过滤掉伪缺血点。pre(x,y,z)表示经过步骤1)后的分割结果中的体素(健康区:pre(x,y,z)=0,脑卒中病灶区:pre(x,y,z)=1)。v(n)表示pre中第n个连通域的体积,Vmax为pre中体积最大的连通域的体积。若v(n)/Vmax<θ1,将第n个三维连通域移除,即将第n个三维连通域内的pre(x,y,z)置为0。
3 实验结果及分析
3.1 客观评估指标
为了评估提出的分割方法性能,本文用Dice系数、精确度(precision)、灵敏度(recall)、平均对称表面距离(Average Symmetric Surface Distance,ASSD)和霍夫曼距离(Hausdorff Distance, HD)来进行分割精度评价。其中,定义正确分割为病灶的区域为真阳性(True Positive, TP);定义被错误分割为病灶区实则为非病灶的区域为假阳性(False Positive,FP);定义被错误分割为非病灶区实则为病灶的区域为假阴性(False Negative, FN)。Dice是分割结果与病灶标签间的一个重叠度度量。Dice、精确度和灵敏度的定义如下:
Dice=2TPTP+FP+FN(4)
precision=TPTP+FP(5)
recall=TPTP+FN(6)
虽然Dice、精確度及灵敏度都可用基于体积的重叠度来度量分割结果的准确度,但两个重叠度大的区域可能在边界上存在差异,这里用基于距离的度量尺度(平均对称表面距离和霍夫曼距离)来衡量此差异,且平均对称表面距离和霍夫曼距离相同,指标越小,代表分割精度越高。设A为病灶标签表面的点,B为分割结果表面的点。平均对称表面距离定义如下所示:
ASD(A,B)=∑ a∈Amin b∈B d(a,b)A(7)
ASSD(A,B)=[ASD(A,B)+ASD(B,A)]/2(8)
其中,ASD(Average Surface Distance)为平均表面距离;d(a,b)为A,B点的欧氏距离且ASD(A,B)≠ASD(B,A)。
霍夫曼距离表示病灶标签和分割结果表面点间的最大距离,可表示分割结果中的离群值。霍夫曼距离:
HD(A,B)=
max{maxa∈Aminb∈B d(a,b),maxb∈Bmina∈A d(a,b)}(9)
3.2 实验结果与分析
本文使用第2章介绍的ISLES2015 SPES数据集进行实验,在ubuntu16.04上使用以tensorflow为后端的keras开源库搭建模型。本文采用自适应优化器Adadelta对损失函数进行优化。训练时,残差模块中的dropout层的随机失活率设为0.2,训练批大小取16,epoch设置为12。
对ISLES2015 SPES数据集进行分割的实验结果表明,本文算法可高精度地分割出脑卒中病灶。经多次实验,在精分割步骤中设定类概率阈值h=0.4,连通域体积比值θ1=0.2可获得最优分割结果。为了进一步论证本文算法的优越性,本文还选用了基于3D UNet的算法[14]作为对比实验,3D UNet在精分割步骤中设定类概率阈值h=0.4,连通域体积比值θ1=0.1可获得最优分割结果。
而为了进一步分析本文提出的算法,还分别选用了基于水平集算法[15]、基于模糊C均值(Fuzzy CMeans,FCM)聚类算法[16]和基于多尺度CNN算法[17]与本文算法比较,分割指标比较如表1、2所示。
实验结果表明:1)本文算法得到的Dice、精确度和灵敏度均高于其他四种算法,具有明显的算法优势;2)本文算法得到的分割距离系数ASSD和HD在四种算法中最优,说明本文算法的分割结果误差最小。
由表1可以看出基于水平集的算法分割效果最差,虽然基于水平集的分割方法具有拓扑适应性强的优点,常用于分割拓扑结构复杂的组织器官及病灶,但由于缺血性脑卒中病灶边界模糊,基于水平集的算法[15]无法准确定位病灶边缘,分割误差较大。基于FCM的算法[16]分割性能虽然有所提高,但该算法是先用FCM的算法进行粗分割,再结合基于水平集的算法进行细化分割,步骤相对复杂、分割效率低。基于多尺度CNN的深度学习算法[17]也可获得较高的分割精度,但该算法用二维的切片图像作为网络的输入,不能很好地利用三维图像的上下文信息,且基于CNN的分割方法是用逐像素的分类完成分割,这样会增加网络计算量,影响分割效率。本文提出的基于3D深度残差网络与级联UNet是一个端对端的训练,在提升分割精度的同时也可保证分割效率,且与3D UNet的分割结果相比,本文算法的分割精度更高,误差更小,分割性能更具优越性。在对ISLES2015数据的分割结果中,一些基于随机森林的方法[18-20]也有较好的分割性能,但随机森林的方法需要人工确定并提取特征,操作复杂不满足全自动分割方式,而本文方法可以高精度地自动分割出病灶,无需人工干预。
测试集和训练集的病灶分割结果图如图5、6所示(图中标注Case17_slice48表示第17个病例中第48层的横断面切片)。
图6将本文方法在训练集上的分割结果与专家的标注结果进行比较,可以看出本文方法的分割结果与病灶标签基本重合,可准确定位出病灶位置并高精度地分割出病灶轮廓。但对于小缺血点的分割(测试患者Case27_slice43所示)仍存在不足,需要在后续研究中用改进预处理等方法提高小病灶的分割精准度。综上所述,本文算法能快速地自动检测并高精度分割出病灶, 可进一步用于基于MRI的缺血性脑卒中的诊断,帮助医生更为快速准确地判定患病情况。
4 结语
本文提出了一种辅助医生诊断急性缺血性脑卒中的新算法。首先对MRI进行图像块提取处理,然后用基于3D深度残差网络与级联UNet的分割网络对采样图像块进行特征提取并得到粗分割结果,最后用基于三维连通域等算法对其进行优化精分割,从而实现对急性缺血性脑卒中MRI的自动分割。本文方法在公开的急性缺血性脑卒中数据集上进行分割,实验结果表明本文算法获得的Dice、精确度、灵敏度和分割距离系数都明显优于对比算法。综上所述,本文提出的基于3D深度残差网络与级联UNet的新算法具有分割精度高、速度快且不需要人工干预等优点,为急性缺血性脑卒中患者设计了一个特定的脑卒中自动分割模型,可进一步帮助医生客观地诊断、评估病灶并规划治疗。
參考文献 (References)
[1]MOZAFFARIAN D, BENJAMIN E J, GO A S, et al. Heart disease and stroke statistics — 2016 update: a report from the American heart association[J]. Circulation, 2016, 133(4):338-360.
[2]ASTRUP J, SYMON L, BRANSTON N M, et al. Cortical evoked potential and extracellular K+ and H+ at critical levels of brain ischemia[J]. Stroke, 1977, 8(1):51-57.
[3]MAIER O, MENZE B H, VON DER GABLENTZ J, et al. ISLES 2015 — a public evaluation benchmark for ischemic stroke lesion segmentation from multispectral MRI[J]. Medical Image Analysis, 2017, 35:250-269.
[4]NEETHU S, VENKATARAMAN D. Stroke detection in brain using CT images[C]// Proceedings of the 2014 Artificial Intelligence and Evolutionary Algorithms in Engineering Systems, AISC 324. Berlin: Springer, 2015:379-386.
[5]KARTHIKEYAN S M. EZHILARASI M. Automatic stroke lesion segmentation from diffusion weighted MRI images[J]. International Journal of Engineering Research & Technology, 2016,7(2):111-115.
[6]ELLWAA A, HUSSEIN A, ALNAGGAR E, et al. Brain tumor segmentation using random forest trained on iteratively selected patients[C]// Proceedings of the 2016 International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, LNCS 10154. Berlin: Springer, 2016:129-137.
[7]SONG B, CHOU C, CHEN X, et al. Anatomyguided brain tumor segmentation and classification[C]// Proceedings of the 2016 International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, LNCS 10154. Berlin: Springer, 2016:162-170.
[8]le FOLGOC L, NORI A V, ANCHA S, et al. Lifted autocontext forests for brain tumor segmentation[C]// Proceedings of the 2016 International Workshop on Brain lesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, LNCS 10154. Berlin: Springer, 2016:171-183.
[9]CHEN L, BENTLEY P, RUECKERT D. Fully automatic acute ischemic lesion segmentation in DWI using convolutional neural networks[J]. NeuroImage: Clinical, 2017, 15:633-643.
[10]LUCAS C, KEMMLING A, MAMLOUK A M, et al. Multiscale neural network for automatic segmentation of ischemic strokes on acute perfusion images[C]// Proceedings of the IEEE 15th International Symposium on Biomedical Imaging. Piscataway: IEEE, 2018: 1118-1121.
[11]KAMNITSAS K, LEDIG C, NEWCOMBE V F J, et al. Efficient multiscale 3D CNN with fully connected CRF for accurate brain lesion segmentation[J]. Medical Image Analysis, 2017, 36:61-78.
[12]GUERRERO R, QIN C, OKTAY O, et al. White matter hyperintensity and stroke lesion segmentation and differentiation using convolutional neural networks[J]. NeuroImage: Clinical, 2018, 17:918-934.
[13]HE K, ZHANG X, REN S, et al. Delving deep into rectifiers: surpassing humanlevel performance on ImageNet classification[C]// Proceedings of the 2015 IEEE International Conference on Computer Vision. Piscataway: IEEE, 2015:1026-1034.
[14]CICEK , ABDULKADIR A, LIENKAMP S S, et al. 3D Unet: learning dense volumetric segmentation from sparse annotation[C]// Proceedings of the 2016 International Conference on Medical Image Computing and ComputerAssisted Intervention, LNCS 9901. Berlin: Springer, 2016: 424-432.
[15]HAECK T, MAES F, SUETENS P. ISLES challenge 2015: automated modelbased segmentation of ischemic stroke in MR images[C]// Proceedings of the 2015 International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, LNCS 9556. Berlin: Springer, 2015:246-253.
[16]FENG C, ZHAO D, HUANG M. Segmentation of ischemic stroke lesions in multispectral MR images using weighting suppressed FCM and three phase level set[C]// Proceedings of the 2015 International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, LNCS 9556. Berlin: Springer, 2015:233-245.
[17]HAVAEI M, DUTIL F, PAL C, et al. A convolutional neural network approach to brain tumor segmentation[C]// Proceedings of the 2015 International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, LNCS 9556. Berlin: Springer, 2015:195-208.
[18]MCKINLEY R, HNI L, WIEST R, et al. Segmenting the ischemic penumbra: a decision forest approach with automatic threshold finding[C]// Proceedings of the 2015 International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, LNCS 9556. Berlin: Springer, 2015:275-283.
[19]MAIER O, WILMS M, HANDELS H. Image features for brain lesion segmentation using random forests[C]// Proceedings of the 2015 International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, LNCS 9556. Berlin: Springer, 2015:119-130.
[20]ROBBEN D, CHRISTIAENS D, RANGARAJAN J, et al. A voxelwise, cascaded classification approach to ischemic stroke lesion segmentation[C]// Proceedings of the 2015 International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, LNCS 9556. Berlin: Springer, 2015:254-265.
This work is partially supported by the National Natural Science Foundation of China (61463035), the China Postdoctoral Science Foundation (2016M592117), the Outstanding Youth Fund Project in Jiangxi Province (2018ACB21038), the Science and Technology Support Project of Jiangxi Science and Technology Department (20151BBG70057), the Science and Technology Project of Jiangxi Education Department (GJJ14137).
WANG Ping, born in 1958, professor. Her research interests include image processing, pattern recognition.
GAO Chen, born in 1993, M. S. candidate. Her research interests include image processing, machine learning.
ZHU Li, born in 1982, Ph. D., associate professor. Her research interests include image processing, machine learning.
ZHAO Jun, born in 1995, M. S. candidate. Her research interests include image processing, machine learning.
ZHANG Jing, born in 1994, M. S. candidate. Her research interests include image processing, machine learning.
KONG Weiming, born in 1984, M. S. candidate. His research interests include medical image analysis.

