中华绒螯蟹野生群体和不同水系人工选育群体的遗传多样性分析*
李晶晶 陈丽梅 耿绪云 董学旺 孙金生
中华绒螯蟹野生群体和不同水系人工选育群体的遗传多样性分析*
李晶晶1,2陈丽梅3耿绪云2董学旺2孙金生1①
(1. 天津师范大学生命科学学院 天津 300387;2. 天津市水生动物疫病预防控制中心 天津 300221;3. 天津农学院水产学院 天津市水产生态及养殖重点实验室 天津 300384)
本研究利用10个微卫星分子标记分析了中华绒螯蟹() 3个不同水系人工选育群体(“长江1号”、“光合1号”和七里海河蟹)和1个海河流域自然群体的遗传多样性和遗传分化水平。结果显示,10个位点在4个群体中的等位基因数()为3~17,平均等位基因数为8.5~9.7,平均期望杂合度为0.720~0.745,平均观测杂合度为0.566~0.661,平均多态信息含量为0.687~0.716,近交系数(is)范围为–0.080~0.827。在40个群体–位点组合中,有13个群体–位点组合显著偏离哈迪–温伯格平衡(<0.05)。遗传多样性分析结果显示,与海河自然群体相比,3个人工选育群体遗传多样性水平略有降低,但仍保持在较高水平,具有较大的选育潜力。遗传分化分析结果显示,群体间遗传分化指数(st)范围为0.015~0.075,遗传相似度为0.7702~0.9401,遗传距离为0.0617~0.2611。基于Nei’s遗传距离构建了群体UPGMA系统进化树,自然群体和“光合1号”聚为一支,而七里海河蟹群体单独聚为一支。综上所述,4个中华绒螯蟹群体间的遗传分化水平较低,群体遗传多样性较高。本研究将为中华绒螯蟹选育繁育和种质资源利用与管理等提供理论基础。
中华绒螯蟹;遗传多样性;遗传分化;微卫星
中华绒螯蟹()俗称河蟹,自然分布于我国长江、黄河、辽河和瓯江等河川及附近地区(Ma, 2007; 肖起珍等, 2017)。自20世纪70年代末突破人工育苗技术难关,经过30余年的发展,中华绒螯蟹养殖遍布我国辽宁、河北、天津、山东、江苏、浙江等沿海省份及广大内陆地区,成为我国重要经济甲壳动物之一(李晶晶等, 2016)。随着中华绒螯蟹养殖产业的规模化发展,过度捕捞野生自然资源,无序引进异地苗种,大大加剧了中华绒螯蟹种质退化和混杂,已成为当前养殖生产发展的瓶颈(王成辉等, 2002)。研究和保护中华绒螯蟹种质资源,对河蟹养殖业的持续稳定发展具有重要意义。近年来,科研院所和养殖企业积极开展中华绒螯蟹良种人工选育工作,已培育出5个国家级新品种(“光合1号”、“长江1号”、“长江2号”、“江海21”和“诺亚1号”)。
天津市地处环渤海中部,具有得天独厚的海、淡水养殖环境条件,中华绒螯蟹养殖历史悠久。辖区内的七里海是我国重要的古海岸与古湿地保护区,独特的生态环境创出了味道鲜美、膏满黄肥的天津市特色农产品品牌——七里海河蟹(朱广奇, 2012; 杜红梅, 2014)。然而,50年代末期,天津海河入海口处建设了防潮闸,切断了中华绒螯蟹的生殖洄游通道,导致本地区自然资源逐年减少。2000年,天津市水生动物疫病预防控制中心采集七里海核心区中华绒螯蟹野生群体作为选育基础群体,利用群体选育技术连续选育6代,获得快速生长的七里海河蟹群体。
在传统人工选育中,由于长期不引进外来亲本,经过连续多代选育获得的新品种或新品系,其种群遗传多样性是否有所降低,有待进行科学评价。一般认为,遗传多样性的大小及其群体遗传结构跟一个物种的进化潜力和抵御不良环境的能力密切相关(李博, 2000)。因此,分析检测育种群体的遗传特征,对于中华绒螯蟹选育效果预测、种质资源保护以及养殖产业健康持续发展意义重大。
目前,天津地区中华绒螯蟹资源主要有人工选育群体“七里海河蟹”、“长江1号”、“光合1号”以及海河流域自然群体。本研究利用10个多态性丰富的微卫星分子标记,比较分析了天津地区4个中华绒螯蟹群体的遗传多样性和群体间的遗传分化,旨在为开展中华绒螯蟹选育和种质资源利用与管理等工作提供理论基础。
1 材料与方法
1.1 实验材料
本实验共采集4个中华绒螯蟹群体,每个群体样品数量为60只(表1)。七里海河蟹群体于2012年采自天津市蟹源水产养殖有限公司,为天津地区海河流域人工选育的第6代成蟹群体;自然群体于2012年采自海河入海口,是天津地区海河流域野生成蟹种群;“长江1号”和“光合1号”于2013年分别采自江苏省淡水水产研究所和盘锦光合蟹业有限公司,为长江水系和辽河水系人工选育幼蟹群体。采集的4个中华绒螯蟹群体同属奇数年群体。分别剪取每只中华绒螯蟹步足肌肉组织,置于–20℃冰箱保存备用。
1.2 微卫星引物
本研究选用了10个已发表的多态性较高的微卫星位点,用于中华绒螯蟹4个群体的遗传分析(Li, 2016)。引物由生工生物工程(上海)股份有限公司合成,引物序列及特征见表2。
1.3 DNA提取与检测
采用天根生化科技有限公司的海洋动物组织基因组DNA提取试剂盒,提取中华绒螯蟹肌肉组织的总DNA,并用1%琼脂糖凝胶电泳检测DNA质量,紫外分光光度法测定DNA浓度和纯度。用灭菌双蒸水将DNA稀释成100 µg/ml后,–20℃保存备用。
1.4 PCR反应体系、反应程序及扩增产物的检测
PCR反应在10 µl体系中进行,包括1×buffer、2 mmol/L MgCl2、0.2 mmol/L dNTP、1 µmol/L引物、0.25 UDNA聚合酶(TaKaRa)和100 ng DNA模板。PCR程序:94℃预变性3 min;94℃变性1 min,退火1 min,72℃延伸1 min,35个循环;最后72℃延伸5 min。PCR扩增产物用6%的变性聚丙烯酰胺凝胶电泳检测,硝酸银法染色,10 bp DNA ladder (Invitrogen)作为Marker检测扩增片段大小。
1.5 数据分析
使用Microsatellite AnalyseR (MSA)软件计算各微卫星位点在4个群体中的等位基因数()、期望杂合度(e)和观测杂合度(o) (Dieringer, 2003)。用公式e=1/∑i2计算有效等位基因数(e) (i表示每个位点第个等位基因的频率) (Kimura, 1964)。FSTAT 2.9.3软件(Goudet, 2001)计算每个群体每个位点的近交系数(is)。应用PIC-CALC 0.6软件计算多态信息含量(PIC)。Genepop 4.2软件检测10个位点的哈迪-温伯格平衡(HWE)偏离情况,并比较分析各位点在群体间等位基因频率(Raymond, 1995)。MSA软件计算st值(Weir, 1984)。应用SPAGeDi 1.1软件(Hardy, 2002)对无效等位基因进行校正,Bonferroni检验对显著性水平的多点检测进行校正(Rice, 1989)。使用Popgene Version 1.31计算Nei’s遗传距离(Nei, 1978),利用MEGA 5软件构建群体间UPGMA系统进化树(Tamura, 2011)。
表1 中华绒螯蟹群体的取样时间、地点、群体类型和样本数

Tab.1 The sampling time, location, population and size of E. sinensis
表2 中华绒螯蟹10个微卫星位点特征
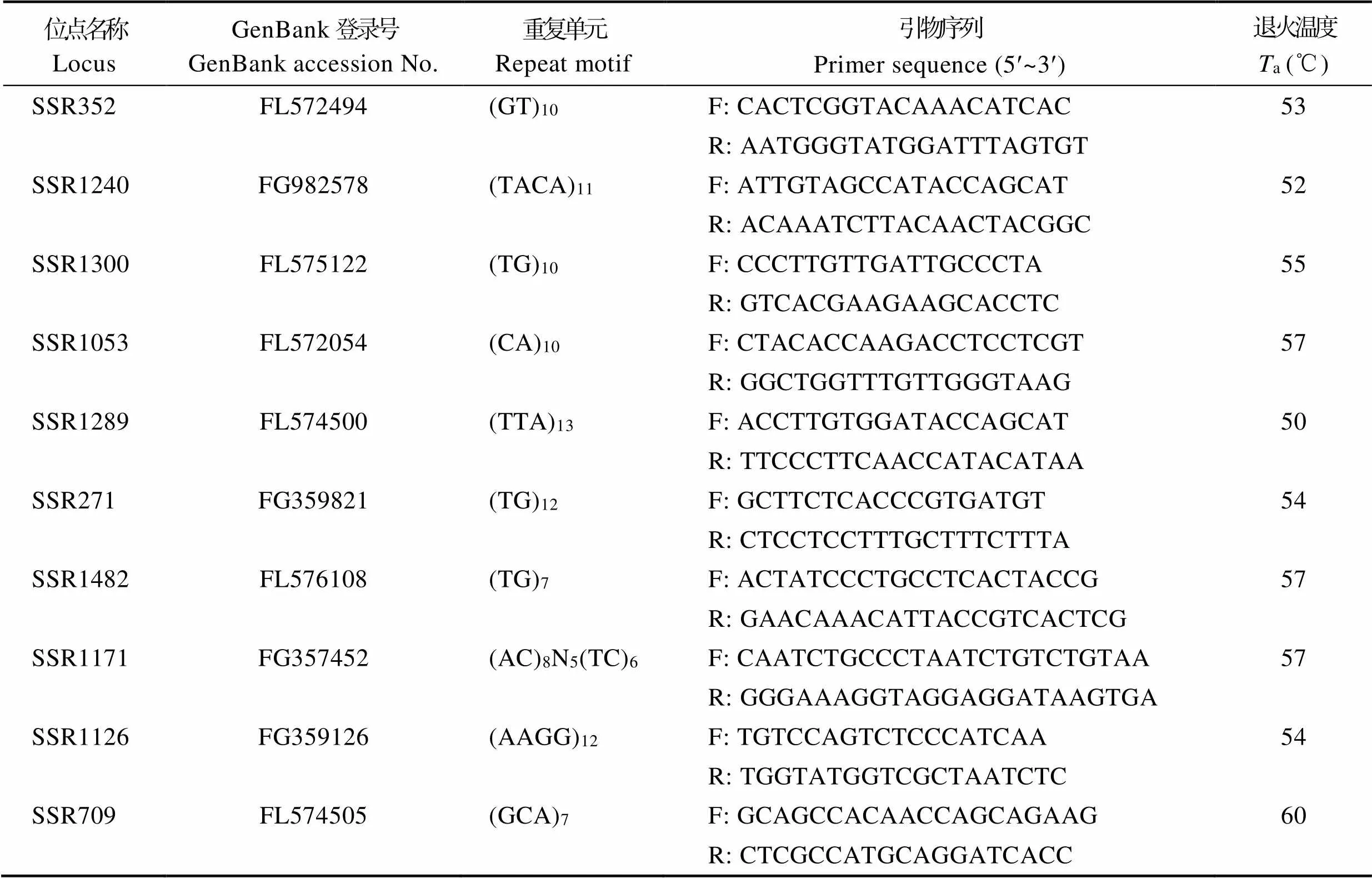
Tab.2 Characteristics of ten microsatellite loci of E. sinensis
2 结果与分析
2.1 微卫星位点多态性及群体遗传多样性
应用选取的10对微卫星引物,对4个中华绒螯蟹群体共240个个体进行扩增分析,遗传多样性参数见表3。等位基因数范围为3~17,其中,位点SSR352和SSR1289等位基因数最多(=17),分别出现在七里海河蟹群体和自然群体中。七里海河蟹、自然群体、“长江1号”和“光合1号”的平均等位基因数分别为9.4、9.7、9.0和8.5;平均观测杂合度为0.658、0.661、0.566和0.618;平均期望杂合度为0.720、0.745、0.732和0.720;平均多态信息含量为0.691、0.716、0.694和0.687,所有参数的最高平均值均出现在自然群体中。所有位点的平均观测杂合度均略低于平均期望杂合度,4个群体均具有较高的平均期望杂合度(>0.5)和平均多态信息含量(>0.5)。
10个位点在4个群体中的近交系数is范围为–0.080~0.827。在七里海河蟹群体中,位点SSR1240、SSR1053和SSR709的近交系数为负值;在“光合1号”群体中,位点SSR1289、SSR1126和SSR709的近交系数为负值;在自然群体中,位点SSR709的近交系数为负值,其余均为正值。经过Bonferroni校正后,10个位点在4个群体中,有13个显著偏离哈迪-温伯格平衡(<0.05)。其中,“长江1号”中偏离HWE平衡的位点最多,为5个,七里海河蟹和海河自然群体均有3个位点出现显著偏离,“光合1号”偏离位点最少,为2个。
2.2 群体间的遗传分化
4个群体间的st值存在显著差异(<0.05),结果如表4所示,七里海河蟹群体与“长江1号”群体、“光合1号”群体的遗传分化程度较高,达到中等水平(st>0.05);七里海河蟹群体与自然群体、自然群体与“长江1号”群体、自然群体与“光合1号”群体、“长江1号”群体与“光合1号”群体的遗传分化程度处于低水平(st<0.05)。分别对每个位点进行等位基因频率的群体间两两比较,60个比较中有28个差异显著(<0.05) (表6)。
表3 4个中华绒螯蟹群体10个微卫星位点的遗传多样性
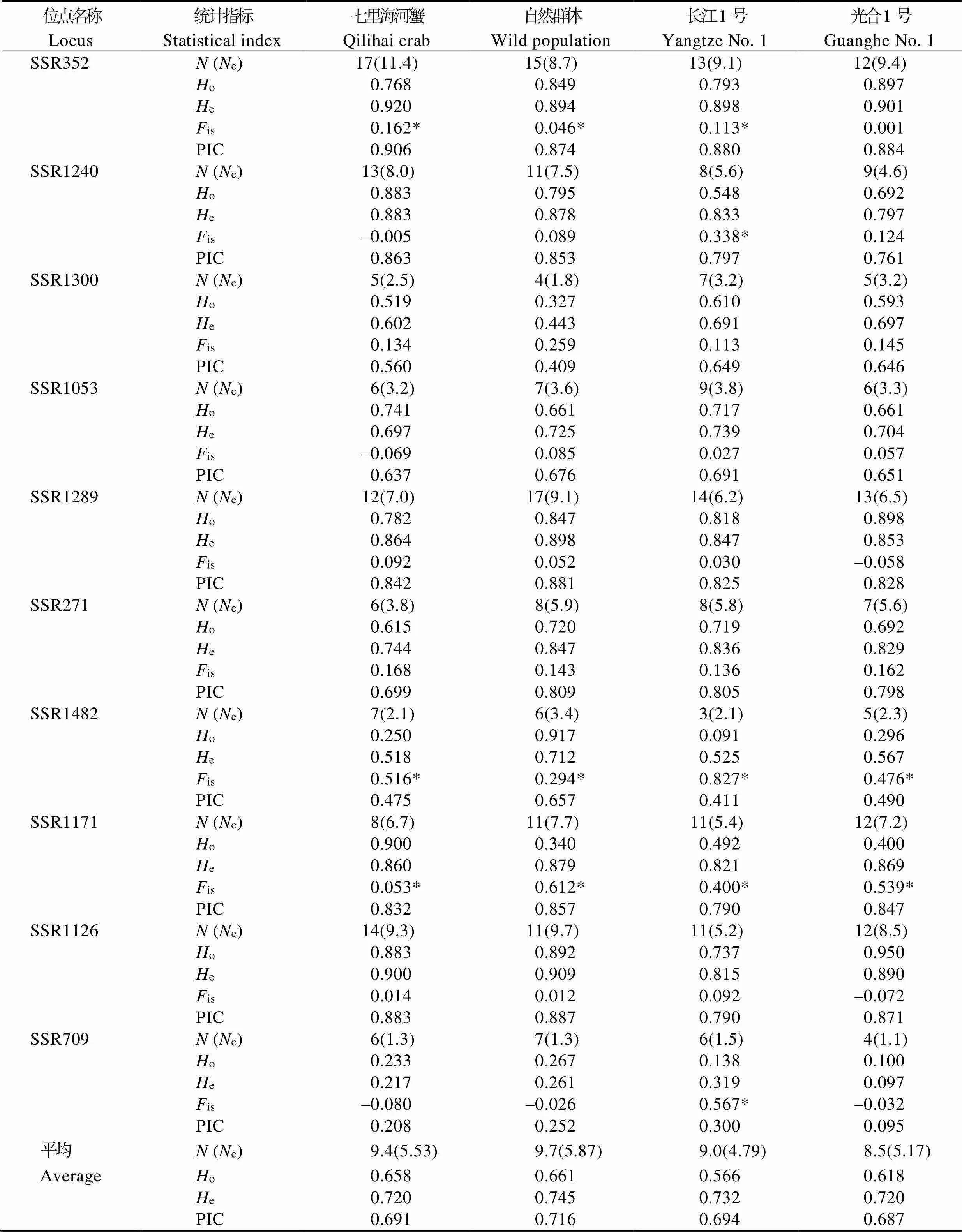
Tab.3 Genetic diversities of ten microsatellite loci in four populations of E. sinensis
注:*表示显著偏离哈迪-温伯格平衡(<0.05)
Note: * means significant deviation from Hardy-Weinberg equilibrium (<0.05)
4个群体两两间的遗传相似度范围为0.7702~ 0.9401,遗传距离(c)范围为0.0617~0.2611,群体间遗传距离越大的遗传相似性越低(表5)。基于Nei’s遗传距离构建的群体UPGMA系统进化树显示,自然群体与“光合1号”聚为一支后,再与“长江1号”聚为一支,而七里海河蟹群体单独聚为一支(图1)。
表4 群体两两间的遗传分化指数(st)
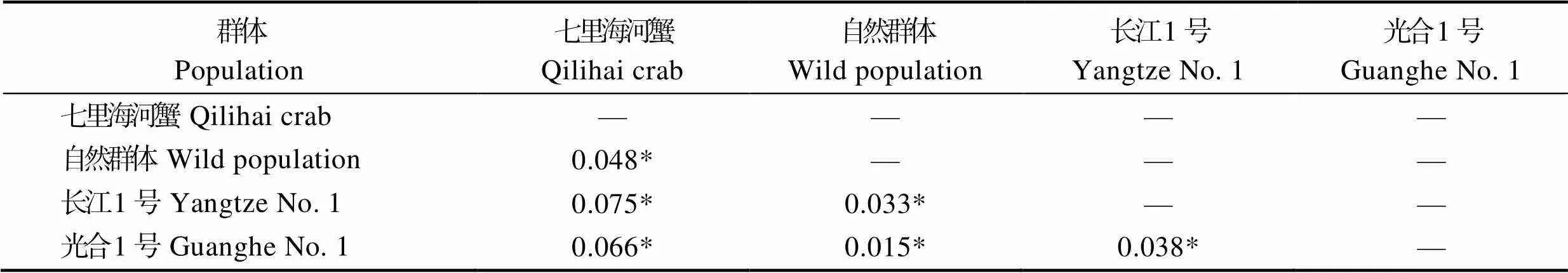
Tab.4 Genetic differentiation index (Fst) between any two populations
注:*表示差异显著(<0.05),下同
Note: * means significant difference (<0.05). The same as below
表5 4个中华绒螯蟹群体的遗传相似度和遗传距离
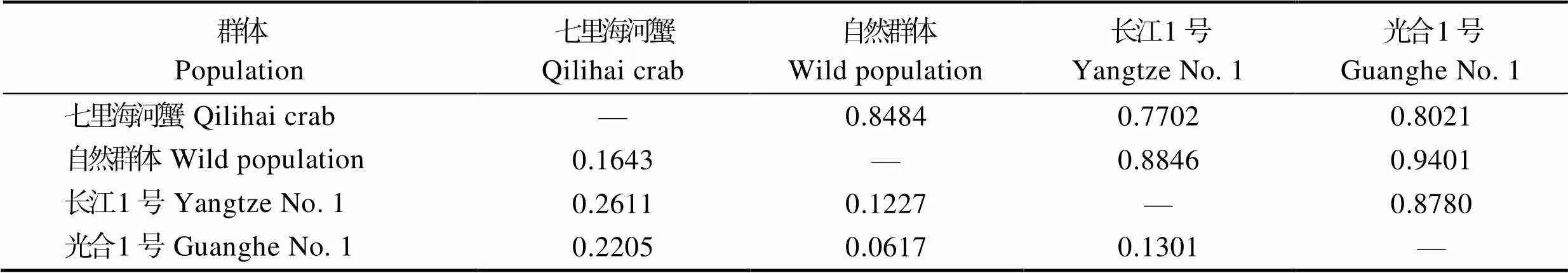
Tab.5 Nei’s genetic identity and genetic distance of the four populations of E. sinensis
注:对角线以上为遗传相似度;对角线以下为遗传距离
Note: The diagonal above was genetic identity; the diagonal below was genetic distance
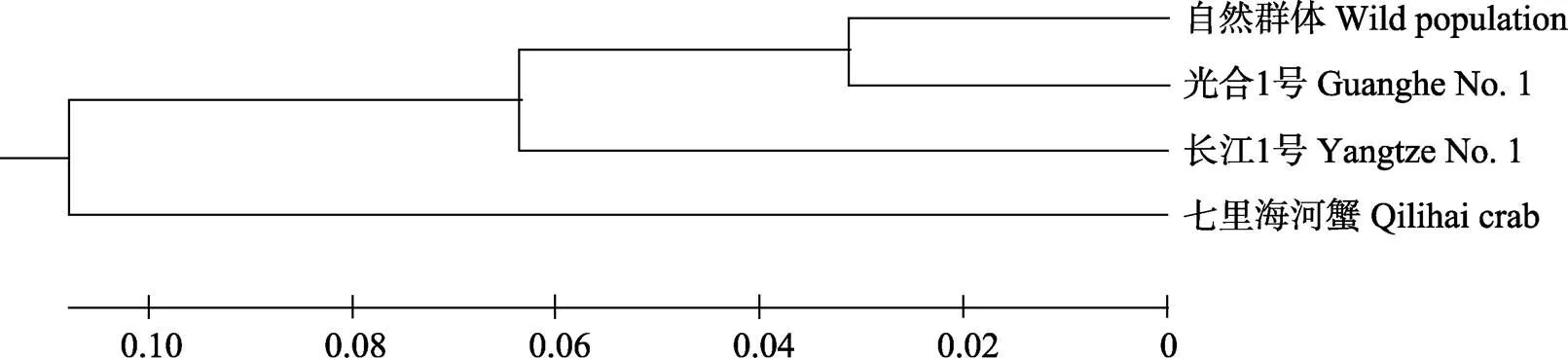
图1 基于Nei’s (1978)遗传距离构建的4个中华绒螯蟹群体的UPGMA系统进化树
表6 10个微卫星位点等位基因频率的群体间两两比较
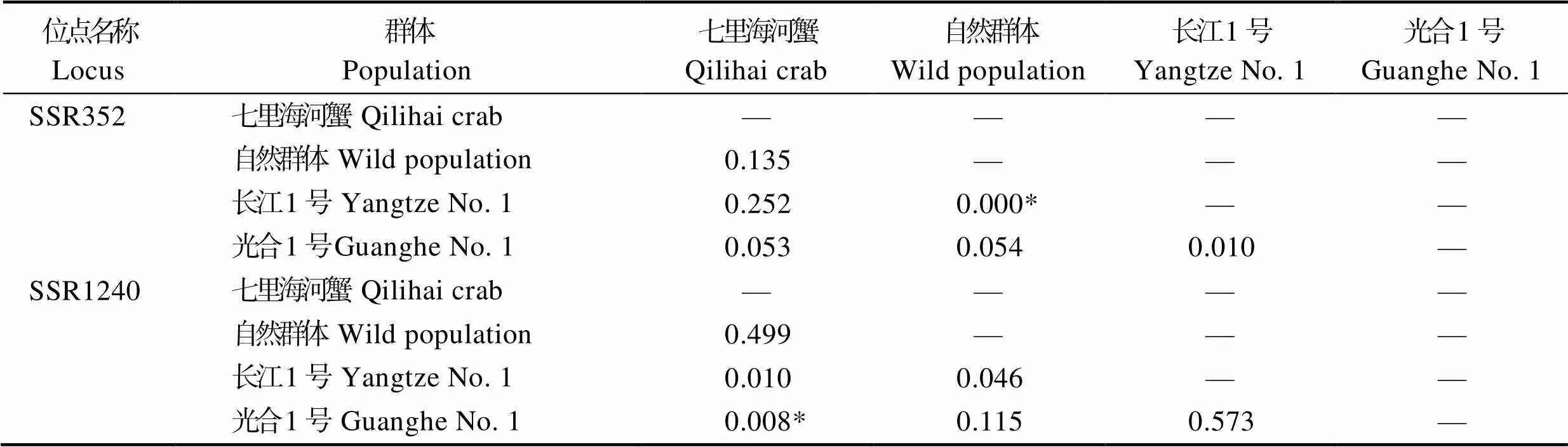
Tab.6 Comparisons of allele frequency in ten microsatellite loci between any two populations
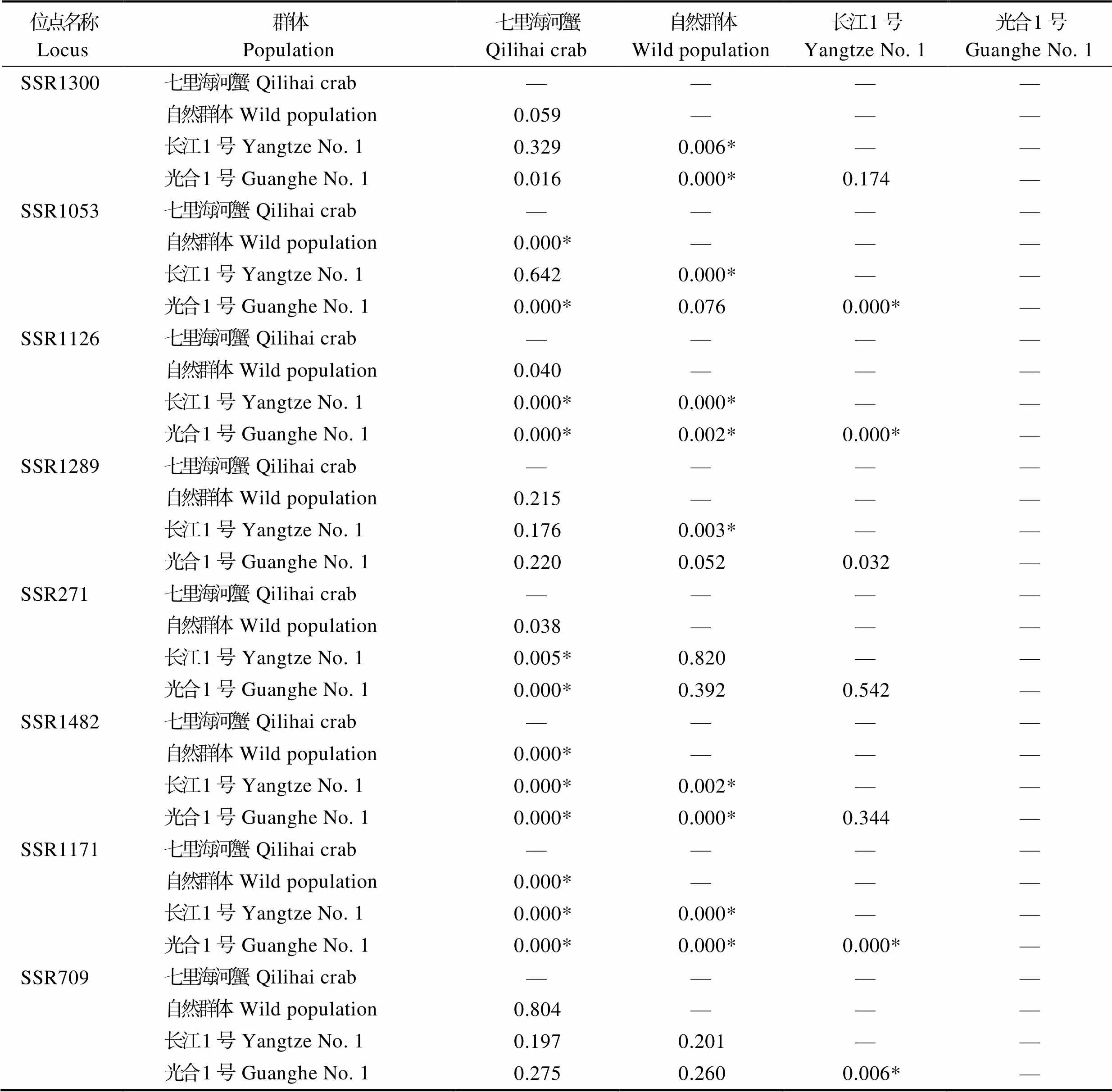
续表6
3 讨论
3.1 中华绒螯蟹自然和人工选育群体遗传多样性
遗传多样性是生物多样性的基础,对物种的生存、繁衍和进化至关重要(张荣良等, 2016)。一个遗传多样性丰富的种群,往往具有较高的适应能力、生存能力和进化潜能,在育种和遗传改良上也更具有潜力,所以对遗传多样性的准确评估是生物多样性保护和可持续利用的基础和前提。人工选择育种就是在保持选育群体具有一定遗传多样性的基础上,筛选获得具有目标性状的选育新品种或新品系(张荣良等, 2016)。大量相关研究表明,养殖群体因人工选择作用和瓶颈效应等原因,其遗传多样性往往低于未经选育的野生群体。张辉等(2010)和王军等(2018)分别应用mtDNA控制区序列和微卫星标记分析了中国对虾()养殖群体与野生群体,显示野生群体的基因多样性高于养殖群体。肖起珍等(2017)利用29对SSR标记对中华绒螯蟹长江水系野生和人工繁育大眼幼体的遗传多样性进行了分析,发现野生群体的平均等位基因数、平均有效等位基因数和平均期望杂合度均高于人工繁育群体,香农信息指数体现的群体遗传多样性野生群体也高于人工繁育群体。在鱼类群体遗传研究中也获得了相似的结果,曾庆凯等(2017)应用SSR标记比较翘嘴鳜()湖南洞庭湖野生群体和广东、安徽养殖群体的遗传多样性,分析结果也显示野生群体的遗传多样性略高于养殖群体。
遗传多样性主要用多态信息含量(PIC)和杂合度()来评估。其中,多态信息含量用于评价标志基因(或标志序列)的多态性大小。PIC>0.5的位点为高多态性位点,0.25 由于连续多代人工选育,不引进外来亲本,所以造成了中华绒螯蟹选育群体遗传多样性的降低,使选育群体的遗传多样性略低于自然群体。但在人工选育过程中,增加有效繁育亲本数量,严格控制选育群体的近交衰退,在一定程度上保证了选育群体的遗传稳定性,使中华绒螯蟹的人工选育群体经过累代繁殖多年之后,依然保持着较高的遗传多样性。 遗传分化指数(st)和遗传距离(c)是衡量群体间遗传差异的重要参数。Wright (1978)认为,st值在0~0.05之间,说明群体间的遗传差异很小;st值在0.05~0.15之间,说明群体间发生了中等水平的遗传分化;在0.15~0.25之间,说明群体间存在较大的遗传差异;当st>0.25时,说明群体间高度遗传分化。本研究中,4个群体两两间比较,七里海河蟹群体与“长江1号”群体、“光合1号”群体间的遗传分化达到中等水平(st>0.05);其他比较组间的遗传分化程度处于低水平(st<0.05)。两两群体间的Nei’s无偏遗传距离为0.0617~0.2611,群体间遗传距离较小。这与肖起珍等(2017)对长江水系中华绒螯蟹野生与人工繁殖群体遗传研究的结果相似。 群体系统进化树显示,自然群体和“光合1号”聚为一支后,再与“长江1号”聚为一支,而七里海河蟹群体单独聚为一支。这可能是由天津地区中华绒螯蟹养殖产业现状导致的。自20世纪中后期,天津海河入海口建设了防潮闸后,海河流域的中华绒螯蟹自然资源逐年减少,同时养殖产业迅速发展。但天津地区遗传育种研究起步晚,育种技术相对落后,本地人工繁育蟹苗量少,远远无法满足当地养殖生产需求。所以,养殖企业大量引进辽河、长江等其他水系苗种,以“光合1号”和“长江1号”居多。在养殖生产过程中,由于养殖群体个体逃逸,可能会与自然群体发生基因交流,从而导致了异地引进群体与自然群体间的遗传距离变小,打破了各水系中华绒螯蟹的原始分布格局。 本研究以中华绒螯蟹3个水系人工选育群体和海河流域自然群体为对象,应用微卫星分子标记技术分析各群体的遗传多样性和群体间的遗传分化。结果表明,4个群体的遗传分化程度处于中低水平,3个人工选育群体的遗传多样性与自然群体相比有一定程度的降低,但仍保持在较高水平上,具有较大的选育潜力。 Botstein D, White RL, Skolnick M,. Construction of genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980, 32: 314–331 Dieringer D, Schlötterer C. Microsatellite analyser (MSA): A platform independent analysis tool for large microsatellite data sets. Molecular Ecology Notes, 2003, 3(1): 167–169 Du HM. Brief discussion of Qilihai crab ecological cultivation. Rural Science and Technology, 2014(3): 29 [杜红梅. 七里海河蟹生态养殖浅谈. 乡村科技, 2014(3): 29] Goudet J. FSTAT, a program to estimate and test gene diversities and fixation indices (version 2.9.3). 2001, Available from http://www.unil.ch/izea/softwares/fstat.html Hardy OJ, Vekemans X. SPAGeDi: A versatile computer program toanalyze spatial genetic structure at the individual or population levels. Molecular Ecology Notes, 2002, 2(4): 618–620 Kimura M, Crow JF. The number of alleles that can be maintained in a finite population. Genetics, 1964, 49(4): 725–738 Li B. Ecology. Beijing: Higher Education Press, 2000, 338–341 [李博. 生态学. 北京: 高等教育出版社, 2000, 338–341] Li JJ, Geng XY, Chen LM,. Identification and characterizationof thirty novel microsatellite DNA markers from the Chinesemitten crabexpressed sequence tags. BMC Research Notes, 2016, 105(9): 1–5 Li JJ, Geng XY, Dong XW,. Evaluation of visible implant elastomer tags in Chinese mitten crab. Marine Fisheries, 2016, 38(4): 400–405 [李晶晶, 耿绪云, 董学旺, 等. 中华绒螯蟹可视荧光标记应用研究. 海洋渔业, 2016, 38(4): 400–405] Ma HT, Chang YM, Yu DM,. Microsatellite variations among four populations of. Zoological Research, 2007, 28(2): 126–133 Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics, 1978, 89(3): 583–590 Nei M. Molecular evolutionary genetics. New York: Columbia University Press, 1987 Qin Y, Shi G, Sun Y. Evaluation of genetic diversity inusing SSR markers. Genetics and Molecular Research, 2013, 12(4): 5833–5841 Raymond M, Rousset F. Population genetics software for exact tests and ecumenicism. Journal of Heredity, 1995, 86(3): 248–249 Rice WR. Analyzing tables of statistical tests. Evolution, 1989, 43(1): 223–225 Takezaki N, Nei M. Genetic distances and reconstruction of phylogenetic trees from microsatellite DNA. Genetics, 1996, 144(1): 389–399 Tamura K, Peterson D, Peterson N,. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular Biology and Evolution, 2011, 28(10): 2731–2739 Wang CH, Li SF. Advances in studies on germplasm in Chinese mitten crab,. Journal of Fishery Sciences of China, 2002, 9(1): 82–86 [王成辉, 李思发. 中华绒螯蟹种质研究进展. 中国水产科学, 2002, 9(1): 82–86] Wang J, Wang QY, Kong J,. SSR Analysis on genetic diversity in breeding and wild populations of. Progress in Fishery Sciences, 2018, 39(2): 104– 111 [王军, 王清印, 孔杰, 等. 中国明对虾人工选育群体与野生群体遗传多样性的SSR分析. 渔业科学进展, 2018, 39(2): 104–111] Weir BS, Cockerham CC. Estimating F-statistics for the analysis of population structure. Evolution, 1984, 38(6): 1358–1370 Wright S. Evolution and the genetics of population variability within and among natural population. University of Chicago Press, Chicago, 1978, 121–124 Xiao QZ, Liu Q, Wu XG,. Genetic diversity analysis of wild and cultured megalopa population offrom Yangtze River. Genomics and Applied Biology, 2017, 36(5): 1935–1945 [肖起珍, 刘青, 吴旭干, 等. 长江水系中华绒螯蟹野生和人工繁殖大眼幼体的遗传多样性分析. 基因组学与应用生物学, 2017, 36(5): 1935–1945] Zeng QK, Sun CF, Dong JJ,. Analysis of genetic diversity in three different populations of. Genomics and Applied Biology, 2017, 36(8): 3241–3250 [曾庆凯, 孙成飞, 董浚键, 等. 翘嘴鳜3个不同群体的遗传多样性分析. 基因组学与应用生物学, 2017, 36(8): 3241–3250] Zhang H, Gao TX, Zhuang ZM,. Comparative analysis of the mitochondrial control region between the cultured and wild population of Chinese shrimp (). Journal of Fisheries of China, 2010, 34(8): 1149–1155 [张辉, 高天翔, 庄志猛, 等. 中国对虾养殖群体与野生群体线粒体控制区序列的比较. 水产学报, 2010, 34(8): 1149–1155] Zhang RL, Wang WJ, Feng YW,. Assessment of genetic variability and microsatellite analysis of Pacific oyster () after artificial selection of the shell width. Progress in Fishery Sciences, 2016, 37(4): 90–96 [张荣良, 王卫军, 冯艳微, 等. 长牡蛎()壳宽快速生长选育群体遗传多样性及遗传结构的微卫星标记分析. 渔业科学进展, 2016, 37(4): 90–96] Zhu GQ. Integration and application of key technologies for ecological cultivation of Qilihai crab. Tianjin Fisheries, 2012(1): 35–39 [朱广奇. 七里海河蟹生态养殖关键技术集成与应用. 天津水产, 2012(1): 35–39] Genetic Diversity Analysis of Wild and Cultured Population offrom Different Water Systems LI Jingjing1,2, CHEN Limei3, GENG Xuyun2, DONG Xuewang2, SUN Jinsheng1① (1. College of Life Sciences, Tianjin Normal University, Tianjin 300387; 2. Tianjin Diseases Prevention and Control Center of Aquatic Animals, Tianjin 300221; 3. Tianjin Key Laboratory of Aqua-Ecology and Aquaculture, Fisheries College, Tianjin Agricultural University, Tianjin 300384) The Chinese mitten crabis one of the most economically important aquaculture species in China due to its taste and nutritional value. In this study, the genetic status of both wild and cultivated stocks ofwas assessed using simple sequence repeat (SSR) markers. Ten microsatellite markers reported in previous studies were selected to estimate the level of genetic diversity within one Haihe natural population and three selected artificial populations (Yangtze No.1, Guanghe No.1, and Qilihai crab) and to compare the degree of genetic differentiation among them. Unique PCR products and high levels of polymorphism were observed for all loci. For the four populations of, the number of alleles per locus ranged from 3 to 17, with a mean allele number of 8.5 to 9.7. The mean observed heterozygosities ranged from 0.566 to 0.661, while the mean expected heterozygosities ranged from 0.720 to 0.745. Additionally, the mean polymorphism information content ranged from 0.687 to 0.716. Furthermore, the inbreeding coefficient (F) ranged from-0.080 to 0.827, and there were 13 cases that significantly deviated from the Hardy-Weinberg equilibrium (<0.05) among the 40 population-locus cases (4 populations × 10 loci). The results of the microsatellite survey indicated that the genetic diversity of the three artificially selected populations was slightly lower than that of the natural population, but still maintained at a high level for further breeding. The analysis of genetic differentiation showed thatFranged from 0.015 to 0.075, genetic similarity ranged from 0.7702 to 0.9401, and genetic distance ranged from 0.0617 to 0.2611. UPGMA phylogenetic analysis divided these populations into two groups. Natural population and Guanghe No.1 stocks were allocated to the same cluster, and the Qilihai crab was allocated to another cluster. In conclusion, the four populations ofpossessed high genetic diversity but low to moderate levels of genetic differentiation. The information on the genetic variation and differentiation obtained in this study will provide a theoretical basis for further breeding and utilization of germplasm resources of. ; Genetic diversity; Genetic differentiation; Microsatellite S968.2 A 2095-9869(2019)06-0105-09 10.19663/j.issn2095-9869.20180831002 http://www.yykxjz.cn/ 李晶晶, 陈丽梅, 耿绪云, 董学旺, 孙金生. 中华绒螯蟹野生群体和不同水系人工选育群体的遗传多样性分析. 渔业科学进展, 2019, 40(6): 105–113 Li JJ, Chen LM, Geng XY, Dong XW, Sun JS. Genetic diversity analysis of wild and cultured population offrom different water systems. Progress in Fishery Sciences, 2019, 40(6): 105–113 * 天津市财政资金项目(七里海优质河蟹繁养技术示范应用)、天津市水产局科研推广项目(J2013-21)、天津市水产产业技术体系专项(ITTFRS2017002)、天津市种业科技重大专项(15ZXZYNC00050)和天津市自然科学基金项目(16JCQNJC09500)共同资助 [This work was supported by Project of Tianjin Financial Funds (Demonstration and Application of Qilihai Crab Breeding and Culture Technology), Research and Extension Project of Tianjin Aquatic Products Bureau (J2013-21), Tianjin Aquaculture Research System (ITTFRS2017002), Major Project for Tianjin Seed Technology (15ZXZYNC00050), and Tianjin Natural Science Foundation (16JCQNJC09500)]. 李晶晶, E-mail: jingjingli206@163.com 孙金生,研究员,E-mail: jinshsun@163.com 2018-08-31, 2018-10-20 SUN Jinsheng, E-mail: jinshsun@163.com (编辑 冯小花)3.2 群体间的遗传分化

