深度迁移学习在花生叶部病害图像识别中的应用
苏婷婷,牟少敏*,董萌萍,时爱菊
深度迁移学习在花生叶部病害图像识别中的应用
苏婷婷1,牟少敏1*,董萌萍1,时爱菊2
1. 山东农业大学信息科学与工程学院, 山东 泰安 271018 2. 山东农业大学化学与材料科学学院, 山东 泰安 271018
运用深度学习方法对花生叶部病害图像进行分类时,需要大量训练样本。而花生叶部病害发生时间较短,多呈地域性分布,为叶部病害图像的采集增加难度。为降低图像采集难度,减少图像标注成本,提出了一种基于微调策略的Inception-v3花生叶部病害图像识别方法。首先迁移Inception-v3模型在茶叶叶部病害图像数据集上进行微调,然后将微调所得模型迁移至花生叶部病害图像数据集上继续训练。实验结果表明,Inception-v3、VGG19、ResNet50、卷积神经网络和微调卷积神经网络在测试集上的准确率分别为94.1%、92.9%、93.2%、80.3%和87.0%,本文提出的方法准确率为95.3%。在花生叶部病害图像数量有限时,本文提出的方法识别准确率显著提高。
迁移学习; 预训练模型; 花生叶部病害; 图像分类
花生是我国重要的油料和经济作物之一,种植范围广泛,其产量和质量对国民经济的发展有重要意义,而病害的发生对其产量和质量造成严重影响,因此花生病害的及时诊断与防治在其生长过程中具有重要意义。目前,常用的花生叶部病害识别的方法主要依靠相关专家、研究人员及务农人员的专业知识和经验进行判断,效率较低且费时费力,在一定程度上影响了花生病害的防治工作。
近年来,深度学习[1]迅猛发展并应用于农作物叶部病害图像识别领域。运用深度学习方法可以取得理想的效果,但前提是需要海量的标签数据。当数据集中含有少量标签数据时,训练集与测试集效果相差较大。而现实中标签数据匮乏,且标注大量数据需要耗费大量人力物力。在农业领域,由于受到季节、地域分布等影响,病害发生时间相对较短,并且部分病害在不同区域存在差异,在较短时间内完成大量病害图像的采集工作比较困难。
迁移学习是一种能够借助已有领域的知识对不同但相关领域问题求解的机器学习方法,用于解决目标任务中标签数据匮乏的问题[2,3]。目前,迁移学习已用于农作物病害图像识别领域。Brahimi等[4]基于番茄叶部病害图像数据集对AlexNet模型进行调整,不仅具有较高的识别准确率,而且在一定程度上减少了由病斑分割对识别效果造成的影响。Sladojevic等[5]运用微调的CaffeNet模型对包含13种病害、1种背景图像和1种正常叶片的15类植物叶片识别,每类正确识别率均在91%以上。
针对当前花生叶部病害图像数量相对较少的问题,引入深度迁移学习思想,提出了一种基于微调策略的Inception-v3(Finetuning-Inception-v3)花生叶部病害图像识别方法。实验结果表明,本文提出的方法在测试集上取得了较好的效果。
1 实验数据
1.1 茶叶叶部病害图像数据集
茶叶叶部病害和花生叶部病害在病害类型与表现特征上存在一定的相似性,因此本文将已构建好的茶叶叶部病害图像数据集作为辅助源域。该数据集中包含炭疽病、机械损伤等多种茶叶叶部病害图像,本文选择正常叶片、叶枯病、炭疽病和茶轮斑病四类,每类选取5000~6000张不等,划分训练集、验证集和测试集。
1.2 花生叶部病害图像数据集
本文将花生叶部病害图像作为目标域。在山东省泰安市周边花生种植地区,人工对花生发病叶片进行采集。以A4纸为拍摄背景,手机为拍摄工具进行拍摄,并按病害种类进行分类。目前已构建完成包含正常叶片及褐斑病等病害图像数据集。本实验选择正常叶片、黑斑病、褐斑病和炭疽病四类。将花生叶部病害图像输入到模型前,需对图像进行预处理。首先按照网络输入图片尺寸要求对所有图片进行裁切,包括224*224和299*299两种。其次由于受到发病率和发病地区等影响,每类病害图像数量400~2000张不等,为减少因数据集中图像数量不均造成的模型泛化能力的影响,对图像数量较少的类别进行图像增强。利用Python对图像进行翻转、旋转等操作以增加样本的多样性。最终获得6700余张图像,并划分训练集、验证集和测试集。
2 卷积神经网络与预训练模型
2.1 卷积神经网络
深度学习本质是通过构建多层神经网络模型,利用海量标签数据,获取数据的特征表示,从而提升分类或预测的准确性[6],已成为当今机器学习领域中最受关注的研究方向。卷积神经网络(Convolutional Neural Networks, CNN)是一种经典的深度学习模型,因具有较好的性能和鲁棒性而广泛应用于图像分类、自然语言分类等领域。传统的CNN通过构建多个隐藏层,根据特定任务,完成对图像从底层细节到高层表征的学习过程[7],特点是局部连接、权值共享和下采样。CNN主要包括卷积层、池化层和全连接层,模型结构如图1所示。

图1 卷积神经网络结构
2.1.1 卷积层卷积层通过卷积运算完成图像特征提取的工作。卷积核以滑动窗口的形式在输入图像上进行逐行扫描,计算图像与网络参数的卷积操作[8],得到图像的特征图表达矩阵,同时再通过激活函数生成下一层的特征图矩阵。其中,底层卷积层用于提取如形状、边缘等低级特征,高层卷积层用于提取如组合性特征等抽象复杂特征。

2.1.2 池化层 池化层又称为下采样层,通常存在卷积层后,通过一系列局部非线性操作对输入特征进行特征选择和降维,以减少模型参数,并提高网络对输入图像平移旋转等抗畸变的能力。池化操作主要有两种:最大池化和平均池化。
2.1.3 全连接层 全连接层出现在多个卷积层与池化层交替堆叠之后,在对提取的特征进行进一步降维,将特征输入到SoftMax层中。
对于输入的样本(,),其中为样本所属标签类别,根据公式(2)得其所属类别。

2.2 预训练模型
CNN模型参数庞大,若针对某一任务训练获得较为理想的模型,需要十万甚至百万标签数据。近年来随着研究的不断深入,出现了预训练模型。预训练模型是针对某一特定任务,基于大型数据集(如ImageNet)上训练而得的模型。当解决类似任务时,不需要构建海量数据集,也不需要重新构建模型,只需将预训练模型迁移到新的任务中,根据任务对模型进行微调即可。
ImageNet数据集模拟人类识别系统建立,是目前世界上最大的图像识别数据库,包含超过1400万张标记图片。Large Scale Visual Recognition Challenge(ILSVRC)使用的公开数据集为Imagenet的子集。ILSVRC中产生如VGG等经典模型。
Inception-v3[9]由Google团队于2015年提出,该模型对GoogleNet模型改进,网络前几层由普通卷积层和最大池化层组成,随后由不同结构的Inception模块堆叠而成,其中Inception模块有35*35,17*17和8*8三种非对称的卷积结构,在节约大量参数、减轻过拟合的同时,增加特征的多样性。
3 迁移学习
迁移学习可以对已有的知识进行利用,解决目标领域中标签数据匮乏的问题。由于茶叶叶部病害与花生叶部病害在病害类型与表现特征上存在一定的相似性,因此本文选择将茶叶叶部病害图像数据集作为辅助源域,一方面可以降低目标任务对标签样本数量的需要,进而达到降低数据采集难度、节省人工标注的目的;另一方面,对已构建完成的茶叶叶部病害图像数据集进行充分利用,在一定程度上提高资源利用率。
根据不同的分类标准,迁移学习有多种分类方式。根据迁移内容的不同,迁移学习可分为基于实例的迁移、基于特征的迁移、基于参数的迁移和基于知识的迁移。本文采用基于参数的迁移,将ImageNet数据集作为源域,茶叶叶部病害图像数据集作为辅助源域,花生叶部病害图像作为目标域。对花生叶部病害图像分类时,模型初始参数来源于基于茶叶叶部病害图像微调所得的模型参数。
4 实验结果与分析
4.1 评价标准
为对模型效果进行评价,采用平均准确率作为评价指标:

其中,n为每批次预测正确的样本数;n为每次迭代的批次数,n为每批次输入的样本总数。
4.2 实验结果及分析
本次实验运行环境为Ubuntu16.04,Tensorflow+Keras框架,训练过程中使用GPU加速。
实验流程如图2所示,首先迁移预训练模型Inception-v3在茶叶叶部病害图像数据集上进行微调,然后将所得模型参数迁移至花生叶部病害图像数据集上进行训练。
采用有监督的方式对模型进行训练,分为前向传播阶段和反向传播阶段。训练前对网络参数进行初始化,通常采用随机初始化和置零法两种,而本文采用基于茶叶叶部病害图像数据集微调后所得模型参数作为网络的初始参数。前向传播阶段,将花生叶部病害图像输入到网络中,由若干卷积层与池化层进行特征提取,并将所得特征输入至Softmax分类器,输入其所对应标签,计算损失函数。反向传播阶段,通过逐层计算损失函数与网络参数的偏导完成权值与偏置的更新。训练过程中不断最小化损失函数以提高分类准确率,直至损失函数收敛。
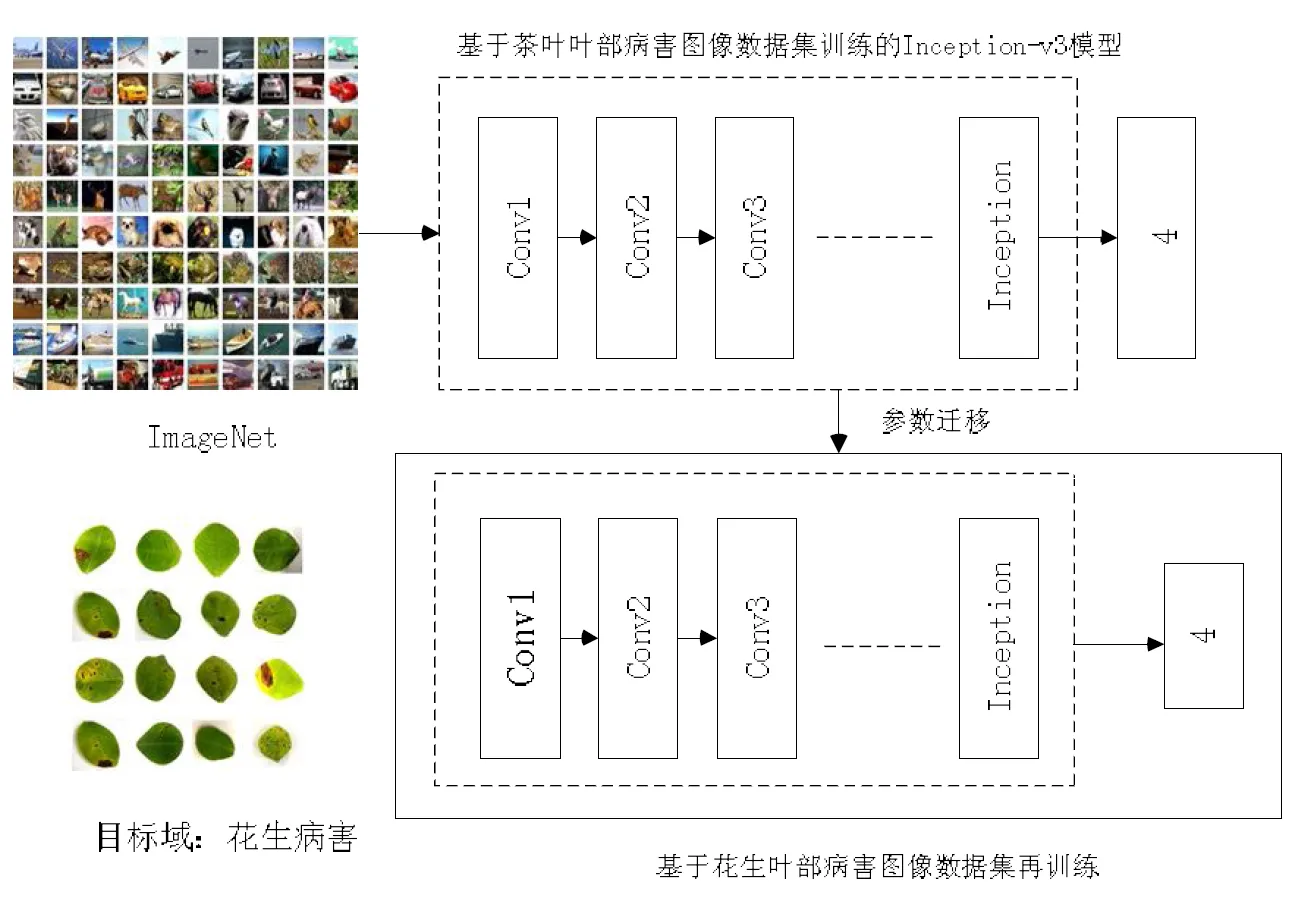
图2 实验流程
为验证本文提出方法的有效性,选择Inception-v3、VGG19[10]、ResNet50[11]、CNN和Finetuning-CNN进行对比。Inception-v3、VGG19、ResNet50为Keras库中的预训练模型。CNN模型含有4个卷积层、2个池化层和3个全连接层,分类器为Softmax,其模型结构如图1所示,模型参数初始化方式为随机初始化。Finetuning-CNN模型结构与CNN相同,首先将CNN模型参数随机初始化,然后在茶叶叶部病害图像数据集上进行训练,最后将训练模型参数迁移至花生叶部病害图像数据集上继续训练。
Finetuning-Inception-v3等模型训练时参数设置如表1所示。训练时,Epoch设置为200,batch_size设置为32,运用SGD方法对损失函数进行优化,而SGD方法容易引起更新参数不稳定的问题,因此引入Momentum作为其历史梯度权重系数,将其值设置为0.9。

表1 Finetuning-Inception-v3等模型参数设置
图3为训练集和测试集每类为750张,测试集为150张时,Finetuning-Inception-v3等模型准确率曲线。Epoch=0时,Finetuning-Inception-v3在训练集和测试集准确率分别为97.8%和79%,均高于Inception-v3等其他模型,经过少量Epoch迭代后,模型即可稳定。Inception-v3等预训练模型和Finetuning-CNN在测试集上的效果均高于CNN,由此可以说明,对模型进行训练时,参数迁移的方法比随机初始化效果好,且源域标签数据量越大,种类越多,泛化能力越强。
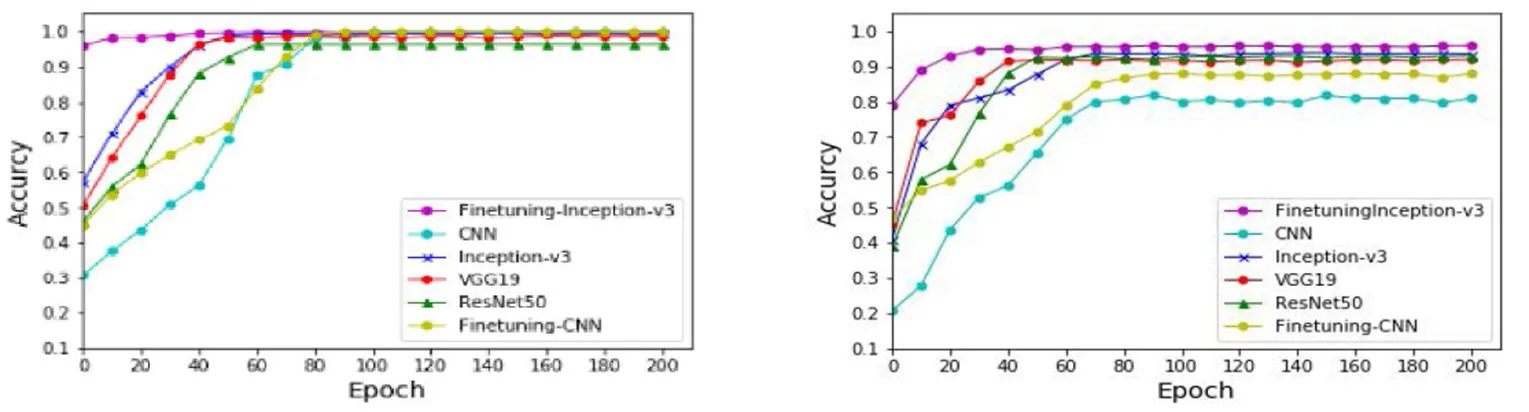
(a)训练集准确率变化曲线 Changing curves of precision in training set(b)测试集准确率变化曲线 Changing curves of precision in test set
表2为训练集和测试集最终准确率。由表2可知,CNN训练集准确率为99.8%,而测试集的准确率为80.3%,训练集与测试集相差略大,此时可认为训练集图像数量较少,模型学习能力较弱,产生过拟合。Finetuning-CNN模型参数源域以茶叶叶部病害图像作为源域的模型,但训练集与测试集效果相差12.9%,效果不理想。直接迁移的Inception-v3等三类预训练模型在测试集上的效果均优于CNN和Finetuning-CNN,三类模型基于ILSVRC使用的数据集训练而得,此数据集中含多达千类,数量超过百万图像,基于此数据集训练所得模型具有较好的泛化能力。而本文提出的Finetuning-Inception-v3效果较好。将Inception-v3模型参数基于茶叶叶部病害图像数据集微调时,尽管茶叶叶部病害图像与花生叶部病害图像在外观上存在一定差距,但两者轮廓、纹理和色彩等某些局部特征具有极大的相似性,因此微调后的模型更加适应于花生叶部病害图像分类。

表2 Finetuning-Inception-v3等模型训练结果
表3为测试集图像数量为150时分类所得的混淆矩阵。

表3 分类混淆矩阵
4 结论
本文提出了一种基于Finetuning-Inception-v3的花生叶部病害图像识别方法,用于解决由于数据采集难度大、标注成本高等原因造成花生叶部病害图像数量较少的问题。首先迁移Inception-v3模型到茶叶叶部病害图像数据集上进行微调,并将微调所得模型参数迁移至花生叶部病害图像数据集继续训练。结果表明,与Inception-v3、VGG19、ResNet50、CNN和Finetuning-CNN相比,本文提出的方法在测试集上效果较好。将茶叶叶部病害图像数据集作为辅助源域时,源域中标签图像种类多且数量庞大,训练所得模型参数具有较好的泛化能力,而在茶叶叶部病害图像数据集中进行微调后,经过小幅度训练即可用于花生叶部病害图像分类。本文提出的方法可较好解决目标任务中病害图像数据量不足的问题,可以减少图像采集和人工标注的工作量。
[1] Hinton GE, Salakhutdinov RR. Reducing the dimensionality of data with neural networks[J]. Science, 2006,313(5786):504-507
[2] Pan SJ, Yang Q. A Survey on Transfer Learning[J]. IEEE Transactions on Knowledge and Data Engineering, 2010,22(10):1345-1359
[3] 庄福振,罗平,何清,等.迁移学习研究进展[J].软件学报,2015,26(1):26-39
[4] Brahimi M, Kamel B, Moussaoui A. Deep Learning for Tomato Diseases: Classification and Symptoms Visualization[J]. Applied Artificial Intelligence, 2017,31(4):299-315
[5] Sladojevic S, Arsenovic M, Anderla A,. Deep Neural Networks Based Recognition of Plant Diseases by Leaf Image Classification[J]. Computational Intelligence and Neuroscience, 2016(6):1-11
[6] Mohanty SP, Hughes DP, Salathé M. Using Deep Learning for Image-Based Plant Disease Detection[J/OL]. Frontiers in Plant Science, 2016(7):1419. doi:10.3389/fpls.2016.01419
[7] 李彦冬,郝宗波,雷航.卷积神经网络研究综述[J].计算机应用,2016,36(9):2508-2515,2565
[8] 张军阳,王慧丽,郭阳,等.深度学习相关研究综述[J].计算机应用研究,2018(7):1-12
[9] Szegedy C, Vanhoucke V, Ioffe S,. Rethinking the Inception Architecture for Computer Vision[J]. 2015:2818-2826
[10] Simonyan K, Zisserman A. Very Deep Convolutional Networks for Large-Scale Image Recognition[C]. Conference paper in ICLR, 2015:1-14
[11] He KM, Zhang XY, Ren SQ,. Deep Residual Learning for Image Recognition[C]. IEEE Conference on Computer Vision and Pattern Recognition (CVPR), 2016:770-778
Application of Deep Transfer Learning in Image Recognition of Peanut Leaf Diseases
SU Ting-ting1, MU Shao-min1*, DONG Meng-ping1, SHI Ai-ju2
1.271018,2.271018,
Massive training samples are needed when classifying peanut leaf disease images using deep learning. Due to the short occurrence time and the regional differences of peanut leaf diseases, it is more difficult to collect abundant leaf disease images. In order to reduce the difficulty of image acquisition and the cost of image labeling, an Finetuning-Inception-v3 image recognition method for peanut leaf diseases is proposed. Firstly, the Inception-v3 model is transferred for fine-tuning on the tea leaf disease image data set. Then, the fine-tuning model is transferred to the peanut leaf disease image data set for further training. The experimental results show that , the accuracy on the test dataset using the proposed method is 95.3%, while the accuracy of Inception-v3, VGG19, ResNet50, Convolutional Neural Networks(CNN) and Finetune-CNN is 94.1%, 92.9%, 93.2%, 80.3% and 87.0%, respectively. Therefore, the recognition rate is significantly improved using the proposed Finetuning-Inception-v3 when the number of peanut disease images is small.
Transfer Learning; pre-trained model; leaf disease of peanut; image classification
S126
A
1000-2324(2019)05-0865-05
10.3969/j.issn.1000-2324.2019.05.028
2018-10-28
2019-01-14
苏婷婷(1991-),女,硕士研究生,主要从事机器学习与人工智能研究及其在农业中的应用. E-mail:m15065806906@163.com
Author for correspondence. E-mail:msm@sdau.edu.cn

