异源表达古菌TRAM基因增强水稻的耐旱性
陈巍,李华丽,邱金龙
异源表达古菌TRAM基因增强水稻的耐旱性
陈巍1, 2,李华丽1,邱金龙1
1 中国科学院微生物研究所,北京 100101 2 中国科学院大学,北京 100049
干旱会直接影响水稻的生长发育,导致其产量和品质下降。在水稻中异源表达细菌RNA分子伴侣Csp能够显著提高水稻的耐旱能力,并且不影响水稻的正常生长。古菌中也发现具有类似细菌分子伴侣Csp功能的TRAM (TRM2 and MiaB) 蛋白,且古菌的DNA复制、转录和翻译等过程与真核生物有着更为相似的调控方式,然而,古菌中RNA分子伴侣蛋白能否调控植物耐旱能力还未见报道。我们选取了嗜冷甲烷古菌R15中两个TRAM蛋白在水稻中进行研究,发现在水稻中过量表达Mpsy_3066和Mpsy_0643两个TRAM蛋白均能显著提高水稻苗期和成株期时对干旱胁迫的耐受能力。同时,我们在水稻原生质体中验证了TRAM蛋白可以发挥其分子伴侣的功能消除RNA的错误折叠对翻译的影响,这可能是TRAM转基因植物发挥其耐旱能力的作用机制。该工作初步展示了异源表达古菌TRAMs可以作为提高水稻耐旱能力的一种有效手段。
水稻,干旱胁迫,RNA分子伴侣,古菌,TRAM
水稻是世界上最重要的粮食作物之一,为世界上超过一半的人口提供了主粮[1]。然而水稻的生长发育需要大量的水分供给,干旱或者供水不足会对水稻的产量和品质产生巨大的影响[2]。特别是近年来全球气候变暖,年降水量不稳定且分布不均,对农业用水特别是水稻等农作物的灌溉用水造成了一定的压力,威胁着粮食安全[3]。因此,提高水稻的抗旱能力是促进粮食增产的一个有效途径。植物在干旱胁迫下会发生一系列的生理应激反应,包括气孔开度变化、胞内活性氧清除酶的合成、渗透调节小分子物质(例如可溶性糖、甘氨酸、脯氨酸) 的积累、叶片卷曲以及叶表面角质层加厚等[4]。基于基因工程的方法将干旱胁迫相关基因编辑使之功能失活或向植物基因组中导入外源基因以得到稳定表达的植株,也可增强植物抵抗干旱胁迫的耐受性[5]。
转录后水平的基因表达调控对真核生物的生长发育和响应外部环境刺激至关重要[6-8]。RNA分子伴侣(RNA chaperone) 是一类广泛存在于动物、植物和微生物中的RNA结合蛋白。RNA分子伴侣通过与非功能性折叠的RNA底物结合,帮助其重排并形成正确的折叠方式或帮助RNase降解错误折叠的RNA,完成正常的RNA代谢过程[9]。在应激状态下,一些RNA分子伴侣的表达量会显著增加,以此来帮助生物体更好地应对胁迫[10-11]。在水稻和玉米中表达细菌中的RNA分子伴侣蛋白——冷激蛋白(Cold shock protein,Csp) CspA和CspB,可以有效地提高其抗冻、抗旱以及抗高温的能力[12]。在拟南芥和小麦中表达密码子优化的CspA和CspB蛋白,也可以提高它们的抵抗干旱和盐胁迫的能力[13]。在水稻中过表达拟南芥RNA伴侣蛋白GRP2和GRP7同样可以显著提高干旱条件下水稻的结实率,从而提高水稻的产量[14]。DEAD-box RNA解旋酶(DEAD-box RHs) 家族成员也具有RNA分子伴侣活性,拟南芥中该家族成员RH8通过与PP2CA互作,对ABA信号和干旱胁迫反应具有重要的调节作用[15]。
古菌作为生物学分类中一个庞大的分支,其RNA分子伴侣蛋白对植物耐旱能力的调控却没有报道。在古菌DNA的复制、转录和翻译过程中,都可以看到与真核生物更为类似的蛋白结构和调控方式[16],且古菌多在极端环境下生存,其本身即具有更强的耐受性。因此,我们希望在古菌中找到类似细菌冷激蛋白的RNA分子伴侣,检测其是否能使植物具有更好的耐受干旱胁迫的能力。虽然Csp类冷激蛋白在细菌中分布广泛,且在同一物种中含有多个同源蛋白,在古菌中却鲜有其分布[17]。有趣的是,在古菌中存在一类与冷激蛋白结构类似的蛋白TRAM (TRM2 and MiaB)。TRAM是一类具有RNA结合功能的相关结构域的总称,包括尿嘧啶甲基化酶TRM2和腺嘌呤硫醇化酶MiaB,这类结构域在包括古菌在内的多种生物体中普遍存在[18]。TRAM蛋白的三维结构已解析,其由5个反向平行的β链组成寡核苷酸/寡糖结合折痕区域(Oligonucleotide/oligosaccharide binding fold,OB-fold),OB-fold结构域一般由70–150个氨基酸组成,通常被认为与核酸识别有关,该结构域家族成员之间序列相似性很低,却具有多个相似的结构[19]。Csp类蛋白也是由类似结构域组成且与TRAM具有结构同源性。体内和体外的实验证明,TRAM蛋白同细菌中Csp蛋白功能相似,可以回补大肠杆菌突变体的冷敏感性。TRAM蛋白与底物RNA的结合无明显序列特异性,且亲和能力在微摩尔级别,这与RNA分子伴侣蛋白相符合,进一步说明其可能就是存在于古菌中的RNA分子伴侣蛋白[10]。
我们从已知的嗜冷甲烷古菌(R15) 4个TRAMs基因中选取了2个可以显著改善大肠杆菌突变体冷敏感性的TRAM基因和,分别在水稻中进行过量表达,检测了其对水稻耐受干旱胁迫的影响,并初步探究了其作用机制。
1 材料与方法
1.1 水稻转基因载体的构建及转化
在 GenBank 数据库中分别获取噬冷甲烷古菌(M. psychrophilus R15) 中两个TRAM蛋白Mpsy_3066 (AFV25265) 和Mpsy_0643 (AFV22852) 的氨基酸序列,参考水稻密码子偏好性,由南京金斯瑞公司对其进行密码子优化并合成相应编码DNA序列。分别将该序列通过SpeⅠ和KpnⅠ酶切位点克隆到双元表达载体pCAMBIA2300-Ubi[20]中,以玉米泛素启动子驱动目的基因的表达,构建的载体命名为pC2300-Mpsy-3066和pC2300- Mpsy-0643。电激法将构建好的载体转入农杆菌AGL1中,利用农杆菌转化日本晴水稻(L. ssp.c.v. Nipponbare) 愈伤组织,并在含有 G418 的筛选培养基上筛选得到阳性转基因植物。以上双元表达载体由中国科学院遗传与发育研究所储成才研究员提供,根癌农杆菌菌株AGL1及日本晴野生型水稻由本实验室保存。
1.2 转基因植株的鉴定
取培养14 d的水稻新鲜叶片,在液氮中冷冻后破碎,用CTAB法提取水稻基因组DNA。设计引物(表1) 用于插入DNA的扩增检测,由华大基因合成,稀释至10 μmol/L。PCR反应采用2×PCR StarMix with Loading Dye试剂盒(康润公司)。反应体系包括10 μL 2×PCR StarMix、正向及反向引物各1 μL、1 μL DNA提取物和7 μL ddH2O。PCR反应条件为:95 ℃ 3 min;95 ℃ 30 s,65 ℃ 30 s,72 ℃ 30 s,35个循环;72 ℃ 3 min。
1.3 转基因植物材料中目的基因的表达分析
取生长14 d的新鲜水稻叶片,迅速加入液氮研磨,使用TRNzol提取试剂盒(天根公司) 提取总RNA。RNA样品反转录采用Maxima H Minus First Strand cDNA合成试剂盒(Thermo公司)。半定量PCR引物设计(表1) 位于检测基因的3ʹ端。PCR反应前先将反转产物稀释5倍,半定量PCR反应体系为10×缓冲液2.5 μL,dNTPs 2 μL,正向及反向引物(10 μmol/L) 各1 μL,cDNA稀释产物5 μL,酶0.5 μL,ddH2O补足至25 μL。半定量PCR反应条件为:95 ℃ 3 min;95 ℃ 30 s,60 ℃ 30 s,72 ℃ 30 s,28个循环;72 ℃ 3 min。以水稻基因表达作为内参。

表1 引物序列
1.4 水稻幼苗对PEG和脱水的敏感性分析
选取籽粒饱满、大小均一的水稻种子。水稻种子经75%酒精消毒1 min,用2.5%次氯酸钠(有效氯含量) 消毒0.5 h后,移至1/2 MS固体培养基上培养。4 d大的水稻苗转移至含有25%的PEG 6000的1/2 MS固体培养基上继续生长,9 d后移到营养液中回复培养2 d后拍照。脱水处理:将消毒后的种子放入37 ℃培养箱中水培催芽24 h,萌发后移入营养液中继续培养,取培养14 d的水稻幼苗至吸水滤纸上干旱处理9.5−10 h,重新复水10 d后拍照。该实验包含3次生物学重复。
1.5 水稻植株干旱处理及分析
消毒后的种子放入37 ℃培养箱中水培催芽24 h,后移入光照培养箱中营养液培养。培养箱生长条件为光周期为13 h光照/11 h黑暗,光照时温度为28 ℃,黑暗时温度为25 ℃。水培一周的小苗移入营养土中,营养土配比为花卉营养土与黄土1∶1,土壤培养温度及光周期条件不变。待植物长到四叶期后,干旱处理7 d,复水10 d后拍照。该实验包含3次生物学重复,所呈现实验结果为3次重复实验的代表结果。
1.6 原生质体瞬时表达载体的构建
水稻原生质体瞬时表达载体pJIT163-Ubi由中国科学院遗传与发育研究所高彩霞研究员提供。Mpsy_3066和Mpsy_0643密码子优化的序列通过d Ⅲ和GⅠ酶切位点分别克隆到pJIT163-Ubi载体中,新构建的载体命名为pJIT163-Mpsy-3066和pJIT163-Mpsy-0643。pJIT163-TrpL-mCherry的TrpL强终止子序列通过引物(表1) 合成直接插入到d Ⅲ和HⅠ 酶切位点之间,即mCherry表达框之前,GFP Y67L表达载体通过引物(表1) 扩增直接引入点突变,新构建的质粒为pJIT163-GFP Y67L。质粒中量提取参考Promega试剂盒Wizard®Midipreps DNA Purification System,调整质粒浓度至1 000 ng/μL。
1.7 水稻原生质体转化及荧光观察和流式细胞分析
原生质体制备及转化主要参考Zhang等[21]的方法并进行了一定改进,水稻MS培养基配制时添加了肌醇(0.1 g/L),水稻幼苗在黑暗条件下生长得到无菌黄化苗。原生质体转化过程中pJIT163-mCherry、pJIT163-TrpL-mCherry质粒的转化量均为500 ng。用于转化后荧光观察的原生质体移入含2 mL WI溶液的35 mm培养皿中孵育。用于转化后流式细胞仪检测的原生质体,置于含200 μL WI溶液的流式管中孵育,转化48 h后进行荧光观察或流式细胞分析。该实验包含2次生物学重复,所呈现实验结果为该重复实验的代表结果。
2 结果与分析
2.1 古菌TRAM编码基因转基因水稻的获得及表达分析
我们对噬冷甲烷古菌R15中两个TRAM蛋白的编码基因和进行了密码子优化,通过农杆菌介导的方法获得了异源表达TRAM的转基因水稻。为了验证TRAMs在水稻中是否正常表达,我们用半定量PCR的方法对不同转基因阳性株系中和基因的表达水平进行检测(图1)。挑选过表达株系进行传代,获得T2代转基因纯合体,对其进行扩繁并用于进一步的实验。
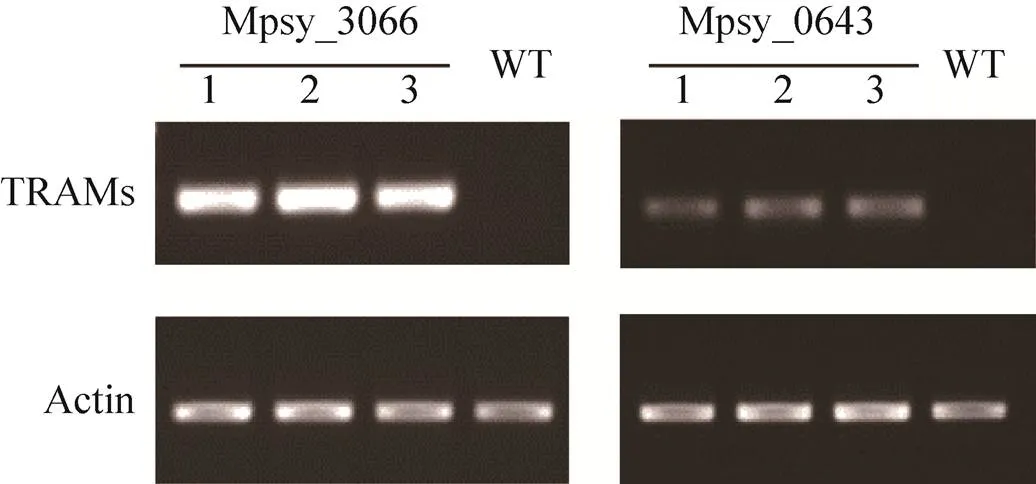
图1 不同转基因水稻株系中TRAM的基因表达水平
2.2 异源表达TRAM增强水稻幼苗耐旱能力
为了检测异源表达TRAM是否改变水稻的耐旱能力,我们首先利用PEG模拟干旱处理,检测水稻幼苗的生长情况。我们将4 d大的野生型和各转基因株系水稻幼苗转移至含有25% PEG6000的培养基或正常培养基上继续生长9 d。结果发现,在没有PEG胁迫处理的情况下,野生型和转基因幼苗生长没有明显区别(图2A、2B);而在PEG处理后,相比于野生型,转基因幼苗的株高均有不同程度的提高(图2C、2D)。

图2 野生型及TRAM转基因水稻幼苗对PEG处理敏感性的分析
同时,我们检测了短期脱水对水稻苗期存活率的影响。我们将在培养液中生长14 d的幼苗置于滤纸上脱水处理9.5 h,复水10 d后统计水稻存活率。结果表明,转基因水稻幼苗存活率明显好于野生型(图3A、3B),表明异源表达TRAM可以显著提高水稻植株对失水的耐受性。

图3 脱水处理后野生型和转基因水稻幼苗的存活率
2.3 异源表达TRAM增强水稻植株期耐旱能力
为了检测水稻植株期耐旱能力,我们将土壤中生长的四叶期水稻植株干旱处理7 d,然后复水10 d,比较野生型和转基因植物的耐旱能力。结果表明,TRAM转基因水稻植株的耐旱能力相比于野生型植株明显提高(图4)。
2.4 古菌TRAM蛋白在水稻细胞中能够解链RNA二级结构
为了进一步研究古菌TRAM蛋白在植物细胞中可能的作用机制,我们以mCherry作为报告基因构建了抗转录终止报告系统,即在mCherry荧光蛋白的正常表达框之前插入了TrpL强终止子序列(图5A)。这个序列可以在转录生成的mRNA上形成双链配对的结构,影响其后mCherry的翻译过程,降低荧光蛋白表达量。我们将构建的该系统在水稻原生质体中进行表达验证,发现在引入TrpL序列后,mCherry荧光蛋白表达量明显下降(图5B),证明了这一报告系统的可靠性。

图4 TRAM转基因水稻植株耐旱能力增强

图5 水稻中抗终止报告系统的建立和验证
基于以上报告系统,我们对转化后的原生质体样品进行流式细胞仪检测并分别统计其荧光强度,结果显示其荧光强度基本呈正态分布(图6A),我们选取转化pJIT163-mCherry和pJIT163- GFP Y67L质粒的原生质体作为对照,标记其峰面积50%为中间值,以该中间值对应的荧光强度为基准,统计不同转化样品高于该荧光强度的峰面积各自所占比例(图6B),结合图6A和6B可以看到共转化或后,其整体荧光强度相比于共转化pJIT163-GFP Y67L质粒的荧光强度明显提高。荧光观察也进一步验证了这一结果(图6C、6D、6E)。以上结果暗示,来自古菌的TRAM蛋白在水稻中可以打开错误折叠的RNA二级结构,具有RNA分子伴侣的功能。
3 讨论
植物可以通过转录及转录后调控响应干旱胁迫。一条成熟的mRNA需经过剪接、加帽、多腺苷酸化等一系列过程才能进入细胞质中进行RNA的翻译。而在RNA的翻译过程中各种RNA二级结构的错误折叠也会影响翻译的进程,使翻译变得缓慢或直接停滞。为了更好地完成生命活动,生物体进化出了多种RNA结合蛋白(RNA binding protein,RBPs)来完成转录后水平的调控。在干旱胁迫时,由于机体的紊乱和受损,RNA分子伴侣的功能的行使显得更为重要。
TRAM结构域是一类具有RNA结合活性的多肽片段。在古菌中该结构域单独存在,直接作为一个功能蛋白发挥着类似于细菌中Csp蛋白RNA分子伴侣的功能,这为TRAM蛋白的开发利用提供了更多的方便性和灵活性。根据已有的报道,细菌中Csp蛋白在植物中异源表达,可以显著地提高植物耐受干旱的能力。因此,我们推测古菌中的TRAMs蛋白可能同Csp蛋白一样在植物中过表达可以提高植物耐受干旱的能力。我们通过实验进一步验证了这一猜想。在PEG模拟干旱的处理条件下,我们统计了植株地上部分的生长能力。与野生型植物相比,过表达TRAM的水稻幼苗植物地上部分生长能力均有不同程度的提高;在对苗期植物脱水处理后复水,水稻幼苗存活率有15%−20%的提高。同时,我们进一步验证了水稻成株期时耐受干旱胁迫的能力,在干旱处理7 d后,野生型植株在复水后枯死较多,而异源表达TRAM的水稻植株仍有半数以上可以继续生长,证明了转基因组水稻植株在成株期的耐旱能力也有所提升。这些结果都支持异源表达TRAM可以提高水稻耐受干旱胁迫的能力。

图6 古菌TRAM在水稻原生质体中对RNA二级结构的解链活性
目前,虽然有大量的文献报道了通过改造植物内源基因或表达异源基因来提高植物抗旱的能力,但是一些基因在提高水稻抗旱能力的同时会影响水稻的生长。例如,由CaMV 35S启动子过表达的一些转录因子(如DREB) 会导致植物生长缓慢,进而导致产量下降[22]。AtRZ-1a过表达拟南芥植株与野生型相比,表现出种子萌发迟缓和幼苗生长变慢的表型。有趣的是,过表达古菌TRAM的水稻植株没有表现出明显的植物营养生长的差异,进一步支持了该类基因在水稻抗旱性遗传改良中的利用价值。
对于TRAM蛋白在植物细胞中可能的作用机制,我们发现TRAM蛋白在植物细胞中具有促进错误折叠的RNA结构改变而抗终止的功能,进一步证明了古菌TRAMs发挥RNA分子伴侣的功能。我们推测TRAM在水稻中也是利用其RNA分子伴侣的功能,在干旱胁迫条件下消除RNA二级结构的错误折叠对蛋白翻译的影响,从而保证机体代谢的正常进行。
在分子水平上,古菌和真核生物相比有更多的相似之处。古菌在转录、翻译及蛋白降解系统的组成元件上,都表现出明显的真核特征[16]。如,古菌中RNA聚合酶比细菌中复杂的多,而且它们的亚基组成与真核生物相似[23];又如,核糖体的三维结构也预示真核生物可能与泉古菌门在进化上的联系[24]。阿斯加德古菌的发现进一步深化了人们对古菌和真核生物关系的认识[25-26]。古菌作为生物学分类中一个庞大的分支,来自于古菌更多的分子元件、代谢产物有待我们开发,相信也将在植物基因工程改造上或者在更大范围获得更好的利用。
总之,本研究阐明了异源表达古菌TRAM基因可以提高水稻耐受干旱的能力,TRAM蛋白在植物细胞中发挥功能可能是通过打开错误配对的二级结构以稳定RNA的正常代谢。本研究也为开发利用古菌中多样的分子资源提供了参考。
致 谢 感谢中国科学院微生物研究所东秀珠研究员为我们提供TRAM的相关信息及材料。
[1] Jiang Y, van Groenigen KJ, Huang S, et al. Higher yields and lower methane emissions with new rice cultivars. Glob Chang Biol, 2017, 23(11): 4728–4738.
[2] Serraj R, McNally KL, Slamet-Loedin I, et al. Drought resistance improvement in rice: an integrated genetic and resource management strategy. Plant Prod Sci, 2011, 14(1): 1–14.
[3] Tardieu F, Simonneau T, Muller B. The physiological basis of drought tolerance in crop plants: a scenario-dependent probabilistic approach. Annu Rev Plant Biol, 2018, 69: 733–759.
[4] Varshney RK, Tuberosa R, Tardieu F. Progress in understanding drought tolerance: from alleles to cropping systems. J Exp Bot, 2018, 69(13): 3175–3179.
[5] Basu S, Ramegowda V, Kumar A, et al. Plant adaptation to drought stress. F1000Res, 2016, 5, doi: 10.12688/f1000research.7678. 1.
[6] Jorrin JV, Maldonado AM, Castillejo MA. Plant proteome analysis: a 2006 update. Proteomics, 2007, 7(16): 2947–2962.
[7] Choudhary MK, Basu D, Datta A, et al. Dehydration-responsive nuclear proteome of rice (L.) illustrates protein network, novel regulators of cellular adaptation, and evolutionary perspective. Mol Cell Proteomics, 2009, 8(7): 1579–1598.
[8] Swamy BPM, Kumar A. Genomics-based precision breeding approaches to improve drought tolerance in rice. Biotechnol Adv, 2013, 31(8): 1308–1318.
[9] Herschlag D. RNA chaperones and the RNA folding problem. J Biol Chem, 1995, 270(36): 20871–20874.
[10] Zhang B, Yue L, Zhou LG, et al. Conserved TRAM domain functions as an archaeal cold shock protein via RNA chaperone activity. Front Microbiol, 2017, 8: 1597.
[11] Goldstein J, Pollitt NS, Inouye M. Major cold shock protein of. Proc Natl Acad Sci USA, 1990, 87(1): 283–287.
[12] Castiglioni P, Warner D, Bensen RJ, et al. Bacterial RNA chaperones confer abiotic stress tolerance in plants and improved grain yield in maize under water-limited conditions. Plant Physiol, 2008, 147(2): 446–455.
[13] Yu TF, Xu ZS, Guo JK, et al. Improved drought tolerance in wheat plants overexpressing a synthetic bacterial cold shock protein gene SeCspA. Sci Rep, 2017, 7: 44050.
[14] Yang DH, Kwak KJ, Kim MK, et al. Expression ofglycine-rich RNA-binding protein AtGRP2 or AtGRP7 improves grain yield of rice () under drought stress conditions. Plant Sci, 2014, 214: 106–112.
[15] Baek W, Lim CW, Lee SC. A DEAD-box RNA helicase, RH8, is critical for regulation of ABA signalling and the drought stress response via inhibition of PP2CA activity. Plant Cell Environ, 2018, 41(7): 1593–1604.
[16] Eme L, Spang A, Lombard J, et al. Archaea and the origin of eukaryotes. Nat Rev Microbiol, 2017, 15(12): 711–723.
[17] Shcherbakova V, Yoshimura Y, Ryzhmanova Y, et al. Archaeal communities of Arctic methane-containing permafrost. FEMS Microbiol Ecol, 2016, 92(10): fiw135.
[18] Anantharaman V, Koonin EV, Aravind L. TRAM, a predicted RNA-binding domain, common to tRNA uracil methylation and adenine thiolation enzymes. FEMS Microbiol Lett, 2001, 197(2): 215–221.
[19] Theobald DL, Mitton-Fry RM, Wuttke DS. Nucleic acid recognition by OB-fold proteins. Annu Rev Biophys Biomol Struct, 2003, 32(1): 115–133.
[20] Christensen AH, Sharrock RA, Quail PH. Maize polyubiquitin genes: structure, thermal perturbation of expression and transcript splicing, and promoter activity following transfer to protoplasts by electroporation. Plant Mol Biol, 1992, 18(4): 675–689.
[21] Zhang Y, Su JB, Duan S, et al. A highly efficient rice green tissue protoplast system for transient gene expression and studying light/chloroplast-related processes. Plant Methods, 2011, 7: 30.
[22] Shen YG, Zhang WK, He SJ, et al. An EREBP/AP2-type protein inwas a DRE-binding transcription factor induced by cold, dehydration and ABA stress. Theor Appl Genet, 2003, 106(5): 923–930.
[23] Huet J, Schnabel R, Sentenac A, et al. Archaebacteria and eukaryotes possess DNA-dependent RNA polymerases of a common type. EMBO J, 1983, 2(8): 1291–1294
[24] Lake JA, Henderson E, Oakes M, et al. Eocytes: a new ribosome structure indicates a kingdom with a close relationship to eukaryotes. Proc Natl Acad Sci USA, 1984, 81(12): 3786–3790.
[25] Spang A, Saw JH, Jørgensen SL, et al. Complex archaea that bridge the gap between prokaryotes and eukaryotes. Nature, 2015, 521(7551): 173–179.
[26] Zaremba-Niedzwiedzka K, Caceres EF, Saw JH, et al.illuminate the origin of eukaryotic cellular complexity. Nature, 2017, 541(7637): 353–358.
Ectopic expression of archaeal TRAM-encoding genes in rice improves its drought-tolerance
Wei Chen1, 2, Huali Li1, and Jinlong Qiu1
1 Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China 2 University of Chinese Academy of Sciences, Beijing 100049, China
Drought stress affects the growth and development of rice, resulting in severe loss in yield and quality. Ectopic expression of the bacterial RNA chaperone, cold shock protein (Csp), can improve rice drought tolerance. Archaeal TRAM (TRM2 and MiaB) proteins have similar structure and biochemical functions as bacterial Csp. Moreover, DNA replication, transcription and translation of archaea are more similar to those in eukaryotes. To test if archaeal RNA chaperones could confer plant drought tolerance, we selected two TRAM proteins, Mpsy_3066 and Mpsy_0643, from a cold-adaptive methanogenic archaeaR15 to study. We overexpressed the TRAM proteins in rice and performed drought treatment at seedling and adult stage. The results showed that overexpression both TRAM proteins could significantly improve the tolerance of rice to drought stress. We further demonstrated in rice protoplasts that the TRAMs could abolish misfolded RNA secondary structure and improve translation efficiency, which might explain how TRAMs improve drought tolerance transgenic rice. Our work supports that ectopic expression of archaeal TRAMs effectively improve drought tolerance in rice.
rice, drought stress, RNA chaperon, archaea, TRAM
March 5, 2019;
March 28, 2019
Jinlong Qiu. Tel/ Fax: +86-10-64807398; E-mail:qiujl@im.ac.cn
2019-04-09
http://kns.cnki.net/kcms/detail/11.1998.Q.20190408.1036.001.html
陈巍, 李华丽, 邱金龙. 异源表达古菌TRAM基因增强水稻的耐旱性. 生物工程学报, 2019, 35(9): 1676–1685.
Chen W, Li HL, Qiu JL. Ectopic expression of archaeal TRAM-encoding genes in rice improves its drought-tolerance. Chin J Biotech, 2019, 35(9): 1676–1685.
(本文责编 陈宏宇)

