恩诺沙星曝露下企鹅珍珠贝肠道微生物多样性及优势菌变化规律
陈明强 李有宁 邓正华 于刚 马振华 郑兴 洪嘉炜 魏海军 王雨
摘要:【目的】明確恩诺沙星曝露下企鹅珍珠贝肠道微生物群落结构尤其是优势菌的变化规律,为企鹅珍珠贝养殖过程中恩诺沙星的科学用药提供参考依据。【方法】针对3个恩诺沙星浓度(0、5和10 mg/L)共9个企鹅珍珠贝肠道样本中的细菌构建16S rRNA文库,再采用Illlumina高通量测序技术从门和属水平上比较分析各处理组间肠道微生物群落结构和多样性的差异。【结果】从9个企鹅珍珠贝肠道样本中共检测到10个菌门,其优势菌门分别是变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、梭杆菌门(Fusobacteria)、螺旋体门(Spirochaetae)、蓝藻门(Cyanobacteria)和厚壁菌门(Firmicutes);共检测到79个菌属,优势菌属主要包括弧菌属(Vibrio)、发光杆菌属(Photobacterium)、嗜冷菌属(Psychrilyobacter)、假交替单胞菌属(Pseudoalteromonas)、疏螺旋体属(Borrelia)、交替单胞菌属(Alteromonas)、微拟球藻属(Nannochioropsis-oceanica)、弓形杆菌属(Arcobacter)、Thalassotalea、Amphritea、Tenacibaculum和微小杆菌属(Exiguobacterium)等。不同恩诺沙星浓度处理下,企鹅珍珠贝肠道的优势菌门与菌属种类丰度差异明显。经恩诺沙星处理后,企鹅珍珠贝肠道变形菌门相对丰度显著升高(P<0.05,下同)、拟杆菌门相对丰度显著降低;弧菌属相对丰度显著升高,微拟球藻属、Aquibacter、BD1-7_clade及希瓦氏菌属(Shewanella)相对丰度则显著降低;此外,弓形杆菌属相对丰度经10 mg/L恩诺沙星处理后显著升高,Psychrobium相对丰度经5 mg/L恩诺沙星处理后显著升高。【结论】经恩诺沙星处理后企鹅珍珠贝肠道微生物的多样性与丰度发生明显变化,且肠道微生物中一些具有特殊功能的优势菌群结构比例也发生变化,该变化规律可为企鹅珍珠贝养殖过程中科学使用恩诺沙星提供参考依据。
关键词: 企鹅珍珠贝;恩诺沙星;肠道微生物;菌群结构;多样性;相对丰度;高通量测序
中图分类号: S968.316 文献标志码: A 文章编号:2095-1191(2019)04-0851-09
Abstract:【Objective】Intestinal microbial diversity and dominant bacteria change analysis of Pteria penguin (Röding) under enrofloxacin exposure were studied to provide reference for scientific administration of enrofloxacin du-ring P. penguin culture. 【Method】The bacterial 16S rRNA library was constructed using the nine P. penguin intestinal bacteria samples under three enrofloxacin concentrations(0,5 and 10 mg/L). The differences in intestinal microbial structure and diversity were analyzed and compared among the groups at phylum level and genus level. 【Result】The analysis showed that, ten phyla were detected from the nine P. penguin intestinal samples, and the core phyla included Proteobacteria, Bacteroidetes, Fusobacteria, Spirochaetae, Cyanobacteria and Firmicutes. And 79 genera were detected, the dominant genera included Vibrio, Photobacterium, Psychrilyobacter, Pseudoalteromonas, Borrelia, Alteromonas, Nannochioropsis-oceanica, Arcobacter, Thalassotalea, Amphritea, Tenacibaculum and Exiguobacterium. After different enrofloxacin concentrations treatments, the abundance of dominant bacteria phyla and genera of P. penguin intestinal tract differed greatly. The relative abundance of Proteobacteria increased significantly under enrofloxacin treatment(P<0.05, the same below), while that of Bacteroidertes decreased significantly. Relative abundance of Arcobacter was significantly increased, and relative abundance of Nannochloropsis, Aquibacte, BD1-7_clade and Shewanella were decreased significantly. Relative abundance of Arcobacter significantly increased after treated by 10 mg/L enrofloxacin, and relative abundance of Psychrobium significantly increased after treated by 5 mg/L enrofloxacin. 【Conclusion】The results indicate that enrofloxacin treatment can greatly change the intestinal microbial diversity and abundance in P. penguin, the proportions of dominant microbial community structure with specific functions also change. The findings in the present study can be served to the scientific administration of enrofloxacin during aquaculture of P. penguin.
Key words: Pteria penguin(Röding); enrofloxacin; intestinal microorganism; microbial community structure; diversity; relative abundance; high-throughput sequencing
0 引言
【研究意義】企鹅珍珠贝[Pteria penguin(Röding)]是一种热带、亚热带的大型海产经济双壳贝类,主要分布于日本九州南部、琉球群岛、菲律宾及我国广东、广西、海南、台湾等沿海地区(栗志民等,2011),具有适应环境能力强、生长和分泌珍珠质速度快的特点(余祥勇等,2005;梁飞龙等,2014),已成为我国培育大型海水附壳珍珠及游离正圆珍珠的主要贝种。但由于养殖海区海水环境的污染恶化及养殖病害的暴发,已严重制约海洋贝类产业健康发展,其中由细菌引起的疾病最明显(李启蒙,2017)。恩诺沙星(Enrofloxacin)属于氟喹诺酮类药物,是一种高效广谱的抗菌药物,已广泛应用于动物细菌性疾病治疗,其作用原理是选择性抑制微生物的DNA拓扑异构酶Ⅱ,使其DNA无法复制。恩诺沙星具有抗菌谱广、组织浓度高、吸收性好、半衰期长等优点,且与其他抗菌药物不会发生交叉耐药,能有效预防多种水产细菌性疾病,现已广泛应用于水产养殖(Balaje et al.,2013)。因此,明确恩诺沙星对企鹅珍珠贝肠道细菌多样性的影响,对指导企鹅珍珠贝养殖过程中科学使用恩诺沙星具有重要意义。【前人研究进展】目前,有关企鹅珍珠贝的研究主要集中在人工育苗(余祥勇等,2000;Wassnig and Southgate,2012)、珍珠人工培育(符韶和梁飞龙,2000;毛勇等,2004)、生殖细胞发生(Arijarasirikoon et al.,2004)、同工酶特征与遗传分析(余祥勇等,2004)、母贝养殖培育(符韶等,2007;郭华阳等,2016)、相关基因克隆及表达响应等方面(邱莹等,2016)。近年来,随着企鹅珍珠贝养殖规模的不断扩大,其病害频繁发生。梁飞龙等(2007)对流沙湾养殖的企鹅珍珠贝多毛类寄生虫病进行调查,结果发现,形笼养殖2~4年的企鹅珍珠贝母贝多毛类寄生虫病感染率为39.13%~44.98%,而开放式养殖2~4年的感染率为26.6%~35.63%,说明企鹅珍珠贝多毛类寄生虫病感染率与养殖方式间存在一定的相关性;程赞等(2011)研究表明,企鹅珍珠贝夏、秋两季的污损生物优势种群有海绵、才女虫、盘管虫、龙介虫、藤壶和海鞘,春、冬两季的污损生物优势种群包含藤壶和草苔虫。至今,国内外研究报道的贝类致病性细菌主要有爱德华氏菌(Edwardsiella tarda)、屈桡杆菌(Flexibacter maritimus)、加氏乳球菌(Lactococcus garvieae)、分枝杆菌(Mycobacterium sp.)、假单胞菌(Pseudomonas plecoglossicida)、溶藻弧菌(Vibrio alginolyticus)等(陆彤霞等,2002;Sakatoku et al.,2018),但有关珍珠贝细菌性疾病方面的研究鲜见报道。【本研究切入点】在水产养殖业使用的抗菌药物中,恩诺沙星是广大养殖户普遍认可的高效抗菌药物之一,但至今鲜见恩诺沙星用于企鹅珍珠贝细菌类病害防治的研究报道。【拟解决的关键问题】利用Illumina平台第三代测序技术研究恩诺沙星对企鹅珍珠贝肠道微生物多样性的影响,明确恩诺沙星处理下企鹅珍珠贝肠道微生物群落结构尤其是优势菌的变化规律,为企鹅珍珠贝养殖过程中恩诺沙星的科学用药提供参考依据。
1 材料与方法
1. 1 试验材料
试验用企鹅珍珠贝购自海南陵水新村市场,运回中国水产科学研究院南海水产研究所热带水产研究开发中心基地,参照蒙钊美等(1996)的管理方法暂养2周。企鹅珍珠贝规格:壳长115.52±3.93 mm,壳高127.16±8.72 mm,壳宽44.99±5.09 mm,体重279.97±64.46 g。
1. 2 试验设计与取样
称取恩诺沙星晶体,溶解于装有砂滤海水的3个玻璃钢桶(体积400 L)中,使恩诺沙星浓度分别为0、5和10 mg/L(Fang et al.,2012),标号为PPA、PPB和PPC;然后将30只健康的企鹅珍珠贝平均分配悬吊在玻璃钢桶中,每桶各10只,试验开始后停止投喂。企鹅珍珠贝在恩诺沙星海水溶液中浸泡24 h。从每处理组中随机挑取3只企鹅珍珠贝,做好标记,其中,0 mg/L处理标号PPA1、PPA2和PPA3,5 mg/L处理标号PPB1、PPB2和PPB3,10 mg/L处理标号PPC1、PPC2和PPC3。用解剖刀切断企鹅珍珠贝闭壳肌,取出其内脏团,以无菌海水将内脏团冲洗干净后无菌条件下采集其肠道,无菌水冲洗干净,采用75%乙醇浸泡3 min,再用无菌水冲洗3次,然后将肠道内容物放入1.5 mL的无菌离心管中,-80 ℃保存备用。
1. 3 PCR扩增及基因文库构建
采用E.Z.N.A® DNA试剂盒(OMEGA Bio-tek)提取企鹅珍珠贝肠道基因组总DNA,采用Qubit® 3.0荧光光度计及琼脂糖凝胶电泳检测DNA含量和质量。PCR扩增以20~30 ng DNA为模板,选取16S rDNA序列V3~V4可变区用于后期的分类分析。针对V3~V4保守区域,以Illumina MiSeq平台设计特异性引物(5'-CCTACGGRRBGCASCAGKVRVGAAT-3'和5'-GGACTACNVGGGTWTCTAATCC-3'),通过PCR扩增肠道细菌16S rDNA序列V3~V4两个高度可变区。PCR扩增产物末端加入适配接头,以便于后期的Miseq上机测序。PCR反应体系25.0 μL:2.5 μL TransStart缓冲液,2.0 μL dNTPs,上、下游引物各1.0 μL,20 ng DNA模板,去离子水补足至25.0 μL。扩增程序:94 ℃预变性3 min;94 ℃ 5 s,57 ℃ 90 s,72 ℃ 10 s,进行24个循环;72 ℃延伸5 min。PCR扩增产物采用1.5%琼脂糖凝胶电泳进行质量检测,然后按照Quick Gel提取试剂盒(QIAGEN公司)进行分离与纯化。3次重复。纯化PCR扩增产物用于基因文库构建及高通量测序。
1. 4 生物信息学分析
基因文库采用Qubit 3.0分光光度计检测浓度,并定量至10 nM,然后按照说明将其上样至Illumina MiSeq设备进行高通量测序(Illumina,San Diego,CA,USA),采用PE 250/300进行配对末端测序,并以MiSeq设备附带的the MiSeq控制软件进行图片分析及碱基核对。测序后加入末端配对的读码框,根据条形码、切断条形码及引物序列对样本进行分类分析(Schloss et al.,2009)。根据连接序列进行质量过滤,对未达以下标准的序列进行抛弃:序列长度<200 bp,无模棱两可的碱基,平均质量分数≥20。然后,根据参考数据库(RDP gold database)进行数据比对,采用Uchime盐酸法推测一些不合理的序列并去除。
原始数据经Flash拼接、Qiime过滤和Uchime Algorithm去除嵌合体后,即得到有效数据(Caporaso et al.,2010),计算每个样品的α多样性。再用Uparse对有效数据在97%水平上的操作分类单元(Operational taxonomic unit,OTU)进行聚类,并用基于Green Gene数据库的RDP Classifier分类工具进行物种注释。稀释曲线采用Mothur v1.30进行分析。
1. 5 统计分析
试验数据采用SPSS 19.0进行分析,并进行单因素方差分析(One-way ANOVA)。
2 结果与分析
2. 1 序列数与OTU数量统计分析结果
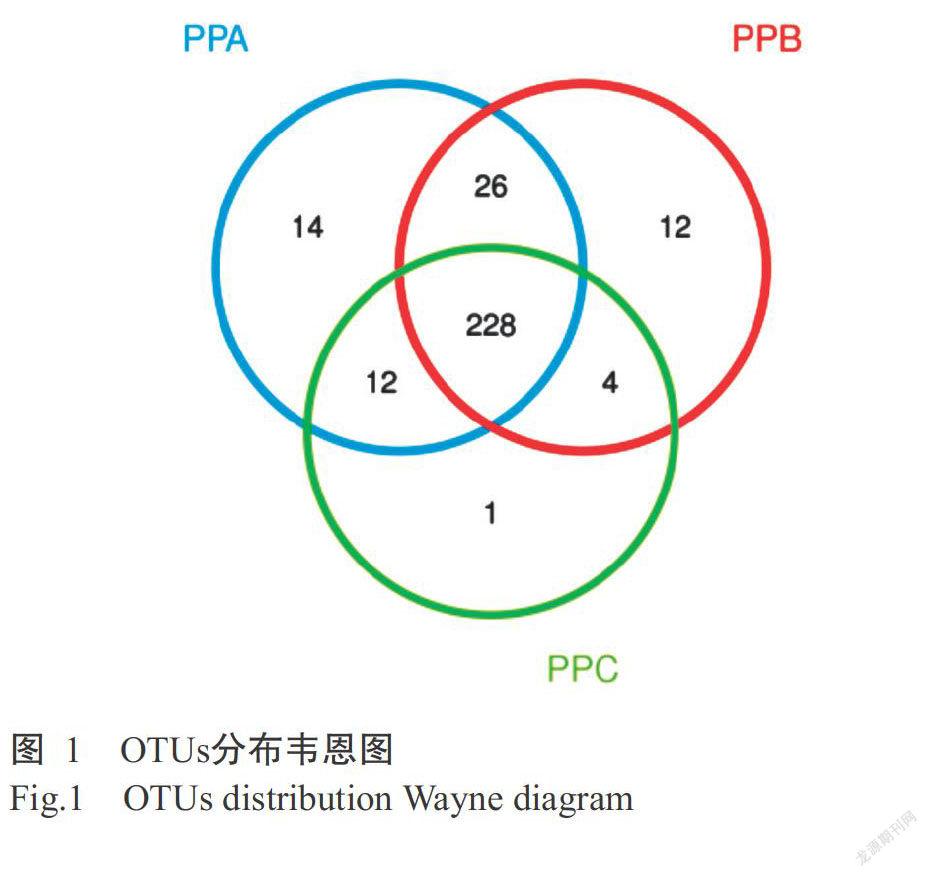
为获得更高质量及更准确的分析结果,3个处理组(PPA、PPB和PPC)共9个样本(PPA1、PPA2、PPA3、PPB1、PPB2、PPB3、PPC1、PPC2和PPC3)的原始数据经Cutadapt(v1.9.1)、Vsearch(1.9.6)及Qiime(1.9.1)分析,去除序列拼接中含有N的序列、引物、接头序列及嵌合体序列,分别获得43345、44283、82654、52772、127402、45194、128256、44999和51991条有效序列,去除嵌合体后有效序列数目与原始PE reads数目的百分比在83.92%以上。将相似水平在97%以上的所有序列进行OTU划分和聚类分析,如图1所示,PPA处理组特有的OTUs为14个,PPB处理组特有的OTUs为12个,PPC处理组特有的OTU为1个,而3个处理组样本共有OTUs为228个。
2. 2 肠道菌群α多样性分析结果
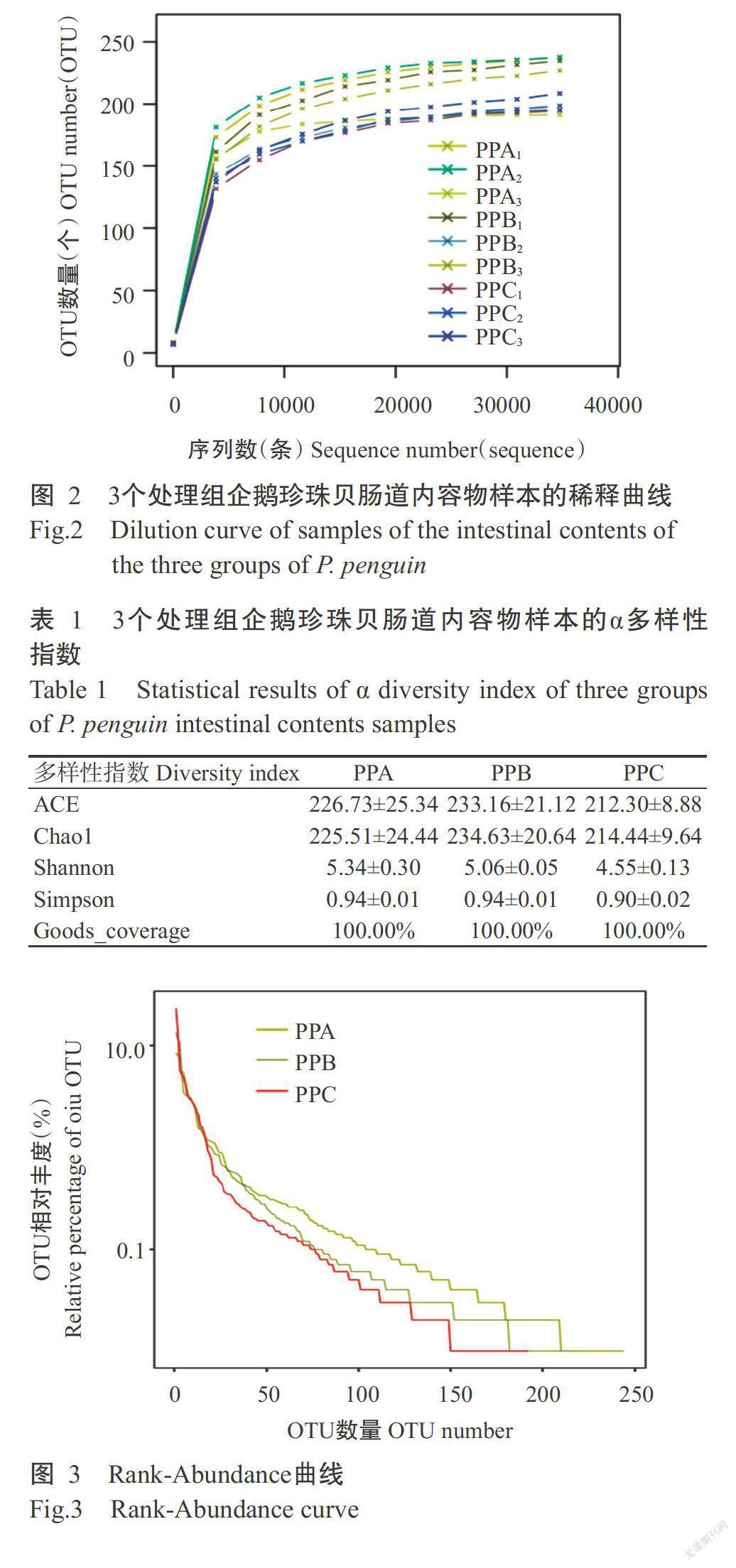
如图2所示,3个处理组9个样本的稀释曲线斜率随测序通量的增大而逐渐降低,最终趋于平坦。同时,以Shannon、Simpson、Chao1和ACE等指数衡量各样本中微生物的多样性,其中3个处理组样本的覆盖率(Goods_coverage)均高达100.00%(表1)。Rank-Abundance曲线反映出PPC处理组企鹅珍珠贝肠道菌群的丰度及均匀度均最高(图3)。综上所述,随测序深度的增加,测序量已饱和且测序数据量合理,说明各样本中几乎所有细菌序列均被检出。3个处理组样品的菌群α多样性因处理不同而存在差异。
2. 3 肠道菌群结构
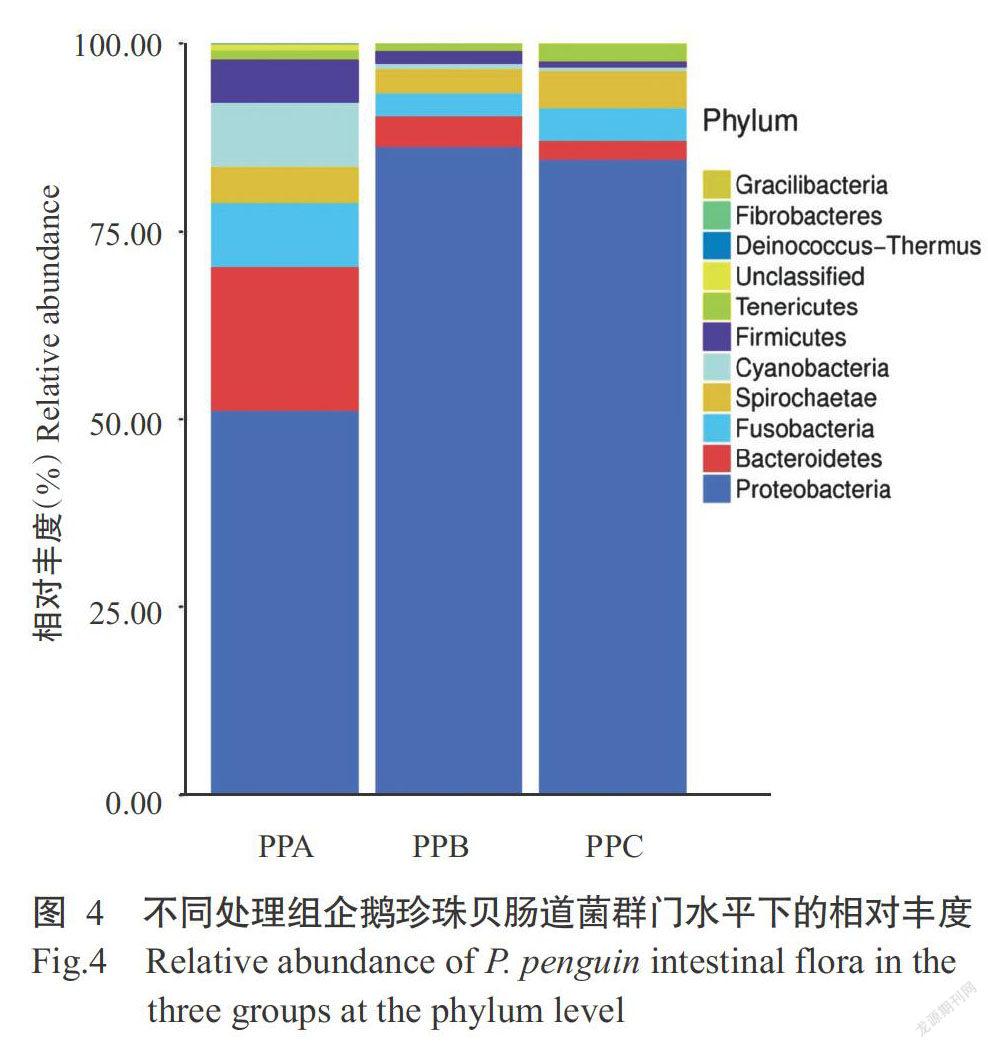
2. 3. 1 门水平下的企鹅珍珠贝肠道菌群结构 3个处理组9个企鹅珍珠贝样本的有效序列分别有74.34%、62.98%和63.40%能注释到门分类水平,共检测到10个菌门,其相对丰度由高到低分别是变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、梭杆菌门(Fusobacteria)、螺旋体门(Spirochaetae)、蓝藻门(Cyanobacteria)、厚壁菌门(Firmicutes)、无壁菌门(Tenericutes)、异常球菌—栖热菌门(Deinococcus-Thermus)、纤维杆菌门(Fibrobacteres)和Gracilibacteria。由图4可见,PPA处理组的变形菌门相对丰度在50.00%以上,而在PPB和PPC处理组中变形菌门的相对丰度均在75.00%以上。变形菌门、拟杆菌门、梭杆菌门、螺旋体门、蓝藻门、厚壁菌门和无壁菌门虽然同为企鹅珍珠贝肠道中的优势菌门,但其相对丰度发生不同程度的改变(表2)。其中,PPB和PPC处理组企鹅珍珠贝肠道变形菌门相对丰度显著高于PPA处理组(P<0.05,下同),拟杆菌门相对丰度则显著低于PPA处理组,但PPB和PPC两处理组间无显著差异(P>0.05,下同);其他优势菌门在不同处理组间也有差异,但差异不显著。
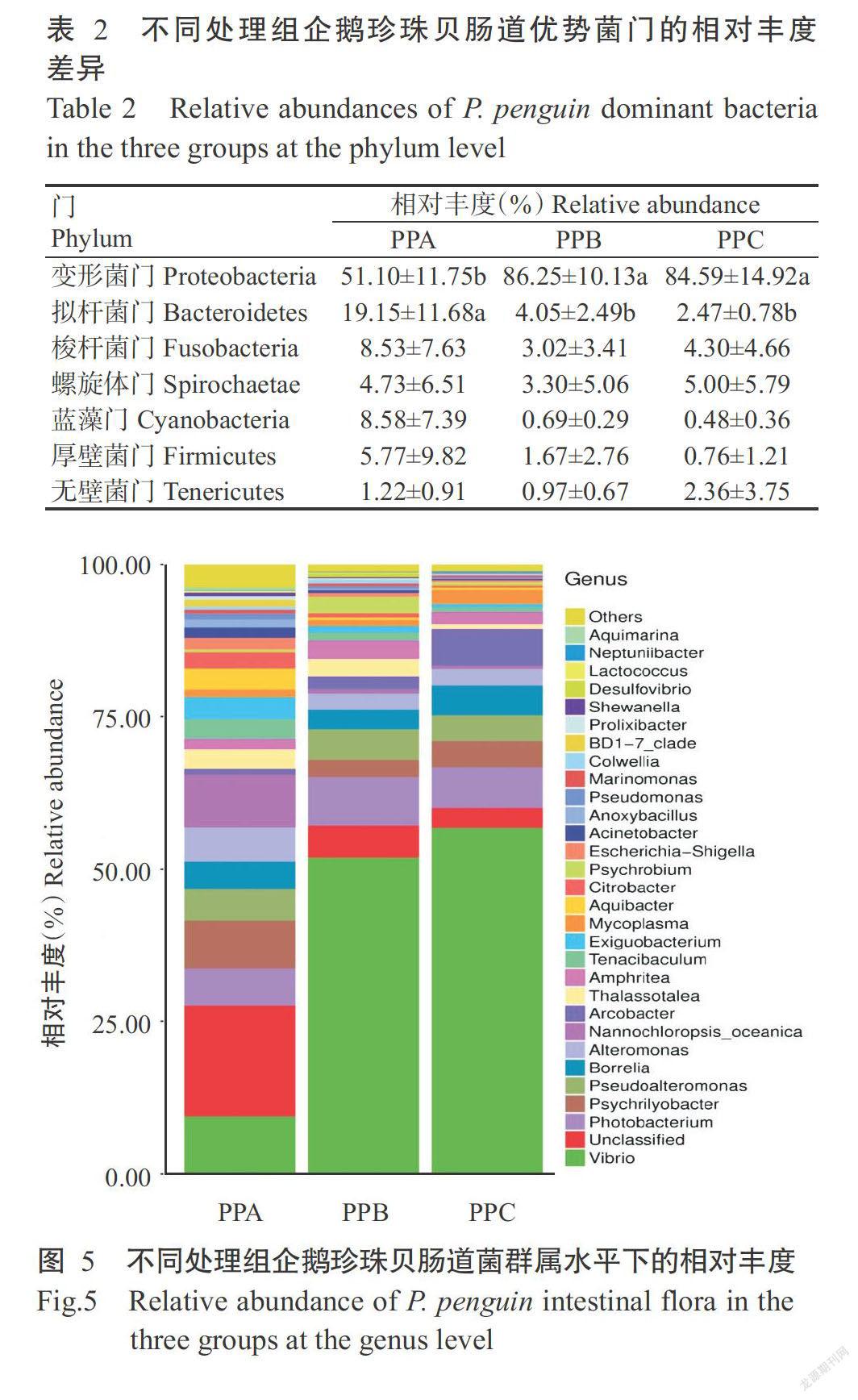
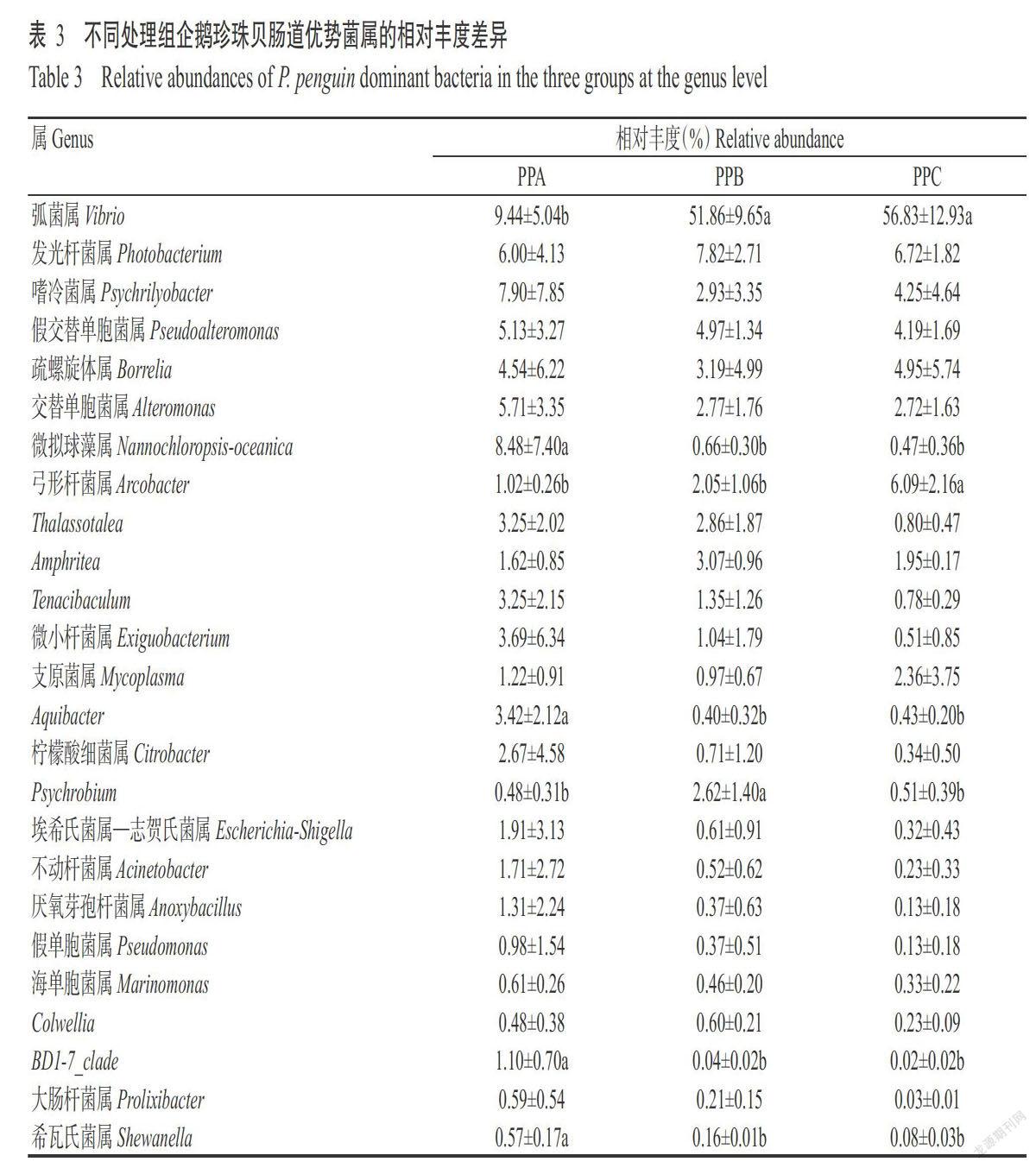
2. 3. 2 属水平下的企鹅珍珠贝肠道菌群结构 3个处理组9个企鹅珍珠贝样本的有效序列分别有77.80%、91.60%和88.90%能注释到属分类水平,共检测到79个菌属,其相对丰度较高的菌属有弧菌属(Vibrio)、发光杆菌属(Photobacterium)、嗜冷菌属(Psychrilyobacter)、假交替单胞菌属(Pseudoalteromonas)、疏螺旋體属(Borrelia)、交替单胞菌属(Alteromonas)、微似球藻属(Nannochioropsis-ocea-nica)、弓形杆菌属(Arcobacter)、Thalassotalea、Amphritea、Tenacibaculum和微小杆菌属(Exiguobacterium)等。弧菌属在PPB和PPC处理组中的相对丰度最高,均在50.00%以上。此外,发光杆菌属、嗜冷菌属、假交替单胞菌属、疏螺旋体属、交替蛋白菌属、微拟球藻属和弓形杆菌属等的相对丰度也较高,均为优势菌属(图5)。由表3可知,经恩诺沙星处理(PPB和PPC处理组)后,企鹅珍珠贝肠道中弧菌属相对丰度显著升高,微拟球藻属、Aquibacter、BD1-7_clade及希瓦氏菌属相对丰度则显著降低。PPC处理组企鹅珍珠贝肠道弓形杆菌属相对丰度显著高于PPA和PPB处理组;PPB处理组企鹅珍珠贝肠道Psychrobium相对丰度显著高于PPA和PPC处理组。
3 讨论
水产动物肠道内部细菌的数量级一般在105~108(Skrodenytė-Arbačiauskienė,2007),主要包括变形菌、厚壁菌、假单胞菌和不动杆菌等,其中部分细菌在水产动物的生长及代谢活动中发挥关键作用(Skrodenytė-Arbačiauskienė et al.,2008;Sullam et al.,2012)。机体肠道中各种微生物与宿主的关系非常密切,对其生长、发育和健康均有重要影响,而肠道微生物的群落结构和丰度又受宿主基因型、日龄及栖息环境等因素的影响(Hooper and Gordon,2001;Savas et al.,2005;Navarrete et al.,2012;Meng et al.,2014)。在正常生长环境下,优势微生物能影响和决定宿主自身微生物的组成、群落结构并维持菌落平衡(Wu et al.,2015),而肠道菌落结构失衡可能会导致宿主疾病发生。本研究结果表明,变形菌门、拟杆菌门、梭杆菌门、螺旋体门、蓝藻门、厚壁菌门、无壁菌门、异常球菌—栖热菌门、纤维杆菌门和Gracilibacteria是企鹅珍珠贝肠道中的优势菌门,即企鹅珍珠贝肠道菌群的多样性规律与鲤鱼(Ye et al.,2014)、虹鳟(Lyons et al.,2015)及金鲳(Chen et al.,2018)肠道细菌多样性规律一致。
变形菌门是养殖海水环境或高盐湖泊环境中最主要的细菌群体之一(Demergasso et al.,2004;Stevens et al.,2005;Wu et al.,2015);拟杆菌门作为海水环境中的重要微生物类群,是溶解性有机物质的主要消费者(Cottrell and Kirchman,2000)。拟杆菌门中的Flavobacteriia纲是海水养殖中高分子有机物的主要降解者,可能是有机物降解菌群的大量存在有利于加快有机物降解,从而减轻水产动物面临的环境压力(陈琼等,2017)。厚壁菌门中95%细菌16S rDNA序列隶属梭菌纲,且大部分16S rDNA序列与丁酸产生菌存在相关性,对肠道上皮的维持与保护非常重要。厚壁菌门作为一种典型的GC碱基低含量菌门,也是老鼠和人类等高等生物肠道菌群中丰度最高的菌门,与能量再吸收和脂肪代谢有关(Ley et al.,2006;Komaroff,2017;赵翔刚等,2017)。变形菌门和厚壁菌门是常见的水产动物肠道微生物,对肠道的生理功能起重要作用(Jumpertz et al.,2011)。本研究发现,3个处理组企鹅珍珠贝肠道优势菌门的相对丰度发生了明显变化。其中,PPA处理组企鹅珍珠贝肠道变形菌门的相对丰度在50.00%以上,在PPB和PPC处理组中变形菌门显著升高,其相对丰度均在75.00%以上;而拟杆菌门相对丰度的变化与其恰好相反,PPB和PPC处理组拟杆菌门的相对丰度显著低于PPA处理组,且PPC处理组拟杆菌门的相对丰度最低。可见,恩诺沙星处理对企鹅珍珠贝肠道优势菌门相对丰度影响明显。
微生物鉴定只有在其种或属的水平上分类才具有意义(Petrosino et al.,2009)。在属分类水平上,弧菌属、发光杆菌属、嗜冷菌属、假交替单胞菌属、疏螺旋体属、交替单胞菌属、微拟球藻属、弓形杆菌属、Thalassotalea、Amphritea、Tenacibaculum和微小杆菌属为企鹅珍珠贝肠道的优势菌属。其中,弧菌属是海洋环境中的常见细菌属,与埃希氏菌属—志贺氏菌属都是变形菌门的主要菌属,属于条件致病菌,在黄鲇鱼、草鱼和对虾肠道中均有发现(Oxley et al.,2002;Wu et al.,2010;Hamilton et al.,2013;Tan et al.,2014)。弧菌在养殖水体有机物降解方面具有重要意义,同时发挥着硝酸盐异化作用,但一些弧菌对珍珠贝等水产动物甚至人类具有潜在的致病性(艾红等,2003;赵晓伟等,2015;毕水莲等,2017)。微小杆菌属也是厚壁菌门的主要菌属之一,能促进凡纳滨对虾的生长与存活,还能产生大量的消化酶类(张莹等,2013;Sombatjinda et al.,2014;Pandey and Bhatt,2015;施兆鸿等,2015)。Ward-Rainey等(1996)从黑刺参肠道内分离获得43株细菌,其中24株为弧菌,为肠道内的核心菌属,其余分别归属于芽孢杆菌属、放线菌和变形杆菌,与本研究中弧菌在企鹅珍珠贝肠道菌属结构中的绝对优势相一致。
假单胞菌可引发海水养殖虾类烂鳃病、红腿病等细菌性疾病,还有研究发现假单胞菌具有直接或间接的杀藻作用,尤其与假单胞菌在养殖水体中保持一定数量时有利于对虾生长;当部分弧菌、假单胞菌和单胞菌等大量存在时,可使对虾患病(陈琼等,2017)。本研究结果表明,在不同恩诺沙星处理下企鹅珍珠贝肠道优势菌属种类的相对丰度差异明显。经恩诺沙星处理后,企鹅珍珠贝肠道内弧菌属相对丰度显著升高,微拟球藻属、Aquibacter、BD1-7_clade及希瓦氏菌属相对丰度则显著降低;此外,弓形杆菌属相对丰度经10 mg/L恩诺沙星处理后显著升高,Psychrobium相對丰度经5 mg/L恩诺沙星处理后显著升高。说明企鹅珍珠贝经恩诺沙星处理后其肠道内菌群结构发生一定程度的变化,该结论可为企鹅珍珠贝养殖生产过程中科学使用恩诺沙星提供参考依据。
4 结论
经恩诺沙星处理后企鹅珍珠贝肠道微生物的多样性与丰度发生明显变化,且肠道微生物中一些具有特殊功能的优势菌群结构比例也发生变化,该变化规律可为企鹅珍珠贝养殖过程中科学使用恩诺沙星提供参考依据。
参考文献:
艾红,李永振,丁彦文. 2003. 海洋珍珠贝病害研究综述[J]. 上海水产大学学报,12(1):61-64. [Ai H,Li Y Z,Ding Y W. 2003. A review of study on diseases in pearl oyster[J]. Journal of Shanghai Fisheries University,12(1):61-64.]
毕水莲,罗永文,关小莺. 2017. PCR衍生技术快速检测副溶血弧菌的研究进展[J]. 江西农业学报,29(8):105-109. [Bi S L,Luo Y W,Guan X Y. 2017. Research advances in rapid detection of Vibrio parahaemolyticus by derivative techniques of PCR[J]. Acta Agriculturae Jiangxi,29(8):105-109.]
陈琼,李贵阳,罗坤,孔杰,莫照兰,栾生,李杰,曹宝祥,张玉玲. 2017. 凡纳滨对虾(Litopenaeus vannanei)亲虾繁殖期水体微生物多样性[J]. 海洋与湖沼,48(1):130-138. [Chen Q,Li G Y,Luo K,Kong J,Mo Z L,Luan S,Li J,Cao B X, Zhang Y L. 2017. Microbial diversity in broodstock waters of the two genders of Litopenaeus vannamei[J]. Oceanologia et Limnologia Sinica,48(1):130-138.]
程贊,王梅芳,万正平,余祥勇. 2011. 企鹅珍珠贝主要污损生物的季节变化及对贝生长的影响[J]. 热带生物学报,2(4):305-309. [Cheng Z,Wang M F,Wan Z P,Yu X Y. 2011. Seasonal variation of major fouling organisms of Pteria penguin(Röding) and its effects on the growth per-formance of the shell[J]. Journal of Tropical Orga-nisms,2(4):305-309.]
符韶,邓陈茂,梁飞龙,黄海立,谢仁政. 2007. 企鹅珍珠贝人工养殖及育珠的研究[J]. 广东海洋大学学报,27(1):34-37. [Fu S,Deng C M,Liang F L,Huang H L,Xie R Z. 2007. Study on technique of round-pearl and artifical cultivation in Pteria penguin(Röding)[J]. Journal of Guangdong Ocean University,27(1):34-37.]
符韶,梁飞龙. 2000. 企鹅珍珠贝附壳珍珠培育的中间试验[J]. 海洋科学,24(2):12-14. [Fu S,Liang F L. 2000. Experimental cultivation of blister pearl with pearl oyster,Pteria(Magmavicula) penguin(Röding)[J]. Marine Sciences,24(2):12-14.]
郭华阳,李有宁,张楠,朱克诚,王雨,张殿昌. 2016. 企鹅珍珠贝早期养殖生长性状的增长规律及生长曲线拟合研究[J]. 南方水产科学,12(5):71-80. [Guo H Y,Li Y N,Zhang N,Zhu K C,Wang Y,Zhang D C. 2016. Study on development rules and growth fitting of early cultured winged pearl oyster(Pteria penguin)[J]. South China Fi-sheries Science,12(5):71-80.]
李启蒙. 2017. 山东省贝类弧菌流行病学调查、药敏试验及毒力基因检测[D]. 泰安:山东农业大学. [Li Q M. 2017. Epidemiology,antibiotic susceptibility test and virulence gene detection of vibrios islotated from shellfish in Shandong Province[D]. Tai’an:Shandong Agricultural University.]
栗志民,刘志刚,王辉,唐西岳. 2011. 企鹅珍珠贝(Pteria penguin)主要经济性状对体重的影响效果分析[J]. 海洋与湖沼, 42(6):798-803. [Li Z M,Liu Z G,Wang H,Tang X Y. 2011. Effects of main economic traits on body weight of Pteria penguin[J]. Oceanologia et Limnologia Sinica,42(6):798-803.]
梁飞龙,刘永,邓陈茂,毛勇. 2007. 广东雷州流沙湾养殖企鹅珍珠贝多毛类寄生虫病的调查[J]. 海洋水产研究,28(2):84-89. [Liang F L,Liu Y,Deng C M,Mao Y. 2007. Survey on polychaete verminosis in farmed pearl oyster (Pteria penguin) in Liusha Bay,Leizhou,Guangdong [J]. Marine Fisheries Research,28(2):84-89.]
梁飞龙,谢绍河,林伟财. 2014. 企鹅珍珠贝珍珠培育技术的研究现状[J]. 水产养殖,35(4):37-41. [Liang F L,Xie S H,Lin W C. 2014. Progress on pearl production of pearl oyster Pteria penguin[J]. Journal of Aquaculture,35(4):37-41.]
陆彤霞,王国良,尤仲杰,郑天伦. 2002. 我国海洋养殖贝类病害研究概况及防治对策[J]. 浙江海洋学院学报(自然科学版),21(2):154-159. [Lu T X,Wang G L,You Z J,Zheng T L. 2002. Advances of studies and the measures of prevention and cure of diseases of marine culture mo-llusc in China[J]. Journal of Zhejiang Ocean University(Natural Science),21(2):154-159.]
毛勇,梁飞龙,符韶,余祥勇,叶富良,邓陈茂. 2004. 企鹅珍珠贝彩虹珠的研究初报[J]. 动物学杂志,39(1):100-102. [Mao Y,Liang F L,Fu S,Yu X Y,Ye F L,Deng C M. 2004. Preliminary studies on rainbow-pearl of Penguin Wing oyster Pteria penguin[J]. Chinese Journal of Zoolo-gy,39(1):100-102.]
蒙釗美,李有宁,邢孔武. 1996. 珍珠养殖理论与技术[M]. 北京:科学出版社. [Meng Z M,Li Y N,Xing K W. 1996. Pearl Culture Theory and Technology[M]. Beijing:Scien-ce Press.]
邱莹,黄桂菊,刘宝锁,范嗣刚,李有宁,陈明强,喻达辉. 2016. 企鹅珍珠贝GLUT1基因全长cDNA克隆及其对葡萄糖的表达响应[J]. 南方水产科学,12(5):81-89. [Qiu Y,Huang G J,Liu B S,Fan S G,Li Y N,Chen M Q,Yu D H. 2016. Clonging of GLUT1 gene from winged pearl oyster Pteria penguin and its expression in response to glucose challenge[J]. South China Fisheries Science,12(5):81-89.]
施兆鸿,王建建,高权新. 2015. 工厂化循环水养殖条件下云纹石斑鱼消化道产酶菌的分离鉴定[J]. 中国水产科学,22(5):941-949. [Shi Z H,Wang J J,Gao Q X. 2015. Isolation and identification of enzyme-producing bacteria from the digestive tract of Epinehelus moara in re-circulating aquaculture systems[J]. Journal of Fishery Scien-ces of China,22(5):941-949.]
余祥勇,王梅芳,梁飞龙,刘永. 2005. 企鹅珍珠贝不同组织同工酶表达的差异[J]. 中国水产科学,12(2):201-206. [Yu X Y,Wang M F,Liang F L,Liu Y. 2005. Differentiation of isozymes from different tissues of Pteria penguin Röding[J]. Journal of Fishery Sciences of China,12(2):201-206.]
余祥勇,王梅芳,刘永,梁飞龙,毛勇,桂建芳. 2004. 企鹅珍珠贝同工酶酶谱特征及其遗传分析[J]. 水产学报,28(4):375-383. [Yu X Y,Wang M F,Liu Y,Liang F L,Mao Y,Gui J F. 2004. The pecaliarities zymogram and their genetic analysis in Pteria penguin[J]. Journal of Fishe-ries of China,28(4):375-383.]
余祥勇,王梅芳,叶富良. 2000. 企鹅珍珠贝个体发生及人工育苗的研究[J]. 海南大学学报(自然科学版),18(3):266-270. [Yu X Y,Wang M F,Ye F L. 2000. Development and artificial propagation of Pteria(Magnaricula) penguin Röding[J]. Natural Science Journal of Hainan University,18(3):266-270.]
张莹,石萍,马炯. 2013. 微小杆菌Exiguobacterium spp.及其环境应用研究进展[J]. 应用与环境生物学报,19(5):898-904. [Zhang Y,Shi P,Ma J. 2013. Exiguobacterium spp. and their applications in environmental remediation[J]. Chinese Journal of Applied Environmental Biology,19(5):898-904.]
赵翔刚,罗衡,刘其根,赵良杰,蔡林荣,戴亮亮,张真. 2017. 稻田养殖沙塘鳢对稻田水体及底泥微生物群落结构及多样性的影响[J]. 淡水渔业,47(4):8-14. [Zhao X G,Luo H,Liu Q G,Zhao L J,Cai L R,Dai L L,Zhang Z. 2017. Influence of the cultured Odontobutis obscurus to the microbial community structure and diversity in rice-fish system[J]. Freshwater Fisheries,47(4):8-14.]
趙晓伟,丁君,窦妍,王梦鸽,常亚青. 2015. 基于Miseq测序技术分析红鳍东方鲀养殖环境菌群多样性[J]. 生态学杂志, 34(10): 2965-2970. [Zhao X W,Ding J,Dou Y,Wang M G,Chang Y Q. 2015. Bacterial diversity in the breeding environment of Takifugu rubripes revealed by MiSeq sequencing[J]. Chinese Journal of Econology,34(10):2965-2970.]
Arijarasirikoon U,Kruatrachue M,Sretarugsa P. 2004. Gametogenic progress in the pearl oyster,Pteria Penguin (Roe-ding 1798)(Bivalvia,Mollusea)[J]. Journal of Shellfish Research,23(2):403-409.
Balaje R M,Sidhu P K,Kaur G,Rampal S. 2013. Mutant prevention concentration and PK-PD relationships of enrofloxacin for Pasteurella multocida in buffalo calves[J]. Research in Veterinary Science,95(3):1114-1124.
Caporaso J G,Kuczynski J,Stombaugh J,Bittinger K,Bushman F D,Costello E K,Fierer N,Peña A G,Goodrich J K,Gordon J I,Huttley G A,Kelley S T,Knights D,Koenig J E,Ley R E,Lozupone C A,McDonald D,Muegge B D,Pirrung M, Reeder J,Sevinsky J R,Turnbaugh P J,Walters W A,Widmann J,Yatsunenko T,Zaneveld J,Knight R. 2010. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods,7(5):335-336.
Chen B,Gao L L,Pan Q. 2018. Woody forages effect the intestinal bacteria diversity of golden pompano Trachinotus ovatus[J]. AMB Express,8(1):29.
Cottrell M T,Kirchman D L. 2000. Natural assemblages of marine proteobacteria and members of the Cytophaga-Flavobacter cluster consuming low- and high-molecular-weight dissolved organic matter[J]. Applied and Environmental Microbiology,66(4): 1692-1697.
Demergasso C,Casamayor E O,Chong G,Gallequillos P,Escudero L,Pedrós-Alió C. 2004. Distribution of prokaryo-tic genetic diversity in athalassohaline lakes of the Ata-cama Desert,Northern Chile[J]. FEMS Microbiology Eco-logy,48(1):57-69.
Fang X,Liu X,Liu W,Lu C. 2012. Pharmacokinetics of enrofloxacin in allogynogenetic silver crucian carp,Carassius auratus gibelio[J]. Journal of Veterinary Pharmacology & Therapeutics,35(4):397-401.
Hamilton M J,Weingarden A R,Unno T,Khoruts A,Sadow-sky M J. 2013. High-throughput DNA sequence analysis reveals stable engraftment of gut microbiota following transplantation of previously frozen fecal bacteria[J]. Gut Microbes,4(2):125-135.
Hooper L V,Gordon J I. 2001. Commensal host-bacterial relationships in the guts[J]. Science,292(5519):1115-1118.
Jumpertz R,Le D S,Turnbaugh P J,Trinidad C,Bogardus C,Gordon J I,Krakoff J. 2011. Energy-balance studies reveal associations between gut microbes,caloric load,and nutrient absorption in humans[J]. The American Journal of Clinical Nutrition,94(1):58-65.
Komaroff A L. 2017. The microbiome and risk for obesity and diabetes[J]. JAMA,317(4):355-356.
Ley R E,Peterson D A,Gordon J I. 2006. Ecological and evolutionary forces shaping microbial diversity in the human intestine[J]. Cell,124(4):837-848.
Lyons P,Turnbull J,Dawson K,Crumlish M. 2015. Exploring the microbial diversity of the distal intestinal lumen and mucosa of farmed rainbow trout Oncorhynchus mykiss (Walbaum) using next generation sequencing(NGS)[J]. Aquaculture Research,48(1):77-91.
Meng H,Zhang Y,Zhao L,Zhao W,He C,Honaker C F,Zhai Z,Sun Z,Siegel P B. 2014. Body weight selection affects quantitative genetic correlated responses in gut microbiota[J]. PLoS One,9(3):e89862.
Navarrete P,Magne F,Araneda C,Fuentes P,Barros L,Opazo R,Espejo R,Romero J. 2012. PCR-TTGE analysis of 16S rRNA from rainbow trout(Oncorhynchus mykiss) gut microbiota reveals host-specific communities of active bacteria[J]. PLoS One,7(2):e31335.
Oxley A P A,Shipton W,Owens L,Mckay D. 2002. Bacterial flora from the gut of the wild and cultured banana prawn,Penaeus merguiensis[J]. Journal of Applied Microbiology,93(2):214-223.
Pandey N,Bhatt R. 2015. Exiguobacterium mediated arsenic removal and its protective effect against arsenic induced toxicity and oxidative damage in freshwater fish,Channa striata[J]. Toxicology Reports,2:1367-1375.
Petrosino J,Highlander S,Luna R,Gibbs R A,Versalovic J. 2009. Metagenomic pyrosequencing and microbial identification[J]. Clinical Chemistry,55(5):856-866.
Sakatoku A,Fujimura T,Ito M,Takashima S,Isshiki T. 2018. Newly isolated bacterium Tenacibaculum sp. strain Pbs-1 from diseased pearl oysters is associated with black-spot shell disease[J]. Aquaculture,493:61-67.
Savas S,Kubilay A,Basmaz N. 2005. Effect of bacterial load in feeds on intestinal microflora of seabream(Sparus aurata) larvae and juveniles[J]. Israeli Journal of Aquaculture Bamidgeh,57(1):3-9.
Schloss P D,Westcott S L,Ryabin T,Hall J R,Hartmann M,Hollister E B,Lesniewski R A,Oakley B B,Parks D H,Robinson C J,Sahl J W,Stres B,Thallinger G G,Van Horn D J,Weber C F. 2009. Introducing mothur:Open-source,platform-independent,community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology,75(23):7537-7541.
Skrodenytė-Arbačiauskienė V,Sruoga A,Butkauskas D,Skrupskelis K. 2008. Phylogenetic analysis of intestinal bacteria of freshwater salmon Salmo salar and sea trout Salmo trutta trutta and diet[J]. Fisheries Science,74(6):1307-1314.
Skrodenytė-Arbačiauskienė V. 2007. Enzymatic activity of intestinal bacteria in roach Rutilus rutilus L[J]. Fisheries Science,73(4):964-966.
Sombatjinda S,Wantawin C,Techkarnjanaruk S,Withyachumnarnkul B,Ruengjitchatchawalya M. 2014. Water quality control in a closed re-circulating system of Pacific white shrimp(Penaeus vannamei) postlarvae co-cultured with immobilized Spirulina mat[J]. Aquaculture International,22(3):1181-1195.
Stevens H,Stübner M,Simon M,Brinkhoff T. 2005. Phylogeny of Proteobacteria and Bacteroidetes from oxic habitats of a tidal flat ecosystem[J]. FEMS Microbiology Ecology,54(3):351-365.
Sullam K E,Essinger S D,Lozupone C A,Óconnor M P,Rosen G L,Knight R,Rilham S S,Russell J A. 2012. Environmental and ecological factors that shape the gut bacterial communities of fish:A meta-analysis[J]. Molecular Ecology,21(13):3363-3378.
Tan W S,Yunos N,Tan P W,Mohamad N I,Adrian T G,Yin W F,Chan K G. 2014. Characterisation of a marine bacterium Vibrio brasiliensis T33 producing N-acyl homose-rine lactone quorum sensing molecules[J]. Sensors,14(7):12104-12113.
Ward-Rainey N,Rainey F A,Stackebrandt E. 1996. A study of the bacterial flora associated with Holothuria atra[J]. Journal of Experimental Marine Biology and Ecology,203(1):11-26.
Wassnig M,Southgate P C. 2012. Embryonic and larval deve-lopment of Pteria penguin(Röding,1798)(Bivalvia:Pteriidae)[J]. Journal of Molluscan Studies,78(1):134-141.
Wu L Y,Wen C Q,Qin Y J,Yin H Q,Tu Q C,Nostrand J D V,Yuan T,Yuan M T,Deng Y,Zhou J Z. 2015. Phasing amplicon sequencing on Illumina Miseq for robust environmental microbial community analysis[J]. BMC Microbiology,15:125. doi: 10.1186/s12866-015-0450-4.
Wu S G,Gao T H,Zheng Y Z,Wang W W,Cheng Y Y,Wang G T. 2010. Microbial diversity of intestinal contents and mucus in yellow catfish(Pelteobagrus fulvidraco)[J]. Aquaculture,303(1-4):1-7.
Ye L,Amberg J,Chapman D,Gaikowski M,Liu W T. 2014. Fish gut microbiota analysis differentiates physiology and behavior of invasive Asian carp and indigenous American fish[J]. The ISME Journal,10(8):2076. doi: 10.1038/ismej.2016.71.
(責任编辑 兰宗宝)

