玉米ZmcpSRP54基因可变剪接分析
关海英 何春梅 王娟 刘铁山 董瑞 刘春晓 刘强 汪黎明

摘要:cpSRP54基因在植物叶绿体发育中发挥重要作用。本研究用生物信息学方法对玉米ZmcpSRP54蛋白进行了进化分析,利用RT-PCR方法对玉米ZmcpSRP54基因全长cDNA进行了克隆,进而对其进行可变剪接分析。结果表明,玉米ZmcpSRP54蛋白与其它7个物种的cpSRP54蛋白有很高的相似性。鉴定到玉米ZmcpSRP54基因25个不同转录本,1个为标准剪接转录本,其它24个为可变剪接转录本。24个可变剪接转录本共使用了17种非标准剪接位点,4种可变剪接方式,主要以可变的3′端位点、可变的5′端位点和外显子跳跃3种可变剪接方式为主。预测出18种不同长度的ORF,编码18种不同长度的多肽。11个多肽其保守结构域氨基酸序列发生部分缺失,6个多肽其保守结构域氨基酸序列发生全部缺失,1个编码全新的多肽。这些结果将为玉米ZmcpSRP54基因的功能研究提供重要信息。
关键词:玉米;ZmcpSRP54基因;RT-PCR;可变剪接 中图分类号:S513:Q781 文献标识号:A 文章编号:1001-4942(2019)06-0001-10
Abstract The cpSRP54 gene plays an important role during chloroplast development in plant. In this study, the bioinformatics method was used for the evolutionary analysis of maize ZmcpSRP54 protein, the RT-PCR method was used for the cloning of full-length cDNA of maize ZmcpSRP54 gene, and then the bioinformatics methods were used for the analysis of alternative splicing. Phylogenetic analysis revealed that the maize ZmcpSRP54 protein was conserved with those cpSRP54 proteins in the other seven species. A total of 25 different transcripts of ZmcpSRP54 were obtained, and 1 was a standard splicing transcript and the other 24 were alternative splicing (AS) transcripts. A total of 17 non-standard splice sites and 4 AS forms were used for the 24 AS transcripts, and alternative 3′ splice site, alternative 5′ splice site and exon skipping were the most prevalent AS forms. A total of 18 ORFs with different lengths were predicted for these 24 AS transcripts, encoding 18 polypeptides with different lengths, and 11 of which had part of the amino acid sequences in the conserved region deleted, 6 of which had all of the amino acid sequences in the conserved region deleted, while 1 of which encoded another new polypeptide. These results would provide important information for the functional study of the ZmcpSRP54 gene in maize.
Keywords Maize; ZmcpSRP54 gene; RT-PCR; Alternative splicing
cpSRP54(chloroplast signal recognition particle 54)是cpSRP(cpSRP54,cpSRP43,cpFtsY和ALB3)途径的重要成员之一,在叶绿体发育中发挥重要作用,该蛋白与其它3个成员相互作用将捕光叶绿素蛋白(LHCPS)从叶绿体基质转运到内囊体膜上[1,2]。拟南芥和水稻中的cpSRP54基因突变后其叶绿体发育均受阻,叶片叶绿素含量显著下降导致植株叶片颜色呈现浅绿色[3-5]。其中水稻ygl14(t)突变体是由于第1个内含子与第2个外显子交界处的第2个碱基A变为T,导致OscpSRP54第一内含子保留,造成氨基酸错译而引起植株叶片颜色发生变异[3]。
可变剪接(alternative splicing,AS)是指一个前体mRNA选择不同的剪接方式(通过不同的内含子剪切和外显子连接方式)产生多种mRNA结构的过程[6]。可变剪接是一种关键的转录后调控机制,能使一个基因产生多个转录本和蛋白异构体,丰富了蛋白质的多样性,也增加了其功能的复杂性[6-8]。前人研究表明,人体基因中约有95%以上都存在可变剪接[9]。1989年,在拟南芥和菠菜中发现1, 5-二磷酸核酮糖羧化加氧酶活化酶(rubsicoactivase)基因发生可变剪接[10]。后经过20多年的研究发现,可变剪接是调控植物基因表達的重要方式[11],参与植物的很多生理代谢过程、信号转导以及对外界生物和非生物胁迫的响应[12-17]。植物基因中大约60%都存在可变剪接[12]。可变剪接方式主要包括外显子跳跃(exon skipping,ES)、内含子保留(intron retention,IR)、可变的5′端位点(alternative 5′splice site,A5SS)、可变的3′端位点(alternative 3′ splice site,A3SS)、互斥外显子(mutually exclusive exon,MEX)等5种基本剪接方式[18]。植物中最多的剪接方式是内含子保留[19-22]。
用水稻OscpSRP54基因序列在玉米中进行同源比对发现玉米ZmcpSRP54基因位于10号染色体上,DNA全长7 526 bp(GRMZM2G113093,B73_RefGen_v3,http://www.maizegdb.org)。该基因有4个不同的转录本(GRMZM2G113093_T01和GRMZM2G113093_T02,B73_RefGen_v3;Zm00001d023431_T001和Zm00001d023431_T002,B73_RefGen_v4,http://ensembl.gramene.org/Zea_mays/Info/Index),其中3个转录本(GRMZM2G113093_T01、Zm00001d023431_T001和Zm00001d023431_T002)包括15个外显子和14个内含子,1个转录本(GRMZM2G113093_T02)包括10个外显子和9个内含子。4个转录本编码3种不同长度的多肽,分别包含556(GRMZM2G113093_T01和Zm00001d023431_T001)、351(GRMZM2G113093_T02)和564(Zm00001d023431_T002)个氨基酸。为了进一步研究该基因是否还有其它可变剪接转录本,本研究通过RT-PCR方法对ZmcpSRP54基因全长cDNA进行克隆,并利用生物信息学软件探索其可变剪接方式,以期为进一步研究玉米及其它物种中该基因的可变剪接机制以及深入研究该基因的功能提供参考依据。
1 材料与方法
1.1 试验材料 试验所用材料是玉米自交系B73,种植于山东章丘龙山基地。开花期取穗位叶,快速置于液氮中速冻,置-80℃冰箱贮存,用于后续RNA提取。
1.2 多序列比对及进化分析 将ZmcpSRP54蛋白序列通过NCBI 数据库进行BLAST比对分析,下载其他物种的同源蛋白序列。利用DNAMAN 5.2.2软件进行多重氨基酸序列比对。利用MEGA 7.0.14 软件对该基因和其他物种来源的同源基因进行氨基酸序列分析,并利用邻接法(NL) 构建系统进化树
1.3 RNA提取及第一链cDNA获得 将玉米叶片组织用液氮研磨至粉末,取100 mg利用RNA提取试剂盒(TIANGEN,北京)提取RNA。通过NanoDrop 2000分光光度计和1%琼脂糖电泳检测RNA纯度和浓度,取约4 μg RNA利用反转录试剂盒(TaKaRa,大连)反转录获得第一链cDNA。所得第一链cDNA稀释4倍后用于后续RT-PCR扩增。
1.4 RT-PCR扩增、测序与可变剪接分析 利用Primer Premier 5.0软件设计扩增ZmcpSRP54基因cDNA全长引物(F: 5′- CTGCTCCCGTACCTCGTCTC-3′; R: 5′- TCCGAGCCCAAGTCCCTAA-3′)。RT-PCR 反应体系(20 μL): TksGflexTM DNA Polymerase (TAKARA,大连)0.3 μL,正反向引物各0.75 μL,模板2 μL,2×TksGflex Buffer 10 μL,加ddH2O补足体系。反应程序:95℃预变性5 min;95℃变性45 s,60℃退火45 s,72℃延伸2 min,共32 个循环,结束反应。用1%琼脂糖凝胶电泳检测PCR扩增产物。PCR产物经回收纯化后连接pEASY-Blunt载体(全式金,北京),转化大肠杆菌(Escherichia coli)感受态细胞DH5α(全式金,北京),挑取单克隆进行PCR阳性鉴定。将鉴定的阳性单克隆进行测序(英潍捷基,北京),获得全长序列。利用Splign(https://www.ncbi.nlm.nih.gov/sutils/splign/splign.cgi)、DNAStar(http://www.dnastar.com)和BioXM 2.6 软件(http://bioxm.software.informer.com/)对这些序列进行比对、外显子/内含子结构分析、ORF预测和编码氨基酸长度分析等。
2 结果与分析2.1 ZmcpSRP54基因进化分析
用ZmcpSRP54蛋白序列在NCBI网站进行BLASTP,搜索高粱、谷子、黍子、水稻、乌拉尔图小麦、二穗短柄草和拟南芥等物种相应蛋白质序列,利用DNAMAN 5.2.2和MEGA 7.0.14 软件进行多序列比对和构建进化树。由图1可見,玉米ZmcpSRP54蛋白PBK10867结构域(信号颗粒识别蛋白)氨基酸序列与其它物种cpSRP54蛋白相应氨基酸序列有很高的相似性。由图2可见,玉米ZmcpSRP54与单子叶植物包括高粱SbcpSRP54、谷子SicpSRP54、黍子PhcpSRP54、水稻OscpSRP54、乌拉尔图小麦TucpSRP54和二穗短柄草BdcpSRP54聚为一个分支,双子叶植物拟南芥AtcpSRP54聚为另一个分支。与玉米ZmcpSRP54亲缘关系最近的是高粱SbcpSRP54,其次为谷子SicpSRP54,其余亲缘关系由近到远依次是黍子PhcpSRP54、水稻OscpSRP54、乌拉尔图小麦TucpSRP54、二穗短柄草BdcpSRP54和拟南
这些结果表明cpSRP54蛋白在植物中是保守的。2.2 RT-PCR扩增获得ZmcpSRP54基因25个转录本
利用设计的扩增ZmcpSRP54基因cDNA全长引物,以玉米自交系B73叶片cDNA为模板进行RT-PCR扩增,发现扩增条带不单一,有多条亮度强弱不等及片段大小不等的条带(图3)。Maizegdb(http://www.maizegdb.org)和Gramene(http://ensembl.gramene.org/Zea_mays/Info/Index)网站报道该基因有4个转录本,我们推测该基因可能有多个转录本。将PCR产物回收纯化后连接克隆载体,64个单菌落经PCR鉴定后得到61个阳性单克隆,将鉴定的阳性单克隆送测序公司测序。所得序列经生物信息学软件分析发现共有25种不同转录本(图3B,表1),分别命名为AS1~AS25,除AS2与GRMZM2G113093_T01相同外,其余24种属于新转录本(A1和A3~A25)。9个克隆所测序列是AS2转录本(14.75%,所占比例最高),其又与GRMZM2G113093_T01相同,推测其为标准剪接方式,其它24个转录本(共52个克隆)为非标准剪接方式。
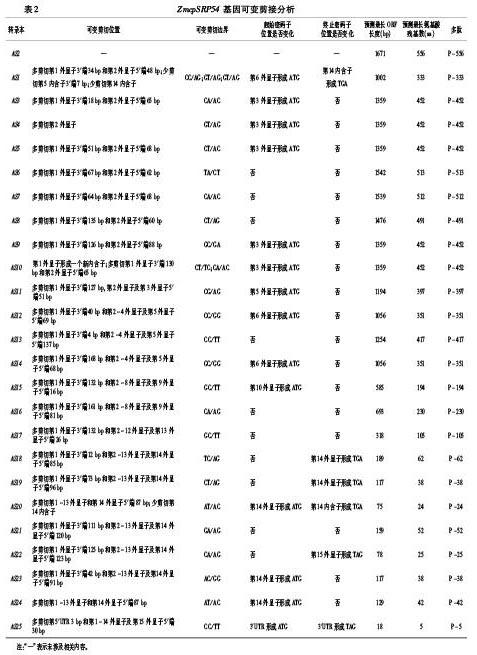
2.3 ZmcpSRP54基因24个新转录本可变剪接分析 以AS2转录本为参照,分析其它24个转录本的可变剪接方式,结果见表2。24个新转录本主要有4种可变剪接方式,包括外显子跳跃、内含子保留、可变的5′端位点和可变的3′端位点。1个转录本(AS4,3个可变剪接克隆)仅使用外显子跳跃一种可变剪接方式;6个转录本(AS3、AS5、AS6、AS7、AS8和AS9,共17个可变剪接克隆)联合使用可变的3′端位点和可变的5′端位点两种可变剪接方式;1个转录本(AS24,1个可变剪接克隆)联合使用可变的3′端位点和外显子跳跃两种可变剪接方式;13个转录本(AS11、AS12、AS13、AS14、AS15、AS16、AS17、AS18、AS19、AS21、AS22、AS23和AS25,共27个可变剪接克隆)联合使用可变的3′端位点、外显子跳跃和可变的5′端位点这三种可变剪接方式;1个转录本(AS1,1个可变剪接克隆)联合使用可变的3′端位点、可变的5′端位点和内含子保留三种可变剪接方式;1个转录本(AS20,2个可变剪接克隆)联合使用可变的3′端位点、外显子跳跃和内含子保留三种可变剪接方式;1个转录本(AS10,1个可变剪接克隆)联合使用可变的3′端位点和可变的5′端位点两种可变剪接方式,同时其第1外显子内部发生42 bp的剪切形成一个新的内含子(表1、表2)。本研究鉴定到的24种可变剪接转录本所使用的4种可变剪接方式中,主要是以可变的3′端位点(23个可变剪接转录本AS1、AS3、AS5、AS6、AS7、AS8、AS9、AS10、AS11、AS12、AS13、AS14、AS15、AS16、AS17、AS18、AS19、AS20、AS21、AS22、AS23、AS24和AS25,共49个可变剪接克隆),可变的5′端位点(21个可变剪接转录本AS1、AS3、AS5、AS6、AS7、AS8、AS9、AS10、AS11、AS12、AS13、AS14、AS15、AS16、AS17、AS18、AS19、AS21、AS22、AS23和AS25,共46个可变剪接克隆)和外显子跳跃(16个可变剪接转录本AS4、AS11、AS12、AS13、AS14、AS15、AS16、AS17、AS18、AS19、AS20、AS21、AS22、AS23、AS24和AS25,33个可变剪接克隆)三种可变剪接方式为主。 24个可变剪接转录本共使用了17种非标准可变剪接位点:①5′-CC..AG-3′(AS1,1个可变剪接克隆);②5′-CA..AC-3′(AS3、AS7和 AS10,共7个可变剪接克隆);③5′-CT..AC-3′(AS5,2个可变剪接克隆);④5′-TA..CT-3′(AS6,4个可变剪接克隆);⑤5′-CT..AG-3′(AS8和AS19,共7个可变剪接克隆);⑥5′-GC..GA-3′(AS9,2个可变剪接克隆);⑦5′-CT..TC-3′(AS10,1个可变剪接克隆);⑧5′-CG..AG-3′(AS11,2个可变剪接克隆);⑨5′-CC..GG-3′(AS12,3个可变剪接克隆);⑩5′-CC..TT-3′(AS13和AS25,共4个可变剪接克隆);5′-GC..GG-3′(AS14,3个可变剪接克隆);5′-GC..TT-3′(AS15和AS17,共5个可变剪接克隆);5′-CA..AG-3′(AS16和AS22,共2个可变剪接克隆);5′-TC..AG-3′(AS18,1个可变剪接克隆);5′-AT..AC-3′(AS20和AS24,共3个可变剪接克隆);5′-GA..AG-3′(AS21,2个可变剪接克隆);5′-AC..GG-3′(AS23,1个可变剪接克隆)。AS10转录本(1个可变剪接克隆)使用了两种非标准可变剪接位点②5′-CA..AC-3′和⑦5′-CT..TC-3′;AS4转录本(3个可变剪接克隆)只是將第2外显子剪切,未使用非标准剪接位点;其它22个转录本(共48个可变剪接克隆)均只使用一种非标准可变剪接位点。 从可变剪接发生的位置与外显子相关来看,与第2外显子有关的转录本最多(全部24个可变剪接转录本),8个转录本第2外显子5′端被部分剪切(AS1、AS3、AS5、AS6、AS7、AS8、AS9和AS10,共19个可变剪接克隆),其余16个转录本第2外显子全部被剪切(共33个可变剪接克隆);其次是第1外显子(除AS4外的其余23个可变剪接转录本),AS20、AS24和AS25转录本第1外显子全部被剪切(共4个可变剪接克隆),AS10转录本第1外显子内部被剪切42 bp形成新内含子,其余19个转录本第1外显子3′端被部分剪切(共44个可变剪接转录本);最少的是第15外显子,只有AS25转录本第15外显子5′端被部分剪切。从可变剪接发生的位置与内含子相关来看,只有第5内含子部分保留(AS1)和第14内含子全部保留(AS1和AS20)。
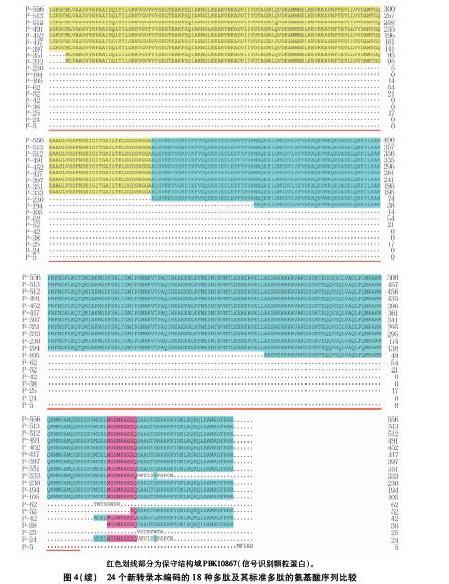
24种可变剪接转录本预测的最长ORF均比AS2转录本的ORF短(表2),其长度短的主要原因有5种:①起始密码子ATG和终止密码子TGA位置均未发生改变,中间区段被部分剪切(AS6,AS7,AS8,AS13,AS16,AS17和AS21,共18个可变剪接克隆);②起始密码子ATG位置未发生改变,终止密码子TGA(TAG)向前移动(AS18,AS19和AS22,共6个可变剪接克隆);③起始密码子ATG向后移动,终止密码子TGA位置未发生改变(AS3,AS4,AS5,AS9,AS10,AS11,AS12,AS14,AS15,AS23和AS24,共24个可变剪接克隆);④起始密码子ATG向后移动,终止密码子TGA向前移动(AS1和AS20,共3个可变剪接克隆);⑤起始密码子ATG和终止密码子TGA均向后移动至原来ORF后,形成全新ORF(AS25,1个可变剪接克隆)。24种转录本预测的18种不同长度ORFs预测编码18种长度不等的多肽,分别包括513(AS6)、512(AS7)、491(AS8)、452(AS3、AS4、AS5、AS9和AS10)、417(AS13)、351(AS12和AS14)、397(AS11)、333(AS1)、230(AS16)、194(AS15)、105(AS17)、62(AS18)、52(AS21)、42(AS24)、38(AS19和AS23)、25(AS22)、24(AS20)、5(AS25)个氨基酸(图4)。24种可变剪接转录本编码的多肽其保守结构域氨基酸序列发生部分缺失或完全缺失(图4)。多肽P-513缺失43个氨基酸(37~79位),后5个氨基酸位于保守结构域;多肽P-512缺失44个氨基酸(38~81位),后7个氨基酸位于保守结构域;多肽P-491缺失65个氨基酸(14~78位),后4个氨基酸位于保守结构域;多肽P-452缺失104个氨基酸(1~104位),后30个氨基酸位于保守结构域;多肽P-417缺失139个氨基酸(58~196位),后122个氨基酸位于保守结构域;多肽P-397缺失159个氨基酸(1~159位),后85个氨基酸位于保守结构域;多肽P-351缺失205个氨基酸(1~205位),后131个氨基酸位于保守结构域;多肽P-333缺失223个氨基酸(1~205位;539~556位),缺失的205个氨基酸其后131个氨基酸位于保守结构域,538位氨基酸提前终止;多肽P-230缺失326个氨基酸(6~331位),后257个氨基酸位于保守结构域;多肽P-194缺失362个氨基酸(1~362位),后288个氨基酸位于保守结构域;多肽P-105缺失451个氨基酸(15~465位),后391个氨基酸位于保守结构域;其它6个多肽(P-62,P-52,P-42,P-38,P-25和P-24)保守结构域氨基酸序列全部缺失;P-5是由于AS25在3′UTR形成短的ORF编码的全新的多肽。
3 讨论与结论
Maizegdb和Gramene网站报道ZmcpSRP54基因有4个转录本,GRMZM2G113093_T01,GRMZM2G113093_T02,Zm00001d023431_T001和Zm00001d023431_T002。本研究通过RT-PCR扩增及测序获得了ZmcpSRP54基因的25个转录本,通过分析发现AS2与GRMZM2G113093_T01相同,没有鉴定到GRMZM2G113093_T02,Zm00001d023431_T001和Zm00001d023431_T002这3个转录本,可能是这几个转录本的表达量比较低,没有被克隆到。植物中可变剪接方式以内含子保留为主[19-22],外显子跳跃出现的频率最低[12]。本研究鑒定到的4种可变剪接方式,主要以可变的3′端位点、可变的5′端位点和外显子跳跃三种可变剪接方式为主,植物中一个转录本也会同时联合使用两个及以上可变剪接方式[23]。本研究发现23个(24个可变剪接转录本中除AS4外)可变剪接转录本同时使用两种及以上可变剪接方式,如果这也是普遍存在的,就会显著增加转录后加工的复杂性。植物内含子序列的识别位点具有高度保守性,约99.24%的剪接位点符合GT-AG规则[24],有的还遵循AT-AC规则[25]。本研究24个可变剪接转录本除了使用GT-AG和AT-AC规则外,还使用其它16种非标准剪接位点。
本研究鉴定到的25个转录本预测编码19个长度不同的多肽,除AS2转录本编码的多肽P-556与GRMZM2G113093_T01和Zm00001d023431_T001编码的多肽相同,AS12和AS14转录本(AS12和AS14转录本与GRMZM2G113093_T02序列不同)编码的多肽P-351与GRMZM2G113093_T02编码的多肽相同,其它17个为新的多肽。除P-556外的18个多肽中,11个多肽其保守结构域氨基酸序列发生部分缺失, 6个多肽保守结构域氨基酸序列发生全部缺失,还有1个长度只有5个氨基酸的全新多肽。保守结构域氨基酸序列发生部分缺失的11个多肽是否具有生物学功能,以及其它6个保守结构域氨基酸序列发生全部缺失的多肽及全新多肽P-5是否完全丧失生物学功能,仍然有待进一步研究。本研究发现的多个可变剪接体也为进一步研究玉米ZmSRP54蛋白功能提供了重要信息。
参 考 文 献:
[1] Aldridge C, Cain P, Robinson C. Protein transport in organelles:protein transport into and across the thylakoid membrane [J]. Febs J., 2009, 276(5): 1177-1186.
[2] Tzvetkova-Chevolleau T, Hutin C, Noel L D, et al. Canonical signal recognition particle components can be bypassed for posttranslational protein targeting in chloroplasts [J]. Plant Cell, 2007, 19: 1635-1648.
[3] 林添资,孙立亭,景德道,等.一个水稻黄绿叶突变体ygl14(t)的鉴定及基因定位[J]. 核农学报,2018, 32(2):216-226.
[4] Amin P, Sy D A C, Pilgrim M L, et al. Arabidopsis mutants lacking the 43- and 54-kilodalton subunits of the chloroplast signal recognition particle have distinct phenotypes [J]. Plant Physiol., 1999, 121(1): 61-70.
[5] Zhang F T, Luo X D, Hu B L, et al. YGL138(t), encoding a putative signal recognition particle 54 kDa protein, is involved in chloroplast development of rice [J]. Rice, 2013, 6:7.
[6] 邢永强,刘国庆,蔡禄. Pre-mRNA选择性剪接的调控及选择性剪接数据库[J].中国生物化学与分子生物学报,2016,32(1): 17-28.
[7] Luco R F, Allo M,Schor I E,et al. Epigenetics in alternative pre-mRNA splicing[J]. Cell, 2011, 144(1): 16-26.
[8] Tress M L, Abascal F, Valencia A. Alternative splicing may not be the key to proteome complexity [J]. Trends Biochem.Sci., 2017, 42: 98-110.
[9] Pan Q, Shai O, Lee L J, et al. Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing [J]. Nat. Genet., 2008, 40:1413-1415.
[10] Werneke J M,Chatfield J M,Ogren W L. Alternative mRNA splicing genernatesthe two ribulose bisphosphate carboxylase/oxygenase activase polypeptides in spinach and Arabidopsis[J]. Plant Cell, 1989, 1:815.
[11] Syed N H, Kalyna M, Marquez Y, et al. Alternative splicing in plants—coming of age[J]. Trends Plant Sci., 2012, 17: 616.
[12] Barbazuk W B, Yan F, McGinnis K M. Genome-wide analyses of alternative splicing in plants:opportunities and challenges [J]. Genome Res., 2008, 18(9):1381-1392.
[13] Xu S H, Zhang Z B, Jing B B, et al. Transportin-SR is required for proper splicing of resistance genes and plant immunity[J]. PLoS Genet., 2011, 7: e1002159.
[14] Mastrangelo A M, Marone D, Laidò G, et al. Alternative splicing:enhancing ability to cope with stress via transcriptome plasticity [J]. Plant Sci., 2012, 185:40-49.
[15] Dubrovina A S, Kiselev K V, Zhuravlev Y N. The role of canonical and noncanonical pre-mRNA splicing in plant stress responses [J]. Biomed.Res. Int., 2013, 2013:264314.
[16] Carvalho R F, Feijo C V, Duque P. On the physiological significance of alternative splicing events in higher plants[J]. Protoplasma, 2013, 250(3): 639-650.
[17] Seo P J, Park M J, Park C M. Alternative splicing of transcription factors in plant responses to low temperature stress: mechanisms and functions[J]. Planta, 2013, 237(6):1415-1424.
[18] Wang Z F, Burge C B. Splicing regulation:from aparts list of regulatory elements to an integrated splicingcode [J]. RNA, 2008, 14(5): 802-813.
[19] Marquez Y, Brown J W S, Simpson C, et al. Comprehensive analysis of alternative splicing in rice and comparative analyses with Arabidopsis [J]. Genome Res., 2012, 22(6): 1184-1195.
[20] Rauch H B, Patrick T L, Klusman K M, et al. Discovery and expression analysis of alternative splicing events conserved among plant SR proteins [J]. Mol.Biol.Evol., 2014, 31(3):605-613.
[21] E Z G, Wang L, Zhou J H. Splicing and alternative splicing in rice and humans [J]. BMB Reports, 2013, 46(9): 439-447.
[22] Reddy A S N, Marquez Y, Kalyna M, et al. Complexity of the alternative splicing landscape in plants [J]. Plant Cell, 2013, 25: 3657-3683.
[23] Chen Q Y, Han Y J, Liu H J, et al. Genome-wide association analyses reveal the importance of alternative splicing in diversifying gene function and regulating phenotypic variation in maize[J]. Plant Cell, 2018, 30(7): 1404-1423.
[24] Lopez A. Alternative splicing of pre-mRNA: developmental consequences and mechanisms of regulation [J]. Annu.Rev.Genet., 1998, 32: 279-305.
[25] Gupta S, Wang B B, Stryker G A, et al. Two novel arginine/serine (SR) proteins in maize are differentially spliced and utilize non-canonical splice sites[J]. Biochimica et Biophysica Acta, 2005, 1728: 105-111.

