扬麦17/宁麦18的F2群体株高QTL定位及分析
胡文静,梁秀梅,高致富,高德荣,程顺和
(1.江苏里下河地区农业科学研究所/农业农村部长江中下游小麦生物学与遗传育种重点实验室,江苏扬州 225007;2.河南农业大学/河南粮食作物协同创新中心,河南郑州 450002)
我国小麦品种中常用的矮秆基因有Rht1、Rht2和Rht8[1-2]。其中Rht1和Rht2来自农林10号,为隐性半致矮基因,是赤霉素反应迟钝型矮秆基因,分别位于小麦染色体4B和4D短臂上,Rht8来自日本赤小麦(Akakomugi),位于小麦2D染色体短臂上,对赤霉素反应敏感[3]。近年来,小麦矮秆基因的遗传基础研究取得了显著进展,Rht1(Rht-B1b)、Rht2(Rht-D1b)、Rht3(Rht-B1c)和Rht10(Rht-D1c)分别被克隆[4-5],其他矮秆基因的研究也取得了一定的进展[6-7],不同学者利用分子标记相继定位出50多个株高相关QTL[8]。不同的矮秆基因对农艺性状具有不尽相同的影响。唐 娜等[9]研究表明,Rht8对小麦品种的有效分蘖以及千粒重没有负面影响,同时还增加了穗粒数,尤其是提高了穗顶部和穗基部的结实率。另有研究发现,赤霉素不敏感型矮秆基因虽然通过增加穗粒数可以增加产量,但却降低了粒重和蛋白质含量[10],一部分矮秆基因在降低株高的同时降低了小麦的水分利用效率[8],另外有一部分矮秆基因会引起小麦生物产量大幅下降[11]。
众所周知,矮秆基因在小麦遗传改良中发挥了重要作用,矮化育种有赖于矮源和矮秆基因的发掘和利用。目前生产中应用的小麦品种遗传背景狭窄,仅有少数矮秆基因能被应用于小麦育种中[12]。因此,深入分析新型矮秆基因的遗传效应,明确其应用价值,创造或筛选新的矮秆种质,对拓宽小麦矮源和促进小麦矮化育种都具有重要的作用。
扬麦17是江苏里下河地区农业科学研究所育成的优质多抗小麦品种,宁麦18是江苏省农业科学院育成的高产抗病小麦品种[13]。本研究以扬麦17和宁麦18创制的310个F2植株为材料构建遗传连锁图谱,并结合F2和F2:3群体的田间表型数据进行小麦株高性状的QTL定位及分析,以期发掘新的株高性状QTL,为矮秆新材料的培育和矮秆新基因的挖掘提供理论和技术 支撑。
1 材料与方法
1.1 材 料
1.1.1 植物材料
供试群体亲本为普通小麦品种扬麦17和宁麦18。扬麦17由江苏里下河地区农业科学研究所育成,株高88~91 cm,茎秆韧性很好,抗倒、抗病、优质、多穗。宁麦18由江苏省农业科学院育成,株高83~86 cm,矮秆、大穗。本研究以宁麦18为父本、扬麦17为母本杂交获得F1代,F1自交得到310株F2植株,F2按单株收,繁殖获得F2:3群体。
1.1.2 引物
参考Roder等[14]、Song等[15](http://www.scabusa.org)、Sourdille等[16]、Guyomarc’h等[17]和Li等[18]提供的序列信息,选取可覆盖小麦大部分基因组的2 403对不同来源的SSR引物。引物由上海捷瑞生物有限公司合成。
1.2 方法
1.2.1 田间试验和表型调查
亲本材料、F2植株及其F2:3家系分别于2015-2016年度、2016-2017年度连续两个生长季节种植于江苏里下河地区农业科学研究所湾头基地试验田。其中,F2群体按单株种植;F2:3家系按株行种植,2次重复;行长1.3 m,行距35 cm,每行点播30粒,栽培管理同大田生产,小麦生长季未见严重病虫害和倒伏。
小麦灌浆后期调查主茎株高。亲本分别调查50株;F2群体调查每个单株;F2:3群体按株行调查,每行随机选取10株,取平均值,并调查2个 重复。
1.2.2 DNA提取和PCR扩增
取亲本和F2群体的苗期叶片,采用CTAB法提取DNA。PCR体系与程序参考Taq酶(北京天根生化科技有限公司)说明书,其中循环数为35,退火温度视不同引物而定,延伸时间为45 s。采用8%的非变性聚丙烯酰胺凝胶电泳分离 PCR扩增产物,银染检测,观察记录结果并拍照保存。
1.2.3 表型数据分析
利用Microsoft Excel 2007软件对表型数据进行整理,利用SPSS 22.0对亲本的株高进行独立样本t测验,并对F2:3群体的株高性状进行描述性统计分析。
1.2.4 连锁图谱的构建和QTL分析
利用JoinMap 4.0软件并采用Kosambi函数构建遗传图谱,使用WinQTLCart 2.5软件选择复合区间作图法进行QTL定位分析[19],若LOD值>2.5,就认为该处存在一个有效的QTL,同时分析其加性效应、显性效应和贡献率。以“Q+性状名称缩写+染色体名称”的方式[20]对QTL进行命名。
2 结果与分析
2.1 表型性状
统计分析显示(表1),两个亲本在株高性状上表现为极显著差异。F2和F2:3群体中的偏度和峰度的绝对值都小于1,即满足正态分布(图1)。同时,株高性状存在明显的双向超亲分离现象,表明均为多基因控制的数量性状,适合进行QTL定位。
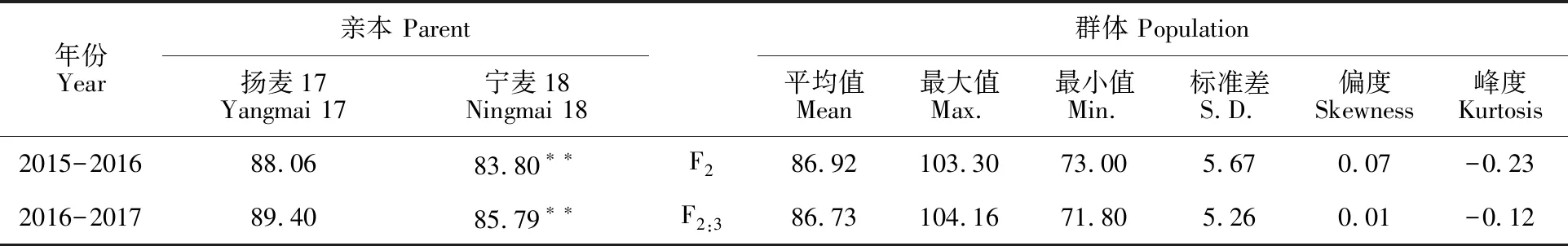
表1 亲本及其F2和F2:3群体的株高表型数据Table 1 Phenotypic values for plant height of two parents,the F2 and F2:3 populations in wheat cm
**:双亲间差异极显著(P<0.01)。
**:Significant difference between parents(P<0.01).

图1 扬麦17/宁麦18 F2:3群体株高的表型分布
2.2 遗传连锁图谱构建结果
参考梁秀梅[21]遗传连锁图谱的构建,利用实验室选取的2 403对SSR引物在扬麦17和宁麦18之间进行多态性筛选,得到330对在亲本间具有多态性的引物,多态性引物检出率为13.7%。采用JoinMap 4.0作图软件构建遗传连锁图谱,将其中的215个SSR标记定位在19条染色体上(1D、6A未覆盖到),该图谱共覆盖1 717 cM,标记间平均距离为7.99 cM[21]。
2.3 QTL定位分析
应用复合区间作图法共定位到8个株高QTL,分别位于小麦的2D、4B、5A、5B和7A上染色体上,其中,QPh-YN-2D.1、QPh-YN-5A.1、QPh-YN-5B.2和QPh-YN-7A四个QTL在两个世代中均检测到,QPh-YN-2D.2、QPh-YN-4B、QPh-YN-5A.2和QPh-YN-5B.1四个QTL仅在一个世代中检测到。
QPh-YN-2D.1位于标记Xcfd53~Xgwm4080之间,染色体上遗传位置是6.8 cM,在两世代中可解释的表型变异分别为2.84%和 6.37%,LOD值分别为2.56和5.75,矮秆效应增效等位基因来自宁麦18。QPh-YN-5A.1位于标记Xwmc415~Xbarc360之间,染色体上遗传位置是44.9 cM,在两世代中可解释的表型变异分别为 14.36%和20.46%,LOD值分别为8.69和12.82,矮秆效应增效等位基因来自扬麦17。QPh-YN-5B.2位于标记Xgpw499~Xmag3262之间,染色体上遗传位置是76.4 cM,在两世代中可解释的表型变异分别为4.26%和5.46%,LOD值均为3.51,显性效应为负,矮秆效应增效等位基因来自宁麦18。QPh-YN-7A位于标记Xmag4044~Xmag3284之间,染色体上遗传位置是16.5cM,在两世代中可解释的表型变异分别为2.15%和3.16%,LOD值分别为2.55和 4.38,矮秆效应增效等位基因来自宁麦18。

图2 小麦株高性状QTL在染色体上的分布
QPh-YN-2D.2仅能在F2世代中检测到,位于标记Xgpw5092~Xgpw4450之间,染色体上遗传位置是100.6 cM,与QPh-YN-2D.1相距93.8 cM,推测是不同的QTL,可解释的表型变异为 5.05%,LOD值为4.59,矮秆效应增效等位基因来自宁麦18。QPh-YN-5A.2也仅能在F2世代中检测到,位于标记Xgwm186~Xbarc303之间,染色体上遗传位置是71.2 cM,与QPh-YN-5A.1相距26.3 cM,推测是相同的QTL,LOD值为3.51,可解释的表型变异为7.72%,矮秆效应增效等位基因来自扬麦17。QPh-YN-5B.1仅能在F2:3世代中检测到,位于标记Xgpw4463~Xgpw5206之间,染色体上遗传位置是65.6 cM,与QPh-YN-5B.2相距10.8 cM,推测是同一QTL,LOD值为2.95,可解释的表型变异为 4.62%,显性效应为负,矮秆效应增效等位基因来自宁麦18。QPh-YN-4B也仅能在F2:3世代中检测到,位于标记Xmag1163~Xgwm538之间,染色体上遗传位置是2 cM,LOD值为4.89,可解释的表型变异为11.24%,显性效应为负,矮秆效应增效等位基因来自宁麦18。
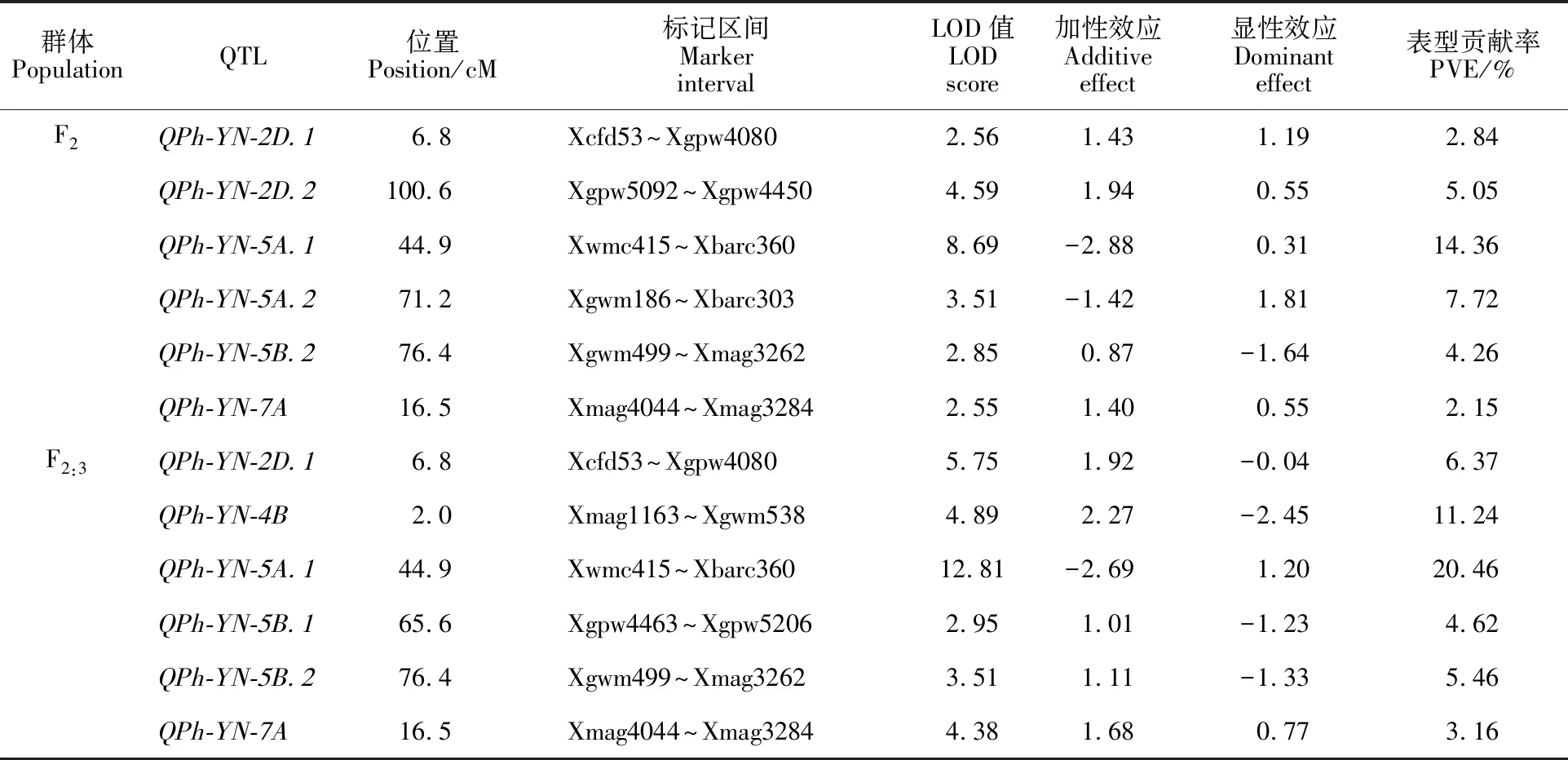
表2 扬麦17/宁麦18的F2和F2:3群体株高的QTL定位结果Table 2 QTL mapping result for plant height in the F2 and F2:3 population of Yangmai 17/Ningmai 18
加性效应:正值表示增效基因来自扬麦17;负值表示增效基因来自宁麦18。显性效应:正值表示杂合子表现值比两纯合子的平均表现值高,负值表示杂合子表现值比两纯合子的平均值表现值低。
Additive effect:Positive additive effect indicates increased effect from Yangmai 17,and negative additive effect indicates increase effect from Ningmai 18.Dominant effect:Positive values indicate that the heterozygotes have the higher phenotypic values than the respective means of the two homozygotes,and negative values indicate that the heterozygotes have the lower phenotypic values than the respective means of the two homozygotes.
3 讨 论
3.1 QTL的比较分析
QPh-YN-5A.1在两世代中可以解释的表型变异为14.36%和20.46%,是一个稳定的主效QTL。已知矮秆基因Rht12位于5AL染色体上,紧密连锁的分子标记是Xgwm291[22],经过比对发现QPh-YY-5A.1与之相距较远[16]。参考Wang等[23]90K芯片图谱和中国春(CS)V1.0版本基因组(http://plants.ensembl.org/Triticum_aestivum/Info/Index)也发现QPh-YN-5A.1与已报道[24]的5A上株高QTL相距较远。因此推测QPh-YY-5A.1是一个新的株高QTL。QPh-YN-5A.2仅能在F2世代中检测到,与QPh-YN-5A.1相距26.3 cM,推测是相同QTL。QPh-YN-2D.1与已报道的2DS上矮秆基因Rht8的特异分子标记Xgwm261[25]遗传距离仅6.8 cM,因此推测此QTL是Rht8的位点。QPh-YN-2D.2仅能在F2世代中检测到,与QPh-YN-2D.1相距93.8 cM,推测是不同的QTL,与中国春比对发现QPh-YN-2D.2位于2DL上,不同于以往报道的株高QTL,推测是一个新的株高QTL。经过与中国春比对发现QPh-YN-5B.2位于5BL上,与叶亚琼等[26]报道的Qph.acs5B相距较远,推测是一个新的株高QTL。QPh-YN-5B.1仅能在F2:3世代中检测到,与QPh-YN-5B.2相距10.8 cM,推测是同一QTL。经过与中国春比对发现QPh-YN-7A位于7AL上,与叶亚琼等[26]报道的QPh.acs-7A.1距离相近,推测是同一个QTL。经过与中国春比对发现QPh-YN-4B位于4BS上。经基因型分析,Rht-B1b的KASP标记在宁麦18和扬麦17中均呈阳性,说明QPh-YN-4B不属于Rht-B1b的位点,该位点未见报道[25,26-27],推测是一个新的株高QTL。
3.2 矮秆基因的显性或者上位性效应
比对结果表明,QPh-YN-4B和QPh-YN-5B分别是两个新的株高QTL,显性效应均为负值,表示后代中杂合子表现值比两纯合子的平均表现值低,说明这两个矮秆基因具有一定的显位效应或者上位性效应。
3.3 QTL的稳定性和表型贡献率
定位到的8个QTL中,只有QPh-YN-2D.1、QPh-YN-5A.1、QPh-YN-5B.2和QPh-YN-7A能够同时在两世代中被检测到,是稳定遗传的QTL;QPh-YN-2D.2、QPh-YN-5A.2、QPh-YN-5B.1、QPh-YN-4B只能在单一世代被检测到,而且大多数株高性状QTL的贡献率较低,只有QPh-YN-5A.1在两世代表型贡献率达到 14.36%和20.46%,QPh-YN-4B在F2:3世代中的表型贡献率达到11.24%,其他株高QTL对表型的贡献率均小于10%,这是因为株高性状属于数量遗传性状,并且供试群体F2和F2:3属于低世代临时性群体,F2世代仅调查单株主茎的表型性状,易受环境影响,表型调查误差大。我们后续会增加重组自交系群体多环境试点以增加结果的可靠性和稳定性,以期寻找到在不同环境下稳定表达的株高性状QTL。
3.4 群体大小和图谱密度对QTL检测结果的影响
以往研究表明,QTL检测结果受图谱密度和图谱长度的影响[28]。本研究所利用的扬麦17/宁麦18群体包括310个F2单株及F2:3家系,基于SSR标记构建的连锁图谱进行QTL检测,最终有215个标记定位到小麦的19条染色体上,图谱覆盖1717 cM,标记间平均图距为7.99 cM,由于SSR标记密度有限,遗传连锁图谱没有完全覆盖到染色体,可能造成部分染色体上QTL的漏检,我们后续会应用覆盖小麦全基因组的55K芯片扫描重组自交系群体构建更加精密的遗传连锁图普以增加QTL定位结果的可靠性。
3.5 QTL位点的紧密连锁
矮秆QTLQPh-YN-5A.1与梁秀梅等[21]在5A上定位到的总小穗数QTL相距11.5~14.5 cM,增效等位基因都来自扬麦17,推测二者是连锁QTL,另一矮秆QTLQPh-YN-2D.1与梁秀梅等[21]定位到的穗长QTLQSl-2D.2相距3~15 cM,增效等位基因都来自宁麦18,推测二者是连锁QTL。利用这些紧密连锁QTL,结合稳定表达的位点,通过回交和分子标记辅助选择,可同时聚合控制多个性状不同位点的等位基因,从而创制出优异的小麦种质资源。
致谢:感谢中国农业科学院作物科学研究所夏先春博士在KASP基因分型方面提供的技术支持。

