枯草芽孢杆菌菌株CGMCC 12426的全基因组测序与分析
董 辉,李轩豪,常静林,贺 欣,侯沁莲,龙 伟
中国医学科学院 北京协和医学院 放射医学研究所天津市放射医学与分子核医学重点实验室,天津 300192
高效聚γ谷氨酸(poly-γ-glutamate,γ-PGA)是一种阴离子、可生物降解的水溶性生物聚合物,可以食用且对人体和环境无害[1]。这种生物聚合物具有多种功能,已广泛应用于食品、化妆品、药品和水处理等工业领域。因此,从工业观点来看,其潜在的应用已被研究[2- 3]。由于D-和/或L-构型的谷氨酸盐通过γ-酰胺键聚合,在每一个谷氨酸单元(γ-L-PGA和γ-D-PGA)中存在手性中心旋光性聚合物,不同的聚合物有不同的用途。那么,为了让这个有前途的生物聚合物用于实际的工业生产中,两个主要问题仍有待解决:如何以更适中的价格大量生产,以及如何控制其结构的多样性[2]。
γ-PGA首先在20世纪初被描述于炭疽菌群(Bacillusanthracis),这种菌株也生产γ-D-PGA。然而,由于其生物毒性,不能直接使用这种微生物。有研究证明,一些细菌(大部分来自芽孢杆菌属)可分泌γ-PGA作为发酵的产物到培养基中[4]。这些细菌中,研究最深入的是枯草芽孢杆菌(B.subtilis)与地衣芽孢杆菌(B.licheniformis)[5- 6]。与地衣芽孢杆菌相比,枯草芽孢杆菌则显示出更高的生产力,因此这种菌株的应用越来越受到人们的重视[7]。枯草芽孢杆菌是一种能够产生内生孢子、需氧的革兰氏阳性菌。γ-PGA由枯草芽孢杆菌(纳豆)所产生,在日本被用作发酵大豆食品纳豆的起动器,具有悠久的历史[8]。最近,从土壤中分离出的枯草芽孢杆菌菌株被命名为枯草芽孢杆菌KH2,已保藏于中国普通微生物培养收集中心(CGMCC № 12426)。研究显示,在含有4%L-谷氨酸的培养基中培养该菌株48 h后,γ-PGA产率达到最大41 g/L。在所用条件下,γ-谷氨酸含量维持在84%~86%,避免了不规则的立体化学问题[2]。本研究描述了枯草芽孢杆菌CGMCC 12426的微生物特征,对其进行了全基因组测序和注释,以期为将来该菌株的代谢和调控机制的深入研究提供实验基础。
材料和方法
分类和特点枯草芽孢杆菌菌株KH2(CGMCC 12426)是革兰氏阳性需氧菌,能够产生内生孢子,为杆状芽孢杆菌,最适生长条件为pH为7.0、30℃(表1、图1)。在LB琼脂培养基上生长时,菌落呈乳白色,表面光滑。能产生过氧化氢酶。菌株KH2能够利用D-葡萄糖、麦芽糖、乳糖和蔗糖作为唯一碳源。基于16S rRNA基因的BLAST结果,菌株KH2与枯草芽孢杆菌物种的相似度最高[9]。菌株KH2和枯草芽孢杆菌DSM 10T之间的16S rRNA基因序列的相似性为100%。在默认设置下,从菌株KH2的基因组序列中得到16S rRNA基因的完整序列,从NCBI数据库中调取芽孢杆菌属的代表性成员序列[10- 15],使用邻接连接法构建系统发育树,分析菌株KH2和芽孢杆菌属其他成员之间的系统进化关系(图2)。
基因组测序选择枯草芽孢杆菌CGMCC 12426用于测序,因为它能生产具有规则立体化学的γ-PGA。基因组序列提交至Genbank公用数据库,存储号为CP018184-CP018185,基因组信息存放在Gp0203613的Genomes on Line Database中[18]。基因组测序和注释由上海中国人类基因组中心进行。该项目的总结如表2所示。
菌株生长条件和基因组DNA制备:将枯草芽孢杆菌CGMCC 12426接种在50 ml LB培养基中,并在转度为200 r/min的摇床中30℃培养12 h。使用QIAamp DNA Mini Kit(Qiagen,CA)提取枯草芽孢杆菌CGMCC 12426的基因组DNA。在Agilent 2100生物分析仪(Agilent,美国)上评估基因组DNA的数量和质量。将基因组DNA储存在-20℃冷冻器中。
基因组测序和组装:使用基因组DNA构建10 kb插入片段SMRT-bell文库,然后在Pacific Biosciences(PacBio)RS Ⅱ测序仪(Pacific Biosciences,CA)的单分子实时(SMRT)DNA测序平台上测序[19]。在1个3 h电影次数的SMRT细胞上共计150 292次聚合酶读数,最终获得总共1 386 478 854个核苷酸碱基。经过滤除去低精度值小于0.8的读数后,以0.866读数质量获得1 256 996 185读数碱基。所有过滤的序列在SMRT分析软件2.3.0版(Pacific Biosciences)中使用RS等级基因组装配过程(HGAP)装配协议2.0进行重新组装[20]。
基因组注释:使用NCBI原核基因组注释管道(prokaryotic genome annotation pipeline,PGAP)进行注释。用KEGG数据库于重建代谢途径[21]。通过RNAmmer和tRNAscan-SE程序预测核糖体RNA和转移RNA[22- 23]。
结 果
菌株CGMCC 12426完整基因组序列由长度为74 165 bp的质粒和长度为4 138 265 bp的环状染色体组成。基因组G + C含量为43.4 mol%。在CGMCC 12426基因组中预测了4581个ORF,30组rRNA(5S rRNA、16S rRNA和23S rRNA)操纵子和87个tRNA(表3)。在总共4581个预测ORFs中,3288个是功能单位,其中1173个被注释为假设蛋白质。当根据COG类别分析生物学作用时,氨基酸转运和代谢蛋白占所有功能分配蛋白质的最大数量(307),其次是转录蛋白(304)和碳水化合物转运和代谢蛋白质(286)。基因在COGs功能分类中的分布见表4。该菌株包含1个质粒,该质粒的重要基因及其功能注解见表5。

表1 根据MIGS建议的枯草芽孢杆菌CGMCC 12426的分类和一般特征[16]Table 1 Classification and general characteristics of Bacillus subtilis CGMCC 12426 suggested by MIGS
证据代码-从直接分析中推断出IDA,TAS可追溯作者陈述(即文献中存在直接报告),NAS不可追溯作者陈述(即,不直接观察活的、孤立的样本,但基于该物种的普遍特性,或轶事证据),这些证据代码来自基因本体项目
Evidence codes-IDA inferred from direct assay;TAS,traceable author statement(i.e.,a direct report exists in the literature);NAS,non-traceable author statement(i.e.,not directly observed for the living,isolated sample but based on a generally accepted property for the species or anecdotal evidence),these evidence codes come from the Gene Ontology project[17]

图1菌株KH2的透射电子显微镜
Fig1Transmission electron microscopy of strain KH2

这些菌株及其16S rDNA序列的GenBank登录号分别为:枯草芽孢杆菌KH2(CP018184)、枯草芽孢杆菌PPL-SC9(KM226924)、淀粉样芽孢杆菌BCRC 11601(NR_116022)、籼型芽孢杆菌SD/3(AJ583158)、偶氮共振菌NBRC 15712(AB363732)、甲烷芽孢杆菌NCIMB 13113(AB112727)、硫杂环杆菌BMP- 1(DQ371431)、硒砷芽孢杆菌SF- 1(AB262082)、柯氏芽孢杆菌DSM 6307(X76437)、蜡样芽孢杆菌ATCC 14579(NR_074540)、砷杆菌cona/3(AJ606700)、结核分枝杆菌MLTeJB(FJ825145)、砷黄杆菌E1H(AF064705)和麻黄芽孢杆菌JMM- 4(AY32601)
The strains and their corresponding GenBank accession numbers for 16S rDNA sequences areBacillussubtilisstrain KH2(CP018184),BacillussubtilisPPL-SC9(KM226924),BacillusamyloliquefaciensBCRC 11601(NR_116022),BacillusindicusSd/3(AJ583158),BacillusazotoformansNBRC 15712(AB363732),BacillusmethanolicusNCIMB 13113(AB112727),BacillusthioparansBMP- 1(DQ371431),BacillusselenatarsenatisSF- 1(AB262082),BacilluscohniiDSM 6307(X76437),BacilluscereusATCC 14579(NR_074540),Bacillusarsenicuscon a/3(AJ606700),BacillusbeveridgeiMLTeJB(FJ825145),BacillusarseniciselenatisE1H(AF064705),andBacillusmacyaeJMM- 4(AY032601)
图2基于16S rRNA基因序列的MEGA 5构建的邻接系统发生树
Fig2Adjacent phylogenetic trees constructed based on the 16S rRNA gene sequence(by MEGA 5 phylogeny software)
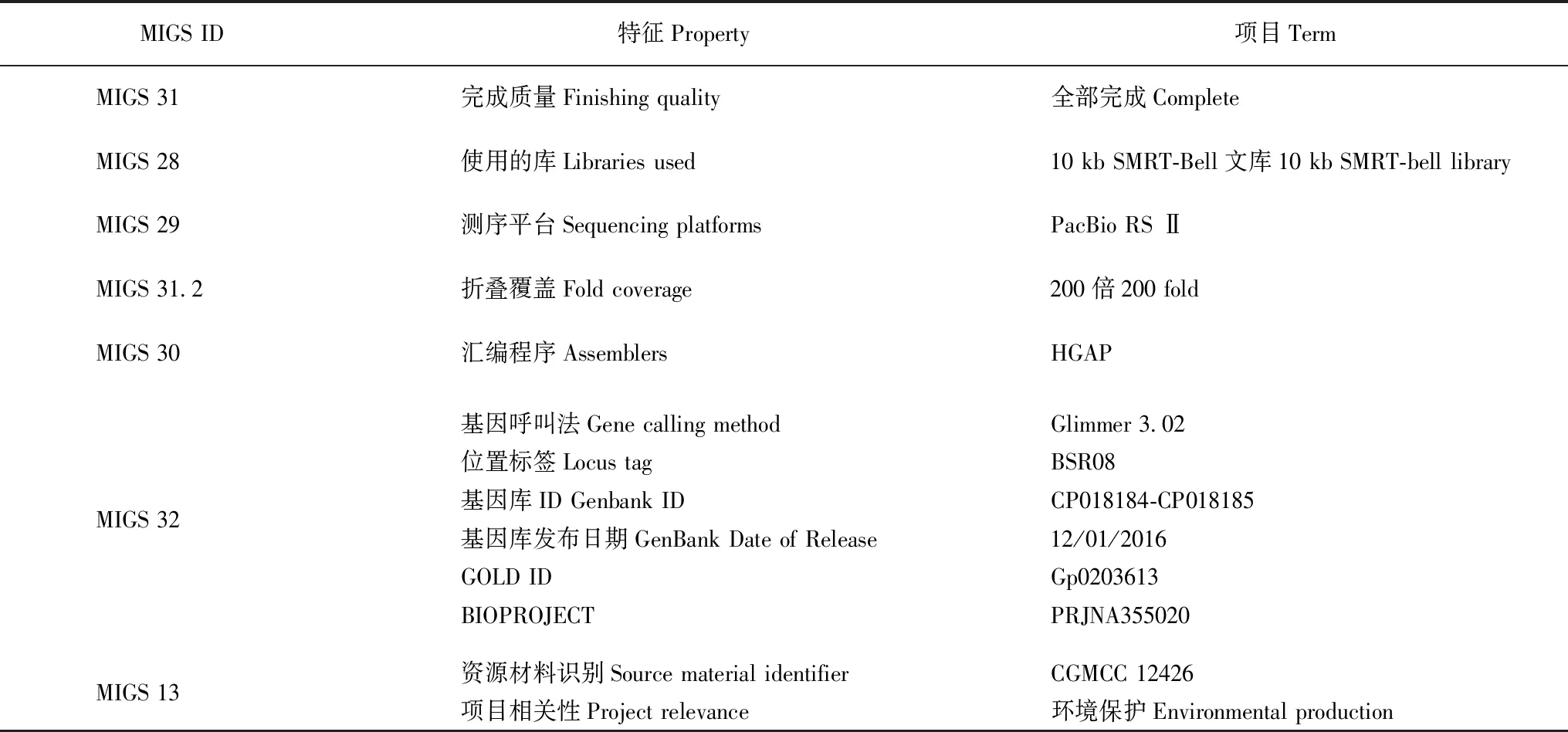
表2 基因组测序项目信息Table 2 Genome sequencing project information

表3 枯草芽孢杆菌菌株CGMCC 12426的基因组统计Table 3 Genome statistics of Bacillus subtilis strain CGMCC 12426
NA:无法使用
NA:not available

表4 与一般COG功能类别相关的基因数量Table 4 The number of genes associated with general COG function categories
总数是根据注释基因组中蛋白质编码基因的总数计算
The total is based on the total number of protein coding genes in the annotated genome

表5 枯草芽孢杆菌菌株CGMCC 12426质粒的重要相关基因及其功能Table 5 Important genes and functions of Bacillus subtilis strain CGMCC 12426
讨 论
枯草芽孢杆菌CGMCC 12426的基因组测序进一步阐明了该菌株的遗传背景和基础,为研究乙醇、有机酸、氨基酸等形成的代谢和调控机制提供了重要理论依据。与枯草芽孢杆菌亚种枯草芽孢杆菌菌株168的扩展研究模型菌株相比,枯草芽孢杆菌菌株KH2的遗传操作效率低得多。基因比对分析发现,KH2中存在1个限制性内切酶,WP_014480489.1 type I restriction-modification system deoxyribonuclease,该酶的存在很可能是KH2这个菌株遗传操作效率比枯草芽孢杆菌菌株168低很多的原因所在。对这两种菌株的基因组特征的比较分析结果发现,两种菌株间只有较小的差异,相似度较高(表6)。该结果有助于鉴定合适的靶基因,可通过系统代谢工程帮助开发具有更高浓度、产量且生产率优良的微生物细胞工厂。
总之,枯草芽孢杆菌CGMCC 12426基因组测序的完成,为其菌株代谢工程改造及调控机制研究提供了大量的生物学信息。该基因组测序为进一步阐明该菌株的遗传背景提供了基础,也为研究代谢和调控机制提供了重要理论依据。使得以适中的价格大量生产γ-PGA,以及控制其结构的多样性成为了可能,随着枯草芽孢杆菌功能基因组学研究的不断深入,将会获得更多优良性状的基因工程菌株,推进生产γ-PGA的产业化进程。

表6 菌株KH2和菌株168之间的基因组特征的比较Table 6 Comparison of genomic features between Strain KH2 and Strain 168

