花生GSTs家族基因的全基因组分析
王 通迟晓元王 冕潘丽娟陈明娜陈 娜 焦 坤
(山东省花生研究所/农业部花生生物学与遗传育种重点实验室,山东 青岛 266100)
谷胱甘肽-S-转移酶(Glutathione-S-transferases, GSTs),是一类重要的多功能超家族酶,普遍存在于植物、动物、真菌和细菌中[1]。植物GSTs最早发现于玉米的除草剂毒性解除反应中[2],之后许多具有抗除草剂功能的GSTs以及GST类似的序列从植物中得到克隆[3]。GSTs作为多基因家族,目前已在17种植物进行研究和报道,其中分别在拟南芥、大豆、玉米、水稻、白杨、番茄和苜蓿中鉴定出25、42、59、81、90、42和73个GST基因[4]。
近年来,大量研究表明,植物GST基因家族可通过亲电取代反应、解毒作用和过氧化物清除过程来调控植物对各类逆境胁迫的适应能力[7-10]。其表达不仅受病原菌侵染[11]、盐害[12]、干旱[13]、冻害[14]、伤害[15]、除草剂[13]和重金属[16]等生物和非生物胁迫诱导,还受到水杨酸(SA)[17]、乙烯(ET)[18]、脱落酸[19]和茉莉酸甲酯(MeJA)[20]等植物激素诱导。在酰胺类除草剂和低温处理下,拟南芥AtGSTU26[21]和水稻OsGSTU5[22]均显著上调表达,而水稻OsGSTU4[23]和大豆GmGSTL1[24]的过表达可提高拟南芥的耐盐性和抗氧化能力。在56个高粱GST家族基因中,有35个受冻害、PEG和高盐诱导而显著表达[26]。
目前,有关花生(ArachishypogaeaL.)GST家族基因的研究还鲜有报道。本研究利用生物信息分析技术对花生基因组数据库中GST家族保守区进行检索,分析花生GST家族基因的数量、结构、系统进化、染色体定位和时空表达特性等信息,为后续的花生GSTs基因在花生中的功能性分析与基因编辑、基因工程等提供理论依据。
1 材料与方法
1.1 实验材料
花生基因组数据库Peanut Base(https://www.peanutbase.org/home),Pfam数据库(http://pfam.sanger.ac.uk/)。拟南芥 GSTs家族的基因序列和蛋白序列[17]。
1.2 花生GST基因家族成员筛选
使用BioEdit软件对获得花生全基因组氨基酸序列建立数据库,利用Pfam数据库中登陆号PF0043、PF02798、PF13409、PF13410、PF13417、PF14497、PF14834、PF17171和PF17172下 载GST家族基因的HMM(Hindden Markov Model,隐马尔科夫模型)文件,用这些GST结构域的氨基酸序列与已建立的花生全基因组氨基酸数据库进行Blastp(E-value=0.001)序列比对,筛选出同源核苷酸序列的候选基因,然后利用Pfam(E-value=1.0)进行分析,去除无GST保守结构域的基因序列。
1.3 花生GST基因的结构与染色体定位分析
花生GST家族基因的基本结构信息均从基因组数据库中提取。等电点与分子质量均使用ExPASy(https://web.expasy.org/protparam/)进行估测分析。利用在线分析工具MEME(http://meme-suite.org/tools/meme)对花生GST蛋白的蛋白质保守基序进行分析。利用GSDS(http://gsds.cbi.pku.edu.cn/)在线工具绘制GST家族基因外显子-内含子结构示意图。根据花生GST家族基因的基因组位置信息,利用R语言chromPlot,绘制花生GST家族基因的染色体分布信息图。
1.4 花生GSTs基因的系统进化树构建
利用MEGA5.0[27]软件进行花生GST基因家族系统发育树的构建,进化树构建方法为邻接法(Neighbor-joining method),分析参数为默认值。
1.5 花生GSTs基因的数字表达分析
从Peanut Base数据库中收集花生GST基序基因匹配的RNA-seq数据(包括22个组织器官)[28],分析GST基序基因在花生不同组织器官的表达情况,并利用pheatmap(R)[29],进行GST的基因表达分析,绘制热图。
2 结果与分析
2.1 花生GSTs家族基因的鉴定
运用 BLASTp 方法对野生种花生(A和B)全基因组注释的蛋白质数据库捜索花生GST序列,后经Pfam分析,结果得到 163 条具有典型GST结构的序列,其中,A基因组中含有76个,B基因组中含有87个。通过R语言可以提取GSTs家族基因的基因编号、在染色体上的位置、基因长度、蛋白质长度、等电点和相对分子质量,结果见表 1。
2.2 花生GSTs基因染色体定位
为了明确花生GST各基因在染色体上的分布,利用花生基因组数据库中的基因位置信息,通过chromPlot制作了161个花生GST基因在染色体上的分布图(图1),H8WP2和F3P8N未能定位到具体的染色体上,分别位于尚未定位的片段重叠群:Adur536和Aipa322。由图2可知,这些基因整体上不均匀地分布于20条染色体上,并且大多分布在染色体的两端。在B02号染色体上分布的基因数目最多有15个,在B03染色体上有14个,在A03、A09和B09各有13个,在A02、A07和B08上各有12个,B05有9个,B01有7个,A05有6个,A04、B04和B07各有5个,A08和A10各有4个,A01、A06、B06和B10分布最少,仅有3个。从染色体上定位情况来看,发现A02、A03、A07、A09、B02、B03、B05、B08和B09号染色体上存在1~2个基因串联复制现象,且串联复制多分布于染色体末端。

表1 野生种花of Arachis duranensis和Ar achis ipaensis的谷甘肽转移酶and 基信息Table 1 Information生glutathione transferases gen e family in Arachis 胱duranensis因Arachis ipaensis基因IDa 编号染体定位色Chr_locb列长度序Gene(aa)c子量分MW/Dad等点电IPe基因IDa 编号染体定位色Chr_locb列长度序Gene(aa)c子量分MW/Dad等点电IPe 1375G A01:91084978-91087490(-)666(334)37.575.25 NQ0MH A06:18108412-18109777(-)705(219)24.716.96 45WUM A01:7617729-7618968(+)465(223)25.996.61 BIU4F A06:21555581-21558066(+)333(219)25.266.92 EK5R9 A01:100193110-100198460(+)978(451)51.308.96 ITV20 A06:9065047-9065310(-)1032(88)9.88 5.73 V7D4Y A02:4742342-4743040(-)669(77)8.69 4.83 ANP5R A07:78058123-78060134(+)342(220)25.446.21 80TQM A02:77441904-77443754(+)648(229)26.515.82 65TC6 A07:78015278-78016792(+)711(220)25.306.91 KZZ3J A02:90181582-90183705(+)624(215)24.315.99 A6W0E A07:78206667-78208242(-)342(221)26.145.76 I2JEZ A02:90176996-90178729(+)264(215)24.355.77 HD4RJ A07:78190526-78192783(-)645(209)24.275.35 0M35T A02:92732875-92737641(-)657(208)22.888.57 E6KMV A07:78145115-78146637(+)645(227)26.425.38 TZ9RJ A02:77355051-77358399(+)648(226)25.937.02 Q2835 A07:78126717-78128488(+)690(222)25.875.22 ZPP4Z A02:77316299-77318404(-)681(225)25.625.35 V3MB3 A07:2681328-2681501(+)429(58)6.26 4.81 73PA3 A02:56088798-56092442(-)687(238)26.975.86 4QM78 A07:78281074-78282743(-)1302(220)25.956.15 8CL43 A02:90193444-90194750(+)246(217)24.507.07 LNV6X A07:8306212-8306553(+)651(114)13.156.90 Q097W A02:8132354-8134688(-)660(422)47.876.38 KRA3S A07:78155197-78158843(-)276(451)52.625.29 KHJ4B A02:87241417-87245156(-)717(216)24.636.21 1H68Z A07:78359555-78361032(-)1002(208)24.726.24 86SPU A02:18217658-18219510(+)669(434)48.489.01 9Z02R A07:4234650-4236143(-)660(216)24.725.39 Z9RFX A03:122739312-122740854(+)1428(216)24.356.06 379GW A08:45881308-45884374(+)648(422)48.125.88 J9XDX A03:103236174-103243890(+)1266(267)30.168.88 K7JNV A08:13756514-13776403(-)618(206)23.175.20 6PF06 A03:27456092-27457963(-)624(222)25.655.59 DB78U A08:10223075-10225595(+)618(268)31.389.37 3602N A03:49329879-49330756(-)663(206)23.555.58 X7GDX A08:2609552-2612062(+)666(344)38.688.89 RFH8Y A03:125335017-125335626(+)471(114)13.046.57 IS6EU A09:102785926-102787475(-)3567(223)25.996.12 P7Y6N A03:27465928-27469023(-)714(220)25.455.31 DAK4F A09:116348316-116350795(+)630(155)17.119.48 R6YAY A03:122810128-122811993(-)801(186)21.416.21 PI6VR A09:116032519-116033614(-)231(239)26.175.33 V4NFM A03:134501937-134516765(+)627(1189)138.17 6.09 51L6N A09:115626185-115627288(+)693(221)25.646.18 444VJ A03:102963465-102965955(+)618(252)28.439.41 WGD8V A09:102671964-102674486(-)1266(225)26.155.37 2C43K A03:125336883-125341375(+)1353(476)53.726.63 1232H A09:102844845-102846748(+)279(230)27.035.82 144BI A03:85906259-85908795(+)1353(143)16.016.02 D7LWC A09:102742028-102744303(-)666(206)23.985.89 C4BD6 A03:122051828-122054933(+)756(220)24.575.99 T56CX A09:102714518-102715968(-)687(223)25.806.32 8J8HS A03:111376334-111377169(-)675(93)10.435.08 TMM2A A09:102749448-102750986(-)678(223)25.976.61 ZZJ9P A04:75280871-75282662(+)660(210)24.155.28 CNF9F A09:102726863-102728357(-)342(222)25.926.11 Z1ELU A04:39314405-39317377(-)804(326)37.195.87 EL1FN A09:49291964-49295024(-)669(195)22.089.69 2TG90 A04:75151503-75152379(+)657(235)25.207.69 7Q2JG A09:5703429-5705440(-)984(111)12.635.10 16Z19 A04:27975627-27979343(-)558(331)37.375.37 S427W A09:9660057-9661661(+)675(216)24.885.58 HA5JM A04:1525418-1533294(-)174(231)25.927.58 WS35U A10:105573425-105573766(+)717(114)13.187.84 ZR4EL A05:34840079-34843453(+)660(237)26.695.57 BK9MN A10:48615500-48615994(+)585(92)10.7610.07 UCD4A A05:34787942-34791007(+)660(239)26.785.08 G9I7U A10:22334853-22335890(-)648(82)9.24 4.83 48DYG A05:94985617-94988890(+)663(229)25.955.49 D1CQS A10:7011683-7013591(-)648(216)24.905.51 DGQ15 A05:34816213-34821474(+)717(239)27.215.27 H8WP2 Adur536:386512-390909(+)678(226)25.585.21 X3TFJ A05:109291604-109295895(+)669(328)36.728.72 F3P8N Aipa322:254186-258240(-)672(122)13.845.58 J14KY A05:67463759-67466088(+)993(157)17.954.56 J0LMG B01:136454199-136456702(+)648(356)39.806.46 C8XNH B01:124474514-124477213(-)984(218)24.657.10 CA6VL B04:2135387-2140234(-)1266(231)25.938.24 XX37D B01:127153433-127153675(-)324(81)9.14 5.73 Y8EUA B05:31736029-31740057(+)546(289)32.605.14

基因IDa 编号染体定位色Chr_locb列长度序Gene(aa)c子量分MW/Dad等点电IPe基因IDa 编号染体定位色Chr_locb列长度序Gene(aa)c子量分MW/Dad等点电IPe Q3KHN B01:124469366-124472255(-)675(224)25.288.93 567W8 B05:31692194-31695453(+)639(239)26.864.99 88ZTU B01:2508292-2509835(-)663(211)24.535.24 62Y2G B05:31712264-31717302(+)672(238)27.095.26 P1LVK B01:35394460-35395349(-)738(204)23.265.97 90W61 B05:32320205-32322000(-)714(221)24.845.54 872NE B01:127161378-127163034(+)669(189)21.377.69 F8RVF B05:132600328-132604274(-)612(229)25.985.61 G700X B02:10546766-10549761(-)894(448)50.996.17 18DX7 B05:28480973-28481863(-)723(76)8.51 5.34 N829R B02:88588945-88591057(+)1347(229)26.435.53 T38YK B05:131962319-131966979(-)675(322)35.968.58 91599 B02:103940167-103942227(+)153(215)24.415.77 20IHP B05:149766624-149770808(+)465(337)37.889.39 L2SQL B02:88565607-88568667(+)654(226)25.927.02 ITF7X B05:13263940-13265611(+)678(271)29.588.93 5HK6K B02:88491209-88492668(-)1212(225)25.655.63 5K82A B06:30102983-30105057(+)804(210)23.845.17 0BL53 B02:36695270-36699385(+)687(238)26.985.28 PK7TM B06:2166201-2171679(+)672(224)25.389.28 J9Q6I B02:36575241-36579730(-)672(239)27.296.00 V9X08 B06:191743-192985(+)687(246)28.059.51 1H19F B02:103947748-103949379(+)660(137)15.135.36 2ES8K B07:122069407-122070286(-)690(218)24.736.00 3NB5C B02:103950859-103952153(+)813(217)24.546.66 7XR4A B07:122033886-122035342(-)630(175)19.986.60 KVQ8U B02:100391639-100395799(-)411(213)24.326.09 IBD1I B07:117706656-117709294(+)966(268)31.369.28 2941I B02:100426610-100428894(-)243(155)17.606.05 QYY53 B07:101832723-101835242(-)1344(328)36.758.88 Q7F6I B02:60847701-60849742(-)630(200)22.168.67 56C70 B07:4111995-4113995(-)954(216)24.745.26 M9I94 B02:21016007-21018179(+)816(449)50.129.06 E9VCF B08:60563749-60566069(+)867(231)26.537.75 NB9CE B02:107416543-107430749(-)690(137)15.075.37 CU8L0 B08:60725824-60727438(+)678(238)27.715.93 FAS74 B02:95006994-95014019(-)675(515)57.097.24 K8T47 B08:105547757-105550237(+)663(377)41.019.13 56NJW B03:123418295-123419836(+)669(216)24.316.06 916M9 B08:60363463-60365020(+)654(220)25.346.15 BHX6F B03:30190474-30191700(-)678(222)25.716.00 TP4A9 B08:61117006-61118620(-)1146(210)24.226.84 D6VSK B03:126165314-126165948(+)654(119)13.416.56 65DGK B08:61074156-61075408(+)648(241)28.195.38 AIV0U B03:110417255-110422126(+)1068(228)25.706.76 4F9Q5 B08:60646212-60647475(+)666(218)25.355.20 P3ATM B03:50352629-50353728(-)693(224)25.666.02 C9SN6 B08:126020683-126023752(+)636(422)48.085.80 Y4CF6 B03:30205526-30208043(-)714(220)25.415.44 PI28L B08:61518238-61519937(-)1545(220)25.875.91 XA49Z B03:135582397-135583130(+)1011(223)26.166.46 PXN4X B08:61080639-61081844(-)525(212)25.055.11 78E4M B03:104953168-104959290(+)639(253)28.447.78 5F7F4 B08:61076627-61078400(-)366(229)26.685.26 Y9MKL B03:135586095-135586943(+)411(230)26.695.91 YG8BB B08:73957266-73958476(+)669(156)17.026.50 G6U08 B03:135585343-135585495(+)651(51)5.76 5.18 PY182 B09:13374341-13376121(+)660(226)26.105.92 B6IF6 B03:135561888-135580850(+)1131(561)65.408.40 HIH8J B09:123979174-123980773(-)567(223)25.996.83 6T20K B03:123499185-123503714(-)312(404)43.535.96 YL5F7 B09:140532768-140535251(-)693(298)33.505.99 NG8TV B03:28619130-28620415(-)663(108)12.769.55 VJ5LB B09:140948321-140951014(+)228(272)30.206.41 KV2BK B03:126167528-126171749(+)600(502)56.756.19 U0SXH B09:141566287-141567402(-)468(221)25.546.00 B5CA6 B04:81084399-81086527(+)717(225)25.935.18 3BP83 B09:123695778-123696912(-)1683(225)26.005.60 H2A5J B04:80761167-80762412(+)759(224)25.706.02 R2FTY B09:124102193-124104048(+)357(226)26.635.65 A0KXQ B04:39343213-39346019(-)678(346)39.516.41 AH1BZM B09:123915410-123917098(-)1038(223)25.896.61 UJ65V B04:25620176-25624055(+)714(382)43.296.28 25PW B09:124077732-124079102(-)687(230)26.915.28 E4FSH B09:123982801-123983593(-)717(221)25.835.02 P02AI B10:122029763-122030249(-)684(104)11.826.83 XN7XG B09:89497871-89499631(-)648(182)20.969.57 ILA3H B10:74013070-74016398(-)633(213)24.268.99 64J14 B09:76021712-76025924(+)645(318)35.198.78 J18II B10:11604667-11606384(-)1506(216)24.855.63 RW4VT B09:49484942-49486442(-)660(226)24.846.10 Note:a:Thegeneaicd ID information in online database;b:Chromosomal localization,and the+and -symbols mean the strand information;c:The values outsite and insite brackets arethelength ofgenomeandaminal sequences;d:Molecular weight;e:Isoelectric point.
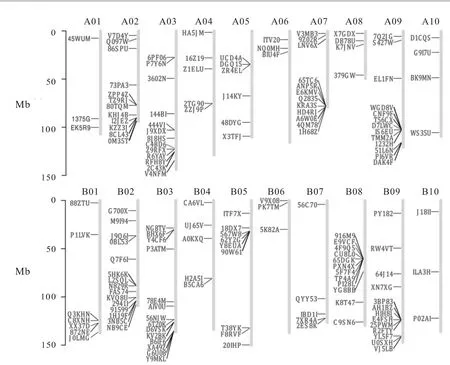
图1 野生种花生A、B基因组GSTs基因的染色体定位Fig.1 Distribution of GSTs genes in A and B chromosomes of wild peanut species
2.3 花生GSTs蛋白的进化树构建
为了解花生GSTs家族蛋白之间的进化关系,使用MEGA7.0 软件构建163个花生GSTs家族成员的蛋白序列的系统进化树(图2)。分析进化树的分枝和保守序列,将花生GSTs家族基因分为6大类:Tau、Theta、Lamda、EF1Bγ、Phi和DHAR。其中Tau类基因数量最多,为91个;其次是Phi,为40个;其余均为8个。对外显子数量分布的统计发现,6大类GST基因间存在显著差异,其中Lamda最多,平均9.4个;其次EF1Bγ平均5.5个;Tau类最少,仅4.0个。结合基因的染色体分布情况,串联重复的基因主要属于Tau和Phi,其4大类中无串联重复基因。
2.4 花生GSTs的基因结构分析
从花生基因组数据库获得163个GST的基因组和编码区序列,使用在线GSDS(http://gsds.cbi.pku.edu.cn/)绘制各GST基因的内含子-外显子结构图,结果显示(图2),该家族基因间的内含子数目及长度存在较大差异,其中EK5R9的外显子数最多,达19个;而G6U08、LNV6X、XX37D、WS35U、ITV20和V3MB3等序列无内含子结构。在K7JNV中的内含子最长,约18 kb,其次是NB9CE和B6IF6,约8 kb。结合进化关系发现,基因结构差异最显著的花生GST家族序列大多分布在Tau类中。
利用在线MEME(http://meme-suite.org/tools/meme)软件对花生GST家族氨基酸的保守结构进行分析(图3),结果显示,花生GST家族中共鉴定出9个保守域,各保守域的氨基酸长度在11~29之间;花生各GST序列中的保守域数量差异较大,其中含有保守域motif 1的基因最多,达138个,其次是含有保守域motif 2的基因,为135个,含有motif 8的基因最少,仅47个。花生GST家族基因间保守结构域的差异可能与其遗传进化有关[4]。

图2 花生GST家族基因的结构与系统进化分析Fig.2 The phylogenetic tree and gene structures of peanut GST gene family
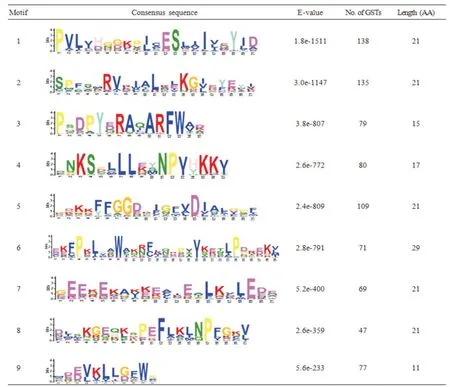
图3 花生GST家族蛋白保守结构域的氨基酸保守性分析Fig.3 The conserved motif of peanut GSTs by logo statistic
2.5 花生GSTs基因表达分析
为研究GST家族基因的各成员在花生中的具体表达模式,对花生22个转录组组织测序结果的pfkm数据[28]分析,去掉各组织中无表达和无差异表达的基因,筛选出80个有表达差异的花生GST基因进行组织表达分析(图3),结果表明,有33个呈现出显著性差异表达(图4中红和绿),其中有6个GST基因的组织表达有极显著差异(图4中标红)。D1CQS在果壳和根瘤结中表达最高,J18II在果壳、根瘤结和花中表达最高,A6W0E仅在花中有较高表达,3602N在果壳中有较高表达,2TG90在果壳和根中表达较高,H2A5J在根中显著高表达。另外,DQCQS和J18II属于Phi亚类,A6W0E,3602N,2TG90和H2A5J属于Tau亚类。说明花生GST家族基因的表达具有一定的组织特异性,其组织表达功能可能与亚家族分类有关。
3 讨 论
在植物GST 家族基因的功能研究中,研究最早、最深入的是其解除除草剂毒害的作用。近年来,随着科研人员对植物GST关注的提高,有关其抗逆和发育调节方面的研究也有大量报道[15,26]。花生中GST 家族的相关研究尚无报道。本研究,对花生A和B野生种基因组进行检索,分别获得76个和87个GST基因序列,总数量多于拟南芥(25个)、大豆(42个)、玉米(59个)、水稻(81个)和苜蓿(73个)[4],这反映出花生异源四倍体的特性。与其他植物相似,花生GSTs家族的蛋白质大小、等电点(pI)等方面存在明显差异,这种差异可能与其基因功能多样有关[4,26,30]。
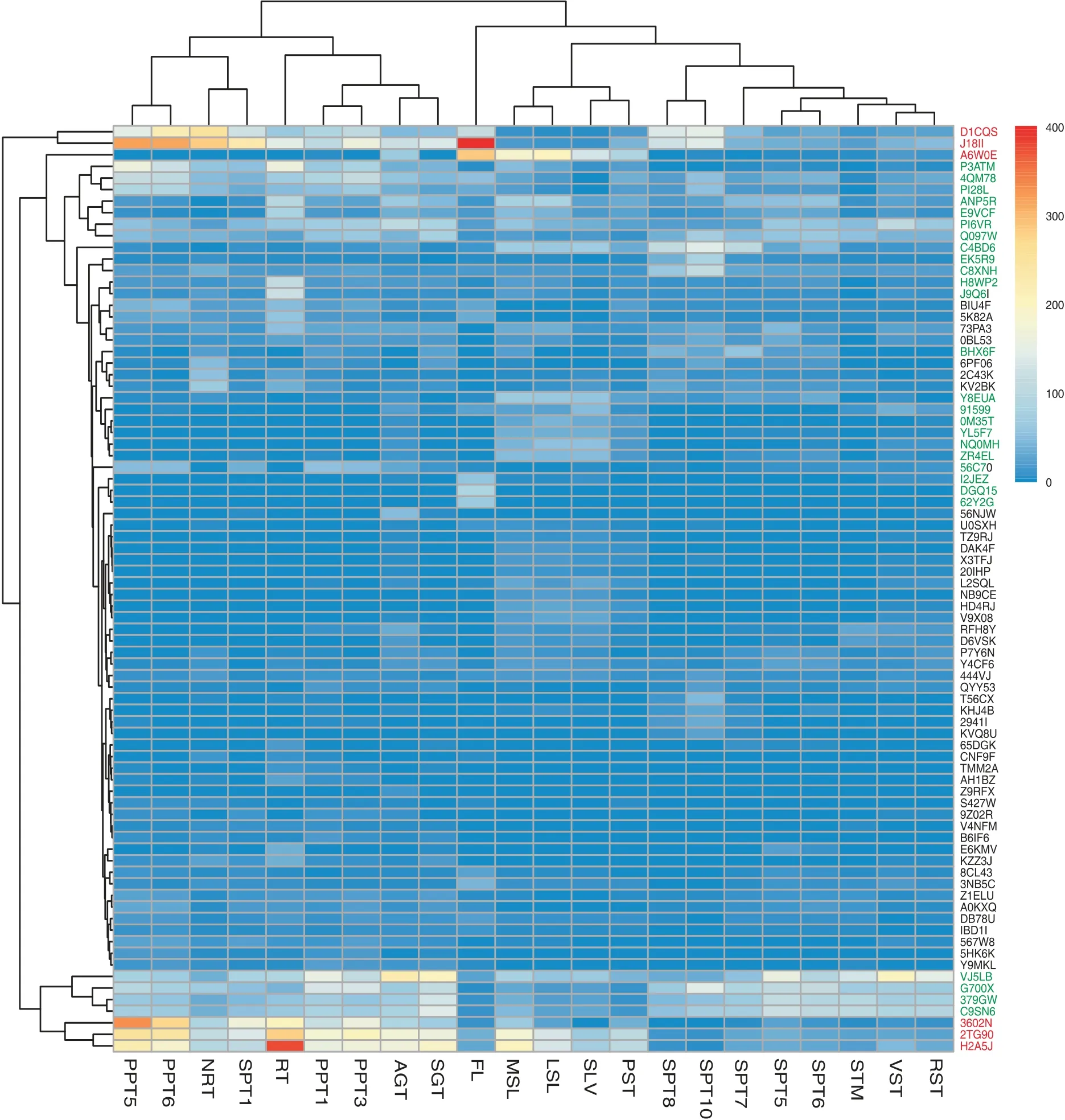
图4 花生GSTs家族基因的组织表达模式Fig.4 Tissues expression profiles of peanut GSTs family
植物基因家族的扩张主要是通过不同的基因复制方式(包括片段复制、串联复制、基因转移和基因组复制等)来实现的[26]。植物中GST家族基因的扩张主要是依靠Tau和Phi亚类的基因串联复制完成的,使得植物特有的Tau和Phi成为成员最多的两个亚类[4,30-31]。基因系统进化分析表明,花生GST家族中Tau和Phi类的成员最多,分别占花生总GST数的55.8%和24.5%,并在A2、A3、A7、A9、B2、B3、B5、B8和B9染色体末端存在GST基因簇位点。推测花生GST家族成员的增加与Tau和Phi类基因的串联复制有关。同一基因簇上的基因具有较近的进化关系和基因结构,这些基因簇上的基因是否为同一基因的串联复制产生,以及它们在花生体内的功能和作用,值得做进一步研究。
植物GST家族各基因的内含子数目及长度存在较大差异,基因的内含子-外显子结构严重异化对其功能分化具有重要意义。花生GST家族各基因的结构差异较大,最多的有18个内含子,最长约18 kb,与已报道其他植物GST家族基因的结构相似[4,31]。
本研究还运用花生基因组数据库中RNAseq数据分析了GST家族基因的组织表达模式,仅有少部分基因的表达量在特定组织中显著上升,其中D1CQS、3602N、2TG90、H2A5J和A6W0E的表达量最高,与南瓜中Phi类GST-Pugf基因相似[32],在花、果壳和根等组织中的表达特异性最强,在幼嫩器官中比成熟器官中丰富,表明这几个基因在花生生殖和发育调节中起到重要作用。本文对花生22个组织中GST基因仅做组织表达分析,并未开展其他生物、非生物和各种激素等处理下的表达分析试验,花生GST家族基因的表达模式还有待更多试验数据支持。

