8个秀珍菇菌株遗传亲缘关系初步分析
王伟科,陆娜
(杭州市农业科学研究院 蔬菜研究所,杭州 310024)
秀珍菇(Pleurotuspulmonarius)隶属于真菌门、担子菌纲、伞菌目、侧耳科侧耳属,菇形秀小、漂亮,口感柔嫩,美味清爽,营养丰富,是近年来浙江省栽培面积最广的珍稀食用菌之一,备受广大消费者喜爱[1]。食用菌种质资源的收集分类鉴定是选育优良品种的前提和基础。但秀珍菇菌株繁多,来源又各不相同,造成异种同名或同种异名现象的普遍出现,而传统的形态学方法很难将秀珍菇菌株区别开来。因此,引入新的秀珍菇品种鉴定分析方法必不可少[2]。
近年来,分子标记技术已广泛应用于药用植物及食用大型真菌的分析。主要手段有ISSR、AFLP、RFLP、RAPD等[3]。ISSR分子标记技术具有操作简单、模板需要量少、多态性丰富、结果记录方便、成本低廉等优点,在物种遗传多样性鉴定、系统进化等多个领域得到普遍运用[4]。我们采用ISSR技术对8个秀珍菇菌株的遗传多样性进行分析鉴定,以期为秀珍菇品种选育中亲本选择提供参考。
1 材料与方法
1.1 供试菌株
菌株S1,杭州市农业科学研究院;菌株S2,浙江省农业科学院;菌株S3,杭州市农业科学研究院;菌株S4,杭州市农业科学研究院;菌株S5,临安鼎新生物科技有限公司;菌株S6,浙江兴森科技有限公司;菌株S7,武义海兴生物科技有限公司;菌株S8,桐乡西岸菌业。
1.2 培养基和培养方法
培养基。采用PDA培养基(葡萄糖20 g,马铃薯去皮200 g,水1 000 mL,pH值自然)。
培养方法。将参试菌株转接于PDA固体培养基中,每个菌株转接3个平皿,于(26±1)℃条件下培养10 d后用于DNA提取。
1.3 基因组DNA的提取
挑取菌丝提取DNA。DNA提取采用CTAB法[5]。提取的DNA经Nanodrop 2000 c定量和琼脂糖凝胶电泳检测纯度后,保存于-20 ℃备用。
1.4 ISSR引物设计及合成
ISSR引物参考哥伦比亚大学公布的序列和Wolfe相关设计[6],从中挑选出16条合适引物,引物由北京擎科生物技术有限公司合成,引物具体序列见表1。
1.5 ISSR分子标记鉴定
ISSR扩增体系根据各自引物退火温度和Mg2+浓度进行优化,优化后体系含10×PCR buffer 2 μL、25 mmol·L-1MgCl21.2 μL、10 mmol·L-1dNTPs 0.5 μL、10 mmol·L-1ISSR引物1 μL、100 ng基因组模板DNA和1 U pfu DNA聚合酶,总体积20 μL。
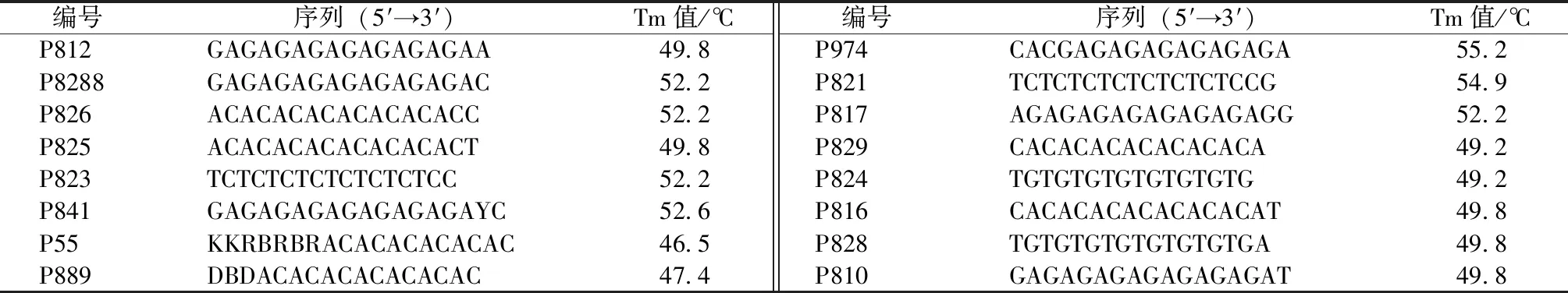
表1 参试菌株ISSR引物参考序列
PCR扩增程序为:94 ℃预变性3 min;94 ℃变性20 s、46~52 ℃退火90 s、72 ℃延伸30 s、35个循环;72 ℃延伸7 min。其中,每个引物的退火温度优先参考值为Tm值下调2 ℃。
1.6 扩增产物的检测
取目标产物10 μL,加入6×上样缓冲液1.5 μL,混匀后于1.5%琼脂糖凝胶上电泳分离,拍照记录。
1.7 数据采集分析
从凝胶成像系统得到的电泳谱带中,选取清晰可辨的电泳条带,以1和0记录条带的有或者无,在相同片段位置上存在扩增带时,记录为1,不存在时记录为0,将图形数据转换成数字数据。并运用NTSYS-pc2.0软件,根据SM相似系数法计算品种间的遗传相似性,UPGMA进行聚类分析。
2 结果与分析
2.1 ISSR多态性
用来扩增秀珍菇的16条ISSR中,有8条获得的ISSR带型较好,各引物扩增获得的条带片段大小范围较大,在500~5 000 bp;共扩增获得207个DNA条带,其中多态性条带119条。引物P828、P974扩增的多态性比率最高,为100%;引物P826扩增的多态性比率最低,为15.8%(表2)。

表2 8条ISSR引物扩增结果
2.2 DNA指纹图谱分析
在秀珍菇ISSR扩增DNA指纹图谱(图1)中:引物P841、P8288分辨力最高,对秀珍菇基因组DNA扩增效果最好;引物P828、P974扩增条带多态性最高,表明用该ISSR引物建立的指纹图谱用来区分秀珍菇遗传背景较好。
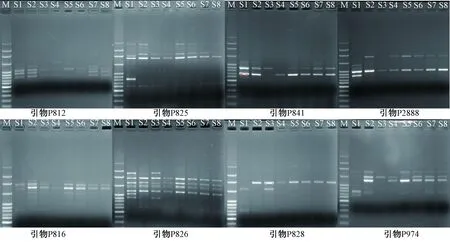
M为DNA marker;S1、S2、S3、S4、S5、S6、S7、S8为8个秀珍菇菌株图1 8个秀珍菇菌株的8条ISSR引物扩增图谱
2.3 聚类分析
用NTSYS-pc2.0的UPGMA对秀珍菇ISSR结果进行聚类分析。结果表明,8份样本可划分为A和B 2大类群,其中,S1为单独一个A类群,剩下为B类群。在B类中,遗传相似系数为0.77处,S3单独聚为一类,其余样本聚为一类。在遗传相似系数约0.82处,8份样本可聚为4类,S1、S3各单独聚为一类,S2、S5、S6聚为一类,S4、S7、S8聚为一类(图2)。
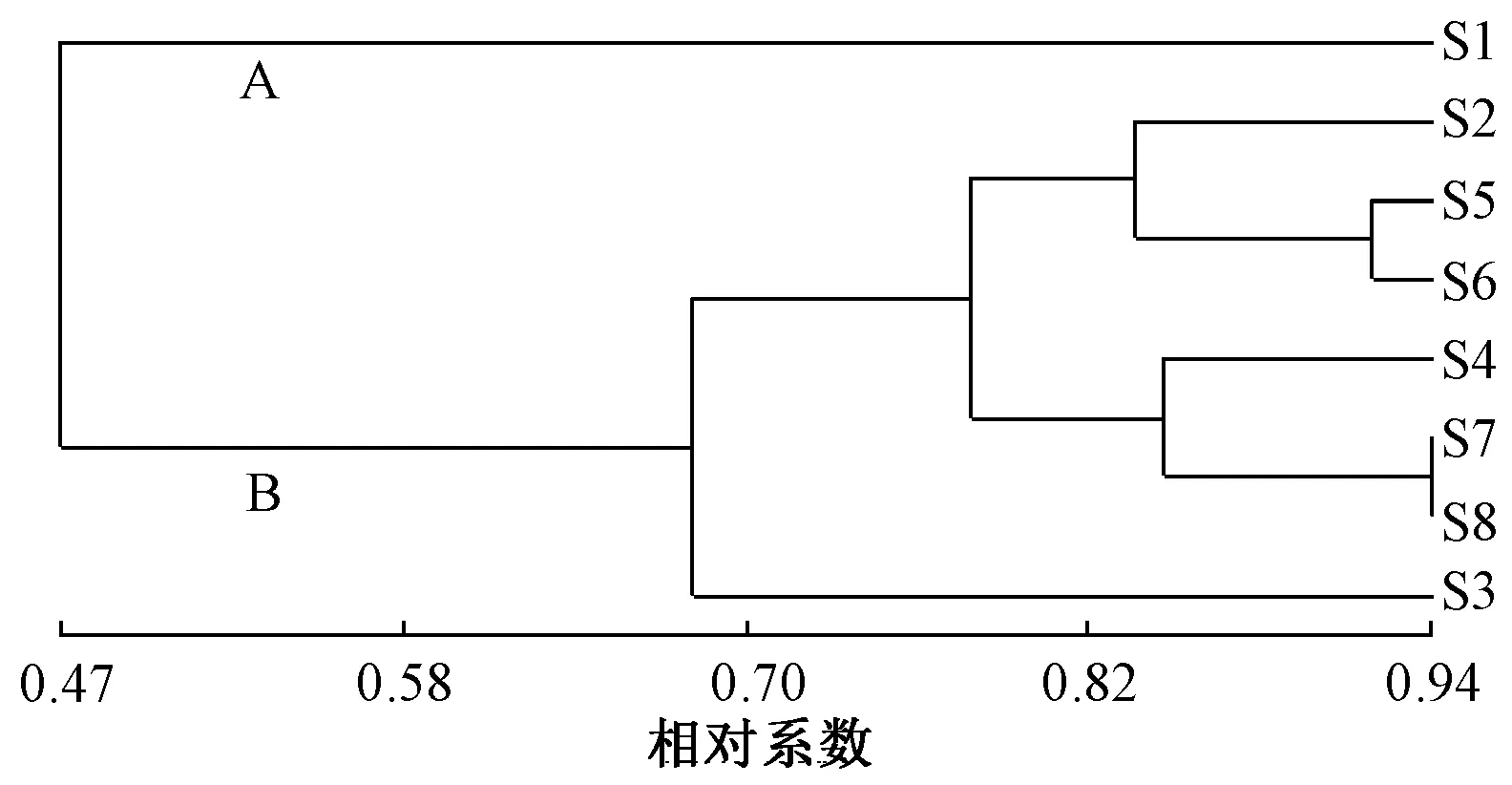
图2 8个秀珍菇样本的UPGMA聚类结果
3 小结
秀珍菇由于长期的人工栽培和相互引种,使得秀珍菇“同种异名”“同名异种”的现象经常出现,仅从子实体外观形态上很难进行准确区分,这不仅影响了秀珍菇品种鉴定的准确性,也会增加菇农选择品种的困惑[7]。因此,菌株或品种间亲缘关系研究和遗传多样性分析对于该属食用真菌育种和生产都具有较为重要的意义,不仅可减少育种工作中亲本选择的盲目性,也能使菇农避免因栽培品种选择错误而造成的经济损失。
16条ISSR引物对8个秀珍菇品种的扩增结果表明,有8条ISSR引物获得的带型较好,多态性丰富,条带清晰,共扩增出207个条带,多态性条带119条,表明秀珍菇种质之间存在丰富的遗传多样性。在DNA指纹图谱中,引物P828、P974扩增条带多态性最高,表明用该ISSR引物建立的指纹图谱可较好地区分秀珍菇遗传背景。
UPGMA聚类分析表明,8份样本可划分为2大类群,其中,S1为单独一类,其余为一类。在遗传相似系数为约0.82处,8份样本可聚为4类,S1、S3各单独聚为一类,S2、S5、S6聚为一类,S4、S7、S8聚为一类。这表明ISSR分子标记可以用于秀珍菇种质资源遗传关系研究,有效地揭示秀珍菇种质资源间丰富的多态性和遗传多样性。

