如何利用TCGA数据库实现医学数据共享
李瑞华,田国祥,郭晓娟,李豹,张军,吕军,5
肿瘤多发病隐匿、预后不良,分子生物学特征复杂,且多种肿瘤发病率逐年上升,已成为严重威胁人类生命和健康的主要疾病之一。《世界癌症报告》预测,全球癌症将由2012年的1400万人递增至2025年的1900万人[1]。一直以来,肿瘤的预防、早期筛检、个体化治疗和预后评估一直是医学界致力解决的关键问题[2]。研究发现,基因变异是所有肿瘤细胞发生的重要的微观分子层面的原因,越来越多的研究者开始从分子遗传学的角度出发进行相关研究[3]。通过测定特定基因表达这一生物学标识,既可以预测肿瘤的生长、扩散及患者存活,同时可根据基因表达情况来制定针对性的诊疗计划[4]。全基因组测序和及生物信息技术的发展为癌症基因组研究提供了新的线索[5],TCGA是2006年由NCI牵头的一个公共资助项目,自2008年开始有阶段性成果发表[6],这三年被认为是TCGA发展的第一个阶段;第二阶段自2009年开始,2009年继续投资2.75亿美元,增加了多种类型的癌症数据,到2014年分析延伸到其他33种类型癌症数据(包括10种罕见肿瘤),来源于11 000多个肿瘤样本,数据量多达255T,包括临床数据、DNA、RNA、蛋白质等多层次的数据。在数据生成方面,该项目取得了无可争议的成功[7]。TCGA的目标是通过大规模、高通量基因组测序和基因芯片技术,整合多维组学数据,研究、定义、发现和分类人类全部肿瘤基因组改变,最终将一套全基因组、多维度癌症基因组图谱绘制出来[8],为肿瘤学研究者提供了大量的基因组数据和相关临床数据,为找到癌症相关基因的微小变异、研究肿瘤生物学机制提供了海量数据基础,从而提高人们对癌症发生的分子层次上的科学认识及预防、诊断和治疗的能力,例如一些研究者利用基因的表达数据与病人存活的数据来探讨两者的关联,进而预测患者的存活状况[9-11]。
1 TCGA的数据产生流程
TCGA包括基因组、蛋白质组、转录组、表观组和临床数据[12]。这些数据由多个组织结构和单位共同支持和维护,比如TSSs(Tissue source site)组织样本和临床数据的来源;BCRs(Biospecimen core resource)中转临床数据和元数据;GCCs(Genome characterization centers)负责基因测序的重头戏,获取组织变异数据;CGHub(Cancer genomics hub)和DCC(Data coordination center)接收GCCs的数据供给研究团队和基因组数据分析中心GDACs(Genome data analysis centers)使用,GDACs分析出的结果再经DCC保存到Community中。其中环节最主要部分是负责测序的GCCs(Genome characterization centers)和负责生物信息分析的GDACs(Genome data analysis centers)。TCGA的数据产生流程如下:
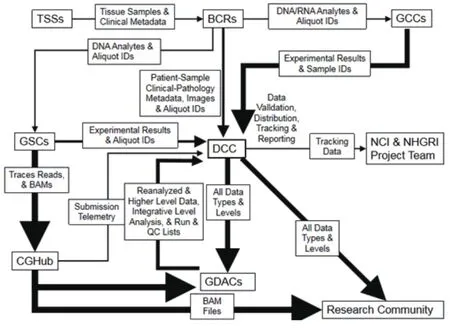
图1 TCGA的数据产生流程[13]
TCGA的数据一般分为4个等级:
Level 1:单个样本的原始测序数据;
Level 2:基因组回帖数据;
Level 3:处理及标准化后的数据,该等级的数据可以直接使用。
Level 4:概括性数据,一般与样本集分析有关。
大部分level 1和level 2属于controlled-access,访问受到限制,需要账号登录才能下载;level 3和Level 4大部分是开放下载的(不需要登录),但公开数据基本能满足临床医生的研究需求,因此无需特意注册账号[14]。
2 TCGA数据库数据提取
2.1 从TCGA网页通过购物车下载此种方法适合于少量数据下载,首先进入TCGA官网(https://cancergenome.nih.gov/),点击“Launch Data Portal”(图2),首次登录会出现“Warning”界面,点击“Accept”进入到数据下载界面,目前有43个project,33096个case(图3)。点击图3中“Repository”进入数据检索界面(图4)。
从TCGA数据库检索界面来看,界面左侧有“Files”、“Cases”,可进行具体的筛选,选择自己想要研究的肿瘤。以女性白人胃癌患者为例,下载与其相关的miRNA数据。在“Cases”栏目下选择 “Primary Site”中的“Stomach”、“Gender”中的“female”及“Race”中的“white”,未进行具体勾选的均默认全选。在“File”栏目下选择“Data Type”的“miRNA Expression Quantification”;还有“Experiment Strategy”的“miRNA-Seq”,这时候界面变成图5所示。

图2 TCGA数据库首页
图3 TCGA数据库下载界面

图4 TCGA数据库检索界面

图5 利用购物车下载数据
结果显示共有106个case,115个文件,这是由于一些case可能对应多个miRNA-Seq样本文件。界面右上角显示所有数据一共只有5.78 MB,可以直接在网页下载。点击购物车标记,购物车按钮变成了绿色(图5),表示该数据已成功放入购物车(系统默认每次显示20个文件,可自行更改为全部添加入购物车),点击桌面右上角的“Cart”。核对需要下载的文件无误后点击右下方“Download”下的“Cart”(图6),

图6 购物车下载rar格式文件
数据将以rar压缩文件的格式保存到默认文件夹里(图7为解压文件),也可自行设定保存路径。
图7 解压后的文件
2.2 通过Data Transfer Tool工具进行下载TCGA购物车下载数据的方法仅适合于少量数据下载,当我们所研究的肿瘤得到的相关数据文件较大或需批量下载数据时,在购物车中直接下载易出现错误,使用下载工具更为便捷。以乳腺癌患者为例,在“Cases”栏目下选择“Primary Site”中的“Breast”、“Program”中的“TCGA”、“Project”中的“TCGA-BRCA”、“Disease Type”中的“Ductal and Lobular Neoplasms”,在“File”栏目下选择“Data Category”中的“Transcriptome Profiling”、“Data Type”的“Gene Expression Quantification”、“Experiment Strategy”的“RNA-Seq”及“Workflow Type”中的“HTSeq-FPKM”(均根据自己的研究需要自行选择),这时候界面变成图8所示。
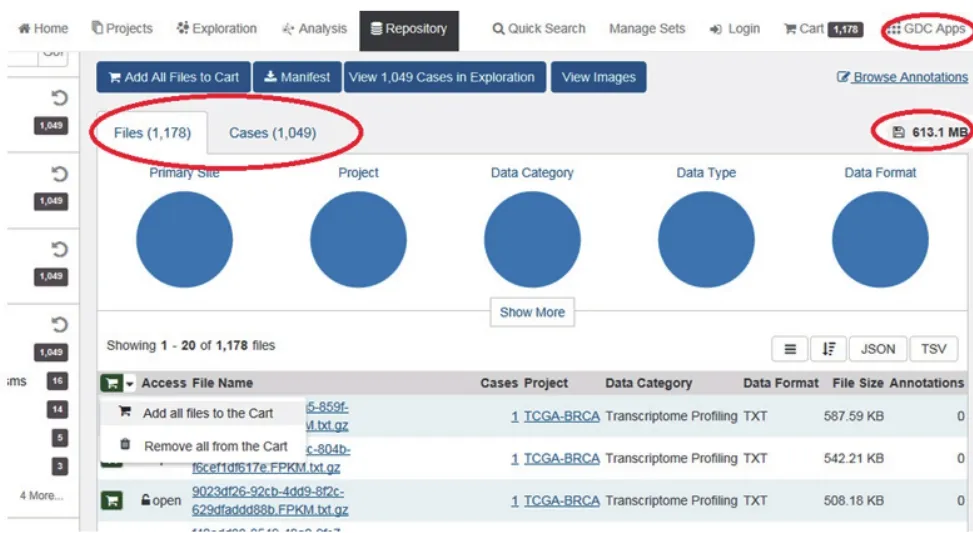
图8 数据检索
显示共有1049个case,1178个文件,所有数据共有613.1 MB,考虑使用下载工具。点击图8右上角所示“GDC Apps”,在下拉菜单里点击“Data Transfer Tool”,找到与电脑操作系统相对应的下载工具安装包(图9)。
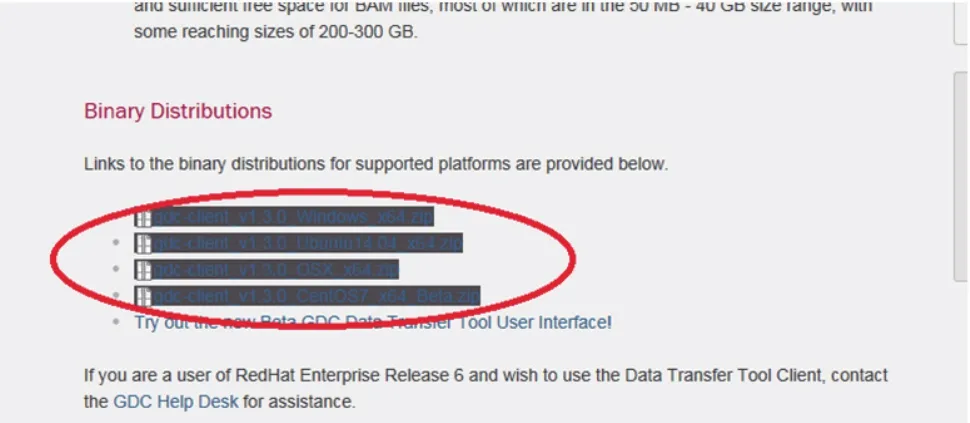
图9 Data Transfer Tool工具下载
下载得到的工具是名为“g d cclient_v1.3.0_Windows_x64(根据电脑系统及软件版本的不同,命名有所差别)”的压缩包,解压后是个“exe”文件(图10),无需安装。如图所示复制该文件所在的路径。
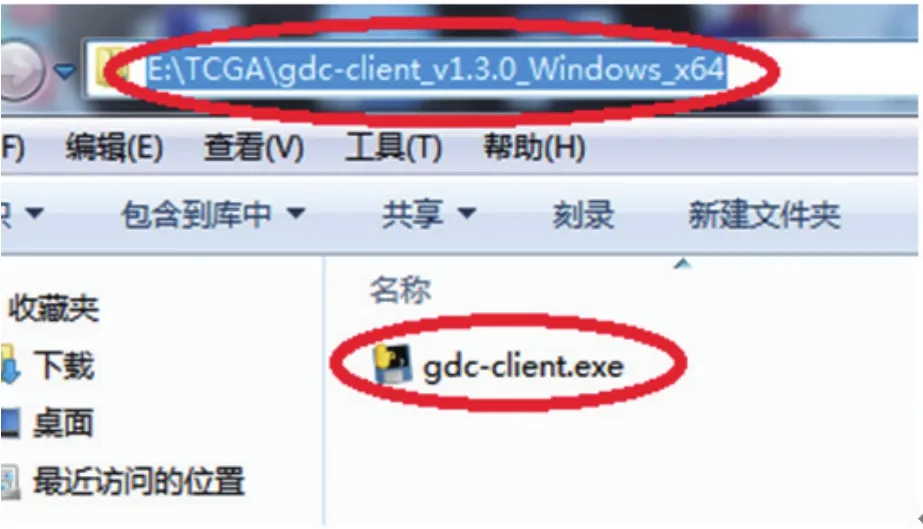
图10 gdc解压文件
打开控制面板 “系统”(或系统与安全)“高级系统设置”,此时界面如图11所示:
点击左下角的“环境变量”,在“环境变量”界面点击“新建”,变量名输入“path”,变量值中输入之前所复制的下载工具所在路径,点击“确定”(依次点图12所示蓝色椭圆框内的三个确定)。
打开计算机命令提示符(方法有很多,这里介绍一种:按“win+r”键打开运行面板,输入“cmd”,点击“确定”),输入“gdcclient-h”(注意“client”和中划线“-”之间有空格),按回车键,将出现以下界面(图13),表示环境变量设置成功。图13所示下方的红色椭圆框内显示将要下载的数据的保存路径。

图11 高级系统设置
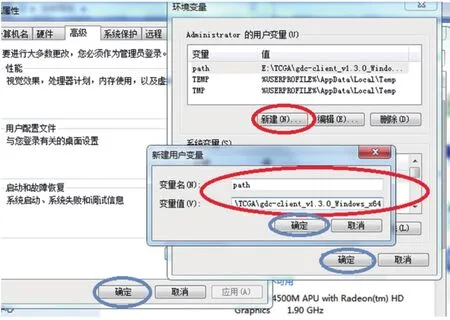
图12 设置环境变量

图13 验证环境变量成功
参数设置成功后,点击图8所示数据检索界面右上角的“cart”,再选择“Download”下的“Manifest”,可下载得到一文本文档(图14)。
然后打开命令提示符,输入命令“gdc-client download-m C:/Users/Adminstrator/Downloads/gdc_manifest_20181208_183307.txt”(根据自己的文件名称不同自行调整)。
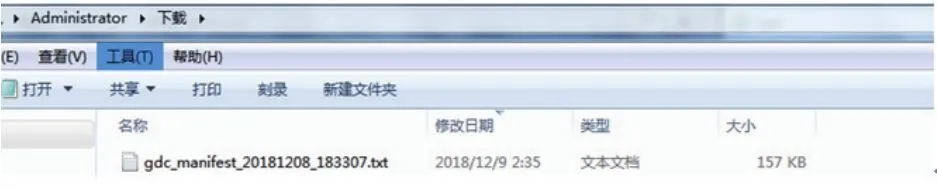
图14 Manifest文件
按回车键后开始下载,并会显示进度(图15)。下载完成后,会在运行目录里找到对应的文件(图16)。
2.3 使用R语言进行数据下载和分析还可利用R语言(RTCGAToolbox)进行数据的下载和分析,操作较方便,但是要求使用者有一定的R语言基础,这里不作具体演示。
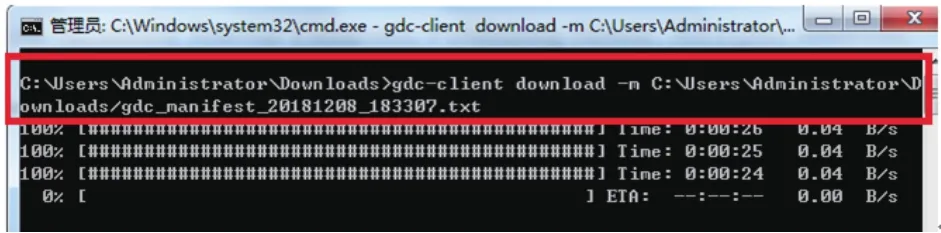
图15 数据下载中
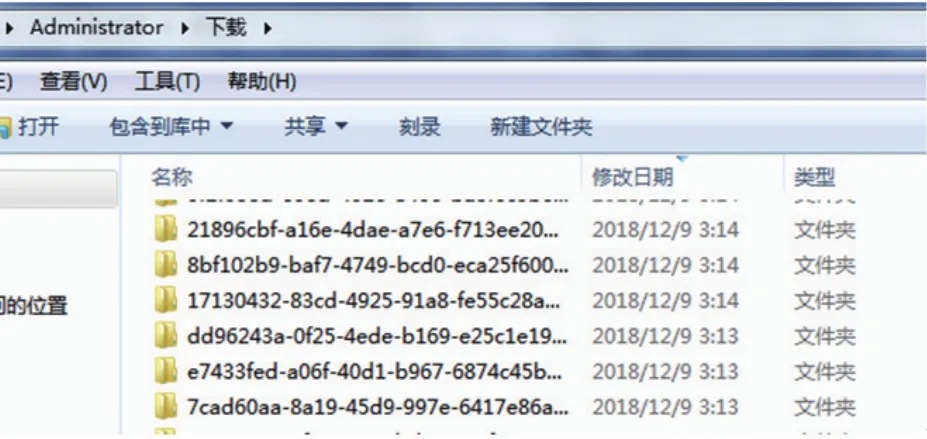
图16 数据下载成功
3 总结
自2003年人类基因组序列图完成,接下来的十几年里,包括肿瘤基因在内的基因测序技术得到飞速发展,人类对肿瘤的分子多样性和各类型肿瘤的共同特点有了更多更深刻的认识。TCGA即肿瘤基因组图谱计划开启了肿瘤分子生物学和精准医学的时代,这样一个前所未有的公开的多维度癌症基因组数据集合,为科研工作者提供了研究癌症的发生发展的新机遇,使我们可以用前所未有的微观视角来看待癌症,从而能一步步更加接近它的全貌。目前,TCGA数据已被用于发现新的突变,确定内在的肿瘤类型,确定泛癌相似性和差异性,同时收集肿瘤演变的证据[1]。目前,越来越多的针对TCGA数据库的生物信息学工具也相继开发出来,更加反映出TCGA数据资源的重要性。
本文通过介绍TCGA项目研究背景、TCGA官方网站架构及数据获取方法,旨在帮助感兴趣的研究者基本了解TCGA项目信息、熟悉网站架构、高效便捷地提取数据资料,提高研究者的工作效率。

